运动发酵单胞菌底盘细胞研究现状及展望
杨永富,耿碧男,宋皓月,何桥宁,何明雄,鲍杰,白凤武,杨世辉
(1 湖北大学生命科学学院,省部共建生物催化与酶工程国家重点实验室,湖北省环境微生物工程技术研究中心,湖北 武汉 430062;2 农业农村部沼气科学研究所,生物质能技术研究中心,四川 成都 610041;3 华东理工大学生物工程学院,生物反应器工程国家重点实验室,上海 200237;4 上海交通大学生命科学技术学院,微生物代谢国家重点实验室,上海 200240)
下一代测序(next generation sequencing,NGS)与质谱(mass spectrometry,MS)技术的发展促进了系统生物学(systems biology)方法在生命科学领域的应用,推动了在基因组层面对生物体“基因-RNA-蛋白-代谢-表型”变化规律与调控机制的深入研究[1]。在系统生物学研究的基础上,通过引入工程学思想策略,并与现代生物学、系统科学及合成科学进行融合,使生物技术系统化和标准化,形成了在理性设计指导下重组或从头合成新的并具有特定功能“人造生命”为目标的“合成生物学”[2-3]。同时,建立了合成生物学“设计-构建-测试-学习”(design-build-test-learn,DBTL)的研究策略,推动了通过理性或半理性改造工业微生物底盘细胞,实现“建物致知”与“建物致用”目标的研究进程。
运动发酵单胞菌(Zymomonas mobilis)是一种兼性厌氧革兰氏阴性菌,具有许多独特的生理特点和优异的工业生产特性,是目前唯一已知能够在厌氧条件下利用Entner-Doudoroff(ED)途径的微生物,具有较高的糖吸收率、乙醇收率和乙醇耐受性等优良特性;近年来被作为纤维素类生物质生物炼制生产生物能源的细胞工厂受到重视[4-5],是美国能源部国家可再生能源实验室(National Renewable Energy Laboratory,NREL)及大湖生物能源研究中心(Great Lakes Bioenergy Research Center,GLBRC)研究的主要底盘细胞之一。
尽管已经利用运动发酵单胞菌开展了一系列包括生理、遗传及代谢工程改造方面的研究,特别是拓宽了其底物利用范围及产物生成种类,在“建物致用”的目标上取得了可喜进展,但是与模式微生物菌株相比,运动发酵单胞菌在生物元件的挖掘与定量、生物元件组装、逻辑线路构建、代谢途径的时空调控以及基因组编辑与优化等“建物致知”领域刚刚起步。本文简述了运动发酵单胞菌作为潜在的细胞工厂实现工业产品生产的独特生理特点,重点总结了其在平台化合物、食品和药品生产等工业领域的应用,系统介绍了运动发酵单胞菌系统生物学研究进展,同时探讨了运动发酵单胞菌现有的遗传学方法、技术与工具等方面的进展及瓶颈,以及进一步开发运动发酵单胞菌作为优良底盘细胞,推动合成生物学理论研究和实践应用发展的研究方向。
1 运动发酵单胞菌的特性及潜在应用
1.1 独特的生理特点
运动发酵单胞菌最初从墨西哥的龙舌兰酒(pulque)中分离得到[6],因其独特的生理特性和乙醇发酵生产能力受到研究人员的重视[7]。运动发酵单胞菌培养过程具有副产物生成少、葡萄糖代谢速率快、对高浓度乙醇(体积分数16%)耐受性好、生长温度(24~45 ℃)和pH 范围(4.0~8.0)广泛且“通常认为是安全的”(generally regarded as safe,GRAS)等特性[8-10](图1)。表型芯片研究结果表明,运动发酵单胞菌的生理特性更接近酿酒酵母而不是细菌[11],但是与酿酒酵母相比,运动发酵单胞菌有着基因组小、对糖的转化率及乙醇收率高等优势[4-5]。另外,研究表明运动发酵单胞菌具有完整的Mo 固氮酶系统及生物固氮能力,可利用空气中的氮气作为唯一氮源生产乙醇且不影响乙醇产量,碳转化率可达到理论最大值的97%,对该氮代谢途径的解析有利于进一步阐明该菌的生理特征[12-13]。
运动发酵单胞菌对高乙醇或高糖的耐受性可能和其独特的细胞膜成分相关,在其细胞膜上含有丰富的磷脂和脂肪酸,膜质中的藿烷(hopanoid)含量可高达总脂质的50%,藿烷与脂肪有助于维持细胞质膜的稳定与通透性,可能是其高乙醇耐受的原因之一[14]。在乙醇浓度上升或温度升高时,运动发酵单胞菌细胞膜的磷脂和脂肪酸的各组分相对比例会发生变化,同时膜蛋白含量也增加,从而降低乙醇或温度升高时对膜流动性的不良影响[15-16]。近期研究表明降低藿烷的含量或改变其极性头部组分会降低其对乙醇的耐受,且高含量的藿烷有助于菌株对低pH 的适应,研究人员结合细菌衍生脂质体的光谱分析推测藿烷可能通过疏水作用和脂质间氢键相互作用来稳定胁迫环境下细菌膜的生理功能[17]。耐低pH 突变株的转录组数据也表明,在酸性pH 环境下,突变菌株中藿烷合成相关的基因表达量高于野生型菌株[18],证明高含量的藿烷成分有助于突变菌株对酸性pH的耐受。
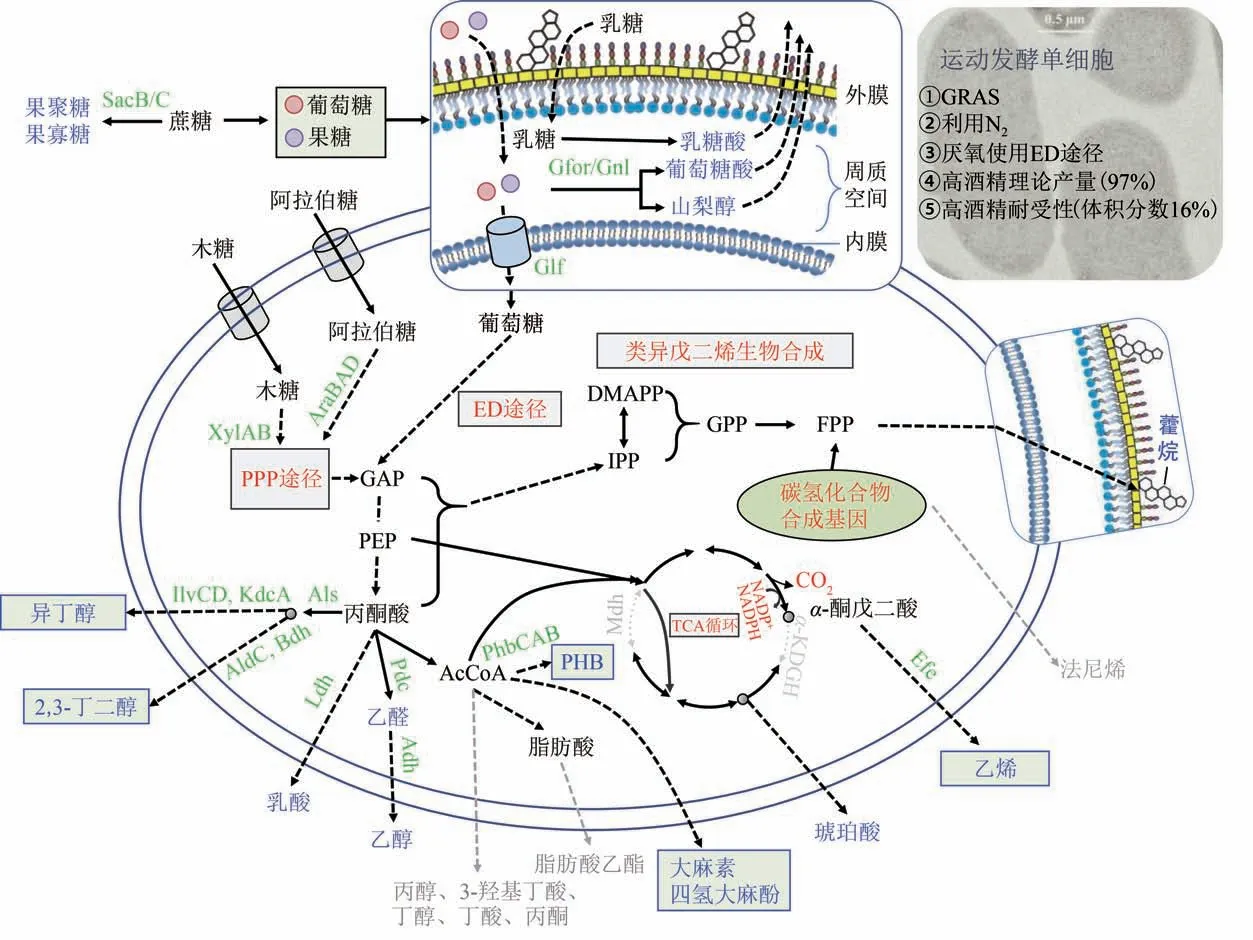
图1 运动发酵单胞菌的重要生理特性及其多样化的应用Fig.1 Physiological characteristics and diverse applications of Z.mobilis
另外,兼性厌氧的运动发酵单胞菌是目前已知唯一可在厌氧条件下通过ED 途径代谢葡萄糖、果糖或蔗糖生产乙醇的微生物。通过ED 途径,运动发酵单胞菌每代谢一分子的葡萄糖或者果糖只产生一分子的ATP,与酿酒酵母等微生物利用的Embden-Meyerhof-Parnas(EMP)途径相比,等量糖代谢ATP 生成减少50%[5]。由于ATP 主要通过细胞生物量的积累而消耗,以维持胞内代谢的能量平衡,ED 途径菌体生物量积累少,大部分碳源(>95%)被转化为产物乙醇。同时,ED 途径的酶占细胞总蛋白的50%左右[19-20],并且每一个与糖降解有关的基因都高效表达,且基本都为组成型表达[21]。这种特异的糖代谢方式可能是在进化过程中为适应高糖环境而形成的,即遗失了EMP 代谢途径中的关键调控酶——磷酸果糖激酶(phosphofructokinase,Pfk)和三羧酸循环(tricarboxylic acid cycle,TCA循环)中的2个关键酶,苹果酸脱氢酶(malate dehydrogenase,Mdh)和酮戊二酸脱氢酶复合体(α-ketoglutarate dehydrogenase complex,α-KDGH)。这一快速糖代谢通路的存在,使运动发酵单胞菌可快速代谢葡萄糖,再加上高效的丙酮酸脱羧酶(pyruvate decarboxylase,Pdc)和乙醇脱氢酶(alcohol dehydrogenaseⅡ,AdhB),使其可以快速生成乙醇。
运动发酵单胞菌这种独特的糖代谢途径可能和其能量产生与生长部分耦联的现象相关,即其能量的产生率和利用率之间不完全关联。运动发酵单胞菌的呼吸链速率很高,比常见的模式微生物大肠杆菌和酿酒酵母要高,但其生物学功能尚未完全解析[22]。目前的研究显示运动发酵单胞菌具有一个结构性呼吸链,包括II 型NADH 脱氢酶(Ndh)、辅酶Q10和细胞色素BD 末端氧化酶,以及一些次要的或仍未确定的成分,呼吸链在有氧条件下使用氧作为末端电子受体[23],运动发酵单胞菌是少数已知可以用NADH和NADPH作为呼吸型NADH 脱氢酶电子供体的细菌之一[24]。另外,葡萄糖和D-乳酸也可以为呼吸链提供电子,分别向与膜结合的葡萄糖脱氢酶和D-乳酸脱氢酶提供电子,这些电子传递给辅酶Q10之后,最终传递到能够将氧气还原为水的末端氧化酶[25]。同时,尽管运动发酵单胞菌中存在的F 型ATP 合成酶(FoF1-type ATPase)可以通过质子梯度产生ATP[26],但其氧化磷酸化效率低下或不参与能量的产生。运动发酵单胞菌这种高效率电子传递链和低氧化磷酸化效率的现象,可以作为细菌中的一种呼吸链系统模式进行研究,揭示其低效率呼吸的原因,理解呼吸链组分与能量产生和胞内氧化还原平衡之间的关系[23]。
运动发酵单胞菌独特的生理特征除有利于其在工业生产中的应用外,还有助于研究人员理解微生物底盘细胞的代谢。如运动发酵单胞菌中的EMP 途径、TCA 循环和磷酸戊糖途径(pentose phosphate pathway,PPP)等途径不完整,可以通过合成生物学手段研究不同能量代谢途径及其组合对底盘细胞代谢的影响。例如,为了验证ATP产量对运动发酵单胞菌生物量积累的影响,研究者尝试补全EMP 途径[27-28],但结果表明糖代谢受到影响、菌株生长受到抑制、且有甘油等副产物生成,分析认为可能是因为内源代谢物浓度改变使外源引入的反应反向进行[28],这些研究为理解底盘细胞的代谢环境如何影响外源基因表达提供了指导。
1.2 广泛的底物利用性与优良的环境适应性
野生型运动发酵单胞菌能利用的底物有限,只包括葡萄糖、果糖和蔗糖,无法利用五碳糖,如来自木质纤维素水解液中的木糖和阿拉伯糖等[5]。为了扩大其底物利用谱,研究人员通过代谢工程及实验室适应性进化(adaptive laboratory evolution,ALE)等不同的策略做了大量的菌株改造工作。如通过引入大肠杆菌中4个与木糖代谢相关的基因xylA/B、tal和tkt,首次得到了可以利用木糖的重组菌株Z.mobilisCP4(pZB5)[29]。在此基础上,采用类似策略通过代谢工程手段引入与阿拉伯糖代谢相关的5个基因,得到了可以利用阿拉伯糖的菌株Z.mobilisCP4(pZB206)[30]。最近一株由Z.mobilisATCC ZW658 通过适应性进化得到的突变株AD50 可以很好地共利用葡萄糖与木糖,其葡萄糖和木糖共利用时的利用速率分别为1.24 g/(g·h)和1.34 g/(g·h)(以干物质计),该突变株是目前最好的葡萄糖和木糖共利用菌株[31]。通过代谢工程手段改造运动发酵单胞菌,提高菌株对五碳糖利用的研究进展可参考此前相关的综述文章[5,10]。
除了可以利用纯糖进行乙醇发酵外,运动发酵单胞菌还可以利用多种生物质原料经过预处理与酶水解等方法释放的单糖进行乙醇发酵[10],如木质纤维素生物质(玉米秸秆等)、能源作物(甘蔗、甜菜、角豆、甘薯和甜高粱等)、能源植物(如柳枝稷)、工业废料(豆粕)、食物残渣、农业废料(玉米芯残渣、米糠、甜高粱秸秆、甘蔗糖蜜、竹残渣、废纸渣等)以及藻体生物质等[32-46]。研究运动发酵单胞菌对多种碳来源原料的利用,特别是来自工业、农业和城市废弃物的原料,将有助于将废弃物转化为有价值的生物燃料或化学品,并促进其在不同领域的应用。
运动发酵单胞菌在由高糖引起的高渗条件下仍能生长,在250 g/L葡萄糖或300 g/L蔗糖条件下都能保持生长,只是会有较长的延迟期[47-48]。研究表明,运动发酵单胞菌可以在高糖环境下生长,可能与其使用无需耗能的协助扩散糖转运系统和由葡萄糖果糖氧化还原酶(glucose-fructose oxidoreductase,Gfor)转化果糖形成的山梨醇有关[49-50]。当细胞处于高浓度蔗糖环境中时,山梨醇在细胞周质内的积累可以高达1 mol/L,帮助降低外部高渗透压对细胞造成的生理伤害[50]。山梨醇除了有利于细胞在由高糖引起的高渗透压环境下生长外,还有助于运动发酵单胞菌对温度和乙醇的耐受[47]。转录组学研究表明,运动发酵单胞菌培养于220 g/L 葡萄糖的高糖条件下时,细胞会通过调节与膜通道和转运体、应激反应机制和代谢途径相关基因的转录水平来实现对高浓度葡萄糖的响应[48]。
运动发酵单胞菌可以适应广泛的pH范围(4.0~8.0),尤其是可以在低pH 环境中生长。2019 年农业农村部成都沼气科学研究所何明雄团队通过多轮常压室温等离子体(atmospheric and room temperature plasma,ARTP)诱变,获得了对乙酸耐受性增强的运动发酵单胞菌突变株AQ8-1 和AC8-9,在5.0~8.0 g/L 乙酸环境下这两个突变株的生长和乙醇产率较野生型均有提升,且通过第3轮ARTP 诱变得到的突变株PH1-29 可以在pH 4.0或3.5 的环境下生长,而野生型菌株基本不生长[51]。通过另外一种策略ALE得到的耐低pH突变株3.5M 和3.6M,与野生型ZM4相比,在pH 3.8下的生长速度提高了50%~130%,发酵时间缩短了4~9 h,乙醇产量提高了20%~63%[18]。这些酸耐受突变菌株的获得为直接利用酸性水解液进行乙醇发酵奠定了基础。
运动发酵单胞菌的兼性厌氧特性为其在工业生产应用提供了便利。此外,由非絮凝菌株Z.mobilisZM4 突变而来的絮凝菌株Z.mobilisZM401 可以在生物反应器培养过程中进行自絮凝,这一特性可以使细胞在连续培养和发酵条件下不随发酵液的排出而流失,提高细胞密度及生物反应器的乙醇生产强度,同时还可增强对抑制物的耐受性,进而降低乙醇工业化生产的成本[4,52]。上海交通大学白凤武课题组近期研究结果表明胞外多糖——纤维素是引起ZM401絮凝的主要物质,通过基因序列比对和实验验证发现,引起ZM401自絮凝的主要原因是ZM401 中的纤维素合成基因bcsA(ZMO1083)的上游基因(ZMO1082)中一个胸腺嘧啶(T)的缺失,使ZM401 中的ZMO1082 与ZMO1083 融合形成了一个新基因bcsA_401,同时纤维素合成操纵子bcsABC在絮凝菌株ZM401 中的表达增高,使得纤维素合成在ZM401菌株中增加,导致细胞发生絮凝[53-54]。
运动发酵单胞菌优良的环境适应性展示了其作为工业底盘细胞的潜力,其对纤维素水解液中抑制物的耐受性也通过不同方法得到了提高。例如过表达内源NADH 氧化酶基因ZMO1885 增强了菌株的糠醛耐受性,且不引起胞内辅因子扰动[55];过表达内源Na+/H+逆向协同转运体基因nhaA(ZMO0119)增强了菌株对钠离子和乙酸钠的耐受[56-57];通过基因组重排技术对菌株进行多轮改造,得到的重组菌株可以耐受7.0 g/L 的乙酸和3.0 g/L 的糠醛[58]。工程菌株的改造提高了运动发酵单胞菌的底物利用及产品生产的鲁棒性,为其在纤维素乙醇生产中的应用奠定了基础[59-60]。
1.3 不同工业领域的应用及研究
与酿酒酵母相比,运动发酵单胞菌细胞体积小导致比表面积高,高细胞比表面积与胞内ED 途径共同影响下,使其葡萄糖代谢速度更快,同时ED 途径产能水平低使生物量积累少,发酵过程乙醇对糖的表观收率高,能耐受高浓度糖和产物乙醇。虽然野生型菌株不能利用木糖等五碳糖,而且木质纤维素水解液中的抑制物对其生长以及乙醇发酵有影响,但是通过代谢工程、合成生物学及实验室适应性进化等手段改造,可以解决这些问题,得到性状优良的菌株[5,60]。NREL 曾与杜邦公司(DuPont)合作,利用运动发酵单胞菌进行纤维素乙醇示范生产技术开发。目前利用运动发酵单胞菌以玉米秸秆为原料通过脱乙酰化和机械研磨处理后的水解液进行纤维素乙醇生产,乙醇浓度达到86 g/L,糖转化率达到理论转化率的73.3%[32,61]。运动发酵单胞菌最近还展示了作为产电菌群的潜力(2.0 mW/m2),在生产乙醇的同时生产电能[62]。
在运动发酵单胞菌中还实现了多种化合物的生物合成,包括聚羟基丁酸(polyhydroxybutyrate,PHB)、D-乳酸、2,3-丁二醇、山梨醇、乙醛、异丁醇和乳糖酸等[63-75](表1)。其中:通过引入PHB 合成操纵子phbCAB,实现了PHB 在运动发酵单胞菌中的积累,可达0.7%的细胞干重[74];通过异源表达优化乙酰乳酸合成酶(Als)、乙酰乳酸脱羧酶(AldC)和丁二醇脱氢酶(Bdh),实现了2,3-丁二醇在运动发酵单胞菌中的生物合成[76],通过进一步敲除乙醇生产途径中的丙酮酸脱羧酶(Pdc)阻断乙醇途径的改造,使2,3-丁二醇的浓度从10 g/L 提高到120 g/L 以上[65];通过敲除过氧化物酶基因(cat)并过表达呼吸链中的NADH 脱氢酶基因(ndh),使得乙醛产量达到1.3 mol/mol(葡萄糖)[66];通过外源引入由组成型强启动子Pgap驱动的2-酮基异戊酸脱羧酶(KdcA),并结合由诱导型启动子Ptet驱动的人工操纵子als-ilvC-ilvD,在运动发酵单胞菌中成功构建了异丁醇生物合成途径,使异丁醇产量达到4.0 g/L[63]。近期,何明雄团队通过引入外源乙烯形成酶(Efe),将运动发酵单胞菌改造为乙烯生产菌株,最高产率可达到12.83 nmol/mL(OD600)[67]。此外,多组学分析还为利用运动发酵单胞菌的甲基赤藓糖醇磷酸(methylerythritol phosphate,MEP)途径生产类异戊二烯提供了参考[25]。
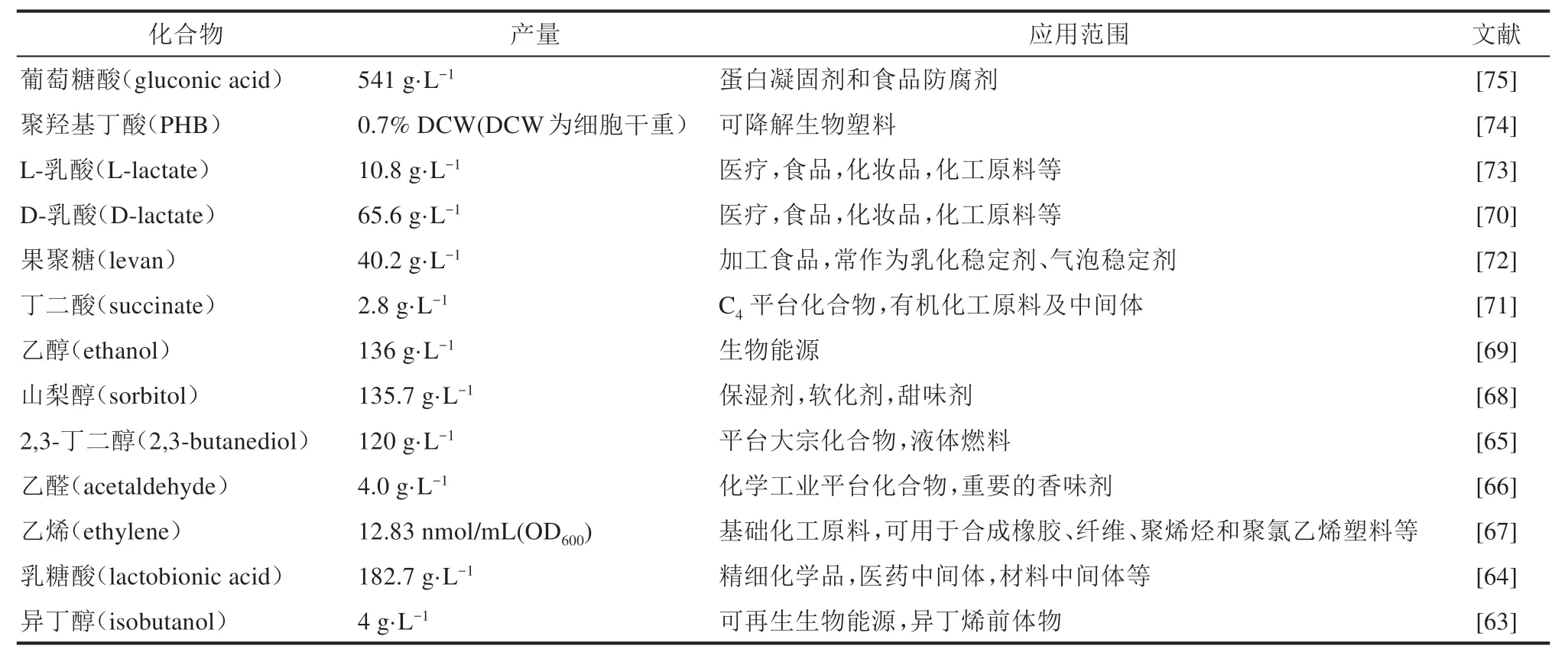
表1 运动发酵单胞菌可生产的不同化合物名单Tab.1 Biochemicals produced by Z.mobilis
除了用于纤维素乙醇及平台化合物生产,运动发酵单胞菌由于其GRAS及副产物低等优点,在食品和药品生产等领域也开始受到重视。在食品应用中,酿酒酵母细胞壁成分被认为是可引起炎症性肠病或克罗恩病(Crohn's disease)的抗原,这种食物过敏反应的存在使寻找酵母发酵替代菌株十分重要[77]。运动发酵单胞菌可以在发酵烘焙食品领域作为酿酒酵母的替代菌株,在对酿酒酵母具有不良反应的人群中具有潜在的应用价值。需要指出的是,野生型运动发酵单胞菌无法利用小麦面粉中的麦芽糖,因此研究人员通过在面团配方中添加运动发酵单胞菌能够利用的蔗糖或与其他可食用微生物共发酵[78-81],对运动发酵单胞菌在面粉发酵中糖的利用和发酵效果进行了研究。日本福山大学的Yuji 和Kenzo[79]报道了在蔗糖添加量为5 g/100 g 面粉时有较好的发酵能力,而较高的蔗糖添加量会降低发酵性能。意大利米兰大学Picozzi 课题组也证明了在每100 g 面粉1 g 或5 g蔗糖的情况下,运动发酵单胞菌可以有效发酵面团[80],并且研究结果表明添加蔗糖显著提高了运动发酵单胞菌的发酵性能,产气率和保留率分别为80%和85%,最大面团高度提高了2.6 倍,说明在添加蔗糖的情况下,运动发酵单胞菌可以有效发酵面团,从而为无酵母发酵产品领域提供了新的途径[78]。
与异型发酵型乳酸菌(Lactobacillus sanfranciscensis)以1∶1比例共发酵,为运动发酵单胞菌无法利用麦芽糖的问题提供了另外一个策略[81]。近期意大利米兰大学Musatti 课题组[82]利用代谢组学,对运动发酵单胞菌在发酵小麦面团时产生的主要挥发性组分等进行了测定,结果表明运动发酵单胞菌能产生传统发酵剂类似的发酵产物,如乙醇、乙酸和4-羟基-2-丁酮(4-hydroxy-2-butanone),另外运动发酵单胞菌还能产生壬酸(nonanoic)、十一酸(undecanoic acid)、十六烯醛(2-undecanoic acid)和酒石酸二乙酯(tartaric acid diethyl ester)等,但添加NaCl 会影响发酵时间和发酵性能,该研究进一步奠定了运动发酵单胞菌在烘焙食品中应用的基础。
近年来,利用益生元与益生菌的特性和功能造福人类健康已受到更多重视并取得了一定突破[83],运动发酵单胞菌在这方面也有很大潜力。运动发酵单胞菌中含有的果聚糖蔗糖酶(SacB)可以被用于生成果聚糖(levan)和果寡糖(fructooligosaccharides,FOS)等功能性或保健产品。果聚糖在食品和医用上具有多种功能,如抗肿瘤[84]、降血压或作为益生元[85]等。FOS 也是一种益生元,它会导致胃肠道微生物群组成和/或活性发生特定变化而有益于宿主健康[86-87]。最近的研究还实现了使用果聚糖蔗糖酶来生产另一种益生元蔗果三糖(kestose),并开发了功能性产品[88]。
运动发酵单胞菌还是开菲尔酒中主要的细菌益生菌成分[89]。开菲尔酒是一种由细菌与酵母共发酵产生的低酒精度碳酸饮品,也是一种功能性保健饮品,在中国又称为菌菇(tibicos),在一项对中国天山地区tibicos进行单分子测序的研究中也发现了运动发酵单胞菌是其中的一种主要微生物[90]。研究人员在一种大鼠模型中评价了Z.mobilisUFPEDA-202的益生菌特性和安全性,结果表明Z.mobilisUFEPEDA-202 为无致病性的安全食用菌株,且对小鼠免疫功能有一定的改善作用[91]。近期一项研究评价了定期服用运动发酵单胞菌发酵液对便秘患者肠道功能的影响,结果表明服用带有菌群的发酵液有助于改善肠道蠕动,而服用不带菌群的发酵液可以有效降低胆固醇和血脂的含量[92]。运动发酵单胞菌的这些特性推动了其作为益生菌的商业化生产,标准化发酵的运动发酵单胞菌原料已经用于制备药用胶囊型益生菌[93-94]。
由于运动发酵单胞菌具有作为底盘细胞发展的优势,在药品开发中也得到了应用。2019 年,德国Farmako公司报道了利用运动发酵单胞菌开发的重组菌株Z.cannabinoidis®利用葡萄糖生产大麻素的研究成果,其四氢大麻酚(tetrahydrocannabinol,THC)的成本约为传统方法的千分之一,大大降低了生产大麻素的成本。与此前报道的利用酵母生产大麻素不同的是,该工艺不需要在合成大麻素后破细胞提取[95],Z.cannabinoidis生产的产物会直接释放到培养环境中,从而可以实现连续培养。Farmako 公司宣称目前该工艺生产了大约180种不同的大麻素衍生物,包括大麻二酚(cannabidiol,CBD)和THC,每克Z.cannabinoidis可以产生4.5千克THC,同时900 h不间断生产。
2 运动发酵单胞菌系统生物学研究进展
2.1 基因组序列与注释的完成与更新
运动发酵单胞菌目前共发现3 个亚种:运动亚种(sp.mobilis)、梨亚种(sp.pomaceae)和弗朗西斯亚种(sp.francensis)。运动发酵单胞菌各亚种生存的温度有所不同[96],运动亚种可于24~45 ℃生存,且由于其发酵性能最佳被作为模式菌株研究。运动发酵单胞菌模式菌株ZM4(ATCC31821)的基因组于2005 年首次完成测序(图2),其基因组由2 056 416 bp 组成,形成一个环状染色体[97];2009 年,利用二代测序技术和系统生物学数据对其基因组进行了修正和更为精准的基因组重注释[98]。
随着测序技术的发展及对ZM4 的进一步深入研究,2018 年,结合多个实验室的研究数据对ZM4 基因组进行了再次更新,重点研究更新了4 个内源质粒信息并进行了更加准确的注释,并探讨了特定生长条件下质粒的表达水平及其与宿主环境适应性的关系[99]。目前,最新的ZM4 基因组包括一个2Mb 的环状染色体(2 058 755bp)和4 个32~39kb 的质粒(pZM32,32 791bp;pZM33,33 006bp;pZM36,36 494bp和pZM39,39 266bp)[99]。同时,木糖利用重组菌株8b 的基因组也得到了报道,其前期改造引入的两个木糖利用表达框被准确定位,其中Peno_talB-tktA-cat表达框插入到pZM36 质粒上,该质粒被重命名为pZM41;而Pgap_xylA-xylB-yiaB'-yiaA'-wecH'-tetA表达框插入到ZMO1237(ldhA)基因内;另外还有65 个SNPs也一同得到鉴定[99]。
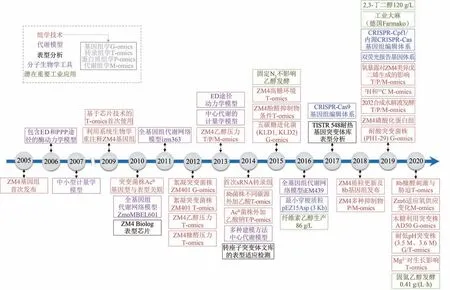
图2 运动发酵单胞菌系统与合成生物学研究进展Fig.2 Research progress of systems and synthetic biology in Z.mobilis
包括模式菌株ZM4 在内,目前运动发酵单胞菌共有29 个基因组测序项目公开,其中13 个菌株的基因组被完整测序组装,另外还有3个菌株基因组为Scaffold 状态,其余为Contig 状态(数据来源于NCBI 网站)。各菌株包含2~8 个质粒不等,基因组大小在2.01~2.22Mb,部分菌株之间的进化关系及基因组之间的差异也被比较分析[5,100]。相比于模式微生物酿酒酵母(12.12Mb)和大肠杆菌(5.15Mb),运动发酵单胞菌(2.14Mb)有基因组小的优势,便于开展基因组精简优化及基因组合成,是一个理想的工业微生物底盘细胞。
2.2 系统生物学方法的建立及应用
基因组测序为比较基因组学的研究奠定了基础,2005 年模式菌株ZM4 基因组测序确定时与Clusters of Orthologous Groups of proteins(COGs)数据库中已测序的其他物种的基因组进行了比较,发现其1668 个开放读码框中有768 个与新鞘氨醇杆菌(Novosphingobium aromaticivorans)高度相似,这一结果与前期16S rRNA 的测序结果一致[97]。比较基因组分析,目前作为常规方法在突变菌株突变位点确定的研究中广泛应用。
基于芯片分析的转录组学技术2009 年在运动发酵单胞菌中首次应用,结合代谢组学分析,开展了氧气对运动发酵单胞菌发酵过程影响的研究[101]。研究发现在有氧与无氧条件下的差异表达基因有166 个,其中ED 途径相关基因在稳定期的表达水平在无氧条件下高于有氧条件,同时微生物生理及代谢组学数据表明在无氧条件下生长快、糖耗快、乙醇产量高、乳酸与乙酸等副产物少,该研究首次利用转录组学并结合代谢组学从全局水平解释了氧气对运动发酵单胞菌碳代谢流及最终产物生成的影响[101]。基于芯片分析的转录组学研究,随后被用于比较运动发酵单胞菌不同生长环境下的基因表达情况,如高糖环境(220 g/L 葡萄糖)、乙醇胁迫(体积分数5%或6%)、糠醛抑制及乙酸毒性等[48,102-108],以及研究突变菌株间基因表达差异,如絮凝菌株ZM401 与非絮凝菌株的差异及乙酸耐受菌株AcR与野生型之间的差异等[53,107](表2)。
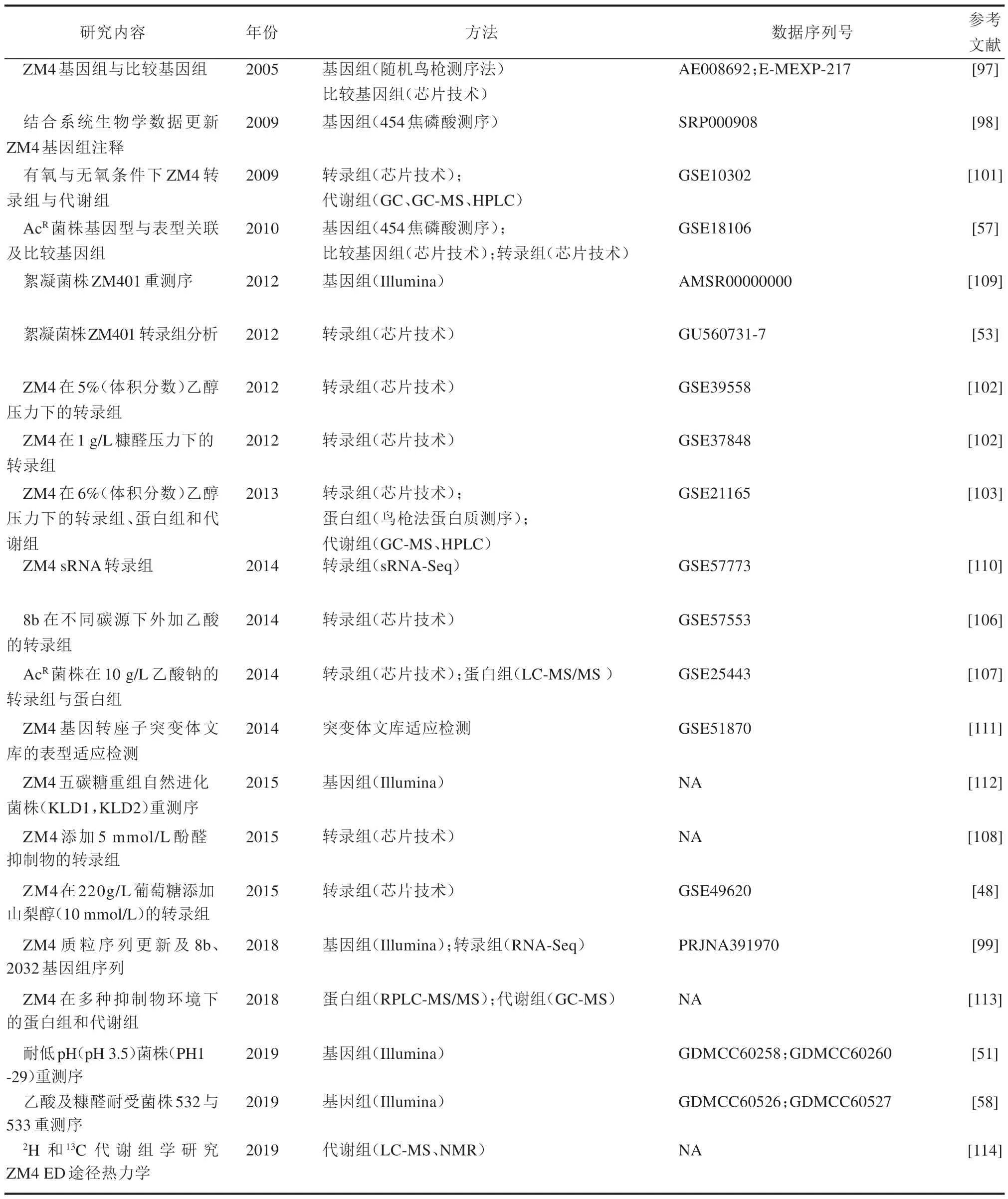
表2 运动发酵单胞菌系统生物学研究进展总结Tab.2 Summary of systems biology research progress in Z.mobilis
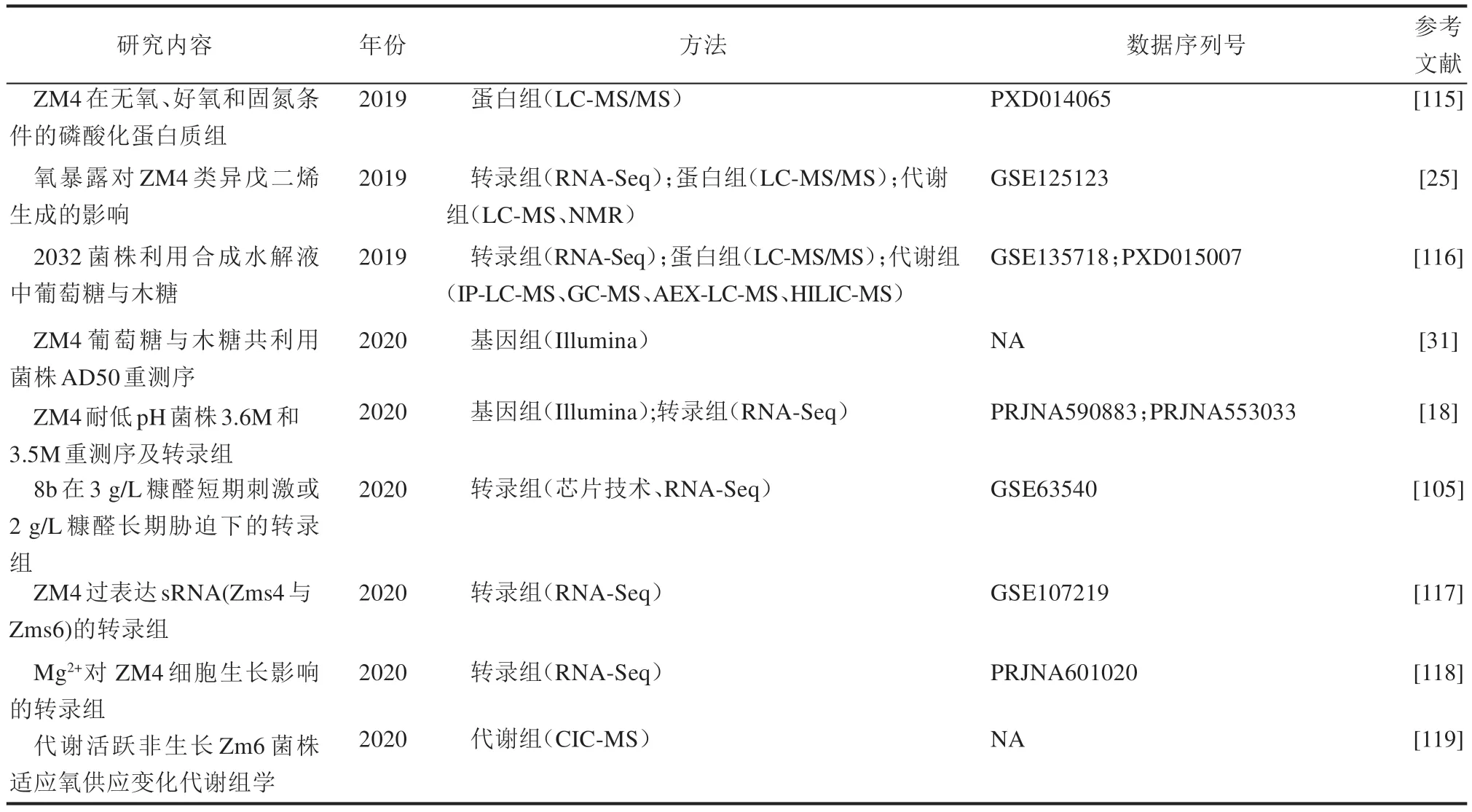
续表
随着NGS 技术的发展,基于RNA-Seq 的转录组学分析得以建立。与基于基因芯片的技术相比,RNA-Seq 获得的信息更多,基因表达的线性范围宽,偏好性更小[120],在运动发酵单胞菌中被逐渐使用,如被用来研究质粒基因在不同条件下的表达水平、氧气对代谢的影响、低pH 的耐受机制、氮源中镁离子浓度对细胞生长的影响等[18,25,99,118]。近期对RNA-Seq 和基因芯片分析两种技术在运动发酵单胞菌中进行了比较研究,对木糖利用菌株8b 在糠醛刺激与胁迫条件下的基因转录水平研究发现这两种转录组技术之间有比较高的相关性,均能用来表征基因表达水平,但RNA-Seq 技术能提供更多转录本结构上的信息[105]。
蛋白质组学与代谢组学相关技术也在运动发酵单胞菌研究中建立。基于全基因组磷酸化蛋白质组研究发现几乎所有ED 途径的酶在有氧情况下磷酸化增加[115],而代谢组学研究也证明了ED 途径在运动发酵单胞菌中为热力学有利的途径,其比大肠杆菌或酿酒酵母中的EMP 途径有近两倍的热力学优势[114];近期一项代谢组学研究表明,有氧培养条件下NADH/NAD+比值降低与磷酸化的糖酵解中间体浓度升高有关[119]。这些不同组学研究结果,促进了对运动发酵单胞菌在不同条件下中心代谢和细胞内氧化还原平衡机制的理解。
除了基因组、转录组、蛋白质组和代谢组分析方法单独使用外,多组学联合分析也被越来越多地用于对运动发酵单胞菌的研究[25,107,113-114,116,119],如比较基因组学与转录组学结合研究运动发酵单胞菌中突变菌株优异表型与基因型之间的关联。基于芯片分析的比较基因组杂交技术(comparative genome hybridization,CGH)与转录组学研究发现了突变菌株AcR乙酸钠耐受的表型,ZMO0117-ZMO0119之间的1.5kb 缺失导致了Na+/H+转运蛋白(NhaA)过表达,进而通过经典遗传学研究将乙酸钠耐受菌株AcR的基因型与表型关联,为工业菌株改造及突变基因型与表型的关联提供了范例[57],随后这一研究思路被用于五碳糖进化菌株以及耐酸进化菌株的基因型变化研究[18,51,112]。转录组、蛋白质组与代谢组等多层次组学技术联用,解释了Z.mobilisZM4 对6%(体积分数)乙醇胁迫反应的分子机制[103]。
目前很多的运动发酵单胞菌研究工作都联合使用了不同的组学技术,如联合使用转录组与蛋白组鉴定条件特异性基因[107],揭示Z.mobilisZM4抗多种抑制物的潜在生物标志物[113]等。近期结合转录组、蛋白组和代谢组等组学技术,以类异戊二烯为目标产物,发现暴露在氧气中会造成4-磷酸甲基赤藓糖醇代谢途径中铁硫辅因子短暂的氧损伤,细胞通过代谢重构将摄取的葡萄糖的18%转化为葡萄糖酸,并将产生的电子传递给电子传递链以降低氧化应激损伤的影响[25];相同的策略也被用于揭示运动发酵单胞菌对水解液中抑制物的多种响应及影响木糖代谢的可能原因[116]。
除此之外,相关的表型分析与表型组研究也有报道。2010 年基于特制96 孔板与Biolog 系统的表型芯片分析技术在Z.mobilisZM4 中得到使用,检测了ZM4 在2000 种不同条件下的表型,表明运动发酵单胞菌的生理特性与酿酒酵母更接近,而与通常的细菌偏离较大[11]。突变体库适应检测技术与表型组学研究也得以开展,美国加州大学伯克利分校Arkin 课题组通过转座子突变构建了带有DNA 编码的运动发酵单胞菌突变体库,通过后续的492 个不同实验检测确定了1586 个基因与水解液中的抑制物耐受相关,该结果表明89%的基因有明显的表型[121],为利用突变适应度对不同菌株进行功能注释提供了蓝图[111]。另外一项研究则利用Tn10 转座子突变体库,分析了运动发酵单胞菌耐热相关基因,发现耐热性机制可能与乙醇耐受机制部分重叠,且发现运动发酵单胞菌的耐热机制与E.coli和Arctocephalus tropicalis相似[122]。这些多组学数据以及表型相关的系统生物学数据联合分析,有利于理解生物过程及相关机制,使系统分析运动发酵单胞菌在不同条件下关键基因及调控因子的表达成为可能,加快菌株代谢工程改造靶点的发现,便于对运动发酵单胞菌进行再设计与改造,强化目标产品生产,同时这些数据与技术方法也在合成生物学DBTL循环的多个环节中发挥重要作用。
2.3 代谢模型的建立与完善
多种组学技术的应用和生理生化实验的开展积累了大量生物学信息,将数据抽象化进行数学建模是理性设计进行代谢工程改造的基础,可以用来预测和理解生物体对设计行为的响应。代谢网络模型的重建,尤其是基因组尺度的代谢网络模型,可以快速实现生物体在不同条件下代谢的假设分析,探索新的科学发现,为设计改造细胞提供全新思路[123]。目前,在包括大肠杆菌和酿酒酵母等多种模式生物中,都建立了高质量的基因组尺度代谢网络模型,得到了广泛的应用[123-125]。
迄今为止,运动发酵单胞菌已建立了5个基于不同约束方法和条件的中小型代谢模型和4个全基因组代谢网络模型[126-134]。此外,还有一个整合胞外代谢组学数据的全基因组代谢网络模型在美国能源部KBase数据库中完成,尚未公开发表。这些模型从不同角度对运动发酵单胞菌的相关生理特性进行了模拟,有助于更好地理解运动发酵单胞菌的细胞代谢,为代谢工程改造靶点提供新的设计策略和指导(表3)。
Pinto 等[135]在2008 年首先提出了运动发酵单胞菌基因组尺度代谢网络模型构建与数据质量及整合相关的问题和解决办法,但没有建立最终模型。随后韩国高级科学技术研究院Lee Sang-Yup团队[128]首次建立了运动发酵单胞菌基因组尺度的计量学代谢网络模型ZmoMBEL601,模拟了多种碳源的利用,进而通过基因敲除模拟,为有效生产琥珀酸提供了策略。之后又有研究者发表了两个基因组尺度代谢网络模型iZM363和iEM439[126-127],其中iEM439 为电荷平衡的模型,该模型通过动态通量平衡分析,提出能量与生长不耦联主要是因为能量用于泵出质子以维持pH[126]。韩国成均馆大学Lee Dong-Yup团队[136]基于基因组代谢网络模型iZM411(基于iZM363 更新)分析发现,敲除pdc(pyruvate decarboxylase)、ldh(lactate dehydrogenase)和pfl(pyruvate formate lyase)基因编码的丙酮酸消耗反应以及cl(citrate lyase)基因可使琥珀酸的摩尔产量增加15 倍,但这些基因组规模的代谢网络模型均无可计算的模型文件,不利于交流更新。最近,伊朗德黑兰大学Marashi和Moghimi课题组[129]基于前期已发表的基因组规模代谢网络模型和最新的注释、文献信息以及生理生化数据库,重新建立了一个更新版的基因组尺度代谢网络模型iHN446,这一模型修正了已发表模型的错误,并提供了可计算的SBML 文件,为后期基因组尺度代谢网络模型的改进奠定了基础。
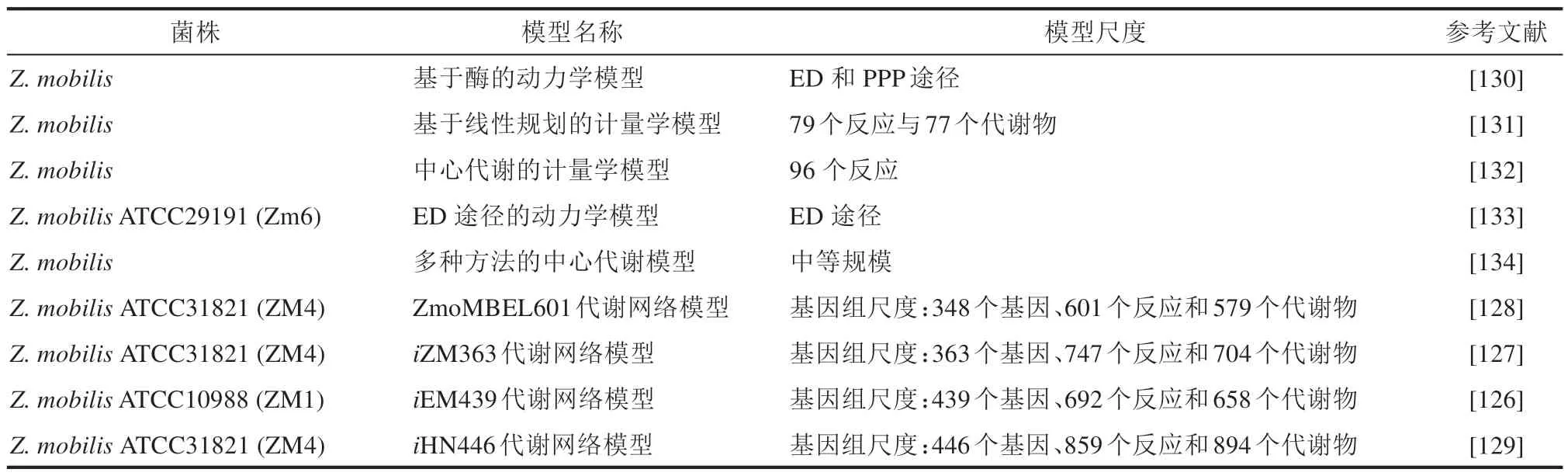
表3 运动发酵单胞菌不同尺度代谢模型Tab.3 Metabolic models at different scales established for Z.mobilis
除全基因组尺度的代谢网络模型外,由于研究目的不同,在运动发酵单胞菌中还建立了多个不同的数学模型。美国科罗拉多大学博尔德分校Kompala 课题组[130]根据运动发酵单胞菌的酶动力学数据建立了一个小型的动力学模型,重点研究了戊糖磷酸途径的异源酶与天然糖酵解途径的相互作用,为运动发酵单胞菌最大化利用葡萄糖和木糖,同时最小化表达外源酶,生产乙醇提供了思路。希腊化学工程科学研究所Klapa 团队[131]基于线性规划(linear programming,LP)构建了一个中小型计量学模型,根据其代谢网络的化学计量连通性,利用LP 对各种生物目标(包括能量最大化、乙醇和生物量生产率等代谢边界)进行了分析。
虽然这些模型可以指导实验设计,但这些简化的模型可能忽略了其他主要代谢途径细节而导致不太准确的预测。随着大量的组学数据的积累及计算领域的发展,将转录组学等系统生物学数据整合到代谢网络模型,构建特定环境的基因组尺度代谢网络模型成为可能[137]。例如通过整合基因组和已发表的转录组数据,江南大学刘立明课题组[138]建立了光滑念珠菌(Candida glabrata)基因组规模的转录调控网络,提高了光滑念珠菌的生长、代谢和基因表达对环境刺激调控行为的理解。蛋白组和代谢组等数据也可以被整合进入代谢网络模型,大肠杆菌最新的iML1515 模型全面整合了多种组学数据,为整合系统生物学数据建立高质量代谢网络模型提供了典范[124]。根据整合数据类型和优化的问题等因素可选择使用不同的算法,相关的数据整合方法可参考近期综述文章[137]。
运动发酵单胞菌研究积累了大量的系统生物学数据,这些数据资源与代谢网络模型的整合有助于建立高质量的全基因组尺度代谢网络模型,帮助系统理解运动发酵单胞菌相关表型现象,为代谢工程与合成生物学理性设计奠定基础[139]。在基因组代谢网络模型中,生物量组成方程十分关键,反映了参与细胞生长和维持生命所需成分的必需物质资源,一般由模拟细胞的实际物质组成决定,通常包括DNA、RNA、蛋白质、脂质和代谢所必需的小分子等[140]。通过通量平衡分析(flux balance analysis,FBA)方法模拟生长表型时,最常用的方法是使生物量目标函数(biomass objective function,BOF)反应通量最大化,以达到最好的模拟效果,而生物质组成与药物敏感性、营养需求和某一物种工业应用的生物合成潜力密切相关,其组成在不同条件下有所不同,因此有必要在不同条件下适当调整生物量组成方程,特别是有机物辅因子[141]。前期已发表的3 个运动发酵单胞菌基因组尺度代谢网络模型的生物量组成方程基本一致,但无法计算生成生物质。最近更新的iHN446 模型虽然生物量组成一致,但基于代谢物连通性做了一些反应更新,使得可以合成生物质[129]。在后期整合更多不同条件下的系统生物学数据、对不同条件下的细胞表型进行模拟时需要对生物量反应方程做出相应的调整,以提高整合系统生物学所建立新模型的准确性。
3 运动发酵单胞菌底盘细胞构建与合成生物学研究进展
3.1 遗传改造
在对运动发酵单胞菌进行研究的过程中开发了多种基因组改造遗传工具,可以分为随机突变和理性设计两类(图3)。随机突变改造属于正向遗传改造方法,通常包括物理诱变、化学诱变、转座子突变(transposon mutagenesis)和ALE 等,诱变后通过筛选得到特定的突变体。希腊雅典大学Typas 课题组[142]1997 年首次发现自杀载体上的转座因子可以用来突变运动发酵单胞菌染色体,进而成功利用微型转座子(mini Mu transposon)构建了稳定的运动发酵单胞菌营养缺陷型。随后其他类型的转座子Tn5、Tn10、Tn951和Tn1725以及插入元件ISZm1068[143]也相继被开发应用,相关进展已被综述[9-10]。
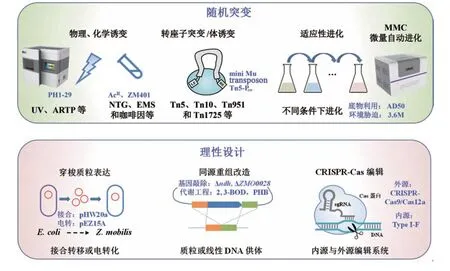
图3 运动发酵单胞菌中使用的遗传改造方法及代表性结果Fig.3 Genetic engineering methods developed in Z.mobilis and representative examples
亚硝基胍(nitrosoguanidine,NTG)、咖啡因(caffeine)和甲基磺酸乙酯(ethyl methane sulfonate,EMS)等化学诱变剂在运动发酵单胞菌中广泛应用,如高乙酸耐受菌株AcR及具有絮凝性状的菌株ZM401 即通过NTG 诱变筛选获得[144-145]。物理诱变方法除了常用的紫外诱变(UV)外,最近一种新型的物理诱变方法ARTP被应用于运动发酵单胞菌中,筛选出了可耐受pH 3.5的菌株PH1-29[51]。ARTP 介导的诱变具有快速、安全且更有效等优点,是传统诱变方法的有力替代。ALE 作为提高菌株生产性状最有效的诱变方法之一,在运动发酵单胞菌中也被广泛应用,并且得到了大量耐受增强或鲁棒性提高的菌株,例如印度理工学院Das课题组[31]通过ALE 筛选出目前葡萄糖与木糖共利用效果最好的菌株AD50,利用ALE 筛选优良菌株的进展可参阅近期综述文章[9-10,60]。何明雄团队[58]将基因组改组(genome shifting)技术用于运动发酵单胞菌,提高乙酸和糠醛耐受性,得到了可以耐受7 g/L乙酸和3 g/L糠醛的突变菌株。
理性设计是基于认知或从系统生物学数据学习推测而来的结论,将候选生物元件、逻辑线路和代谢途径转入底盘宿主细胞或直接对底盘细胞进行改造,通常会使用质粒过表达或基因编辑等方法实现。使用质粒载体进行内、外源基因的表达是最常使用且方便简单的方法,在运动发酵单胞菌中有大量的成功案例,且相关质粒构建组装方法如Gibson 组装和BioBrick 组装等也已经在运动发酵单胞菌中建立[5]。此外,通过Golden Gate方法建立了“One-POT”多片段组装方法,该方法将含有IIS 型限制内切酶BbsI 识别和酶切序列的特异性接头添加到启动子Padh2、终止子Tpdc和绿色荧光蛋白基因gfp序列两侧,实现了绿色荧光蛋白表达的定向组装并成功使绿色荧光蛋白得以表达,这一多DNA 片段无痕连接技术为在运动发酵单胞菌中多基因多途径组装奠定了技术基础[146]。
同源重组是基因组编辑中基因插入失活或基因替换的常用方法,在运动发酵单胞菌中被用于构建多株基因失活的菌株,如ndh失活菌株、sacC失活菌株和限制修饰(restriction and modification,R-M)系统失活菌株等[147-150],同时研究人员使用FLP 重组酶[151]或SacB 筛选的方法[54]消除引入的抗性基因。最近,美国威斯康星大学麦迪逊分校Kiley 课题组[152]在运动发酵单胞菌中开发了基于同源重组和质粒丢失的两步无痕编辑技术,成功敲除了编码乳酸脱氢酶基因(ldh)和纤维素合成酶操纵子(bcsABC),插入了链孢红素操纵子,实现了无抗性基因菌株的构建。
然而,这些方法步骤较为繁杂,时间周期长,而且编辑效率不高。CRISPR-Cas 技术作为近年发展起来的高效快捷基因编辑技术,已被开发作为运动发酵单胞菌基因编辑的方法之一。2017 年四川大学谭雪梅课题组[153]将CRISPR-Cas9 引入运动发酵单胞菌,利用从化脓性链球菌(Streptococcus pyogenesCICC 10464)中克隆的Cas9 基因,构建了pSUZM2a-Cas9 质粒,结合T7 启动子带动表达的单链RNA,消除了运动发酵单胞菌中的内源质粒。近期利用链霉菌(Francisella novicida)中的Cas12a(Cpf1)蛋白在Z.mobilisZM4 中构建了可诱导表达Cas12a 的重组菌株,可以完成RNA 引导的双链DNA 在靶位点的断裂切割,从而建立了一种高效方便的基因组编辑工具,利用该方法做到了内源质粒消除、基因敲除、基因插入和单碱基替换,该工具还用于代谢工程,在ZM4 中建立了乳酸生物合成途径[154]。
基于外源编辑酶的CRISPR-Cas 系统对宿主菌株具有一定的毒性,开发内源CRISPR-Cas 编辑系统是解决这一问题的有效方法。我们最近表征了Z.mobilisZM4 中内源I-F 型CRISPR-Cas 系统,建立了一种有效的基于I-F 型CRISPR-Cas 内源基因组编辑方法,实现了包括基因删除和替换(效率为100%)、原位修饰(100%)、大片段删除(>10kb)及同时进行多个基因编辑(18.75%)等多种基因组改造[155],提高了运动发酵单胞菌基因编辑改造效率,为合成生物学底盘细胞的构建提供了有力的技术支撑,也为其他物种内源CRISPRCas编辑系统的建立提供了范例[156]。
运用CRISPR-Cas 技术进行基因编辑后,编辑质粒的快速消除成为一个限制多轮基因编辑的瓶颈。传统上常用的编辑质粒消除方法是基于编辑质粒的非必需性或毒性,在无抗性培养基中连续传代,最终得到基因编辑后编辑质粒消除的菌株,但这一方法操作烦琐、效率低且耗时长,制约了CRISPR-Cas基因组编辑的高效、快速和连续应用。为了解决以上问题,不同的编辑质粒消除方法也在不断尝试完善,策略之一是将编辑质粒的复制子替换为温敏复制子,通过温度升高抑制温敏复制子的复制来消除编辑质粒[157]。通过运用逻辑线路来诱导控制编辑质粒gRNA表达,也是实现编辑质粒消除的可行策略,并在其他菌株中得到了应用[158],但目前的诱导型启动子系统具有不同程度的渗漏,通过突变筛选更加严谨的诱导型启动子或寻找新的诱导型启动子[159],以及建立结合转录与翻译多层次调控的逻辑线路[160],可能是实现编辑质粒快速消除的更有效策略。
3.2 多种穿梭质粒的构建与应用
作为基因工程改造的工具,质粒载体应用改变了分子生物学的发展,通过使用质粒将DNA 运输传递到宿主细胞中,使外源基因直接在质粒上表达或通过同源重组整合到宿主基因组,促进了代谢工程和合成生物学的发展[161]。质粒分为广泛宿主质粒和穿梭质粒两大类,在运动发酵单胞菌的研究中,已经构建并不断改进优化了多种可以在运动发酵单胞菌中使用的穿梭质粒,推动了运动发酵单胞菌研究的开展。
穿梭质粒构建最关键的两点是具有在大肠杆菌和运动发酵单胞菌中复制的复制子以及合适的筛选标记。穿梭质粒通常在大肠杆菌中构建和扩增,因此经典质粒的复制子常被采用,例如pUC系列及pBR 系列质粒的复制子[162-163]。运动发酵单胞菌中复制子的寻找一直是研究重点,常用的是对各菌株染色体复制子或内源质粒复制子的克隆和尝试,例如pSUZM1、pSUZM2和pSUZM3质粒的构建就分别采用了Z.mobilisZM4 菌株染色体和两个内源质粒上的复制子[163]。运动发酵单胞菌中可用的筛选标记主要分为两大类:抗性筛选和营养缺陷型筛选。由于运动发酵单胞菌对一些常用抗生素的耐受性较高,目前采用的抗生素有壮观霉素(Spe)、四环素(Tc)、氯霉素(Cm)与卡那霉素(Km),其中Km 需要在高于200 mg/L 浓度下使用[164-165]。营养缺陷型标记筛选的代表是ZM4的泛酸营养缺陷型,由于缺乏编码天冬氨酸脱羧酶的单一基因panD,使得该营养缺陷型菌株必须在添加有β-丙氨酸的培养基中或者异源表达panD基因的情况下才可以正常生长,这一营养缺陷型的发现有助于在运动发酵单胞菌中通过营养缺陷型标记筛选突变菌株[166]。
目前应用于运动发酵单胞菌的质粒主要有两类来源:一种是将来源于大肠杆菌和运动发酵单胞菌的质粒进行整合,得到一个可以同时在大肠杆菌和运动发酵单胞菌复制的穿梭质粒,这样的质粒可以在这两种宿主中存在并繁殖[167-168],但是这类质粒往往因为骨架太大,导致转化效率不高,在早期研究中使用偏多[169];另一种是基于革兰氏阴性细菌的宿主广泛性质粒[33,136,170-172],这类质粒含有在革兰氏阴性细菌内均可以识别的复制子及mob基因,可以采用接合转化法将其转化至运动发酵单胞菌中[57,152],如常用的pBBR1MCS-2 质粒。近年来,根据不同研究目的,在运动发酵单胞菌中构建了一系列穿梭质粒。随着对质粒及抗性基因研究的深入,质粒越来越小,例如目前广泛使用的pEZ15Asp合成穿梭质粒,其骨架大小仅为3 kb,含有在大肠杆菌和运动发酵单胞菌复制的复制子,壮观霉素抗性筛选基因以及BioBrick酶切位点[76],方便了遗传操作与代谢途径构建[21,105,162,173-179]。
3.3 影响转化效率的因素及转化效率的提高
高效遗传转化方法在基因工程改造与基因组精简改造中起着不可或缺的作用,研究人员在运动发酵单胞菌中建立了多种遗传转化方法,常用的是接合转移法和电转化法[5,180]。早在20 世纪80 年代,研究人员就开发了可以在运动发酵单胞菌中使用的接合转移转化法,但是该方法费时且转化效率有菌株特异性[180-181]。2011 年上海交通大学钟建江课题组[182]设计了一种新型载体pHW20a,将接合转移的转化效率提高了两个数量级,且较之前用于接合转移的质粒载体具有更好的稳定性及操作性。随着电转化技术在其他菌株中的应用,研究人员也在运动发酵单胞菌中建立了简单方便的电转化方法,其转化效率在103CFU/μg量级[183]。
低转化效率不利于重组转化子的获得及相关突变体文库的构建,澳大利亚新南威尔士大学Rogers课题组[184]曾尝试改变电转电压等电转参数及电转后的复苏培养基与复苏时间以提高转化效率,但未获成功。影响转化效率的因素是多样的,不同遗传转化技术的转化效率不同,同一技术(如电转化法)不同参数的选择、感受态细胞制备的质量(如细胞收集时期、制备方法和最终细胞浓度等)以及转化后复苏培养基的选择等都会带来转化效率的差异。山东大学张友明课题组[185]使用室温方法制备大肠杆菌感受态细胞,发现其转化效率比在冰上制备的感受态细胞高出10 倍,这一方法也在蜡样芽孢杆菌(Bacillus cereus)中得到验证[186],说明该方法值得在包括运动发酵单胞菌在内的其他菌株中进行尝试。此外,细胞的物理屏障如细胞壁或特殊的膜结构也可能妨碍电转化效率,如在制作感受态细胞时使用溶菌酶处理阪崎肠杆菌(Cronobacter sakazakii)可以提高其电转化效率[187]。运动发酵单胞菌特殊的细胞膜组分中高含量藿烷在提高其对外界环境耐受的同时,或许是影响其转化效率的一个因素,在未来的研究中可以作为研究方向寻找转化效率的突破,如在制备感受态细胞时尝试降低藿烷成分等。
限制修饰R-M 系统可以保护细菌免于外来DNA 的入侵,即可以破坏外来DNA 在体内的存留[188]。对R-M 系统的破坏,特别是核酸内切酶基因的缺失,使得外源DNA 进入细胞后不会由于核酸内切酶的识别而被大量的切割而降解,从而可以改变细菌对外源DNA 的摄取能力。运动发酵单胞菌前期研究表明,对R-M 系统相应基因进行敲除会影响质粒的电转化率[149-150]。进一步研究表明,转化效率受到R-M 系统的影响,不仅仅是转化方法与过程等转化条件参数。为了防止质粒受到宿主R-M 系统的限制,Rogers 课题组[170]在Z.mobilisZM4 的转化过程中使用I 型限制酶抑制剂,发现可以小幅度提高转化效率,但其转化效率仍处于103CFU/μg 量级。进一步研究发现将运动发酵单胞菌中IV 型R-M 系统编码甲基转移酶和限制性内切酶的基因mrr(ZMO0028)失活,可使甲基化质粒的电转化效率提高60 倍,且不影响生长率和生物量;而使R-M 系统I 型负责识别未甲基化或错误甲基化DNA 的S 亚基编码基因ZMO1933失活,可使非甲基化质粒的电转化效率提高30 倍,但会影响细胞生长[150]。何明雄团队[149]的研究结果表明,在突变株Zmmrr和Zm1933中,甲基化穿梭质粒pBBR1MCS-tet 的电转化率分别提高了17 倍和2 倍。白凤武课题组[54]在ZM4 与絮凝菌株ZM401 中实现了ZMO0028和ZMO1933的双敲除,使得甲基化和非甲基化质粒pHW20a 的电转化效率均大幅提升,且双敲除菌株的生长与乙醇生成以及ZM401 的絮凝性能未受影响。
这些研究结果之间可能因为使用的质粒大小与浓度不同存在一些差异,但可以确定的是运动发酵单胞菌中R-M 系统会影响甲基化与非甲基化外源质粒的电转化效率,可以通过控制内源R-M 系统或者外源DNA 甲基化状态提高转化率。另外值得一提的是,我们在研究Z.mobilisZM4 内源CRISPR-Cas 系统的过程中发现,用pEZ15Asp 载体转化ZMO0028基因敲除菌株ΔZMO0028时,转化效率为野生型菌株的90 倍,同时在ΔZMO0028的背景下所有基因组编辑操作的效率都得到了显著提高,单基因敲除的编辑效率均达到100%(16/16),而大于10 kb 片段敲除的编辑效率从12.5%(2/16)提高到50%(8/16),表明R-M 系统可能不仅影响外源DNA 的转化效率,而且影响CRISPR-Cas 技术相关的编辑效率[155]。
3.4 生物元件的预测与鉴定
在合成生物学的“设计”和“构建”环节中,编码和非编码区域的模块化生物元件信息是生物系统设计的重要基础。在运动发酵单胞菌中,前期通过经典的分子生物学方法研究确定了一些启动子[180]。基因组序列的发表以及大量系统生物学数据的积累为生物功能与调控元件的挖掘及鉴定奠定了基础[5,60]。sRNA 作为一种反式作用元件,在基因表达的多个过程中发挥作用,如细胞在氧化应激、乙醇、温度或pH 变化等胁迫条件下调节多种代谢途径[189]。美国得克萨斯大学奥斯汀分校Contreras 课题组[110]通过sRNA 转录组学及计算机预测分析,发现了运动发酵单胞菌15 个新的sRNA,并且通过实验研究表明其中Zms2、Zms6和Zms18 与调节应对乙醇胁迫相关。该课题组[190]也通过高通量测序技术探索sRNA 可能的作用机制,结合已有转录组学数据,开发了运动发酵单胞菌REFINE(RNA-seq examiner for phenotype-informed network engineering)系统,为理解sRNA与其他生物元件之间的关系与作用机制及其在代谢工程实践中的应用提供了有效工具。
转录本内的5'UTR 可以在各种生物体中参与mRNA 表达水平的调控,以应对各种代谢物或环境条件变化,例如sRNA 会与目标基因的5'UTR相作用,调控下游基因的表达[191]。Contreras 团队与作者课题组合作,利用生物信息学手段对转录组数据进行分析,发现了运动发酵单胞菌中36 个5'UTR 可能与乙醇、乙酸或木糖胁迫相关,其中UTR_ZMO0347 被证明可调控其下游基因hfq的表达,使其在乙醇胁迫条件下的表达下调[192],这一结果为研究运动发酵单胞菌在胁迫环境下细胞响应调控网络提供了新的思路。进一步研究揭示了运动发酵单胞菌中sRNA-sRNA 及sRNAprotein 之间相互作用的调控网络,解析了两个影响乙醇生成sRNA(Zms4 和Zms6)的共调控网络[117]。该研究在运动发酵单胞菌中首次揭示了sRNA 作用网络,通过一组不同的mRNA 靶点和大量的sRNA 协同,调控多个参与乙醇代谢相关的通路。
最近,基于系统生物学数据及双荧光报告基因系统对运动单胞菌进行生物元件预测、筛选与鉴定的策略得以建立。通过流式细胞仪检测包括EGFP、mCherry和RFP等5 种报告基因的荧光强度,建立了基于流式细胞术的双荧光报告基因系统:该系统含有EGFP和opmCherry两个报告基因,其中EGFP用以表征待测的调控元件,opmCherry作为内参以消除内外噪声;利用该系统进一步表征了基于系统生物学数据预测和生物信息学方法分析预测的38 个启动子和4 个核糖体结合位点(ribosome binding site,RBS)的强度,实验结果与系统生物学预测的相关性较高[21]。与早期着眼于重要功能基因启动子的研究[193]不同的是,基于系统生物学数据可以预测筛选不同强度的启动子,供不同的需要使用。这一研究为利用系统生物学数据筛选潜在调控元件以及使用双荧光报告基因系统定量鉴定调控元件提供了新的策略与方法,我们利用这一策略筛选鉴定了3个内源乙醇诱导性启动子[159]。此外,双荧光报告基因系统还被用于表征sRNA 与靶基因5'UTR 之间的相互作用[117]。
美国威斯康星大学麦迪逊分校Landick 课题组[194]近期利用RNA-Seq、TSS-Seq、Term-Seq与Ribo-Seq 等多组学技术系统鉴定了运动发酵单胞菌转录起始位点和转录终止位点,并分析确定了运动发酵单胞菌中σA启动子−10 和−35 区域的保守序列为TANNNN 和TTGNNN。与大肠杆菌的−10 区域保守序列相比,运动发酵单胞菌的−10 区域缺少在大肠杆菌中的T-7位点,这与新月柄杆菌(Caulobacter crescentus)相似,说明大肠杆菌−10 区域的T-7位点不具有普遍性。这些转录调控系统研究结果可结合双荧光报告基因系统,定量化鉴定运动发酵单胞菌非编码区域的相关生物调控元件与逻辑线路,增加可用于合成生物学研究的生物元件与逻辑线路的种类与数量,推动运动发酵单胞菌在合成生物学领域的进一步发展。
4 运动发酵单胞菌底盘细胞构建展望
4.1 底盘细胞构建
运动发酵单胞菌具有基因组小、遗传操作工具基本完备、安全性高、环境胁迫耐受性强及副产物低等特点,已作为底盘细胞用于开发包括纤维素乙醇在内的多种平台化合物的生产(图1,表1)。碳、氮作为地球生物化学循环的重要组成部分,也与地球的能源与环境相关,利用生物进行碳、氮的固定具有重要意义。
运动发酵单胞菌具有完整的Mo 固氮酶系统,可以利用氮气为唯一氮源生长并发酵生产乙醇[12-13]。天然生物固氮过程受到复杂的调控作用,并且进化出了独特的氧保护机制,虽然目前关于固氮的合成生物学有了一定的进展,但还属于理论探索阶段[195]。我们在开发运动发酵单胞菌内源I-F 型CRISPR-Cas 系统时成功敲除了固氮基因簇中的10kb 基因组片段(ZMO1815-ZMO1822),敲除菌株生长未受到明显影响[155]。美国威斯康星大学麦迪逊分校Amador-Noguez 课题组[115]提出了运动发酵单胞菌可能的固氮调控机制,为固氮调控的研究奠定了理论基础。利用运动发酵单胞菌基因组小及遗传操作系统成熟等优势,将其作为底盘细胞推进固氮系统中氧保护机制、固氮基因簇简化、固氮相关催化元件和调控元件的挖掘与鉴定等固氮合成生物学的研究,是未来值得拓展的研究方向之一。
在运动发酵单胞菌能够固氮的前提下,引入外源的固碳途径,将有可能实现氮气和二氧化碳的共同利用,利用合成生物学的手段创造出一种目前自然界尚不多见的可同时固定碳、氮的微生物。过去50 年,人类活动使大气中CO2的浓度从300 μL/L 以下上升到了413 μL/L,预计到2045 年会上升至500 μL/L[196]。CO2浓度的不断升高,将使全球温度上升,导致冰山和积雪的融化,从而引起海平面的上升,严重影响全球气候和环境。利用生物固定CO2进行生物能源的生产或绿色生物制造,有利于降低大气中CO2浓度及由此带来的各种不利影响。最近以色列魏茨曼科学研究院的Milo 研究团队[197]通过外源引入卡尔文循环中的Rubisco 酶同时共表达磷酸核酮糖激酶(phosphoribulokinase,Prk)对大肠杆菌进行改造,并通过实验室进化使其可以利用CO2为唯一碳源。蓝藻中固碳羧化体相关基因及蛋白结构的确定,使其得以在大肠杆菌中实现人工合成,也为固定二氧化碳提供了另一种思路[198-200]。北京化工大学谭天伟教授课题组[201]提出了基于CO2为底物,进行第三代生物炼制的绿色生物制造概念,系统分析了目前生物CO2固定途径和细胞能源供应新模式的两大关键问题,为细胞工厂工程化改造提出了前瞻性的指导意见。
运动发酵单胞菌本身拥有完整的固氮基因簇,利用内、外源CRISPR-Cas 基因编辑技术以及双荧光报告基因生物元件鉴定系统等研究工具将运动发酵单胞菌作为底盘细胞进行改造,使其利用二氧化碳和氮气生产目标化合物有很大的潜力(图4)。需要指出的是,碳、氮的固定都是高耗能的生物过程,而运动发酵单胞菌的氧化磷酸化产能效率较低,在此过程中,能量的供应将是一个具有挑战性的难点。
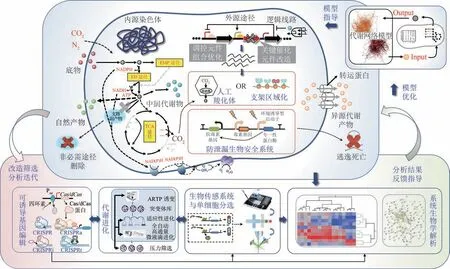
图4 运动发酵单胞菌底盘细胞构建展望Fig.4 Perspectives on developing Z.mobilis as a chassis cell
4.2 CRISPR-Cas技术的发展
CRISPR-Cas 技术可以快速实现基因组的编辑,是目前广泛使用的高效基因组编辑技术。2017 年美国科罗拉多大学博尔德分校Gill 课题组[202]开发了CREATE(CRISPR-enabled trackable genome engineering)技术,可实现对基因组上多基因和多位点的同时编辑,且便于追溯和追踪基因型与表型的关系,进一步提升了编辑效率。天津大学王智文课题组[203]将CREATE 技术用于编辑改造大肠杆菌全局调控因子,并与转录组分析结合研究大肠杆菌的耐药性。这些研究展现了CRISPR-Cas 技术多基因同时编辑的能力,但由于CRISPR-Cas 技术可编辑位点受到PAM 位点的限制,制约了基因组编辑位点的选择。近期报道的两种人工改造的Cas 酶(SpG 和SpRY)中的SpRY几乎可以不受PAM 位点序列限制进行编辑,这一突破极大扩展了CRISPR-Cas 系统的编辑能力,同时也为其他类型CRISPR-Cas 系统的改造提供了良好的思路[204]。利用编码在转座子中的CRISPR-Cas系统,研究人员实现了高效特异的DNA 片段插入,为利用CRISPR-Cas 技术的基因插入操作提供了又一高效解决方案[205-206]。
基于CRISPR-Cas 开发的多种CRISPRi/a 基因沉默或激活方法也被广泛使用。湖北大学张桂敏课题组[207]在枯草芽孢杆菌中建立了dCas9-α/ω 系统,通过设计位点特异性gRNA可实现同时激活和抑制不同基因的表达,展现了CRISPR-Cas 技术多基因、多方向和多维度同时精准调节基因表达的强大能力,为其在其他物种中的扩展应用提供了借鉴。除了作为基因表达调控的工具,CRISPRCas 还可以用来进行基因组尺度的基因型鉴定[208]。清华大学邢新会和张翀课题组[209]建立了基于CRISPRi的全基因组高通量基因功能研究技术,该技术通过建立全基因组gRNA文库,高效研究包括转录非编码RNA(non-coding RNA,ncRNA)在内全基因组所有基因的功能。Arkin 课题组[210]最近发表的预印本研究表明,通过构建一个紧凑和全面的CRISPRi 文库(约33 000 个sgRNA),可覆盖基因组的所有元件,包括蛋白编码基因、ncRNA、启动子和转录因子结合位点等。天津大学宋浩课题组[211]最新发表的预印本将CRISPRi与组学分析相结合,确定在多个细胞过程中起作用的潜在目标基因,提升E.coli中脂肪酸的产量,对得到的56 个有益基因进行组合改造,使得脂肪酸最终发酵产量达到21.6 g/L,为目前E.coli重组菌株中最高。
这些基于CRISPR-Cas 发展起来的技术方法,与系统生物学或高通量检测等其他技术结合后,如CREATE 与转录组学的结合[203],会进一步释放其强大的功能,是将来菌株改造的发展方向。目前运动发酵单胞菌中的内、外源CRISPR-Cas 技术均已建立[153-155],尤其是内源系统的建立会减少外源编辑酶对细胞的毒性,实现快速的基因编辑,目前已有少量相关研究开展[156]。但是,内源系统较为复杂,发挥作用时需要多个相关蛋白的辅助作用,可能会影响基于内源系统的CRISPRa/i或单碱基编辑工具的开发,所以应借鉴以上优秀应用实例,同时利用内源和外源CRISPR-Cas 体系开发完善运动发酵单胞菌基因编辑工具,为CRISPR-Cas 技术在运动发酵单胞菌中的进一步应用提供可能。
4.3 高效筛选平台的建立与应用
合成生物学细胞工厂设计研究过程中会有大量中间突变体产生,快速高效分选到理想菌株十分关键。传统筛选方法往往以目标代谢物为主,通量低、效率低、费时、费力且昂贵。高通量或自动化筛选平台与检测技术的建立,可以显著提升工作效率,减少人为误差,提高准确性。目前,已在运动发酵单胞菌中建立了基于Bioscreen C 的微量生长检测系统,可同时鉴定200种不同条件对菌株生长的影响[212];基于Biolog 表型芯片技术可快速进行表型鉴定,一次最多可同时进行48 块96孔板的鉴定[11]。此外,基于微流控的微生物液滴培养仪(microbial microdroplet culture system,MMC)全自动传代筛选技术也在运动发酵单胞菌中建立,可用于改造后优势菌株的快速自动化进化筛选[213]。
除了基于细胞生长等表型进行筛选的技术外,近年来,基于生物传感的高通量筛选技术也被广泛开发利用,美国伊利诺伊大学厄巴纳-香槟分校赵惠民课题组[214]利用关键代谢物水平转换为荧光信号的遗传传感器,应用酿酒酵母中的丙二酰辅酶A 传感器FapR 及其相应的操纵子fapO,监测活细胞内丙二酰辅酶A 的水平,为酵母的高通量遗传筛选提供了有效的工具。江南大学李江华和张娟课题组[215]利用赤藓糖醇反应性转录因子EryD,在解脂耶氏酵母构建了能够在动态范围特异性响应的传感器-调节器系统,用于快速筛选和鉴定突变体文库,构建了基于生物传感的赤藓糖醇高通量筛选方法。上海交通大学冯雁和杨广宇课题组[216]利用细胞膜表面半乳糖透酶(LacY)对底物及糖基化产物通透性的差异建立了首个针对岩藻糖基化酶的单细胞超高通量筛选技术平台。这些基于生物传感器的高通量筛选方法是目前在运动发酵单胞菌中所缺乏的,相关方法的建立可广泛用于运动发酵单胞菌突变体和遗传文库的筛选、微生物生理学研究、发酵条件优化和系统生物学研究,加快生物元件、逻辑线路、代谢途径及细胞工厂性能的检测鉴定。
单细胞精度的快速分选也是高通量筛选平台的重要构成[217]。基于流式细胞术的荧光激活细胞分选(fluorescence-activated cell sorting,FACS)技术在微生物代谢工程中应用较多,但由于技术限制,利用FACS 进行细胞分选的结果尚存在较高的背景噪声,尤其是应用于细胞大小为10 μm 以下的原核细胞时,无法高效实现高精度的单细胞分选,往往需要在分选后再次通过孔板或平板筛选。另外,FACS 技术只能根据细胞内或细胞表面的荧光信号进行分选,不能根据胞外物质进行分选。基于微流控技术的荧光激活液滴分选系统(fluorescence activated droplet sorting system,FADS)则可以根据胞外分泌物进行分选,并方便与其他检测技术整合[218]。
中国科学院青岛生物能源与过程所徐健团队通过耦合单细胞拉曼光谱和液滴微流控技术,开发了拉曼激活单细胞液滴分选技术(Ramanactivated single-cell droplet sorting,RADS),该技术能够在在无标记、无损伤的前提下进行单细胞精度的分选。以生产虾青素的雨生红球藻为研究对象,筛选通量可以达260 个细胞/min,分选准确率高达98.3%,且分选出来的细胞92.7%能够正常分裂增殖[219]。近期,该团队基于自主研发的介电单细胞捕获/释放的拉曼激活液滴分选技术pDEPRADS研制了高通量流式拉曼分选仪FlowRACS,可以实现基于分子光谱、非标记式、单细胞精度、高通量流式的酶活筛选[220]。该团队近期还开发了一种高覆盖度的单细胞拉曼分选测序技术(Ramanactivated gravity-driven single-cell encapsulation and sequencing,RAGE-Seq),可实现对单个细菌细胞的分选与测序,覆盖度>95%,意味着可以将单个细菌细胞的高覆盖度基因与代谢表型直接关联,为细菌单细胞水平的基因型与表型的关联提供了技术支持[221]。另一个值得关注的是使用光激活细胞成像和分类技术的伯克利之光(Berkeley Lights‘Beacon’)。细胞在该系统中被隔离培养在体积为皮升的纳米流体设备“笔”上用于生长和分析,每个芯片包含多达14 000 支“笔”,其中容纳了许多单细胞或菌落,通过荧光定量产物及标准光学技术进行细胞计数,该技术可以在纳米流体设备上对单细胞进行克隆分离、细胞选择和适当的细胞编辑等操作[222]。
包括运动发酵单胞菌在内的原核生物,对单细胞精度分选的要求更高,以上这些高通量自动化技术的扩展应用,特别是单细胞精度的细胞分选及基于生物传感的高通量筛选在运动发酵单胞菌中的应用,将大大提升运动发酵单胞菌候选菌株的分离筛选效率,推动运动发酵单胞菌高效筛选平台的建立以及在单细胞水平的研究。
4.4 精准代谢调控的建立
原核生物中有多种精细调控机制,保证其对环境良好的适应性,包括基因水平、转录水平、转录后水平、翻译水平和翻译后水平调控等。在传统的代谢工程应用中,以使用启动子在转录水平和RBS 在翻译水平的调控居多,即使用特定强度的启动子或RBS 对特定的基因进行调控。最近,研究人员提出了在翻译层面新的调控机制。江南大学陈坚课题组[223]通过改变基因N 端编码序列的45 bp 碱基以调控基因的翻译起始效率,应用于N-乙酰神经氨酸(N-acetyl-neuraminic acid,NeuAc)的合成调控,成功将NeuAc 的产量提高了3.21 倍。中国科学院天津工业生物技术研究所张学礼团队[224]首次提出了一种利用起始密码子组合调控(combinatorial modulation of initial codons,CMIC)的新策略,在翻译层面调节优化代谢途径,将其应用于大肠杆菌中玉米黄素合成途径的3 个基因crtZ、crtY和crtI,得到一株玉米黄素产量为出发菌株10倍的重组菌株,CMIC策略只需改变3 bp的序列,将缩小库容量,从而减小工作量和缩短实验时间。
以上这类代谢调控一般称为静态调控,即无需感知细胞内外的环境,直接使用组成型元件对调控基因进行过表达或抑制,甚至还可以直接敲除,但这一过程可能会造成胞内稳态环境的破坏,且无法对细胞内外环境的改变作出即时的响应,因此研究人员开发了动态调控策略,即通过感应相关信号分子,如外界诱导信号或体内代谢物水平,以动态调节代谢通路中基因的表达水平[225],如大肠杆菌中被深入研究的多种诱导型启动子系统,包括乳糖操纵子、色氨酸操纵子以及来源于λ噬菌体的T7转录系统和PLPR-Clts857 温敏启动子系统等[226]。这类系统在发酵工业中得到很好的应用,例如内蒙古农业大学职业技术学院曹志军课题组[227]将PLPR-Clts857 温敏型启动子系统用于两步法发酵生产丙酮酸,发酵前期在40 ℃下使温敏开关启动noxE基因的表达,以消耗NADH 促进生物量和丙酮酸的积累,而后在34 ℃关闭开关抑制细胞生长,提高了丙酮酸的产量,最终使丙酮酸的产量达到了93.0 g/L。
除了高温诱导调控外,近期清华大学陈国强课题组和中国科学院微生物所娄春波团队合作开发了冷诱导型开关系统,该系统使用一种热失活的蛋白酶和一种冷失活的TEV 敏感转录因子在转录和翻译后两个水平调控基因的表达,且为了提高系统的严谨性,研究者还向系统引入了一个额外的蛋白水解模块,以专门降解残留蛋白或由于泄漏表达的蛋白,该工作建立了一个高性能的冷诱导系统,用于严格和快速控制基因表达,使表达热不稳定重组蛋白成为可能,同时该系统在基础研究、工业和生物医学应用方面也具有巨大的潜力[160]。基于CRISPR-Cas 的可诱导控制的基因编辑、激活与干扰系统也被实现[211,228-229],如通过使用诱导型启动子控制dCas9的表达或光诱导控制gRNA 的活性[230],为动态控制基因实现时空调控提供了又一强有力的工具。最近,研究人员利用光敏核苷酸替换了部分gRNA序列,通过光来实现对DNA 序列的切割,从而实现基因编辑在时间和空间上的调控,该技术被称为cfCRISPR,为靶标DNA精准切割的时空调控提供了参考[231]。
除了人工诱导动态调控系统外,细胞自主诱导调控系统也被逐渐开发应用。日本金泽大学Tsuge 课题组[232]在谷氨酸棒杆菌中开发了一个利用厌氧特异性启动子调节碳代谢流的系统,使用乳酸脱氢酶基因(ldhA)的厌氧特异性启动子替换葡萄糖-6-磷酸异构酶基因(pgi)的启动子,实现了在有氧条件下碳流重定向到PPP 途径生成大量NADPH 供1,5-戊二胺(1,5-diaminopentane)生产使用的氧响应开关,使1,5-戊二胺的产量、收率、生产速率分别提高了4.6 倍、4.4 倍和2.6 倍,在有氧生长后pgi会自动在厌氧条件下被诱导,使碳流转向糖酵解途径,继续生产琥珀酸,这一方法使通过单一菌株两步发酵分阶段生产不同化合物成为可能,降低了生产成本。中山大学刘建忠课题组[233]在大肠杆菌中使用群体感应(quorum sensing,QS)系统动态调控4-羟基苯乙酸(4-hydroxyphenylacetic acid,4HPAA)代谢途径,使4HPAA产量达到17.39 g/L±0.26 g/L。
在运动发酵单胞菌中关于静态调控相关的研究和使用相对较多,如常用的强启动子Pgap、Ppdc、Peno和PadhB等,通常用于过表达目的基因,以调控目标产物的产量或提高对抑制物的耐受性[5,60]。Contreras 教授与作者课题组合作在解析sRNA 和5'UTR 的调控方面开展了相关研究[110,117,190,192],并于最近提出了sRNA-sRNA 及sRNA-protein 之间相互作用的调控网络[117]。关于运动发酵单胞菌动态调控方面的研究目前尚少,成功应用的有四环素诱导型启动子Ptet,该诱导型启动子首次在运动发酵单胞菌中应用于调控2,3-丁二醇的生产[76],随后也被用于双荧光报告基因的构建[21]和异丁醇生产途径的调控[63]。最近,Landick课题组[234]报道了使用lacIq基因和PT7A1-O34启动子在运动发酵单胞菌中实现了对发酵代谢途径中的关键酶丙酮酸脱羧酶(Pdc)基因的诱导控制,达到了对运动发酵单胞菌碳流控制的目的。
在运动发酵单胞菌中建立精准的代谢调控,将有利于更好地在时间和空间上调控相关代谢途径,发挥其所拥有的优势性能。基于组学数据筛选与双荧光报告基因系统鉴定的策略[21],筛选鉴定了运动发酵单胞菌中内源乙醇诱导型启动子,但对其具体诱导机制及应用还需进一步研究[159]。在其他菌株中开展的相关研究及进展,为填补运动发胞单胞菌在这一领域的不足提供了研究思路,是未来值得投入的研究方向。
总之,运动发酵单胞菌具有安全、环境适应性强、糖及乙醇耐受性高、可在厌氧条件下通过ED 途径进行乙醇发酵、糖代谢速率快及乙醇收率高等优点,已经在生物能源、平台化合物、食品和药品等不同工业领域取得了一系列成果。同时,运动发酵单胞菌基因组小,具有一系列包括内、外源CRISPR-Cas 基因编辑系统在内的遗传工具及系统与合成生物学研究方法,积累了大量的生理、生化及系统生物学数据,有望发展为一个高效的工业底盘细胞,尤其是基于丙酮酸为前体物质生产多种平台化合物的高效细胞工厂。但是,我们仍需在目前研究较少的基因时空表达网络与动态调控机制等基础理论研究以及高通量基因编辑与自动化筛选技术等方面取得更大突破;进一步理解制约遗传转化效率的分子机制,建立提高转化效率的方法策略;揭示影响基因组编辑效率的因素,建立可循环开展对基因组多位点同时编辑的高效CRISPR-Cas 基因编辑体系及单碱基编辑方法;结合生物信息学及机器学习等方法从多组学数据中挖掘生物元件,优化包括双报告基因系统在内的生物元件定量检测体系,丰富生物催化与调控元件库;建立生物元件、逻辑线路、代谢途径高通量组装与性能检测方法;整合运动发酵单胞菌生理、生化及多组学数据,建立数字细胞模型,指导包含防泄漏生物安全体系在内的安全、高效细胞工厂的理性设计及应用。
——一道江苏高考题的奥秘解读和拓展

