生物分子序列的人工智能设计
王也,王昊晨,晏明皓,胡冠华,汪小我
(清华大学自动化系,合成与系统生物学研究中心,教育部生物信息学重点实验室,北京信息科学与技术国家研究中心,北京 100084)
随着合成生物学与生物信息技术的迅猛发展,促成了生命密码从对自然的探索到人工合成的质变[1],使得人工分子的设计与合成生物系统的构建成为了可能。近年来人工合成的生物分子序列,例如药物小分子、DNA调控元件、蛋白质分子等,在医疗[2-4]、化工[5-6]、农业[7]等领域有着广泛的应用[8]。早期的生物序列设计手段主要聚焦于对天然序列进行随机突变[9-10]或者基于功能模块的组合进行筛选[11-12],存在一定的局限性:一方面,潜在的序列随着序列长度的增加构成了一个指数增长的空间。以DNA 调控序列为例,仅100 个碱基长度的DNA 序列的潜在的碱基组合达到了4100,即存在约1060种潜在的待测序列,远超出目前任何实验文库(约106~8复杂度)的筛选能力[13]。蛋白质序列由于氨基酸的丰富组成,潜在的序列空间更广阔,同时还需考虑折叠构象等复杂约束,通过局部的修改来获得新功能十分困难[14]。另一方面,由于人工突变后的序列与天然序列间存在很高的相似度,易与宿主细胞产生相互影响,通过随机突变的搜索方式难以保证合成生物系统的绝缘性和遗传稳定性[11]。
近年来,人工智能技术的迅猛发展为生物序列的智能设计提供了新的机遇。由于生物数据本身的高维特性以及数据中隐含模式的复杂性,深度学习算法在挖掘重要生物学特征、探求特征之间隐含的复杂关系等方面表现出了独特的优势[15]。随着各类生物组学数据的不断积累,基于深度学习的预测模型在生命科学领域已展现出广阔的应用前景[16]。如在处理基因组数据场景下DNA 序列motif 的识别发现[17],基因元件相互作用的预测[18],基因表达量预测[19-20]以及基因调控网络的预测等[21]。
在人工智能研究领域,以变分自编码器(variational auto-encoder,VAE)[22]、生成对抗网络(generative adversarial network,GAN)[23]等为代表的深度生成式模型的研究近年来取得了重大突破。深度生成式模型可以从高维数据样本中提取重要的特征与特征组合规律,并据此生成海量的全新样本,在图像、音频数据的生成中已取得了重大进展[24-26]。基于人工智能的设计模型,已逐渐被应用于药物研发[27-28]、对未知化学反应的探索[5]等方向,成功实现了小分子药物[29-30]、基因调控序列[31]新型人工蛋白质以及基于CRISPR 编辑技术的guide RNA设计[32-34]等的合成设计[35-36]。
不同于传统的设计手段在天然序列的局部进行小范围探索,智能算法可提取生物数据的复杂特征并与寻优算法相结合,利用生物特征的低维表示,针对特定生物学功能进行定向优化[37]。通过对潜在序列空间的探索与寻优,实现生物序列的智能化、自动化设计(图1)。在降低了搜索实验负担的同时,提高了生物分子序列设计和优化的效率[38]。因此,深入研究生物序列设计的智能算法,有利于在更广阔的空间中高效设计生物分子,帮助促进生物分子的快速进化。
本文主要综述了智能算法在生物序列设计中的应用,重点介绍在生物分子设计中常用的深度生成式模型,包括生成对抗网络[23]、变分自编码器[22]、递归神经网络等。在此基础上,系统总结了各类生物分子的智能寻优策略与评估方法,以及将智能算法应用于生物数据中的挑战与发展方向。
1 人工智能算法设计生物序列
从模式识别角度分析生物序列设计中的共性的问题:前人的研究发现,特定功能的生物分子序列会形成高维序列空间中的低维流形[39]。例如,2018 年的一篇关于氨基酸序列的研究证实,来自不同细菌的氨基酸序列组成的序列空间中,大肠杆菌的同一氨基酸家族的突变体序列会形成低维流形[40];针对药物小分子的研究也发现,具有视黄醇受体活性的脂肪酸分子,在高维序列空间中可形成低维流形[30];在DNA 序列的设计中也有报道发现编码抗菌肽的DNA 序列在化学性质空间中形成低维流形[41]等。因此,生物序列的设计问题从算法上可归结为从潜在的高维序列空间中,寻找由特定功能的生物序列组成的低维流形问题。
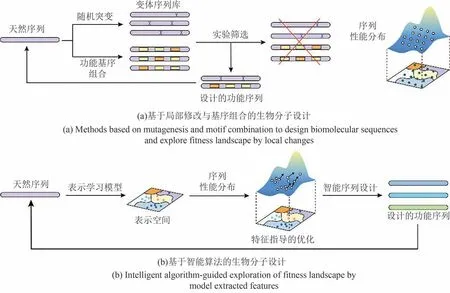
图1 是否利用智能算法指导进行生物分子设计的比较Fig.1 Biomolecular design with or without machine learning-guided search
利用智能算法进行全新生物分子序列设计的基本思路是将离散、高维的生物分子序列空间映射到低维、连续的特征表示空间,通过表示空间对潜在人工分子进行寻找和筛选[42-43]。这一方面可以降低直接对生物分子序列和结构进行设计的复杂度,同时提高人工分子设计的有效性比例,降低大规模文库搜索的实验成本;另一方面,与定量评估以及寻优算法相结合,可对具有特定优良性能的生物分子进行定向优化。由于生物分子具有复杂的序列模式,除了每个位置独立的原子或碱基特征以及两两间的相互作用以外,其远距离相互作用特征往往难以被准确捕捉和描述。而利用机器学习算法的特征提取能力,可捕获生物分子的基本单位如碱基、氨基酸或原子间的远距离相互作用[40]。以此为指导,可高效探索表示空间中潜在的分子序列,从而设计人工分子。除此以外,通过对生成的分子建立定量评价体系,将智能设计的人工分子扩充到天然生物分子序列库中,可优化性能预测模型,进一步缩短对新分子探索的周期[39,44]。
在人工智能领域中,深度生成式模型由于具有强大的模拟数据分布的能力,可通过从低维数据表示中采样和寻优设计全新的人工样本[45],因此近年来在生物序列的智能设计中有着广泛的应用。生物分子设计中常用的深度生成式框架主要包括生成对抗网络,变分自编码器和递归神经网络等(图2),我们将探讨这些模型各自的特点与在生物序列设计中的应用。
1.1 生成对抗网络
生成对抗网络(generative adversarial network,GAN)由Goodfellow等[23]在2014年提出,其通过生成器和判别器的对抗来估计概率分布并生成同训练样本位于类似分布中的新样本。在生物序列设计中,生成对抗网络框架已应用于核酸序列[41]、蛋白质[46]和小分子药物[47]等的设计。生成对抗网络不能获得生物序列在高维序列空间的显式分布,但通过生成器与判别器的自我博弈,可生成与天然生物分子位于类似分布的全新人工分子序列。在DNA 序列设计与药物分子设计中,研究人员通过将生成对抗网络与t-SNE[48]、主成分分析等的降维方法相结合,对生物序列的物理化学特征如长度、带电量等进行降维,可观察到算法生成的全新序列与天然生物分子具有相似的化学特征分布[30,41]。例如,在2019 年的一篇文献[41]中,作者利用GAN 设计编码蛋白的人工DNA 序列:以服从低维正态分布的向量作为生成器的输入,生成器产生的DNA 序列与来自Uniprot[49]数据库中超过3655 条的天然蛋白编码序列共同作为判别器的输入,训练GAN 生成编码蛋白的人工DNA 序列。之后在序列的物理化学特征表示空间中对序列做t-SNE 降维,发现新设计的序列与天然序列位于相似的空间分布中。作者结合抗菌性能预测模型与GAN 进行了的抗菌剂编码序列的循环优化设计,每轮模型生成的排名靠前的人工序列再次作为真实序列输入判别器。通过在独立预测器上进行人工序列的性能评估,得到最终设计的人工序列中40.2%为具有抗菌活性的编码序列。

图2 生物分子序列生成任务中常用的深度生成模型Fig.2 Deep generative models commonly used in biomolecule sequence generation
1.2 变分自编码器
变分自编码器(variational auto-encoder,VAE)是利用具有自编码器结构的神经网络构造的有向概率图模型。在生物序列的设计中,Gómez-Bombarelli 团队[43]首次将基于VAE 的生成式模型引入小分子药物序列的设计中。在VAE 的基础上,药物设计领域已开发出针对不同的分子序列表示方式(如药物的SMILES 结构[50]、3D 结构[51]、原子的三维立方网格[51]、分子特征的二值向量[52]等)不同输入形式的药物序列设计算法,以及针对多靶标设计的条件变分自编码器[53]。例如,2018 年Lim 等[53]使用化合物的油水分配系数、氢键供体性质等性质作为条件向量输入,利用分子序列(对分子的SMILES表示进行独热编码,获得输入向量)与条件向量成对输入到VAE 中,最终生成了分别达到5类不同理化性能指标的人工化合物分子。通过引入对抗思想形成对抗自编码器(adversarial auto-encoder,AAE)框架,可进一步提高在结构上合理药物序列的比例[54]。
1.3 循环神经网络
循环神经网络(recurrent neural network,RNN)是自然语言处理中经典的序列数据生成模型[55]。其中长短期记忆结构(long-short term memory,LSTM)可学习并决定过去的信息保留与否[56]。门循环单元(gate recurrent unit,GRU)的网络结构则更为简化,通常可获得与LSTM类似的效果[55]。在生物序列的设计问题中,研究发现,以化合物分子的SMILES 表示作为输入,基于RNN 的方法可学习到分子序列语法与化学空间的低维分布[57]。对于长度分布差异较大的序列,RNN 具有独特的优势。例如,2019 年Alley 等[58]利用基于LSTM的模型,通过对神经网络各层信息的平均化整合,获得了病毒、细菌、植物、哺乳动物等物种中各类蛋白质序列的特征表示,从而形成通用的蛋白序列表示空间,并利用该表示空间与绿色荧光蛋白的荧光强度模型相结合,进行绿色荧光蛋白序列的人工设计,对于蛋白质序列的优化具有重要的意义。
现有的深度生成式模型具有不同的优势与特点,因此在生物序列设计中适应于不同的应用方向。生成对抗网络可以生成比变分自编码器更加尖锐的数据分布[23],生成与原序列位于相似高维分布中的人工序列。但不能获得显式的数据分布,并且易出现模式崩溃现象,即生成的样本之间相似度过高,影响人工生物序列的多样性[59]。对抗自编码器则将生成对抗网络的对抗思想引入变分自编码器,尽量使得隐层的分布与先验分布接近。但AAE 和VAE 利用最大似然法拟合分子的整体分布[22],分布拟合任务的收敛较困难[60]。循环神经网络可灵活处理不定长的生物序列,但通常模型规模大,所需训练时间长,且生物分子序列相比于自然语言生成的场景,缺乏成熟的语义嵌入网络。在生物序列生成时容易出现碱基的重复,因此相对于仅含4类碱基的核酸,更适合于化合物分子团等单元数目较多的生物序列的生成。
如何合理整合与利用不同的智能模型的优势,针对各类生物分子序列的特点,提取重要的生物学特征,形成生物序列的特征表示空间,对于各类生物分子序列的智能设计与优化具有重要的意义[61]。
2 利用寻优算法进行定向优化
为了对特定性能进行优化,在通过智能算法获得低维的特征表示空间后,可与迁移学习、强化学习等算法相结合,在表示空间针对特定性能对生物分子进行寻优。同时,对于不同类型的分子,由于其结构对功能的影响,也存在不同的分子输入表示形式(表1)。
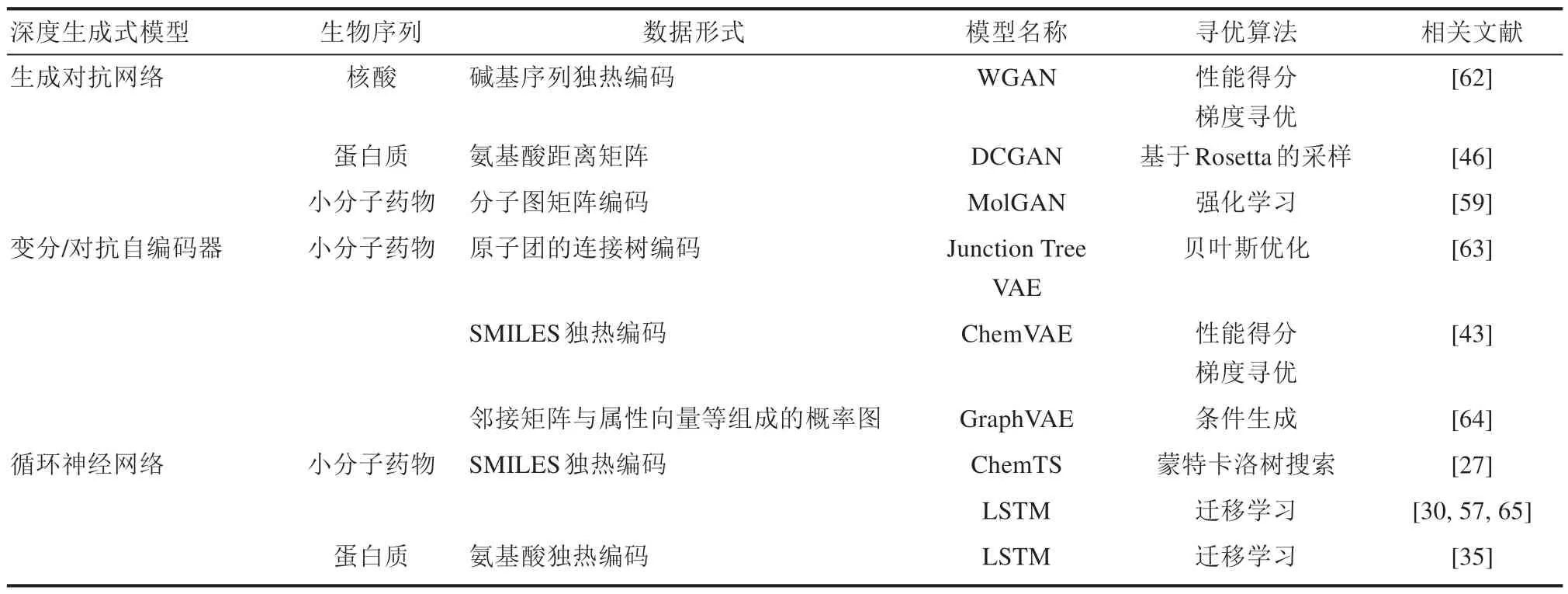
表1 深度生成式模型与优化算法结合的应用研究Tab.1 Applications for deep generative models combined with optimization algorithms
2.1 基于迁移学习的定向优化
迁移学习是一类常用的机器学习方法,其通过将针对某一任务开发的模型、策略作为初始点,经过微调重新使用在另一任务的模型中。在一些场景下,具有特定功能的分子往往数据量较小,无法直接进行预测模型的训练与优化。迁移学习方法将生物数据库中各类生物序列整合,对模型进行预训练,再针对特定性能的分子,如特定疾病的靶向药物或分子抑制剂等对模型进行精调(fine-tuning),从而对这些分子进行扩充设计与探索。例如,研究人员利用RNN 对数据库中约10 万条无性能偏好的生物分子进行了预训练,并迁移学习到约1 万条具有活性的分子上进行精调,最终可以再发现416 种已证明具有活性的药物分子[57]。在一些药物设计的案例中,特定的靶标化合物数目非常有限的情况下,迁移学习进行定向优化也取得了较好的效果。例如,研究人员在丰富的药物数据库上预训练生成器,之后迁移学习到25 种维甲酸和过氧化物酶体增殖受体激动剂上进行精调,最终成功合成了5 种新的有效药物分子[30]。
迁移学习可以灵活地适配不同的智能设计框架,为人工分子的性能优化提供了重要的思路。但基于迁移学习的优化方法获得的人工分子,将与现存特定性能的分子位于类似分布中,最终性能优化提升的效果会受到一定的限制。
2.2 基于强化学习的定向优化
利用生物分子性能的预测模型,可在强化学习框架下,对特定性能的生物序列进行定向优化[66]。例如,有研究利用药物性能预测网络对人工分子性能进行打分,在强化学习的框架下,对药物分子的溶解温度与其作为JAK2 抑制剂的性能进行定向优化,成功获得了一批性能超越天然分子的药物分子序列[39]。2019 年,研究人员基于GAN 框架,利用靶向和非靶向的药物作为正负样本进行强化学习,在表示空间针对DDR1的抑制能力进行定向优化设计,历时仅21 天设计出了DDR1抑制剂的新药候选分子。对在实验室合成的6 个潜在新药分子进行生化初筛后,对4 个具有活性的分子进行体外细胞实验,其中2个化合物展现了显著的DDR1抑制能力[67]。
近年来,基于强化学习的优化算法在小分子药物设计中展现出了巨大的潜力。伴随着各类生物分子的性能预测模型准确性的逐步提高,未来基于强化学习的框架在不同生物序列中的优化将成为重要的研究方向。
2.3 基于蒙特卡洛树搜索的定向优化
对于生物分子序列,可使用基于决策树搜索的方法进行生成与优化。其中,蒙特卡洛树搜索(Monte Carlo tree search,MCTS)是一种在缺乏强启发的情况下常用的基于树的序列搜索方法[68],主要包含4个步骤:选择,拓展,模拟和反向传播更新。利用蒙特卡洛搜索可以从头开始同时生成与优化生物分子:选定当前最可能的决策,如碳原子或其他化学结构组成单位如苯环,随后采用随机搜索进行模拟,用以模拟完整序列的延伸结果。通过对多次采样的结果进行平均,反向传播回相应的节点,可学习获得决策成功的概率。因此,在训练完成后可通过基于策略的抽样生成新的序列。
在生物分子序列设计中,有研究通过MCTS与RNN、VAE 相结合,利用不同的神经网络来进行分子有效性预测,成功实现了药物序列的设计。例如通过利用RNN 网络进行延伸模拟,实现了对药物分子性能(如正辛醇-水分配系数等)的优化[27]。在化学反应的智能设计中,利用蒙特卡洛搜索与人工神经网络相结合的方法,以高于传统设计方法3 倍的搜索效率,成功设计了2 倍数目的目标分子生成反应[5]。
基于树搜索方法的序列设计可实现生物序列的生成与寻优,但其依赖于性能预测模型的统计得分作为序列生成的指导,且往往不考虑表示空间的分布,因此更适用于具有较为准确的性能预测模型[69],但表示空间分布的可解释性较弱的生物序列设的设计。
2.4 基于贝叶斯优化的定向优化
贝叶斯优化方法可针对任意连续表示空间进行建模。以分子在连续隐空间的表示作为输入,通过多次重复高斯过程进行探索,计算采样函数的值,最终以将采样函数最大化值,作为优化分子。例如,研究人员利用稀疏高斯过程[70]对VAE 生成的隐空间进行了贝叶斯优化[63],结果显示人工分子与天然分子在相对位置比对的相似度大于0.6 的情况下,人工分子的设计成功率超过83%。
基于贝叶斯优化的分子寻优方案,不依赖于具体的预测模型指导,但需要在分子具有良好的连续隐空间表示基础上,进行基于高斯过程的探索和寻优。因此通常适用于基于自编码器的深度生成式模型(如VAE和AAE)。
2.5 基于性能得分梯度回传的定向优化
将性能预测模型与生物分子表示相结合,可以实现对连续隐空间基于梯度的定向优化。利用生成模型获得的表示空间,与预测模型相连接,计算分子的性能分值对于分子表示的梯度,并将梯度回传到表示空间,可以指导表示空间的寻优方向。例如在药物设计中,研究人员将VAE 与性能预测器相结合,利用编码器将化合物序列映射到低维表示空间,使用预测器对隐空间中化合物的性能进行预测。最终利用性能得分对于分子表示的梯度,指导隐空间的寻优方向进行定向优化[43]。在基因元件设计中,研究人员测定了基于酵母中元件基序(Motif)设计的数十万条启动子序列的表达活性,以此训练卷积神经网络预测模型。之后利用基于基因表达活性的梯度回传的方式指导启动子设计,产生了大量的具有特定功能与序列多样性的启动子元件[71]。
综上,包括迁移学习、强化学习等在内的离散与连续寻优的方法,在生物分子的序列中均取得了较大的进展。在蛋白质设计领域,基于Rosetta 算法评分[72]的采样方式是常用的蛋白质序列与骨架的优化方法。在实际的生物分子应用中,可将不同的寻优方案进行整合优化。例如,将迁移学习的精调与强化学习的框架相结合,在精调到特定的性能分布后,利用强化学习进一步优化提升生物分子的性能[67]。除此以外,在对单一性能进行优化的基础上,通过设计各类条件输入,例如目标状态下重要基因的表达谱[73]、现有特定功能的分子序列[74]、目标性能值[75]等,生成式模型可设计不同类型的生物分子,形成依赖条件输入的生物分子定向设计[53,76]。
为了对生物序列进行准确的预测与优化,可以综合各种组学数据的信号输入[77],利用进化过程中的序列进行数据增强[78]。另外,深度生成式模型也可作为对训练数据进行增强的数据生成器[79],通过从不同角度丰富训练数据,进一步提高预测模型的准确性[80]。从计算层面,可以发挥智能算法强大的特征提取能力的优势[81],在样本数量受限的情况下,进行无监督或半监督的序列特征提取[82],通过模型解析可以帮助人们发现新的重要生物学特征[83-84],为人工生物系统的构建提供重要支撑。
3 生成序列的计算评价指标
利用深度生成式模型进行定向优化,需要筛选多样性较高、与天然分子相似性较低、满足目标需求(如表达量、靶点或组织特异性)的人工分子。合适的评估体系的设计有利于提高目标分子的设计成功率和筛选效率[85]。下面列出的是目前在生物序列设计领域中一些常用的评估指标,可为其他评估尚未成熟的生物序列设计问题提供思路(表2)。
3.1 基于分布的评估
从生成的生物分子是否与天然分子位于类似分布的角度,可从人工分子的合理性、多样性、新颖性等方面进行评估。在药物设计领域,对设计的序列进行性能预测具有相对标准化的定量评估指标,如基于二维分子印迹[86]的Tanimoto 距离[87],可以衡量设计的生物序列之间的相似性。RDkit 包可以初步检测是否为结构合理的药物序列[88]。在蛋白设计领域,可以利用Rosetta 算法对设计的人工蛋白进行评估、筛选与优化[89]。除此以外,可使用在性能预测等任务中表现鲁棒的神经网络对智能设计的生物序列进行评价。例如类似于图片生成领域中可利用图片分类问题常用的特征提取网络InceptionNet[90]对生成结果进行评价,药物设计领域则利用预训练的药物深度神经网络ChemNet 的隐层对生成结果进行评价。研究人员据此提出利用Frechet ChemNet Distance(FCD)来衡量生成式模型设计的药物序列p(·)与天然药物序列pw(·)之间的距离[91]。为了获得每个分子的数学表示,以ChemNet的倒数第2层作为序列的分布。假设隐层表示满足多维高斯分布,计算模型设计药物序列的高斯分布p(·)的均值和方差则为(m,C),天然药物序列的高斯分布pw(·)均值和方差则为(mw,Cw)。可由此计算出二者的Frechet Distance(Wasserstein-2 Distance),用于评估智能设计的生物分子的多样性以及是否与真实分子之间有类似的化学性质。与之对应的,在人工蛋白设计的问题中,可以利用大量实验测试绘制的经验性蛋白适应度分布(fitness landscape),对设计的人工蛋白质与多肽链进行分布一致程度的评估[92-93]。
3.2 基于优化目标的评估
针对优化性能目标的评估,可利用单独训练的预测模型对生成的生物序列进行评价。除此以外,从生物序列再发现的角度,计算重设计的恢复比例,即从训练集中分出一部分生物序列作为测试集,计算生成的序列与测试集序列的重合比例。例如,在2018 年的一篇文章中[57],作者使用了1239 个药物序列训练循环神经网络生成药物分子,其中28%的分子可在测试集中出现,并与测试集的药物分子位于相似的低维流形上,验证了药物分子设计的有效性。
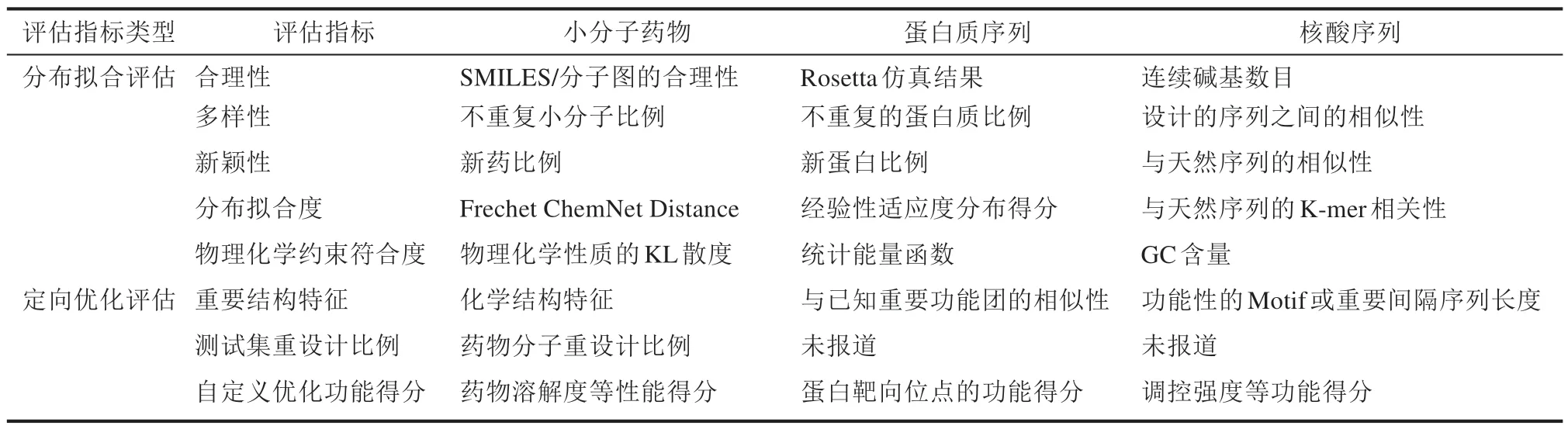
表2 深度生成式模型进行生物序列设计的常用评价指标Tab.2 Evaluation criteria for deep generative model designed biomolecular sequences
3.3 计算模拟与生物实验相结合
对于满足计算评估指标的生物分子,可通过分子生化方法进行人工合成,利用批量生化实验,如大规模平行报告系统[94]、细胞外给药技术等,测试智能设计的分子的有效性。2018 年有研究人员[74]将条件对抗自编码器设计的300 000 个候选JAK2 激酶抑制剂进行docking 筛选[95]、分子动力学筛选后获得了100个潜在序列。之后利用专家知识筛选后的抑制剂分子,通过胞外给药曲线测定的方法,成功获得了1 种具有JAK2 激酶抑制剂活性,且同时不具备其他对照蛋白(如JAK3 激酶)活性的药物分子序列。
目前生化实验筛选的通量与潜在的序列空间相比仍非常有限。利用智能算法与生化实验测试相结合的方式,搭建智能算法与生化测试的循环优化设计框架,可以提高生物序列的设计效率[96]。例如,我们利用GAN 设计大肠杆菌启动子序列的设计,经过第1 轮计算筛选与生化实验测试后,利用测得的人工启动子的活性结果,对启动子活性预测模型进行迭代优化,最终智能设计的人工启动子序列设计成功率超过了70%[31]。进一步,我们基于深度学习模型搭建了启动子设计软件Gpro,提供了启动子智能化、模块化的设计平台[97]。
4 总结与展望
为了更加精确、稳定地调控细胞内的生化反应,满足对不同生命活动调控的需求,需要对生物序列进行人工设计与优化,这是合成生物学面临的重要的科学问题[98]。由于生物序列的复杂性,智能算法在该类问题中具有独特的优势:不同于传统的设计手段在天然序列的局部进行小范围探索,智能算法可以通过提取生物数据的复杂特征,并在这些生物特征的指导下,实现自动化、批量化、端到端的智能设计。因此,伴随着智能算法的发展,与生物序列测试数据的积累,在数据与模型的共同驱动下,生物分子的设计将打开全新的篇章。
针对不同的生物序列,利用智能算法进行自动设计,面临着不同的挑战。表3从数据量、常用的智能设计模型等角度比较了在这些应用领域进行智能设计的挑战与潜在的发展方向。在药物小分子序列设计领域,计算评估的指标相对比较完善,但分子的有机合成需考虑的因素仍较为复杂。尽管有研究利用整合智能算法与先验规则的框架,对化合物分子的合成线路进行自动设计[101],对设计出的分子进行有机合成仍然是药物开发的限速步骤。因此如何综合各类生化指标,将药物分子合成线路的设计融入到生物序列的智能设计中,是未来重要的研究方向。在特定功能蛋白质的设计中,由于对其三维折叠的构象等性能的预测仍缺乏准确性,目前探索的范围仍然有限。如何利用蛋白质序列与结构的表示空间,结合物理化学约束模型,进行蛋白质的智能设计与优化,仍然是尚未解决的重要问题,在未来具有广阔的应用前景。在核酸序列的设计中,核酸序列的合成约束相对小分子化合物与蛋白质更少,但各类核酸序列的生物学功能迥异,并且与细胞内复杂的蛋白质调控网络存在相互作用,同时尚缺乏系统规范的性能评估体系。其中,DNA 序列设计的研究主要关注于转录调控序列和用于微芯片的DNA 探针、针对编码抗菌肽基因的DNA 序列等的设计。因此,如何对基因组的顺式调控元件与反式作用因子等不同层次的信号进行建模整合,成为研究的关键。
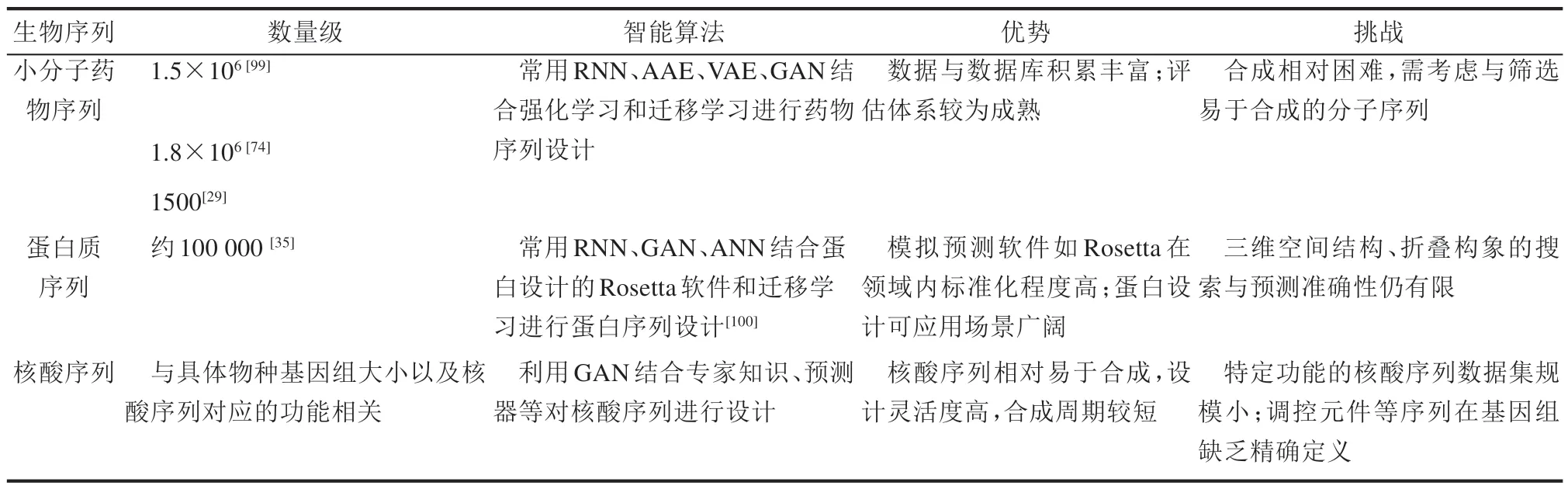
表3 对药物分子、蛋白质和核酸序列进行智能设计的优势与挑战Tab.3 Advantages and challenges of intelligent design for drug molecules,proteins and nucleic acid sequences
在生物序列设计问题中,一方面不同生物序列的智能设计面临着各自的挑战;另一方面,人工生物序列作为合成生命系统的重要信息写入载体,其如何与胞内复杂的多层次调控之间相互影响,尚待研究。未来人工生物序列的智能设计需充分考虑生物系统具有多层次的调控高度耦合的复杂特性,通过对不同层次的生物序列进行设计,从系统的角度利用生物序列对天然生物系统中不同层次的调控进行干预,实现生物序列与系统底盘环境的整体智能适配与优化。这将为人工生物序列的设计与合成生命系统的构建向着高通量、智能化、自动化的方向发展提供重要支撑。

