细菌GntR家族转录调控因子的研究进展
刘国芳,王欣欣,苏辉昭,陆光涛
细菌GntR家族转录调控因子的研究进展
刘国芳1,王欣欣2,苏辉昭2,陆光涛2
1. 广西民族大学海洋与生物技术学院,广西多糖材料与改性重点实验室,南宁 530008 2. 广西大学生命科学与技术学院,南宁 530004
GntR家族转录调控因子是细菌中分布最为广泛的一类螺旋–转角–螺旋(helix-turn-helix,HTH)转录调控因子,此家族转录调控因子包含两个功能域,分别是N端的DNA结合结构域和C端的效应物结合结构域/寡聚化作用结构域。DNA结合结构域的氨基酸序列是非常保守的,但效应物结合结构域/寡聚化作用结构域的氨基酸序列却存在很大的差异性。目前许多GntR家族的转录调控因子已经被鉴定,这些转录因子调控细菌许多不同的细胞过程,如运动性、葡萄糖代谢、细菌的耐药性、病原细菌的致病力等。本文主要阐述了GntR家族转录调控因子的发现、二级结构、生物学功能、调控模式等方面的研究进展,旨在为研究者全面、深入地了解GntR家族转录调控因子的功能及作用机理提供帮助。
GntR家族转录调控因子;二级结构;生物学功能;调控模式
细菌经常暴露在不断变化的环境中。为了生长和生存,细菌能够根据环境中可利用营养的数量和类型,开启不同的调控系统,使细胞代谢过程能够适应环境的变化。细菌的转录调控是细菌快速繁殖适应变幻莫测环境的主要机制之一[1],其中最常见的转录调控方式是通过转录调控因子与启动子的结合,来调控基因转录的起始,进而调控基因的转录水平。
在细菌中,已鉴定出许多转录调控因子,根据它们所结合DNA的模式以及自身保守的基序,人们将这些转录调控因子分为若干类群[2]。其中最为常见的是螺旋–转角–螺旋(helix-turn-helix, HTH)类群,此类群的转录调控因子含有一个保守的α螺旋–转角–α螺旋DNA结合结构域[3]。根据DNA结合结构域的序列,人们又将此类群的转录调控因子分为多个不同的家族[2],其中GntR家族转录调控因子是分布最为广泛的一类HTH转录调控因子。本文主要阐述了GntR家族转录调控因子的发现、二级结构、生物学功能、调控模式等方面的研究进展,将为转录调控因子调控机理的研究和动植物病原菌致病机理的研究提供新的知识和基础。
1 GntR家族转录调控因子的发现和基本结构
第一个发现的GntR家族转录调控因子是枯草芽孢杆菌()中的葡萄糖酸盐操纵子抑制子(lucoae operonepressor, GntR)[4]。GntR家族转录调控因子包含两个功能域(图1)分别是N端的DNA结合结构域(DNA-binding domain, DBD)和C端的效应物结合结构域/寡聚化作用结构域(effector- binding and oligomerization domains, EBD)。DNA结构域的氨基酸序列在GntR家族成员中是非常保守的,但效应物结合结构域/寡聚化作用结构域的氨基酸序列存在很大的差异性。根据C端氨基酸序列的不同,GntR家族转录调控因子分为7个亚家族(图2),分别是FadR (attycidegradationegulator)、HutC (istidineilization repressor)、MocR (genes of GntRegulator)、YtrA (operon transcriptional regulator)、AraR (L-binose operon transcriptionalepressor)、DevA (elopment transcriptional regulator)、PlmA (asmidaintenance)[5~7],其中FadR、HutC、MocR、YtrA亚家族是GntR家族中比较重要的亚家族。
FadR亚家族是GntR家族转录转录调控因子中成员最多的一类,占约40%。其成员中的C端结构域包括6~7个α螺旋,由150~170个氨基酸组成。C端结构域可与效应物或者配体结合,如羧酸,结合后,蛋白的构象发生变化,进而影响与DNA的结合[8]。该亚家族转录调控因子是在大肠杆菌()中首次发现。中的FadR参与调控脂肪酸的合成与分解代谢,是一个酰基辅酶A结合蛋白,以二聚体的形式存在。其中N端是DNA结合结构域,C端是配体结合结构域,可以与酰基辅酶A结合[9]。
仅次于FadR亚家族的是HutC亚家族,约占GntR家族的30%。该亚家族成员的C端结构域由170个氨基酸组成,包括α螺旋和β折叠[10]。此亚家族的第一个转录调控因子是在恶臭假单胞菌()中发现。的HutC是一个组氨酸利用操纵子调节因子[11]。结构研究发现,此亚家族的转录调控因子C端的效应物结合结构域/寡聚化作用结构域的折叠方式与中发现的单核分支酸裂解酶很像,均以二聚体的形式存在并且在HutC亚家族中是非常保守的[12]。
MocR亚家族中的转录调控因子的典型特征是C端有一个比较大的效应物结合结构域/寡聚化作用结构域,平均由350个氨基酸组成。折叠方式属于I型吡哆醛5ʹ-磷酸盐(pyridoxal 5ʹ-phosphate, PLP)依赖性酶,此折叠类型的原型酶为天冬氨酸转移酶(aspartate aminotransferase, AAT)[13]。此家族转录调控因子的N端DNA结合结构域和C端AAT结构域是通过多肽来连接的。多肽链的长度在MocR亚家族中不同成员之间是不相同的。

图1 GntR家族蛋白结构示意图

图2 GntR家族转录调控因子亚家族成员C端二级结构的示意图
与GntR家族中其他6个亚家族相比,YtrA亚家族中的成员的肽链组成较短,一般由120~130个氨基酸组成。其中大约50个氨基酸是位于C端结构域,形成2个α螺旋[14]。YtrA最早是在枯草芽孢杆菌()中发现的[15]。中的基因与其他5个基因构成一个操纵子。YtrA由130个氨基酸组成,负向调控操作子中基因的表达。绝大多数编码YtrA亚家族转录调控因子的基因与ATP-binding cassette (ABC)转运系统相关基因组成了操纵子,可以调控ABC (ATP-binding cassette)转运系统相关基因的表达[10]。由于YtrA亚家族成员氨基酸组成较少,肽链的长度较短,给其结构的研究造成了很大的困扰。
随着研究的深入,GntR家族蛋白越来越多的被鉴定出来。在PfAM数据库中,GntR家族序列由2008年的8561条增长到89733条。GntR家族在细菌中广泛分布,主要分布在变形菌门Proteobacteria,厚壁菌门Firmicutes 和放线菌门Actinobacteria等5958种细菌中(图3)。根据各亚家族C端的差异构建系统发育树,结果发现,MocR和PlmA在进化上虽然比较接近,但GntR家族各亚家族蛋白在整体进化上距离较远(图4),说明各亚家族在进化上变化较大。
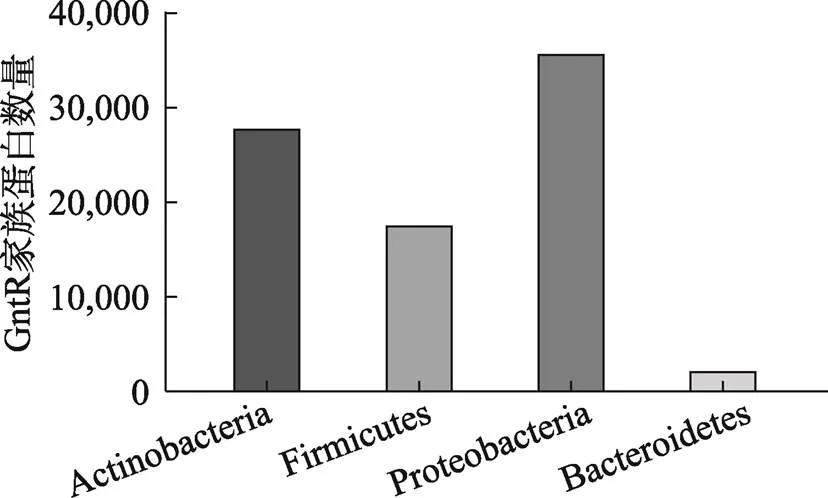
图3 GntR家族蛋白在细菌类群中的分布
放线菌门:Actinobacteria;厚壁菌门:Firmicutes;变形菌门:Proteobacteria;拟杆菌门:Bacteroidetes。

图4 GntR家族蛋白系统发育树
2 GntR家族转录调控因子的生物学功能
许多GntR家族转录调控因子已经被鉴定。这些转录因子调控细菌许多不同的细胞过程[5],包括运动性[16]、发育[17]、葡萄糖代谢[18]、细菌的耐药性[19]、细胞的毒性[20]、病原细菌的致病力[21,22]等。
2.1 动物病原细菌中GntR家族转录调控因子的生物学功能
目前,与动物病原菌GntR家族转录调控因子相关的研究报道有很多。本文将以已报道的几个动物病原菌中的GntR转录调控因子为例,阐述动物病原菌中GntR家族转录调控因子的生物学功能。
病原物中的FadR转录调控因子是一个全局调控因子,可调控脂肪酸合成、水解、转运等相关基因的表达。其N端的DNA结合结构域可与靶基因特定的DNA序列结合;C端可与配体结合,其生理配体是LC脂肪酰基辅酶A硫酯。当FadR与配体结合后,FadR就会从靶基因的结合位点上解离下来,进而调控靶基因的表达[23](图5)。FadR转录调控因子能够抑制脂肪酸转运相关基因(和)、编码分解代谢相关酶基因(、、、、)的表达[9]。
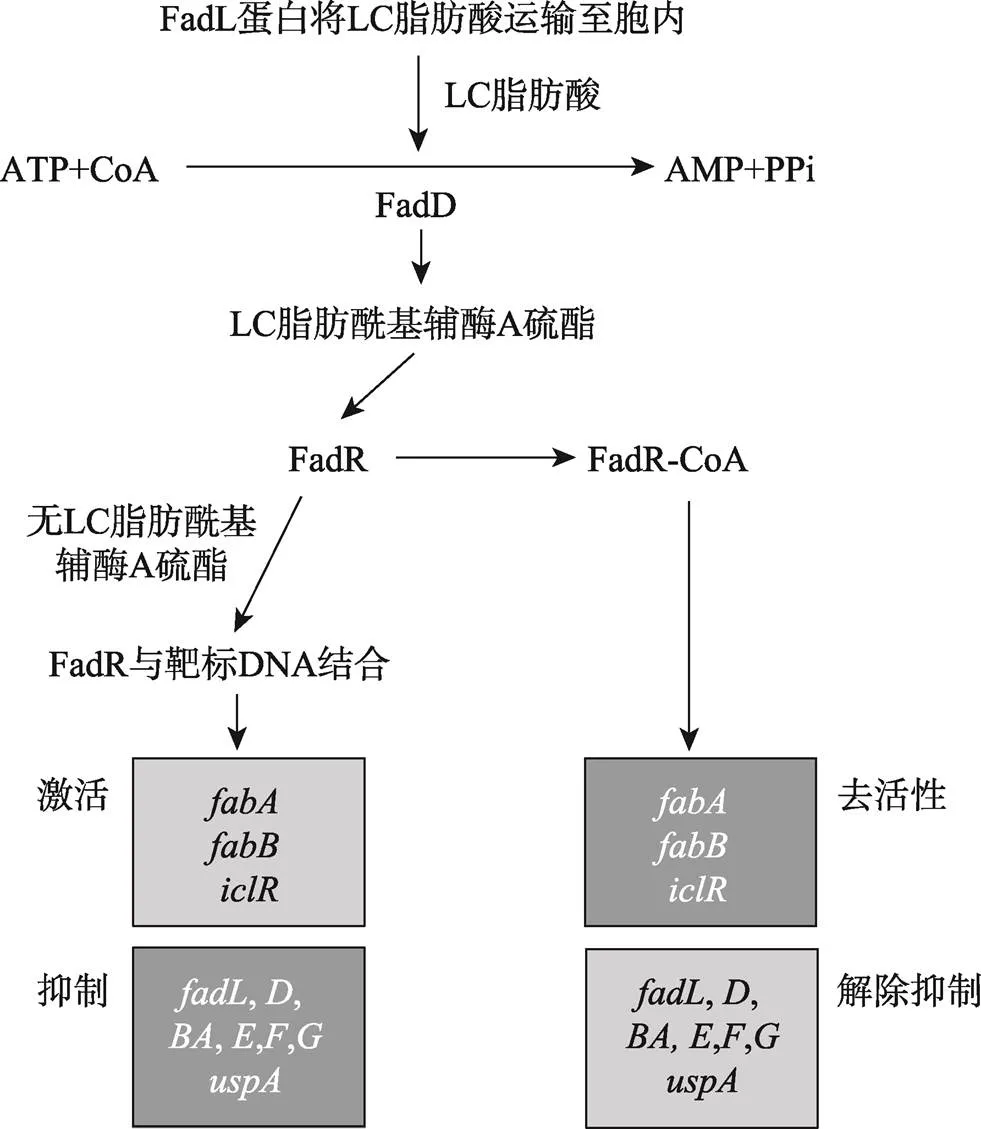
图5 大肠杆菌中FadR调控脂肪酸代谢的模式图
铜绿假单胞菌()是一种条件致病菌,是一种医院内感染的主要致病菌,感染此菌后会引发一些疾病,如肺炎、细菌病、尿道和外科感染[24]。在此菌中发现,GntR家族转录调控因子不仅抑制自身基因的表达,而且参与该菌的许多代谢过程,包括抑制葡萄糖代谢过程中的关键酶葡萄糖酸盐透性酶GntP的表达[24];参与调控2-羟基喹啉和氨基苯甲酸盐的代谢[25]。
溶血弧菌()是一种食源性病原体,有时可在受污染的海鲜中发现,可引致人类急性肠胃炎和出血性败血症。此菌中存在两个鞭毛系统参与细菌的运动,在许多细菌中,运动性与病原细菌的致病、生物膜的形成和环境的适应相关[16]。在此菌中鉴定到的一个GntR家族转录调控因子参与调控细胞的运动性。变形链球菌()为革兰氏染色阳性球菌,是龋病的主要致病菌。在此菌中所鉴定到的GntR家族转录调控因子StsR,是通过调节糖转运相关基因的表达,来影响该菌早期生物膜的形成和胞外多糖(extracellular polysaccharide, EPS)的合成[26]。已知生物膜的形成和EPS往往与病原细菌的致病密切相关。所以,StsR极有可能参与调控变形链球菌的致病过程。肺炎链球菌()是一种重要的人类病原体,通常存在于呼吸道上部,可引致肺炎、中耳炎、脑膜炎、败血症等疾病。在此菌中鉴定到的GntR家族转录调控因子CpsR可调控荚膜多糖的合成和致病过程[27]。金黄色葡萄球菌()是一种重要的人类病原体,可引致多种感染,如脓肿、败血症、中毒性休克综合症和心内膜炎。在此菌中鉴定到的GntR家族转录调控因子NorG是一个全局转录调控因子,调控参与多种不同细胞过程的基因[28]。布鲁氏菌(spp.)能够引起布鲁氏菌病,此病是一种人畜共患的传染病,对公众健康及安全构成了极大危害。研究发现,布鲁氏菌侵染巨噬细胞期间,GntR转录调控因子影响IV型分泌系统(type IV secretion system, T4SS)和群体感应系统相关基因的表达[20]。
综上所述,动物病原菌中目前鉴定的GntR家族转录调控因子许多是全局转录调控因子,可调控许多细胞过程,包括运动性、生物膜的形成、胞外多糖的合成、群体感应等。这些全局性转录调控因子通过调控与动物病原菌致病相关的细胞过程,进而影响病原细菌的致病过程。
2.2 植物病原细菌中GntR家族转录调控因子的生物学功能
相较于动物病原菌,植物病原菌中关于GntR家族转录调控因子的研究相对较少。十字花科黑腐病菌(pv.,)能够引起十字花科植物发生黑腐病。在此菌中鉴定的GntR家族转录调控因子HpaR1是一个全局转录调控因子,调控许多细胞过程,包括致病过程、III型分泌系统(type III secretion system, T3SS)相关基因的表达、致病因子胞外多糖、胞外纤维素酶相关基因的表达等[29~31]。柑橘溃疡病菌(ssp.)是一种对柑橘危害很大的致病菌,引发柑橘溃疡病,导致世界范围的柑橘产量减少。此菌中鉴定的GntR家族转录调控因子YtrA调控细菌致病过程、T3SS和III型效应物(type III effectors, T3Es)相关基因的表达、细菌在寄主和培养基中的生长等多个细胞过程[32]。
除以上动植物病原菌之外,在一些非致病菌中也鉴定到了GntR家族转录调控因子。灰色链霉菌()中鉴定的GntR家族转录调控因子DasR参与调控N-乙酰氨基葡萄糖的代谢[33];天蓝色链霉菌()中鉴定的GntR家族转录调控因子DevA与该菌的发育相关[17];慢生型大豆根瘤菌()中鉴定的GntR家族转录调控因子MocR与该菌的运动性、生物膜的形成等相关[34]。
3 调控模式
转录调控因子通过识别基因启动子区的特定序列并与其结合,进而调控基因的转录。根据调控机制的不同分为正转录调控(转录激活)和负转录调控(转录抑制)两类,GntR家族转录调控因子绝大多数是转录抑制子。
基因转录抑制主要包括3种模式[35]:(1)转录因子结合在启动子区的−10区附近,使得RNA聚合酶(RNA polymerase, RNAP)无法与启动子结合,进而抑制基因的转录;(2)转录因子结合在启动子的特定位置,使启动子区的构象发生改变,形成一个环,封闭了启动子的−35区和−10区,使得RNAP无法与启动子结合,阻止了转录的起始;(3)某些基因的转录需要转录激活因子,而转录抑制因子通过与转录激活因子相互作用,使得激活因子无法起到激活转录的作用,进而阻止转录的起始。
在植物病原细菌中所鉴定的GntR家族转录调控因子HpaR1,其功能及调控模式不同于以往报道的GntR家族转录调控因子。HpaR1是一个全局转录调控因子,属于YtrA亚家族,正调控多种不同的细胞过程,包括胞外多糖、胞外酶活性、运动性以及抗逆性等多种细胞过程[29]。其调控的EPS的合成,是通过正调控一簇与EPS合成相关的基因操纵子来实现的。HpaR1结合在基因启动子区的−35区和−10区之间。一般而言,转录调控因子结合在基因启动子的−35区和−10区内,会阻断RNAP沿DNA模板链的移动,从而抑制基因的转录,因而是起到一个转录抑制子的作用。但是HpaR1却是通过与RNAP蛋白间的相互作用,增强了RNAP与基因启动子区的结合,从而增强了基因的表达。因此,HpaR1对基因的调控模式是一种新的调控模式[30]。
目前的研究报道主要集中在某个转录调控因子的调控模式的研究,但是细胞内的转录调控因子往往是形成一个调控网络发挥作用的。中的HpaR1和另一个全局性转录调控因子Clp,能够以一种特有的模式共同调控胞外纤维素酶主校基因()表达(图6)。HpaR1和Clp均正调控的转录。HpaR1结合在启动子区的−35区上游包括−35区序列(HpaR1 binding site, HBS);Clp在启动子区中有两个结合位点,CBSI (Clp binding site I)位于启动子区的−35区上游,CBSII (Clp binding site II)位于启动子区的−35区上游包括−35区序列,其中CBSII与HBS是部分重叠。HpaR1对转录激活效率低于Clp,但对启动子区的结合能力比Clp更强。因此HpaR1能将Clp从CBSII结合位点驱逐出来,从而与Clp一起,精细地调控的转录水平[31]。
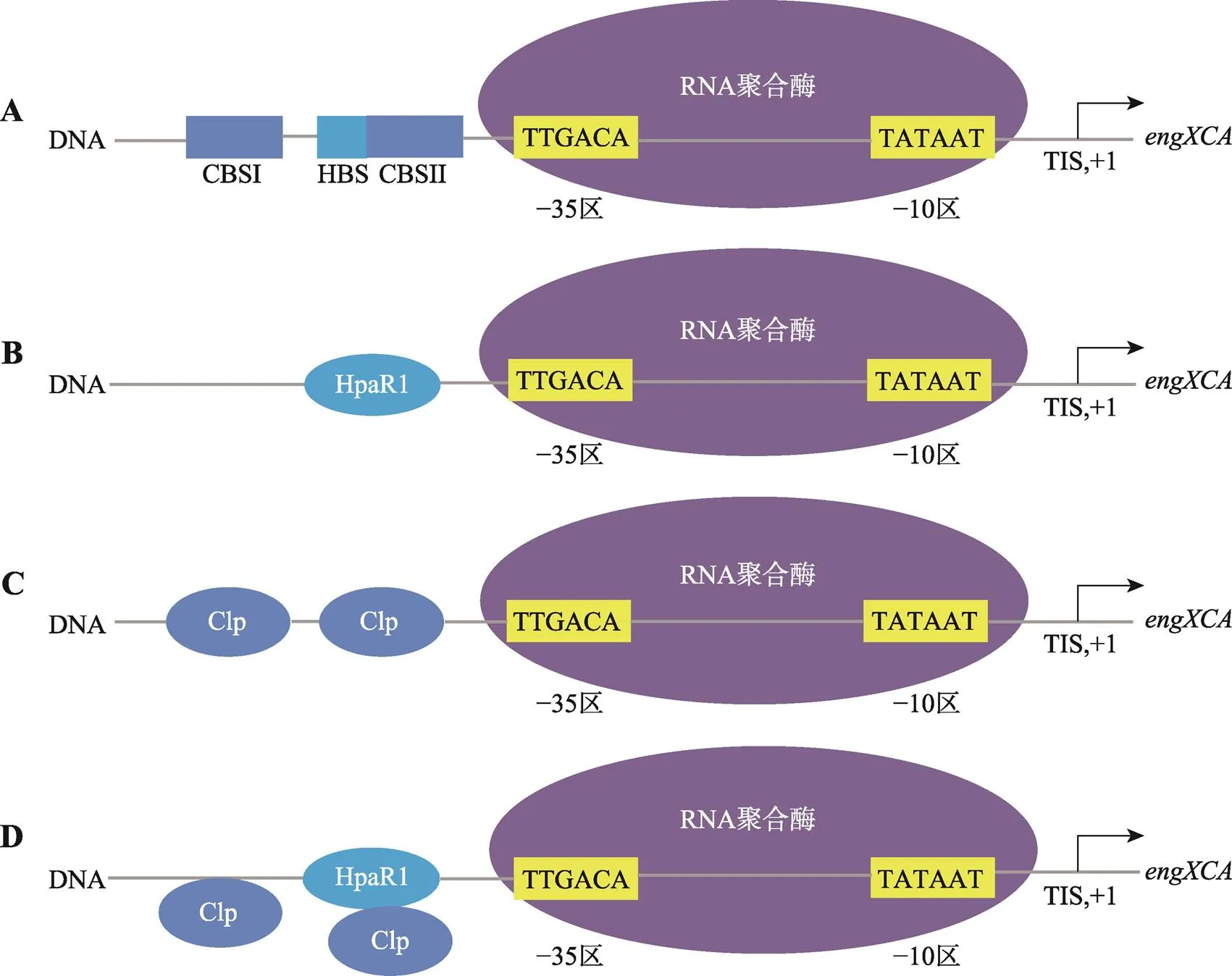
图6 HpaR1、Clp调控engXCA的模式图
A:HapR1、Clp均能与启动子区结合,Clp与启动子区有两个结合位点,其中CBSII与HBS是重叠的;B、C:HpaR1、Clp在体外均能激活基因的转录;D:HpaR1与Clp相比,HpaR1与启动子区有更高的亲和力,并且能将Clp从其结合位点CBSII驱逐下来[31]。
4 结语与展望
随着功能基因组学的发展,转录调控因子的研究取得了极大的进展。越来越多的GntR家族转录调控因子被鉴定。但是目前关于GntR家族转录调控因子的研究主要还是集中在其生物学功能的研究,对于其结构、配体以及两个或者两个以上转录调控因子共同调控一个靶基因的研究相对比较少。因此,GntR家族转录调控因子与其它转录因子共同调控靶基因的作用机理是一个有趣的研究方向。我们对中两个全局转录调控因子HpaR1和Clp共同调控一个靶基因进行研究,已筛选出的一些候选靶基因与该菌的致病相关,包括编码致病因子胞外纤维素酶与胞外多糖等基因。这些研究将有利于病原菌致病网络的构建,给人们防治动植物病害提供理论基础。
另外,目前只有很少的GntR家族蛋白的晶体结构被解析,如中的FadR、谷氨酸棒状杆菌()中的LldR、中的YvoA等。GntR家族中的转录调控因子的晶体比较难获得,与靶标DNA共结合的晶体就更加难获得。结构得不到解析,对该家族转录调控因子功能的研究也造成了很大的影响。已知GntR家族转录调控因子C端结构域与配体结合后,使转录调控因子与靶基因结合或者解离,进而调控靶基因的转录水平。但目前只筛选到了很少的配体,主要是因为目前还没有快速大量筛选配体的方法,而细菌生活的环境又是复杂多变的,环境中的许多小分子物质都有可能是GntR家族转录调控因子的配体。只有快速筛选配体技术的发展,才能获得大量的配体。GntR家族转录调控因子往往可调控致病菌的致病过程。因此,通过鉴定其配体,可以此作为药物靶标,来防治动植物病害。
[1] Majidian P, Kuse J, Tanaka K, Najafi H, Zeinalabedini M, Takenaka S, Yoshida KI.GntR regulation modified to devise artificial transient induction systems., 2017, 62(6): 277–285.
[2] Pabo CO, Sauer RT. Transcription factors: structural families and principles of DNA recognition., 1992, 61: 1053–1095.
[3] Haydon DJ, Guest JR.A new family of bacterial regulatory proteins., 1991, 63(2–3): 291–295.
[4] Fujita Y, Fujita R. New diagnostic systems--technics, efficiency and limitations. Cholangioscopy. a) Peroral cholangioscopy., 1987, 45(7): 1466–1471.
[5] Hoskisson PA, Rigali S. Chapter 1: Variation in form and function the helix-turn-helix regulators of the GntR superfamily., 2009, 69: 1–22.
[6] Suvorova IA, Korostelev YD, Gelfand MS. GntR family of bacterial transcription factors and their DNA binding motifs: structure, positioning and co-evolution., 2015, 10(7): e0132618.
[7] Vindal V, Suma K, Ranjan A. GntR family of regulators in: a sequence and structure based characterization., 2007, 8: 289.
[8] Zheng MY, Cooper DR, Grossoehme NE, Yu MM, Hung LW, Cieslik M, Derewenda U, Lesley SA, Wilson IA, Giedroc DP, Derewenda ZS.Structure ofTM0439: implications for the mechanism of bacterial GntR transcription regulators with Zn2+-binding FCD domains., 2009, 65(Pt 4): 356–365.
[9] van Aalten DM, DiRusso CC, Knudsen J, Wierenga RK.Crystal structure of FadR, a fatty acid-responsive transcription factor with a novel acyl coenzyme A-binding fold., 2000, 19(19): 5167–5177.
[10] Rigali S, Derouaux A, Giannotta F, Dusart J. Subdivision of the helix-turn-helix GntR family of bacterial regulators in the FadR, HutC, MocR, and YtrA subfamilies., 2002, 277(15): 12507–12515.
[11] Allison SL, Phillips AT. Nucleotide sequence of the gene encoding the repressor for the histidine utilization genes of., 1990, 172(9): 5470– 5476.
[12] Fillenberg SB, Friess MD, Körner S, Böckmann RA, Muller YA. Crystal structures of the global regulator DasR from: implications for the allosteric regulation of GntR/HutC repressors., 2016, 11(6): e0157691.
[13] Milano T, Angelaccio S, Tramonti A, Di Salvo ML, Contestabile R, Pascarella S.Structural properties of the linkers connecting the N- and C- terminal domains in the MocR bacterial transcriptional regulators., 2016, 3: 8–18.
[14] Gao YG, Yao M, Itou H, Zhou Y, Tanaka I. The structures of transcription factor CGL2947 fromin two crystal forms: A novel homodimer assembling and the implication for effector-binding mode., 2007, 16(9): 1878–1886.
[15] Yoshida KI, Fujita Y, Ehrlich SD. An operon for a putative ATP-binding cassette transport system involved in acetoin utilization of., 2000, 182(19): 5454–5461.
[16] Gu D, Meng HM, Li Y, Ge HJ, Jiao XN. A GntR family transcription factor (VPA1701) for swarming motility and colonization of., 2019, 8(4): 235.
[17] Hoskisson PA, Rigali S, Fowler K, Findlay KC, Buttner MJ. DevA, a GntR-like transcriptional regulator required for development in., 2006, 188(14): 5014–5023.
[18] Daddaoua A, Corral-Lugo A, Ramos JL, Krell T. Identification of GntR as regulator of the glucose metabolism in., 2017, 19(9): 3721–3733.
[19] Truong-Bolduc QC, Hooper DC. The transcriptional regulators NorG and MgrA modulate resistance to both quinolones and beta-lactams in., 2007, 189(8): 2996–3005.
[20] Li ZQ, Wang SL, Zhang H, Zhang JL, Xi L, Zhang JB, Chen CF. Transcriptional regulator GntR ofregulates cytotoxicity, induces the secretion of inflammatory cytokines and affects expression of the type IV secretion system and quorum sensing system in macrophages., 2017, 33(3): 60.
[21] Zhou XF, Yan Q, Wang N.Deciphering the regulon of a GntR family regulator via transcriptome and ChIP-exo analyses and its contribution to virulence in., 2017, 18(2): 249–262.
[22] Zhou Y, Nie RN, Liu XY, Kong JH, Wang XH, Li JQ. GntR is involved in the expression of virulence in strainP1/7., 2018, 365(14).
[23] Gao RS, Li DF, Lin Y, Lin JX, Xia XY, Wang H, Bi LJ, Zhu J, Hassan B, Wang SH, Feng YJ. Structural and functional characterization of the FadR regulatory protein from., 2017, 7: 513.
[24] Huang WL, Wilks A. A rapid seamless method for gene knockout in., 2017, 17(1): 199.
[25] Wang TT, Qi YH, Wang ZH, Zhao JR, Ji LX, Li J, Cai Z, Yang L, Wu M, Liang HH. Coordinated regulation of anthranilate metabolism and bacterial virulence by the GntR family regulator MpaR in., 2020, 114(5): 857–869.
[26] Li ZB, Xiang ZT, Zeng JM, Li YQ, Li JY. A GntR family transcription factor inregulates biofilm formation and expression of multiple sugar transporter genes., 2019, 9: 3224.
[27] Wu KF, Xu HM, Zheng YQ, Wang LB, Zhang XM, Yin YB.CpsR, a GntR family regulator, transcriptionally regulates capsular polysaccharide biosynthesis and governs bacterial virulence in., 2016, 6: 29255.
[28] Truong-Bolduc QC, Dunman PM, Eidem T, Hooper DC. Transcriptional profiling analysis of the global regulator NorG, a GntR-Like protein of., 2011, 193(22): 6207–6214.
[29] An SQ, Lu GT, Su HZ, Li RF, He YQ, Jiang BL, Tang DJ, Tang JL. Systematic mutagenesis of all predictedgenes inpv.reveals a GntR family transcriptional regulator controlling hypersensitive response and virulence., 2011, 24(9): 1027–1039.
[30] Su HZ, Wu L, Qi YH, Liu GF, Lu GT, Tang JL. Characterization of the GntR family regulator HpaR1 of the crucifer black rot pathogenpathovar., 2016, 6: 19862.
[31] Liu GF, Su HZ, Sun HY, Lu GT, Tang JL. Competitive control of endoglucanase geneexpression in the plant pathogenby the global transcriptional regulators HpaR1 and Clp., 2019, 20(1): 51–68.
[32] Zhou XF, Yan Q, Wang N. Deciphering the regulon of a GntR family regulator via transcriptome and ChIP-exo analyses and its contribution to virulence in., 2017, 18(2): 249–262.
[33] Chi WJ. Retracted: DasR, a GntR-family global regulator, regulates N-acetylglucosamine metabolism in., 2017.
[34] Taw MN, Lee HI, Lee SH, Chang WS. Characterization of MocR, a GntR-like transcriptional regulator, in: its impact on motility, biofilm formation, and soybean nodulation., 2015, 53(8): 518–525.
[35] Browning DF, Busby SJ. The regulation of bacterial transcription initiation., 2004, 2(1): 57–65.
Progress on the GntR family transcription regulators in bacteria
Guofang Liu1, Xinxin Wang2, Huizhao Su2, Guangtao Lu2
In bacteria, GntR family transcription regulators are the widespread family of transcription factors. Members of this family consist of two functional domains, a conserved N-terminal DNA-binding domain that contains a typical helix-turn-helix (HTH) motif and a C-terminal effector-binding or oligomerization domain. Usually, the amino acid sequences of N-terminal DNA-binding domains are highly conserved, but differ in the C-terminal effector-binding or oligomerization domains. In the past several decades, many GntR family transcription regulators have been characterized in a number of bacteria. These regulators control a variety of cellular processes such as cell motility, glucose metabolism, bacterial resistance, pathogenesis and virulence. In this review, we summarized the discovery, C-terminal domains, biological function and regulation mode of GntR family transcription regulators. This review will help researchers to obtain more knowledge about the functions and mechanisms of the GntR family transcriptional regulatory factors.
GntR family transcription regulators; C-terminal domains; biological function; regulation mode
2020-09-18;
2020-12-07
广西民族大学科研基金项目(青年基金项目,编号:2019KJQN004) 和广西科技重大专项项目(桂科,编号:AA18242026) 资助 [Supported by Guangxi University for Nationalities Scientific Research Fund (No. 2019KJQN004),and Science and Technology Major Project of Guangxi (No. AA18242026)]
刘国芳,博士,副教授,研究方向:植物保护。E-mail: lgf8411@126.com
陆光涛,博士,研究员,研究方向:植物保护。E-mail: lugt@gxu.edu.cn
10.16288/j.yczz.20-245
2020/12/11 15:29:42
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20201211.0900.001.html
(责任编委: 梁海华)

