选择性多聚腺苷酸化的生物学效应及其调控机制研究进展
徐海冬,宁博林,牟芳,李辉,王宁
选择性多聚腺苷酸化的生物学效应及其调控机制研究进展
徐海冬1,2,3,宁博林1,2,3,牟芳1,2,3,李辉1,2,3,王宁1,2,3
1. 农业农村部鸡遗传育种重点实验室,哈尔滨 150030 2. 黑龙江省普通高等学校动物遗传育种与繁殖重点实验室,哈尔滨 150030 3. 东北农业大学动物科学技术学院,哈尔滨 150030
真核生物基因的前体mRNA (pre-mRNA)及一些lncRNA在成熟过程中其3'端会发生剪切和多聚腺苷酸化反应(cleavage and polyadenylation, C/P),C/P的发生需要多聚腺苷酸化信号(polyadenylation signal, PAS)的存在。选择性多聚腺苷酸化(alternative cleavage and polyadenylation, APA)是指具有多个PAS的基因,在其mRNA 3ʹ端成熟过程中,由于选择不同的PAS,导致产生出多个3'UTR长度和序列组成不同的转录异构体。3ʹUTR长度和序列的不同会影响mRNA的稳定性、翻译效率、运输和细胞定位等,因此APA是真核生物的一个重要转录后调控方式。近年来,对大量动物、植物及酵母的基因组测序分析发现,APA在真核生物广泛存在,针对APA的生物学效应和调控机制开展了一系列研究。目前已鉴定出许多APA调控的顺式调控元件和反式作用因子。本文重点介绍了APA生物学效应和调控机制的最新研究进展,并探讨了未来APA调控的研究方向。
基因表达调控;选择性多聚腺苷酸化;多聚腺苷酸化信号;顺式调控元件;反式作用因子
真核生物的mRNA前体(pre-RNA)和一些长链非编码RNA前体(pre-lncRNA)在成熟过程中其3ʹ端会被核酸内切酶剪切(cleavage),然后由模板非依赖的RNA聚合酶—poly(A)聚合酶(PAP)在3ʹ端合成一个多聚腺苷酸尾巴(polyadenylation)。真核生物mRNA前体3ʹ端的剪切和多聚腺苷酸化反应(cleavage and polyadenylation, C/P)需要多聚腺苷酸化信号(polyadenylation signal,PAS),PAS通常位于剪切位点上游10~30个核苷酸处。选择性多聚腺苷酸化(alternative cleavage and polyadenylation, APA)是指含有多个PAS的基因,在mRNA 3ʹ端成熟过程中,不同PAS的选择利用将导致一个基因产生多个转录异构体,这些转录异构体具有长短不一的3'末端[1]。近年来,由于高通量测序技术的发展和研究的深入,人们发现APA在真核生物中普遍存在,例如人类超过70%的基因都存在APA现象[2]。APA在不同类型的基因中分布不同,其中,在蛋白编码基因中分布最为广泛,其次为lncRNA和假基因,而在小RNA (small RNA)和微小RNA (microRNA)中的分布最少[3]。大多数APA仅会导致mRNA含有不同长度的3ʹUTR,但有少数APA还会改变mRNA编码区序列,导致编码不同的蛋白。3ʹUTR存在大量基因表达的顺式调控元件,这些顺式调控元件广泛参与mRNA的稳定性、翻译、运输和细胞定位[1]。APA使得同一基因的不同转录本具有不同的顺式调控元件,因而导致不同转录本的表达不同。目前已知APA是真核生物基因表达调控的重要调控机制之一,它在许多生物学过程中发挥重要的调控作用;其调控异常会导致癌症等多种疾病发生[4~7]。近年来,由于APA功能的多样性及其调控的重要性,针对APA的研究引起了人们广泛关注。本文综述了APA调控研究的最新进展,旨在为深入解析APA的调控机制和生物学效应提供参考。
1 C/P反应的顺式调控元件和反式作用因子
C/P反应是mRNA前体成熟的一个重要步骤,关系到mRNA的核外运输和胞质翻译过程。C/P反应需要位于mRNA前体的顺式元件及许多反式作用因子的参与。目前已鉴定出许多C/P反应的顺式调控元件、反式作用因子以及多种核酸酶(图1)[1,8~10]。
1.1 C/P反应的顺式调控元件
PAS是mRNA前体进行C/P反应的信号,它位于mRNA前体的3ʹ端。根据PAS的序列特征和功能,可将其组合并归类为核心元件和辅助元件两部分。其中,核心元件包括距剪切位点上游约20~22个核苷酸处的A-rich六聚体、剪切位点下游约10~30个核苷酸处的U/GU-rich元件和临近剪切位点的CA (或UA)序列[9]。AAUAAA是典型的A-rich六聚体序列,其单碱基突变所形成的非经典序列仍具有AAUAAA的部分活性[5]。下游U/GU-rich元件的单个核苷酸突变体同样也具有一定的活性。CA (或UA)是临近剪切位点前常见的两个核苷酸,它们控制剪切效率。辅助元件包括剪切位点下游的G-rich元件和剪切位点上游的UGUA元件[9]。其中G-rich元件影响剪切效率;UGUA元件促进C/P反应[10]。此外,剪切位点上游的U-rich元件也参与剪切调控。U-rich元件有两类:一类是位于A-rich六聚体和剪切位点之间的U-rich元件;另一类是位于A-rich六聚体上游的U-rich元件,这两种U-rich元件都可以与CPSF复合体互作,促进C/P反应[10]。
1.2 C/P反应的反式作用因子
目前已知有4种蛋白复合物和多个蛋白质单体参与C/P反应。参与C/P反应的4种蛋白质复合物分别是剪切多聚腺苷酸化特异因子(cleavage and polyadenylation specificity factor, CPSF)复合物、剪切激活因子(cleavage stimulation factor, CSTF)复合物、剪切因子复合物I(cleavage factor I m, CFIm)和剪切因子复合物II(cleavage factor II m, CFIIm)[1,9,11]。
CPSF复合物主要由CPSF160 (CPSF1)、CPSF100 (CPSF2)、CPSF73 (CPSF3)、CPSF30 (CPSF4)、PAP互作因子(factor interacting with PAP, FIP1)和WD重复域33 (WD repeat domain 33, WDR33) 6种蛋白构成[9,11]。CPSF160通过β螺旋结构域特异性结合AAUAAA[12];CPSF30的N端含有5个保守的锌指结构域,其中第2个和第3个锌指结构域能与PAS上游的U-rich特异性结合;CPSF73是一种锌依赖性核酸内切酶,其金属-β-内酰胺酶结构域和β-CASP结构域可精准识别CA(或UA)位点并对其临近的位点进行精准剪切[9]。FIP1的C端精氨酸富集区可与PAS下游的U-rich序列结合,并同时招募PAP[9,12]。WDR33具有富含β螺旋的WD40保守结构域,能与AAUAAA直接结合,在蛋白–蛋白和蛋白-RNA互作中发挥重要功能[12,13]。
CSTF复合物是由CSTF77 (CSTF1)、CSTF64 (CSTF2)和CSTF50 (CSTF3) 3种蛋白质亚基组成的六聚体复合物[1,11]。CSTF64和CSTF64τ (CSTF64的旁系同源体)的N端具有U-/GU-rich的识别基序(RNA recognition motif, RRM)[9]。CSTF77蛋白C端的脯氨酸富集结构域(monkey-tail, MT)与RRM附近的铰链区(hinge结构域)相互作用,促进CSTF64与RNA的结合[9];CSTF77的N端具有一个螺旋HAT结构域,可分别结合CSTF64和CSTF50,可作为支架蛋白为其他反式作用因子提供结合平台。CSTF50的保守性较低,仅存在于少数高等动物中,可通过WD40结构域与CSTF77的MT结构域相互作用,也与RNAP II的羧基末端域(carboxy-terminal domain, CTD)互作,增强其他反式作用因子的活性[1]。
CFIm复合物主要包括CFIm25、CFIm59(或CFIm68)[9,11]。CFIm25具有RRM结构域,可特异性识别PAS上游的UGUA元件,进而招募CFIm59/ CFIm68,形成CFIm复合物[9]。CFIm59的RRM结构域与CFIm25二聚体结合,增强CFIm25的RNA结合活性,同时也可促进RNA环化[10]。CFIIm复合物主要包括PCF11 (PCF11 cleavage and polyadenylation factor subunit)和CLP1 (CLP1 cleavage factor polyribonucleotide kinase subunit 1)两种蛋白质,PCF11结合在PAS的下游,在RNAP II转录终止中发挥作用,CLP1可作为连接CPSF复合物和CFIm复合物的衔接蛋白[11]。
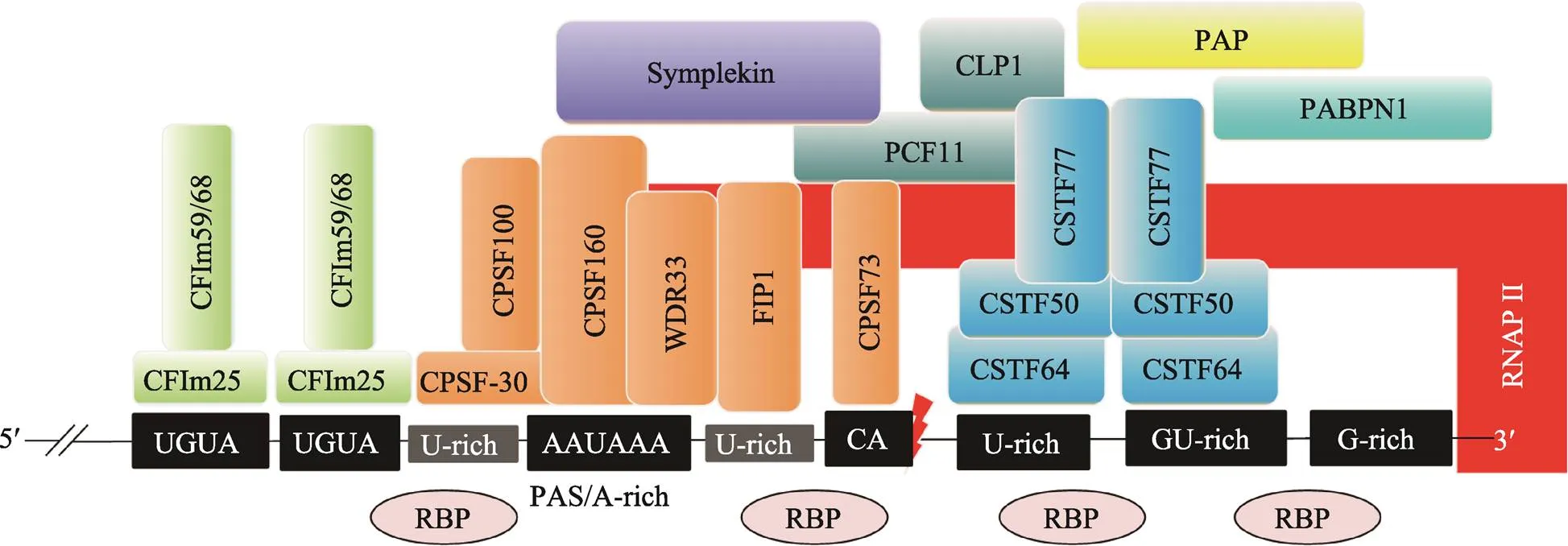
图1 C/P反应的顺式调控元件和反式作用因子
CPSF:剪切多聚腺苷酸化特异因子(cleavage and polyadenylation specificity factor);CSTF:剪切激活因子(cleavage stimulation factor);CFIIm:剪切因子复合物I (cleavage factor I m);RBP:RNA结合蛋白(RNA binding protein);RNAP II:RNA聚合酶II(RNA polymerase II);PAP:poly(A)聚合酶(poly(A) polymerase);PABPN1:核poly(A)结合蛋白(nuclear poly(A)-binding protein 1);WDR33:WD重复域33 (WD repeat domain 33);FIP1:PAP互作因子(factor interacting with PAP);CLP1:C/P因子亚基1 (cleavage factor polyribonucleotide kinase subunit 1);PCF11:C/P因子亚基(cleavage and polyadenylation factor subunit);PAS:多聚腺苷酸化信号(polyadenylation signal)。根据参考文献[1, 8~10]总结绘制。
参与C/P反应的蛋白质单体包括偶对蛋白(symplekin)、RNA聚合酶II(RNA polymerase II,RNAP II)、poly(A)聚合酶(poly(A) polymerase, PAP)和核poly(A)结合蛋白(nuclear poly(A)-binding protein 1, PABPN1)等。RNAP II的C端结构域(CTD)能招募多个蛋白复合物,为C/P反应提供酶促反应的平台。
另外,有些反式作用因子蛋白本身不能直接结合RNA,但它们可以作为支架蛋白结合上述各复合物,从而发挥调控C/P的作用,例如,Symplekin作为连接CPSF和CSTF复合物的连接蛋白。
1.3 转录与C/P反应
真核生物基因的转录延伸、RNA前体的选择性拼接以及C/P反应相偶联。RNAP II在转录延伸、选择性拼接以及C/P反应中都发挥重要作用。RNAP II的N端控制RNA的合成,RNAP II的CTD结构域可募集C/P反应所需的多种反式作用因子,为mRNA前体的C/P反应提供平台。RNAP II的CTD结构域与C/P反应的反式作用因子的结合是一个动态过程,其中CPSF复合物中的WDR33和CPSF30在其他蛋白质的参与下特异性识别AAUAAA保守基序[9]。同时CFIm25和CSTF64分别与PAS上游UGUA元件和下游U/GU-rich元件等保守基序特异性结合,并在CSTF77、Symplekin、CFIm68/CFIm59和FIP1等蛋白的作用下,CSTF、CPSF和CFIm等形成一个复合体,随后CPSF73对PAS下游剪切位点进行精准剪切[9]。CPSF和FIP1互作可招募PAP,招募的PAP会在剪切后的前体mRNA的3'端缓慢合成10~15个腺苷酸残基。此后,PABPN1会结合到短的poly(A)序列上,同时锚定PAP,快速合成poly(A),形成长约200个腺苷酸的poly(A)尾巴[1]。转录延伸与C/P反应偶联保证了RNA的精细化加工,这是一种快速精确的调控方式,以最小的耗能方式为RNA转录加工提供保障。
1.4 胞质多聚腺苷酸化
除了上述细胞核内多聚腺苷酸化,还存在胞质多聚腺苷酸化(cytoplasmic polyadenylation)。与核内多聚腺苷酸化不同,胞质多聚腺苷酸化发生于胞质内成熟的mRNA,它不与转录相偶联,也不存在剪切反应。这类mRNA没有翻译活性,它们的poly(A)尾巴通常都很短,大约有20多个核苷酸。胞质多聚腺苷酸化能延长这类mRNA的poly(A)尾巴,从而激活其翻译活性。胞质多聚腺苷酸化是调控mRNA翻译的一个重要机制,多见于卵子发生和早期发育胚胎。例如,在卵母细胞受精后,卵母细胞质存留的无翻译活性的mRNA因发生胞质多聚腺苷酸化而激活其活性,表达出合子基因组表达所需的酶和转录因子[14,15]。在胚胎期晶状体纤维分化过程中,由于晶状体蛋白相关基因的mRNA发生胞质多聚腺苷酸化,从而提高了这些mRNA的翻译效率[16]。
与核内多聚腺苷酸化相同,胞质多聚腺苷酸化也同样受到许多顺式调控元件和反式作用因子的调控,其中,顺式调控元件包括胞质多聚腺苷酸化元件(cytoplasmic polyadenylation element, CPE)、CPE上游的Pumilio结合元件(pumilio binding element, PBE)以及CPE下游的PAS[17,18];已知的反式作用因子有CPE结合蛋白(CPE binding proteins, CPEBs)、Pumilio蛋白以及CPSF160/100/30[19]。反式作用因子CPEBs能结合CPE元件,Pumilio能结合PBE元件,而CPSF160/100/30能结合PAS元件。非磷酸化的CPEB1结合Pumilio,招募脱腺苷化酶和翻译起始抑制因子,进而抑制翻译;但磷酸化的CPEB1会与脱腺苷酸化酶和翻译起始抑制因子解离,转而与CPSF互作,从而招募胞质poly(A)聚合酶PAPD4(GLD-2),快速合成80~250个核苷酸的poly(A)尾巴[17]。胞质poly(A)结合蛋白(cytoplasmic poly(A)-binding protein 1, PABPC1)结合poly(A)尾巴,它同时与mRNA 5ʹ端的帽结构复合物亚基eIF4G互作,形成闭锁环复合体(closed loop complex),从而促进翻译[19]。胞质多聚腺苷酸化与核内多聚腺苷酸化的调控有相似之处,也有不同之处。两者的相似之处,例如,两者都需要顺式调控元件PAS和反式作用因子CPSF和symplekin的参与。两者的不同之处,例如,与核内多聚腺苷酸化不同,具有多个PAS的mRNA在胞质多聚腺苷酸化中,其近端PAS可远程调控mRNA 3'末端的多聚腺苷酸化[18]。
2 APA的生物学效应
根据所在位置不同,PAS可以分为非编码区PAS (untranslated region PAS, UTR-PAS)和编码区PAS (coding region PAS, CR-PAS)两大类[1,20],其中以UTR-PAS最为多见。一个基因如果有多个PAS,不同PAS的选择利用会使同一个基因产生3ʹ末端序列组成和长度均不同的转录本。基因如果选择不同的UTR-PAS,形成的转录异构体之间3ʹUTR的序列组成和长度均存在差异,但所编码的蛋白质序列不会改变[20]。如果选择位于外显子或内含子的CR-PAS,所形成的转录本会编码出截短型蛋白质(图2)[1,20]。
真核生物mRNA的3ʹUTR存在许多的基因表达顺式调控元件,例如miRNA、lncRNA和RNA结合蛋白(RNA binding protein, RBP)的结合位点(图2)。这些调控元件在mRNA稳定性、运输、翻译以及细胞定位中发挥重要作用。APA导致同一个基因产生多个具有不同3ʹUTR的转录本,由于3ʹUTR的差异,使得这些转录本具有不同的mRNA稳定性、翻译效率、运输以及细胞定位,并最终导致它们具有不同的生物学功能。
2.1 APA产生差异性蛋白亚型
基因利用CR-PAS会编码出C端不同的蛋白异构体,有些蛋白异构体可能会缺失跨膜区或DNA结合区等重要功能结构域(图2),不同蛋白异构体的功能可能相近也可能相反[1]。有研究发现,鸡生长激素受体(growth hormone receptor,)基因存在CR-PAS和UTR-PAS。基因利用UTR-PAS会产生完整的GHR蛋白,完整的GHR定位于细胞膜,作为GH-GHR-IGF1信号通路的重要分子;而利用CR-PAS则会形成疏水跨膜域缺失的分泌蛋白—生长激素结合蛋白(growth hormone binding protein, GHBP)[21,22],主要参与维持体内GH水平的稳定[23]。在B细胞和T细胞激活过程中,多数基因选择利用CR-PAS,使得编码的蛋白质由膜锚定型转向分泌型[24]。视网膜母细胞瘤结合蛋白(retinoblastoma- binding protein 6, RBBP6)利用CR-PAS产生截短型蛋白ISO6,它与全长的RBBP6竞争性参与PAS剪切[25]。
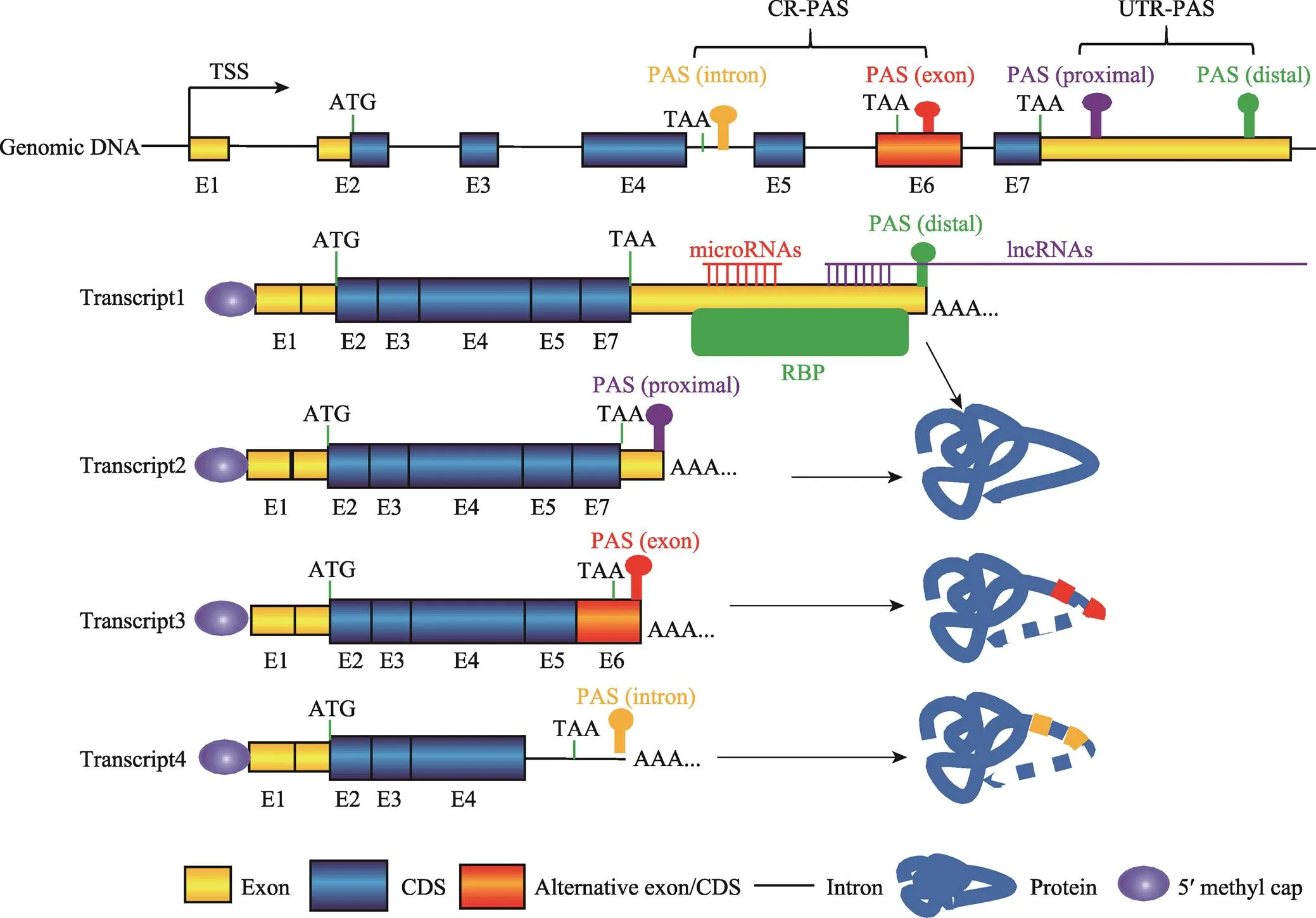
图2 PAS类型及其APA示意图
PAS:多聚腺苷酸化信号(polyadenylation signal);CR-PAS:编码区PAS (coding region PAS);UTR-PAS:非编码区PAS (untranslated region PAS);TSS:转录起始位点(transcriptional start site);CDS:蛋白质编码区(coding sequence);RBP:RNA结合蛋白(RNA binding protein);lncRNA:长链非编码RNA (long non-coding RNA)。
2.2 APA影响mRNA的稳定性
绝大多数APA可导致同一个基因转录出多个不同长度的3ʹUTR转录本。因为3ʹUTR存在调控稳定性的顺式调控元件,如miRNA的结合位点(图2),因此,同一个基因的不同转录本具有不同的稳定性[9]。一般来说,短3ʹUTR的转录本具有较高的稳定性和翻译效率,这类转录本多见于增殖期细胞;而长的3ʹUTR转录本则有利于转录后的精细化调控,这类转录本多见于细胞分化和组织发育等复杂生物学过程[26]。
干扰素调节因子5 (interferon regulatory factor 5,)是系统性红斑狼疮的候选基因。双链RNA结合蛋白STAU1能结合长3ʹUTR亚型转录本的Alu元件,导致mRNA快速降解;而短3ʹUTR亚型的转录本不存在Alu元件,因此稳定性较高[27]。是调节肌细胞和肝细胞增殖的关键基因,其长3ʹUTR亚型转录本含有结合位点,而其短3ʹUTR亚型转录本没有,因此的短3ʹUTR亚型转录本可逃避介导的基因沉默,稳定性较高[28,29]。
尽管短3ʹUTR亚型转录本可显著提升mRNA的翻译效率,但是,3ʹ端测序和蛋白定量质谱分析发现小鼠和人的T细胞中短3'UTR对蛋白质表达丰度的影响有限[30~32],提示并非所有APA引起的短3ʹUTR亚型都可提高mRNA的翻译效率。
2.3 APA影响mRNA的运输和定位
mRNA的出核转运和胞质定位受到位于3ʹUTR的定位顺式调控元件(zip codes)和反式作用因子(zip codes结合蛋白)的调控[33]。同一基因3ʹUTR序列长度和组成不同的转录本可能具有不同的定位信号,这会导致同一个基因的不同转录本在细胞中的分布不同和功能差异。
研究表明,出核蛋白(Aly/REF export factor, ALYREF)通过与PABPN1和CSTF64互作,并结合mRNA的3ʹUTR促进mRNA出核转运[34]。另外,研究发现,核RNA输出因子1 (nuclear RNA export factor1, NXF1)可与CFIm68互作,并结合于成熟的长3ʹUTR亚型转录本的UGUA元件协助其出核转运[35,36]。脑源性神经营养因子(brain-derived neurotrophic factor,)[37]和肌醇单磷酸酶(inositol monophosphatase 1)[38]的长3ʹUTR亚型转录本主要定位在神经细胞树突中,而其短3ʹUTR亚型转录本则主要分布在神经元胞体内[37]。长3ʹUTR亚型转录本3ʹUTR的U-rich元件可结合Hu antigen R (HuR),HuR会招募SET蛋白,促进SET与新翻译出的CD47蛋白互作,并同时激活Rac家族GTP酶1 (Rac family small GTPase 1, RAC1),从而使CD47向质膜转移;短3'UTR亚型转录本的3'UTR则缺少U-rich元件,不能与HuR结合,无法招募SET蛋白和激活RAC1,故造成CD47蛋白滞留在内质网中[39]。、整合素α1 (integrin α1,)和肿瘤坏死因子受体超家族成员13C (TNF receptor superfamily member 13C,)的不同蛋白质亚型也存在这种细胞定位机制[39]。虽然长3'UTR亚型转录本在细胞质中易降解,但却具有高效的出核方式,这保证了长3'UTR亚型转录本在细胞质中发挥作用,也保证了其转录后的精细调控。
3 APA调控机制
简单来说,一个具有多个PAS的基因,其mRNA前体在成熟过程中PAS的选择利用取决于各PAS发生C/P反应的时机。当mRNA前体具有两个PAS时,PAS的选择涉及Tp1、Tp2和Tt三个关键时间变量,Tp1和Tp2分别是在近端PAS和远端PAS进行C/P反应的时间,Tt是转录复合体通过两个PAS间的时间[40]。如果Tp1 < Tt + Tp2,近端PAS的利用率增加,会产生短3ʹUTR亚型转录本;如果Tp1 > Tt + Tp2,则远端PAS的利用率增加,产生长3ʹUTR亚型转录本[40]。这3个关键时间参数受到顺式调控元件信号强弱和反式作用因子作用强度等多个因素的调控。目前已知APA的调控受到PAS序列、PAS序列间的距离、染色质的修饰、转录酶的延伸速度、蛋白质-RNA互作强度和核心反式作用因子浓度的影响[1,9]。
3.1 APA顺式调控
PAS是C/P反应的顺式调控元件,PAS序列信号的强弱影响其在APA中是否被选择和利用。远端PAS常具有经典的AAUAAA、U/GU-rich等元件[26,40],而近端PAS常含有这些元件的非经典序列,其剪切活性较远端PAS低,故在APA中通常选择和利用远端PAS,这种现象普遍存在于在果蝇、小鼠和人的大多数基因[2]。
两个PAS间的序列也会影响PAS的选择。研究发现,在两个PAS间插入外源片段可提高近端PAS的利用[41]。两个PAS之间的DNA甲基化也会影响PAS的选择利用。研究发现,作为甲基化特异绝缘蛋白的CCCTC结合因子(CCCTC-binding factor, CTCF)会与DNA甲基化位点结合,并招募黏连蛋白复合物(cohesin complex)形成染色质环形结构,阻止RNAP II的转录延伸,从而促进近端PAS的选择利用[42]。
染色质空间结构也可影响PAS的选择,处在染色质较为松散区域的PAS,易被选择和利用。研究发现,酿酒酵母的组蛋白H3K4和H3K36甲基化酶SET1和SET2可调控APA,这是由于这两个甲基化酶修饰组蛋白H3后,H3K4me1和H3K36me3可增加核小体乙酰化,从而减少了核小体的位阻效应,使染色质处于松散的开放状态,有利于选择远端PAS[43]。还有研究发现APA的调控也与mRNA 3'UTR的腺嘌呤的甲基化(6-methyladenosine, m6A)和RNA二级结构有关[44,45]。
3.2 APA反式调控
APA受到前述参与C/P反应的4个蛋白质复合物的核心蛋白因子、RNA拼接因子以及其他一些RNA结合蛋白等的调控。
其中,RNAP II是APA的重要反式调控因子。黑腹果蝇中有一种突变型RNAP II,它的转录延伸效率较低,导致基因短3ʹUTR亚型转录本的表达量增加[46]。在紫外线诱导的酵母DNA损伤修复模型中,紫外线引起的RNAP II的C端高度磷酸化,在降低转录延伸效率的同时还会影响剪接体蛋白复合物的结合[47],结果导致基因利用近端PAS,产生短3ʹUTR亚型转录本[48]。
反式作用因子浓度的改变是调控APA的一个重要因素。有研究发现CSTF64/CSTF64τ或FIP1的缺失可增加多数基因远端高活性PAS的选择利用[49~51],敲低CFIm25和CFIm68会增加多数基因近端低活性PAS的选择利用[52]。在紫外线诱导DNA损伤修复的模型中,BRCA1/BARD1结合并隔绝CSTF50,使得CSTF50的浓度显著降低,造成较多基因选择利用远端PAS[53]。胚胎干细胞(ESCs)分化过程中,FIP1和CSTF各亚基的蛋白水平呈逐渐下降的趋势,同时较多的基因选择利用远端PAS。与此相一致,FIP1缺失会导致ESC细胞较多基因选择利用远端PAS[50]。降低RNA结合蛋白Nudt21会促进细胞重编程相关基因选择利用近端PAS,形成短3'UTR亚型转录本,提高其稳定性和编码能力,促进已分化细胞发生去分化,形成具有多能性的祖细胞[54]。
反式作用因子和顺式调控元件的互作强度减弱或丧失也是影响APA的重要因素。Robert等[27]发现基因近端PAS的经典序列AAUAAA突变为非经典AAGAAA后,CPSF160与其互作的能力下降,导致由选择近端PAS转而选择远端PAS,从而产生长3ʹUTR亚型的mRNA。异质性核糖核蛋白F(heterogeneous nuclear ribonucleoproteins F, hnRNP)和H/H[49]、多聚嘧啶序列结合蛋白(PTB)[55]和性别致死因子(sex lethal)[56]可结合CR-PAS下游的GU-rich元件,竞争性抑制CSTF64与GU-rich元件结合,导致UTR-PAS型选择利用增加。有报道发现PABPN1与近端PAS结合并抑制CPSF160与其互作,导致远端PAS的选择利用增加[57,58]。异质性核糖核蛋白K (heterogeneous nuclear ribonucleoptotein K, hnRNP K)可阻止CFIm68与CFIm25结合,抑制CFIm复合物与的近端PAS上游的UGUA结合,导致远端PAS的选择利用增加,从而促进长3ʹUTR亚型lncRNA_2的表达[59,60]。αCP胞嘧啶结合蛋白(αCP poly(C) binding protein, αCP PCBP)能结合C-rich元件,促进C-rich元件临近的PAS的选择利用[61]。
许多APA的调控因子自身也受到APA的调控。例如人和果蝇基因利用CR-PAS产生没有编码能力的mRNA[62],导致CSTF77的表达降低,CSTF77表达下降使其对CR-PAS的选择利用减少,UTR-PAS选择利用增加,产生具有编码能力的长亚型转录本,提高细胞内CSTF77蛋白水平[63],而高水平的CSTF77会促进CR-PAS的选择利用,产生没有编码能力的mRNA。有报导表明CSTF64和CSTF64τ也存在这种表达互抑现象[64,65]。这种通过APA调节mRNA长度的机制可以维持C/P反应所需蛋白的生理浓度,并为其他基因的APA调控提供了保障。
4 APA异常与疾病
目前已知绝大多数的真核生物基因都存在APA。作为基因表达的一个重要调控机制,APA的异常与人类疾病密切相关。目前发现,血液、免疫、神经、癌症等多种疾病与APA的异常相关[4~7]。研究发现,在地中海贫血患者中,和基因近端PAS的突变和缺失造成了远端PAS选择利用的增加[4];同样发现,在静脉血栓患者中,近端PAS突变引起的凝血酶原(prothrombin)基因远端PAS的利用增加[66]。在急性粒细胞白血病患者中,CPSF160的表达显著高于健康人群,造成白血病相关的融合基因产生高稳定性的短3'UTR亚型转录本,促进白血病细胞生长[67]。在动脉粥样硬化、II型糖尿病、结肠炎和关节炎等慢性炎症疾病患者中,炎症反应因子NLRP3的短3ʹUTR亚型转录本的表达水平较高,NLRP3的短3ʹUTR亚型转录本可逃避Tristetraprolin(TTP)和的负调控,导致NLRP3蛋白表达升高,诱导机体发生炎症反应[68]。胶质母细胞瘤的CFIm25的表达增加,导致致癌基因产生高稳定性的短3ʹUTR亚型转录本,增强了恶性胶质瘤细胞的增殖和侵袭能力[69]。在肌萎缩性脊髓侧索硬化症(amyotrophic lateral sclerosis, ALS)和额颞叶痴呆(frontotemporal dementia, FTD)患者中,抑微管装配蛋白()基因利用CR-PAS所产生的转录本不能翻译出具有功能的蛋白,导致运动神经元轴突的再生被抑制[70]。在阿尔兹海默症(Alzheimer's disease,AD)患者中,微管相关蛋白的基因选择利用近端PAS,避免了的抑制作用,导致TAU蛋白的表达增加,加剧神经元内TAU蛋白凝聚,造成神经纤维缠结[71]。在帕金森疾病(Parkinson's disease, PD)患者中,细胞内多巴胺会促进α-突触蛋白(α-synuclein)基因产生长3'UTR亚型转录本,增强了α-突触蛋白转录本的稳定性和蛋白表达水平,造成α-突触蛋白由突触末端向线粒体和细胞体转移,蓄积的α-突触蛋白形成路易小体(lewy body)[72]。慢性淋巴细胞白血病和多发性骨髓瘤细胞的3ʹ端转录组测序分析发现,肿瘤细胞的抑癌基因较多地选择利用CR-PAS,产生缺失DNA结合结构域或跨膜结构域的蛋白,这些蛋白往往丧失抑癌作用[73,74]。
5 结语与展望
近来大量的研究表明,APA是真核生物中广泛存在的一个重要基因表达调控机制,但目前人们对于许多基因APA的生物学效应和调控机制了解还不够,特别是对特定组织和细胞在特定发育阶段的APA及其APA的生物学效应和调控尚不清楚。未来有必要开展以下4个方面的工作:(1)建立高通量、高准确率的APA测序分析平台。虽然目前已建立了多种3ʹ端富集测序分析技术,但是这些方法仍存在不足之处,分析结果不够准确。未来,有必要采用第三代测序和单细胞测序技术开展特定组织或细胞在特定发育阶段的APA研究。(2)开展APA的调控机制研究。尽管目前人们已鉴定出了许多APA的顺式调控元件和反式作用因子,但还需进一步开展不同组织和细胞的特异性APA顺式调控元件和反式作用因子(包括非编码RNA)的鉴定和功能分析。(3)开展表观遗传修饰对APA的调控研究。目前已有研究发现,DNA、RNA和组蛋白的表观修饰影响基因的APA,但其作用机制尚不清楚,未来有必要开展DNA甲基化、组蛋白修饰以及RNA前体甲基化等表观修饰对APA的影响和作用机制的研究。(4)开展APA的生物学效应和调控机制的体内研究。目前,APA研究主要局限于体外试验,APA在体内真正的生物学效应和调控机制还有待阐明,未来需要利用基因敲除和基因编辑等技术,开展体内APA的生物学效应和调控机制研究。随着APA研究的不断深入,相信许多生物学过程的分子作用机制和一些人类疾病的发生机制终将会被阐明。
[1] Tian B, Manley JL. Alternative polyadenylation of mRNA precursors., 2017, 18(1): 18–30.
[2] Derti A, Garrett-Engele P, Macisaac KD, Stevens RC, Sriram S, Chen RH, Rohl CA, Johnson JM, Babak T. A quantitative atlas of polyadenylation in five mammals., 2012, 22(6): 1173–1183.
[3] Brutman JN, Zhou X, Zhang YZ, Michal J, Stark B, Jiang ZH, Davis JF. Mapping diet-induced alternative polyadenylation of hypothalamic transcripts in the obese rat., 2018, 188: 173–180.
[4] Curinha A, Oliveira Braz S, Pereira-Castro I, Cruz A, Moreira A. Implications of polyadenylation in health and disease., 2014, 5(6): 508–519.
[5] Gruber AJ, Zavolan M. Alternative cleavage and polyadenylation in health and disease., 2019, 20(10): 599–614.
[6] Patel R, Brophy C, Hickling M, Neve J, Furger A. Alternative cleavage and polyadenylation of genes associated with protein turnover and mitochondrial function are deregulated in Parkinson's, Alzheimer's and ALS disease., 2019, 12(1): 60.
[7] Nourse J, Spada S, Danckwardt S. Emerging roles of RNA 3'-end cleavage and polyadenylation in pathogenesis, diagnosis and therapy of human disorders., 2020, 10(6): 915.
[8] Jafari Najaf Abadi MH, Shafabakhsh R, Asemi Z, Mirzaei HR, Sahebnasagh R, Mirzaei H, Hamblin MR. CFIm25 and alternative polyadenylation: conflicting roles in cancer., 2019, 459: 112–121.
[9] Neve J, Patel R, Wang ZQ, Louey A, Furger AM. Cleavage and polyadenylation: ending the message expands gene regulation., 2017, 14(7): 865–890.
[10] Zheng DH, Tian B. RNA-binding proteins in regulation of alternative cleavage and polyadenylation., 2014, 825: 97–127.
[11] Knipe DM, Howley PM. Fields virology. 6th ed. 2013: Lippincott Williams & Wilkins Publishing.
[12] Chan SL, Huppertz I, Yao CG, Weng LJ, Moresco JJ, Yates JR 3rd, Ule J, Manley JL, Shi YS. CPSF30 and Wdr33 directly bind to AAUAAA in mammalian mRNA 3' processing., 2014, 28(21): 2370–2380.
[13] Hamilton K, Sun YD, Tong L. Biophysical characterizations of the recognition of the AAUAAA polyadenylation signal., 2019, 25(12): 1673–1680.
[14] Sha QQ, Zhang J, Fan HY. A story of birth and death: mRNA translation and clearance at the onset of maternal- to-zygotic transition in mammals., 2019, 101(3): 579–590.
[15] Winata CL, Łapiński M, Pryszcz L, Vaz C, Bin Ismail MH, Nama S, Hajan HS, Lee SGP, Korzh V, Sampath P, Tanavde V, Mathavan S. Cytoplasmic polyadenylation- mediated translational control of maternal mRNAs directs maternal-to-zygotic transition., 2018, 145(1): dev159566.
[16] Shao M, Lu T, Zhang C, Zhang YZ, Kong SH, Shi DL. Rbm24 controls poly(A) tail length and translation efficiency ofmRNAs in the lenscytoplasmic polyadenylation., 2020, 117(13): 7245–7254.
[17] Villalba A, Coll O, Gebauer F. Cytoplasmic polyadenylation and translational control., 2011, 21(4): 452–457.
[18] Dai XX, Jiang JC, Sha QQ, Jiang Y, Ou XH, Fan HY. A combinatorial code for mRNA 3'-UTR-mediated translational control in the mouse oocyte., 2019, 47(1): 328–340.
[19] Charlesworth A, Meijer HA, de Moor CH. Specificity factors in cytoplasmic polyadenylation., 2013, 4(4): 437–461.
[20] Chen W, Jia Q, Song YF, Fu HH, Wei G, Ni T. Alternative polyadenylation: methods, findings, and impacts., 2017, 15(5): 287–300.
[21] Lau SL. Molecular characterization of the chicken growth hormone receptor gene. Hong Kong: Hong Kong University, 2005.
[22] Lau JS, Yip CW, Law KM, Leung FC. Cloning and characterization of chicken growth hormone binding protein (cGHBP)., 2007, 33(1): 107–121.
[23] Dehkhoda F, Lee CMM, Medina J, Brooks AJ. The growth hormone receptor: mechanism of receptor activation, cell signaling, and physiological aspects., 2018, 9: 35.
[24] Cheng LC, Zheng DH, Baljinnyam E, Sun FZ, Ogami K, Yeung PL, Hoque M, Lu CW, Manley JL, Tian B. Widespread transcript shortening through alternative polyadenylation in secretory cell differentiation., 2020, 11(1): 3182.
[25] Di Giammartino DC, Li WC, Ogami K, Yashinskie JJ, Hoque M, Tian B, Manley JL. RBBP6 isoforms regulate the human polyadenylation machinery and modulate expression of mRNAs with AU-rich 3' UTRs., 2014, 28(20): 2248–2260.
[26] Turner RE, Pattison AD, Beilharz TH. Alternative polyadenylation in the regulation and dysregulation of gene expression., 2018, 75: 61–69.
[27] Graham RR, Kyogoku C, Sigurdsson S, Vlasova IA, Davies LRL, Baechler EC, Plenge RM, Koeuth T, Ortmann WA, Hom G, Bauer JW, Gillett C, Burtt N, Cunninghame Graham DS, Onofrio R, Petri M, Gunnarsson I, Svenungsson E, Rönnblom L, Nordmark G, Gregersen PK, Moser K, Gaffney PM, Criswell LA, Vyse TJ, Syvänen AC, Bohjanen PR, Daly MJ, Behrens TW, Altshuler D. Three functional variants of IFN regulatory factor 5 (IRF5) define risk and protective haplotypes for human lupus., 2007, 104(16): 6758–6763.
[28] Boutet SC, Cheung TH, Quach NL, Liu L, Prescott SL, Edalati A, Iori K, Rando TA. Alternative polyadenylation mediates microRNA regulation of muscle stem cell function., 2012, 10(3): 327–336.
[29] de Morree A, Klein JDD, Gan Q, Farup J, Urtasun A, Kanugovi A, Bilen B, van Velthoven CTJ, Quarta M, Rando TA. Alternative polyadenylation of Pax3 controls muscle stem cell fate and muscle function., 2019, 366(6466): 734–738.
[30] Gruber AR, Martin G, Müller P, Schmidt A, Gruber AJ, Gumienny R, Mittal N, Jayachandran R, Pieters J, Keller W, van Nimwegen E, Zavolan M. Global 3' UTR shortening has a limited effect on protein abundance in proliferating T cells., 2014, 5: 5465.
[31] Spies N, Burge CB, Bartel DP. 3' UTR-isoform choice has limited influence on the stability and translational efficiency of most mRNAs in mouse fibroblasts., 2013, 23(12): 2078–2090.
[32] Spangenberg L, Shigunov P, Abud APR, Cofré AR, Stimamiglio MA, Kuligovski C, Zych J, Schittini AV, Costa ADT, Rebelatto CK, Brofman PRS, Goldenberg S, Correa A, Naya H, Dallagiovanna B. Polysome profiling shows extensive posttranscriptional regulation during human adipocyte stem cell differentiation into adipocytes., 2013, 11(2): 902–912.
[33] Jambhekar A, Derisi JL. Cis-acting determinants of asymmetric, cytoplasmic RNA transport., 2007, 13(5): 625–642.
[34] Shi M, Zhang H, Wu XD, He ZS, Wang LT, Yin SY, Tian B, Li GH, Cheng H. ALYREF mainly binds to the 5' and the 3' regions of the mRNA., 2017, 45(16): 9640–9653.
[35] Ruepp MD, Aringhieri C, Vivarelli S, Cardinale S, Paro S, Schümperli D, Barabino SML. Mammalian pre-mRNA 3' end processing factor CF I m 68 functions in mRNA export., 2009, 20(24): 5211–5223.
[36] Chen SL, Wang RJ, Zheng DH, Zhang H, Chang XY, Wang K, Li WC, Fan J, Tian B, Cheng H. The mRNA export receptor NXF1 coordinates transcriptional dynamics, alternative polyadenylation, and mRNA export., 2019, 74(1): 118–131. e7.
[37] An JJ, Gharami K, Liao GY, Woo NH, Lau AG, Vanevski F, Torre ER, Jones KR, Feng Y, Lu B, Xu BJ. Distinct role of long 3' UTR BDNF mRNA in spine morphology and synaptic plasticity in hippocampal neurons., 2008, 134(1): 175–187.
[38] Andreassi C, Riccio A. To localize or not to localize: mRNA fate is in 3'UTR ends., 2009, 19(9): 465–474.
[39] Berkovits BD, Mayr C. Alternative 3' UTRs act as scaffolds to regulate membrane protein localization., 2015, 522(7556): 363–367.
[40] Davis R, Shi YS. The polyadenylation code: a unified model for the regulation of mRNA alternative polyadenylation., 2014, 15(5): 429–437.
[41] Marsollier AC, Joubert R, Mariot V, Dumonceaux J. Targeting the polyadenylation signal of pre-mRNA: a new gene silencing approach for facioscapulohumeral dystrophy., 2018, 19(5): 1347.
[42] Nanavaty V, Abrash EW, Hong CJ, Park S, Fink EE, Li ZY, Sweet TJ, Bhasin JM, Singuri S, Lee BH, Hwang TH, Ting AH. DNA methylation regulates alternative polyadenylationCTCF and the cohesin complex., 2020, 78(4): 752–764. e6.
[43] Michaels KK, Mohd Mostafa S, Ruiz Capella J, Moore CL. Regulation of alternative polyadenylation in the yeast Saccharomyces cerevisiae by histone H3K4 and H3K36 methyltransferases., 2020, 48(10): 5407–5425.
[44] Yue YN, Liu J, Cui XL, Cao J, Luo GZ, Zhang ZZ, Cheng T, Gao MS, Shu X, Ma HH, Wang FQ, Wang XX, Shen B, Wang YZ, Feng XH, He C, Liu JZ. VIRMA mediates preferential m6A mRNA methylation in 3'UTR and near stop codon and associates with alternative polyadenylation., 2018, 4: 10.
[45] Patraquim P, Warnefors M, Alonso CR. Evolution of Hox post-transcriptional regulation by alternative polyadenylation and microRNA modulation within 12 Drosophila genomes., 2011, 28(9): 2453–2460.
[46] Pinto PAB, Henriques T, Freitas MO, Martins T, Domingues RG, Wyrzykowska PS, Coelho PA, Carmo AM, Sunkel CE, Proudfoot NJ, Moreira A. RNA polymerase II kinetics in polo polyadenylation signal selection., 2011, 30(12): 2431–2444.
[47] Maita H, Nakagawa S. What is the switch for coupling transcription and splicing? RNA Polymerase II C-terminal domain phosphorylation, phase separation and beyond., 2020, 11(1): e1574.
[48] Yu LJ, Volkert MR. UV damage regulates alternative polyadenylation of the RPB2 gene in yeast., 2013, 41(5): 3104–3114.
[49] Nazim M, Masuda A, Rahman MA, Nasrin F, Takeda JI, Ohe K, Ohkawara B, Ito M, Ohno K. Competitive regulation of alternative splicing and alternative polyadenylation by hnRNP H and CstF64 determines acetylcholinesterase isoforms., 2017, 45(3): 1455–1468.
[50] Lackford B, Yao CG, Charles GM, Weng LJ, Zheng XF, Choi EA, Xie XH, Wan J, Xing Y, Freudenberg JM, Yang PY, Jothi R, Hu G, Shi YS. Fip1 regulates mRNA alternative polyadenylation to promote stem cell self- renewal., 2014, 33(8): 878–889.
[51] Hwang HW, Park CY, Goodarzi H, Fak JJ, Mele A, Moore MJ, Saito Y, Darnell RB. PAPERCLIP identifies microRNA targets and a role of CstF64/64tau in promoting non- canonical poly(A) site usage., 2016, 15(2): 423– 435.
[52] Zhou ZJ, Qu J, He L, Zhu Y, Yang SZ, Zhang F, Guo T, Peng H, Chen P, Zhou Y. Stiff matrix instigates type I collagen biogenesis by mammalian cleavage factor I complex-mediated alternative polyadenylation., 2020, 5(3): e133972.
[53] Fontana GA, Rigamonti A, Lenzken SC, Filosa G, Alvarez R, Calogero R, Bianchi ME, Barabino SML. Oxidative stress controls the choice of alternative last exonsa Brahma-BRCA1-CstF pathway., 2017, 45(2): 902–914.
[54] Brumbaugh J, Di Stefano B, Wang XY, Borkent M, Forouzmand E, Clowers KJ, Ji F, Schwarz BA, Kalocsay M, Elledge SJ, Chen Y, Sadreyev RI, Gygi SP, Hu G, Shi YS, Hochedlinger K. Nudt21 controls cell fate by connecting alternative polyadenylation to chromatin signaling., 2018, 172(1–2): 106–120. e21.
[55] Roy D, Bhanja Chowdhury J, Ghosh S. Polypyrimidine tract binding protein (PTB) associates with intronic and exonic domains to squelch nuclear export of unspliced RNA., 2013, 587(23): 3802–3807.
[56] Gawande B, Robida MD, Rahn A, Singh R.Sex-lethal protein mediates polyadenylation switching in the female germline., 2006, 25(6): 1263–1272.
[57] Jenal M, Elkon R, Loayza-Puch F, van Haaften G, Kühn U, Menzies FM, Oude Vrielink JAF, Bos AJ, Drost J, Rooijers K, Rubinsztein DC, Agami R. The poly(A)- binding protein nuclear 1 suppresses alternative cleavage and polyadenylation sites., 2012, 149(3): 538–553.
[58] Mohibi S, Zhang J, Chen XB. PABPN1, a target of p63, modulates keratinocyte differentiation through regulation of p63α mRNA translation., 2020, 11(140): 2166–2177.
[59] Naganuma T, Nakagawa S, Tanigawa A, Sasaki YF, Goshima N, Hirose T. Alternative 3'-end processing of long noncoding RNA initiates construction of nuclear paraspeckles., 2012, 31(20): 4020–4034.
[60] Naganuma T, Hirose T. Paraspeckle formation during the biogenesis of long non-coding RNAs., 2013, 10(3): 456–461.
[61] Ji XJ, Wan J, Vishnu M, Xing Y, Liebhaber SA. αCP Poly(C) binding proteins act as global regulators of alternative polyadenylation., 2013, 33(13): 2560–2573.
[62] Juge F, Audibert A, Benoit B, Simonelig M. Tissue- specific autoregulation of Drosophila suppressor of forked by alternative poly(A) site utilization leads to accumulation of the suppressor of forked protein in mitotically active cells., 2000, 6(11): 1529–1538.
[63] Luo WT, Ji Z, Pan ZH, You B, Hoque M, Li WC, Gunderson SI, Tian B. The conserved intronic cleavage and polyadenylation site of CstF-77 gene imparts control of 3' end processing activity through feedback autoregulation and by U1 snRNP., 2013, 9(7): e1003613.
[64] Yao CG, Biesinger J, Wan J, Weng LJ, Xing Y, Xie XH, Shi YS. Transcriptome-wide analyses of CstF64-RNA interactions in global regulation of mRNA alternative polyadenylation., 2012, 109(46): 18773–18778.
[65] Yao CG, Choi EA, Weng LJ, Xie XH, Wan J, Xing Y, Moresco JJ, Tu PG, Yates JR, Shi YS. Overlapping and distinct functions of CstF64 and CstF64τ in mammalian mRNA 3' processing., 2013, 19(12): 1781–1790.
[66] Ceelie H, Spaargaren-van Riel CC, Bertina RM, Vos HL. G20210A is a functional mutation in the prothrombin gene; effect on protein levels and 3'-end formation., 2004, 2(1): 119–127.
[67] Shima T, Davis AG, Miyauchi S, Kochi Y, Johnson DT, Stoner SA, Junichiro Y, Miyamoto T, Zhou JH, Ball ED, Akashi K, Zhang DE. CPSF1 regulates AML1-ETO fusion gene polyadenylation and stability in t(8; 21) acute myelogenous leukemia., 2017, 130(Suppl.1): 2498.
[68] Haneklaus M, O'neil JD, Clark AR, Masters SL, O'neill LAJ. The RNA-binding protein Tristetraprolin (TTP) is a critical negative regulator of the NLRP3 inflammasome., 2017, 292(17): 6869–6881.
[69] Masamha CP, Xia Z, Yang JX, Albrecht TR, Li M, Shyu AB, Li W, Wagner EJ. CFIm25 links alternative polyadenylation to glioblastoma tumour suppression., 2014, 510(7505): 412–416.
[70] Melamed Z, López-Erauskin J, Baughn MW, Zhang OY, Drenner K, Sun Y, Freyermuth F, Mcmahon MA, Beccari MS, Artates JW, Ohkubo T, Rodriguez M, Lin NW, Wu DM, Bennett CF, Rigo F, Da Cruz S, Ravits J, Lagier- Tourenne C, Cleveland DW. Premature polyadenylation- mediated loss of stathmin-2 is a hallmark of TDP-43- dependent neurodegeneration., 2019, 22(2): 180–190.
[71] Dickson JR, Kruse C, Montagna DR, Finsen B, Wolfe MS. Alternative polyadenylation and miR-34 family members regulate tau expression., 2013, 127(6): 739–749.
[72] Rhinn H, Qiang L, Yamashita T, Rhee D, Zolin A, Vanti W, Abeliovich A. Alternative α-synuclein transcript usage as a convergent mechanism in Parkinson's disease pathology., 2012, 3: 1084.
[73] Lee SH, Singh I, Tisdale S, Abdel-Wahab O, Leslie CS, Mayr C. Widespread intronic polyadenylation inactivates tumour suppressor genes in leukaemia., 2018, 561(7721): 127–131.
[74] Singh I, Lee SH, Sperling AS, Samur MK, Tai YT, Fulciniti M, Munshi NC, Mayr C, Leslie CS. Widespread intronic polyadenylation diversifies immune cell transcriptomes., 2018, 9(1): 1716.
Advances of functional consequences and regulation mechanisms of alternative cleavage and polyadenylation
Haidong Xu1,2,3, Bolin Ning1,2,3, Fang Mu1,2,3, Hui Li1,2,3, Ning Wang1,2,3
During the maturation of pre-mRNAs and some lncRNAs, their 3'ends are cleaved and polyadenylated. The cleavage and polyadenylation (C/P) require the presence of a polyadenylation signal (PAS) at the RNA 3ʹend. Most eukaryotic genes have multiple PASs, resulting in alternative cleavage and polyadenylation (APA). APA leads to transcript isoforms with different coding potentials and/or variable 3ʹUTRs. The 3'UTR affects mRNA stability, translation, transportation, and cellular localization. Therefore, APA is an important mechanism of posttranscriptional gene regulation in eukaryotes. In recent years, whole genome sequencing of animals, plants and yeast has revealed that APA is pervasive in eukaryotes, and the functional consequences and regulation of APA have been studied. To date, many-acting regulatory elements and-acting factors for APA regulation have been identified. In this review, we summarize the recent advances in the functional consequences and regulation of APA and discuss the future directions, aiming to provide clues and references for future APA study.
gene expression regulation; alternative cleavage and polyadenylation; polyadenylation signal;- acting regulatory element;acting factor
2020-07-07;
2020-10-19
国家自然科学基金项目(编号:31572392,3187131154)和农业部产业体系项目(编号:CARS-41)资助[Supported by the National Natural Science Foundation of China (Nos. 31572392, 3187131154), and the China Agriculture Research System (No. CARS-41)]
徐海冬,在读博士研究生,专业方向:动物遗传育种与繁殖。E-mail: 1689512382@qq.com
王宁,博士,教授,博士生导师,研究方向:动物遗传育种与繁殖。E-mail: wangning@neau.edu.cn
10.16288/j.yczz.20-200
2020/12/14 14:04:37
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20201211.1654.003.html
(责任编委: 杨昭庆)

