基于单细胞ATAC测序技术对18-三体综合征染色质开放性区域转录因子的分析
邱晓芬,汤冬娥,虞海燕,廖秋燕,胡芷洋,周俊,赵鑫,何慧燕,梁灼健,许承明,杨明1,,戴勇
基于单细胞ATAC测序技术对18-三体综合征染色质开放性区域转录因子的分析
邱晓芬1,2,3,汤冬娥3,虞海燕3,廖秋燕3,胡芷洋3,周俊3,赵鑫3,何慧燕3,梁灼健3,许承明2,杨明1,2,戴勇3
1. 广西师范大学生命科学学院,桂林 541006 2. 中国人民解放军联勤保障部队第924医院妇幼与生殖中心广西代谢性疾病研究重点实验室,桂林 541002 3. 暨南大学第二临床医学院和南方科技大学第一附属医学院(深圳市人民医院)临床医学研究中心,广东省自身免疫性疾病精准医疗工程技术研究中心,深圳市自身免疫病工程研究中心,深圳 518020
18-三体综合征是常见的常染色体非整倍体疾病之一,长期以来人们对18-三体综合征疾病的临床表型(如智力障碍、心脏、肾脏异常等)发生及发展相关的基因调控知之甚少。为探索影响该疾病表型的调控因子,本研究利用单细胞ATAC测序技术,对18-三体综合征和对照组的脐带血单个核细胞的开放性染色质区域的转录因子进行分析。捕获11,611个细胞构建的单细胞文库鉴定得到7种主要免疫细胞群,细胞数量统计的结果提示18-三体综合征的免疫系统异常。通过分析开放性染色质区域,筛选得到14个转录因子(<0.05,|FC|>1.2)。采用实时荧光定量PCR验证其中4个转录因子(TEAD1、TEAD2、TEAD4、Twist2)的相对表达量的结果符合预期。上述研究结果表明这4个转录因子可能与18-三体综合征患者心脏和骨骼发育的异常相关,为18-三体综合征表型的发生及发展的机制研究提供候选分子。
18-三体综合征;单个核细胞;单细胞ATAC测序;转录调控;转录因子
18-三体综合征(trisomy 18 syndrome)于1960年首次被遗传学研究者爱德华(Edwards JH)等[1]和史密斯(Smith DW)等[2]报道,因此又常被称为爱德华兹综合征(Edwards syndrome,ES),由于当时染色体鉴定技术尚不完善,第3条18号染色体被认为是17号染色体。ES是由于存在额外的18号染色体而导致的一系列不同身体器官和系统异常的疾病,根据患者核型的不同可分为游离型(症状典型,占90%以上)、嵌合型和易位型,活产率1/6000~1/8000,总体患病率1/2500~1/2600[3],活产率远低于总体患病率的原因是ES有很高的胎儿丢失率和死产率[4],同时早产率高于平均水平[5]。活产儿约50%在出生一周内死亡,其余大多数在一年内死亡,死亡通常是由于中枢性呼吸暂停,上呼吸道阻塞,呼吸功能不全,心力衰竭等综合因素;活产患儿平均寿命为10天[6]。ES的临床特征包括严重的和轻微的异常,产前和出生后生长缺陷,明显的精神和认知障碍。典型的轻微异常包括特征性的头面部特征(后枕部突出,耳位低,耳廓形状异常,眼裂短,小颌),拳头紧握,压指,指甲发育不良,踇趾短,胸骨短,腹股沟疝,骨盆小。最常见的重大畸形是心脏和肾脏畸形,心脏室间隔缺损,马蹄肾,肾盂缺水[3]。ES是染色体非整倍性疾病,目前无法根治,主要依靠产前筛查进行预防,对于已出生的患儿,可采用外科手术等进行延长寿命的干预措施[7]。超声波检查,人类染色体鉴定技术和高通量测序等技术的发展有效地增加了对胎儿染色体异常疾病的早期筛查力度,对预防出生缺陷具有重要意义。FitzPatrick等[8]将来自21-三体综合征和13-三体综合征病例羊水样本原代培养细胞的mRNA与来自正常细胞的mRNA的cDNA阵列进行对比杂交,当阵列cDNA按染色体位点分组时,可以清楚地识别出相关三体染色体显示出最显著的转录失调,与正常细胞相比,三体染色体上基因的平均转录水平仅提高了1.1倍。他们的数据显示大多数(>95%)>±2SD异常表达的基因没有定位到三体染色体上,并且在13-三体综合征中比21-三体综合征中发生了显著的差异表达更普遍。这暗示着基因组调控的机制可能比以往认为的更远距离上起作用,即一个染色体的增加可能会影响其他染色体基因的转录水平,三体染色体上转录因子的差异表达将产生全基因组转录失调的影响,他们得出一个结论:三体染色体上基因的略微上调导致了继发性的、广泛性的和更极端的转录失调,这种错误调控的程度可能决定了表型的严重程度。因此,探究18-三体综合征染色质的开放性区域的转录因子,不仅可以加强对18-三体综合征临床表型的发生和发展中调控因子变化的理解,而且也可为疾病的诊治提供潜在的候选分子。
随着单细胞测序技术的发展,近几年相继出现单细胞转录组测序技术、单细胞染色质可及性测序等技术。单细胞测序技术克服了传统的高通量测序技术对细胞的平均水平的基因表达情况进行分析的局限性。2015年出现了基于转座酶的高通量染色质可及性单细胞测序法scATAC-seq (single cell assay for transposase accessible chromatin using sequencing)[9],该方法建库过程不包含读段的长度筛选,可以同时检测开放性DNA区域和相应的转录因子,是基于Tn5转座酶可及染色质的开放性区域的单细胞测序的高通量测序技术。传统测序建库的过程包括DNA片段化、末端修复、接头连接、文库扩增、多次纯化分选等步骤,耗时长,但是将Tn5转座酶用于测序文库构建时,可将DNA片段化、末端修复、接头连接等多步反应转变为一步反应,缩短建库时间。染色质的可及性,也称为染色质的可接近性,是指细胞核内一些参与DNA复制或转录的大分子能够与染色质中DNA所能发生物理接触的程度,由核小体或其他染色质结合因子在染色质上的占据情况决定,也可以通过染色质中DNA对DNase的敏感性评估[10]。不同类型的细胞或在不同的生理条件或外界刺激下,细胞核中的染色质会呈现出不同的结构和状态,并且在发育期间对外部刺激的响应表现为动态变化,这些差异或动态变化的状态的表现形式之一就是染色质可及性的变化[11]。表观遗传调控的重要机制之一是通过改变染色质可及性来调控基因表达的,简而言之,染色质可及性的改变对基因的表达起着重要的调控作用。
为了定位细胞水平上与18-三体综合发生相关的调控因子,揭示相关的转录因子,本研究利用scATAC-seq技术对18-三体综合征的脐带血单个核细胞以及对照组的脐带血单个核细胞进行无细胞差异的测序及分析,探索性的对18-三体综合征的转录前调控水平进行初步的构建,揭示疾病相关活性基因的定位以及细胞特异性的相关基因作用程度。
1 材料与方法
1.1 人脐带血单个核细胞提取
选取深圳市人民医院接诊的经人类染色体核型鉴定的脐带血进行研究。纳入本次研究的实验组(N=1)材料的人类染色体G显带鉴定结果为18-三体综合征,核型结果是:47,XY,+18;对照组(N=1)材料的人类染色体G显带鉴定结果为:46,XY。2~3 mL脐带血于EDTA抗凝管中,采用淋巴细胞分离液提取脐带血单个核细胞保存备用,检测细胞活性大于80%后进行单细胞文库构建。本研究由深圳市人民医院医学伦理委员会批准,且所有孕妇签署了知情同意书。
1.2 scATAC-seq文库构建及细胞类型聚类与鉴定
ScATAC-seq使用10×微流控测序平台。本研究样本在10× Chromium平台上生成脐带血单个核细胞scATAC-seq数据的所有步骤,包括细胞核提取和悬浮、文库构建、仪器和测序设置,都遵循官方推荐,可在此下载:https://support.10xgenomics.com/ single-cell-atac。细胞核提取简要步骤:单个核细胞用PBS+0.04%BSA清洗1~2遍,(1)加入10,000~ 1,000,000个细胞至2 mL离心管,4℃ 300×离心5 min;(2)移除上清,加入100 μL冷的裂解液,吹打10次混匀,冰上孵育3~5 min;(3)在裂解的细胞中加入1 mL冷的Wash Buffer,吹打混匀5次,4℃ 500×离心5 min;(4)移除上清,根据步骤(1)的细胞浓度,设细胞溶解过程中有50%的细胞核丢失,用冷的细胞核稀释缓冲液重悬,放置在冰上。(5)细胞核计数。得到细胞核后进行核悬浮转座: (1)准备转座Mix;(2)等温孵化;(3)配制Master Mix;(4) Load Chromium Next GEM芯片;(5)准备Master Mix转座后的核,准备凝胶珠;(6)上机,运行仪器后将GEMs转移出来。得到测序的原始数据后,用Cell Ranger ATAC通过Latent Semantic Analysis (LSA)[12]进行聚类和t-SNE分析。使用官方软件Cell Loupe进行细胞类型鉴定,通过细胞开放区域特异性的细胞标志基因的特定表达确定细胞类型。我们使用MOODS (https://github.com/jhkorhonen/MOODS)的Python库,在Cell Ranger ATAC中包装,扫描每个峰(peak)是否与基团位置匹配,用JASPAR数据库[13]注释peak。
1.3 实时荧光定量PCR检测差异表达的转录因子
为了验证这些转录因子是否在疾病组中是否差异表达,采用实时荧光定量PCR (quantitative real- time PCR, qPCR)检测TEAD1、TEAD2、TEAD4和Twist2这4个转录因子在疾病组和对照组中的表达量,设置3个实验重复。步骤如下:(1) Trizol法提取细胞悬液的RNA;(2)使用提取的RNA合成cDNA:采用反转录试剂VAZYME R222-01进行cDNA逆转录合成,冰上配制逆转录体系(20 μL):RNA加1 pg~ 1 μg,5×HiScript®II qRT SuperMixa加2 μL,加入RNase-free H2O将体系的体积配至20 μL,50℃温育15 min,85℃高温灭活5 s,收集反转录的cDNA,用于荧光定量检测;(3)以反转录的cDNA为模板,分别加入引物T-β-actin,xw-0000291,caspase-3,Bcl-2,Beclin1,Bax进行相对定量分析,反应体系如表1,将96-PCR板置于Realtime PCR仪(Biometra Tone)上进行PCR反应。所有的指标均按以下程序进行:95℃,30秒;40个PCR循环(95℃,5 s;60℃,34 s (收集荧光))。为了建立PCR产物的熔解曲线,扩增反应结束后,按95℃,15 s;60℃,60 s;95℃,15 s进行反应;并从60℃缓慢加热到99℃ (仪器自动进行Ramp Rate为0.05 ℃/s),反应的引物序列见表2。各样品的目的基因和管家基因分别进行Realtime PCR反应,根据测得的各样品目的基因和管家基因的Ct值进行ddct法的相对定量分析。
2 结果与分析
2.1 聚类鉴定得到7类主要免疫细胞群
通过10× Genomics平台的微流控系统对实验组(ES)和对照组(NC)进行scATAC-seq。在去除低质量和不合格的细胞后,一共捕获了11,611个细胞,其中ES组5296个细胞,NC组6315个细胞。通过对单细胞测序得到的数据进行聚类分析后得到7个不同类别的类群。对这些细胞类群进行无抗体的细胞标记基因鉴定,确定了7类主要的免疫细胞,然后用已知的细胞标记基因进行鉴定[14,15]。用和鉴定T细胞(T),用鉴定B细胞(B),用鉴定自然杀伤细胞(natural killer cell, NK),用和鉴定树突状细胞(dendritic cell, DC),用鉴定单核细胞(monocyte),用和鉴定CD4+T细胞,用鉴定CD8+T细胞(图1),每种颜色峰的高度代表对应细胞类型染色质的开放程度。统计各细胞群数量的结果显示,ES组中,T细胞,NK细胞和DC细胞的细胞数量显著减少,B细胞和CD4+T细胞的细胞数量显著增加(图2)。研究结果提示,18-三体综合征患儿在子宫内免疫系统发育异常。让人兴奋的是,早在1994年,就有研究人员[16]研究了18-三体综合胎儿血液的免疫系统,在妊娠20~36周时通过脐带穿刺术从8个18-三体综合征的胎中获得的胎儿血液中淋巴细胞亚群,使用流式细胞仪分析并计数,得出结论:与染色体正常胎儿的相比,在18-三体综合征中,平均T细胞和NK细胞计数显著降低。
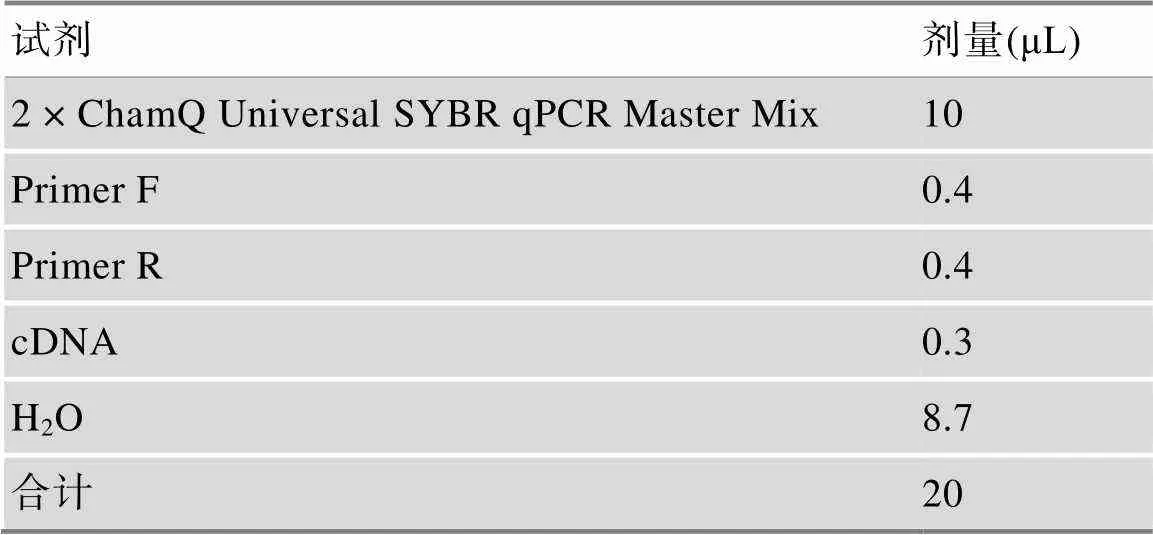
表1 qPCR的反应体系

表2 qPCR的引物序列
2.2 可及性位点差异转录因子
在细胞层面上对18-三体综合征进行了差异可及性位点的motif分析,motif是转录因子结合的基序,通过motif可以找到结合该motif的转录因子。以<0.05, |FC|>1.2为筛选阈值,其中B细胞筛选得到11个转录因子:Esrra、PBX1、TEAD1、TEAD2、TEAD4、Twist2、HOXC12、ZNF410、SREBF1、FOSL2::JUN、FOS::JUNB;单核细胞(Monocyte)筛选得到7个转录因子:Esrra、PBX1、TEAD1、TEAD2、HOXC12、TBX20、ZNF410。T细胞筛选得到6个因子:Esrra、PBX1、TEAD1、TEAD2、TEAD4、MYBL1。自然杀伤细胞(NK)筛选得到1个差异转录因子:SREBF2;树突细胞群(DC) 1个差异转录因子:HOXC12;CD8+T细胞群筛选得到1个转录因子:TEAD1;CD4+T细胞群没有得到筛选到转录因子(图4)。通过整理,一共得到筛选得到14个的转录因子:Esrra、PBX1、TEAD1、TEAD2、TEAD4、Twist2、HOXC12、ZNF410、SREBF1、FOSL2::JUN、FOS::JUNB、SREBF2、TBX20、MYBL1。随后我们对不同细胞类群的差异转录因子调控的基因进行了GO功能分析。图5展示了B细胞群差异转录因子调控基因功能分析的结果,其他细胞群的结果见附加材料(附图1~5)。结果显示在已鉴定到的7类主要免疫细胞的主要生物进程功能是转录调控,信号转导;细胞组分功能主要集中在细胞核、细胞膜和细胞质;分子功能主要是蛋白质结合、金属离子结合、DNA结合。
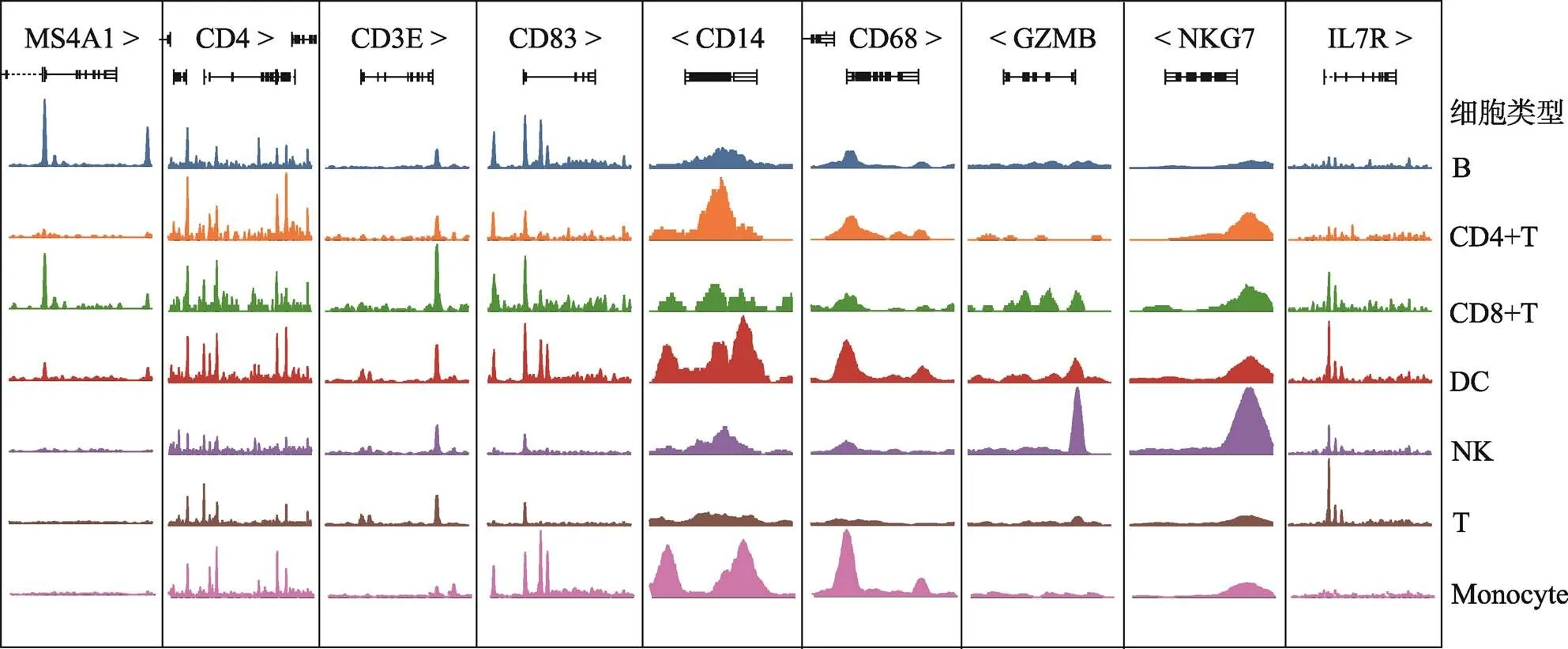
图1 部分特异性细胞标记基因对应染色质可及性开放程度

图2 脐带血单个核细胞t-SNE聚类与鉴定
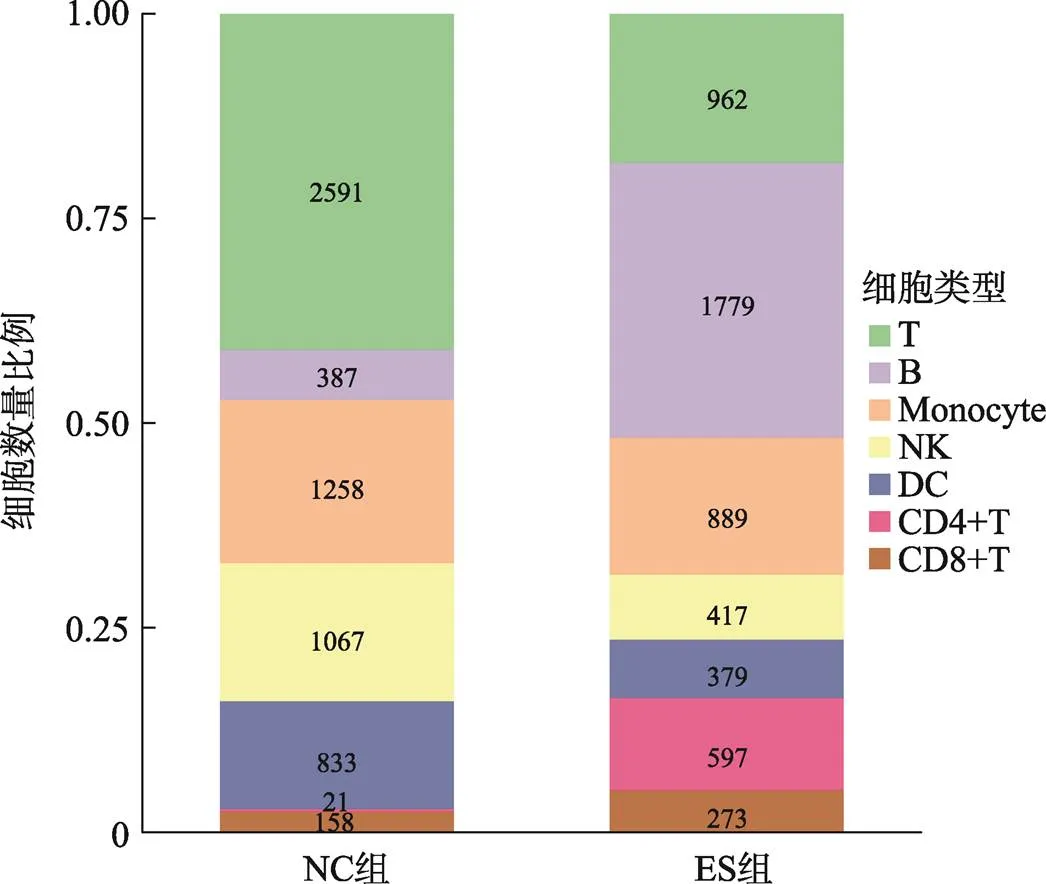
图3 7类细胞群数量及比例
2.3 转录因子qPCR相对表达量
qPCR实验验证了4个转录因子相对表达量的结果(图6),4个实验组(ES)的转录因子(TEAD1、TEAD2、TEAD4、Twist2)的相对表达量低于对照组(NC),符合实验预期的结果。
3 讨论
单细胞ATAC-seq技术能在单细胞水平上捕获每种细胞类型的开放性染色质区域,实现无标记鉴定细胞类型特异性的顺式和反式调控元件和与疾病相关转录因子活性的定位。本研究得到了疾病组和对照组的单细胞文库,鉴定得到7类主要的免疫细胞群,筛选得到14个转录因子,选取了4个通过qPCR验证的转录因子进行讨论。本研究的不足是样本数量不足。采用单细胞测序技术,得出7种主要的免疫细胞数量比例的结果以及差异转录因子,对18-三体综合征疾病的认识有意义。
已有研究者通过对18-三体综合征胎儿的脐带血进行流式细胞仪分析后报道[16]18-三体综合征胎儿宫内时期的免疫系统的异常情况。本次研究通过单细胞测序技术,进行无抗体的细胞群鉴定,发现了18-三体综合征胎儿脐带血单个核细胞比例与对照组的差异。随后我们发现了疾病组差异表达的转录因子,验证了其中4个(TEAD1、TEAD2、TEAD4、Twist2)转录因子的相对表达量与实验结果相符。
转录增强关联域(transcriptional enhanced associate domain, TEAD)蛋白家族由4个旁系转录因子组成,其功能是调节基因表达,以响应Hippo信号通路,Hippo信号通路是调控器官发育、细胞生长、增殖和组织稳态和再生的重要转录信号通路[17~19]。2019年Akerberg BN等研究发现TEAD1是心脏转录调控网络的核心组成部分,控制心脏调控区域和心肌细胞特异性基因功能[20]。Joshi S等[21]的研究表明因子在肌细胞分化中具有特定作用,TEAD1是已知的Hippo信号转导的转录因子,参与心脏发育。Wen T等[22]的研究揭示了TEAD1在小鼠心血管发育中的关键作用,并确定了TEAD1在遗传调节层次的上游起作用,以促成平滑肌收缩。Osman I等[23]的研究表明TEAD1通过转录诱导SLC1A5促进血管平滑肌细胞增殖,从而激活mTORC1信号传导并促进新血管内膜形成。同时,Liu R等[24]的研究表明TEAD1在维持正常的成人心脏功能中具有非常重要的作用。
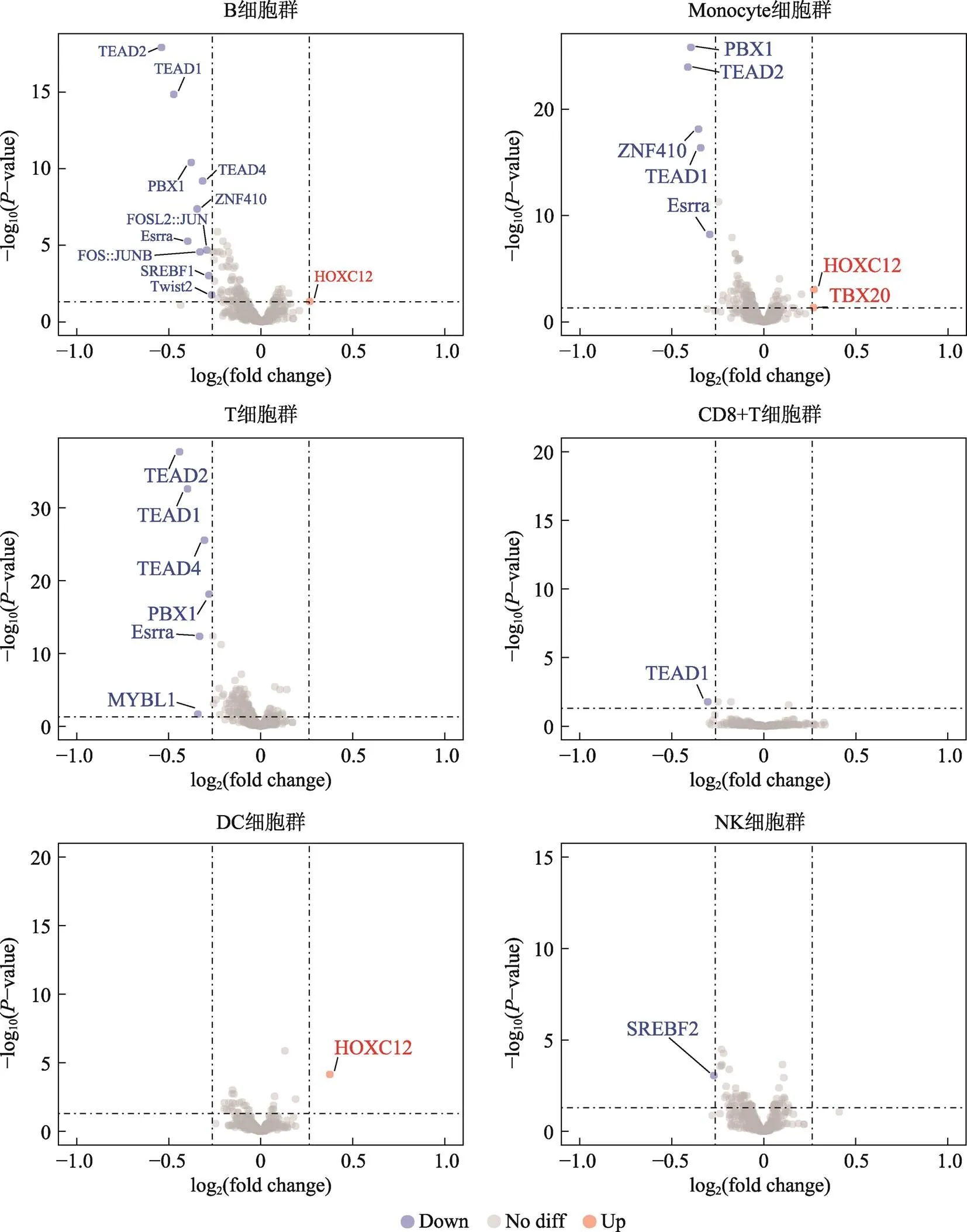
图4 不同细胞群转录因子差异表达火山图
Twist2是Twist子家族的高度保守成员,负责间质细胞谱系中发育程序的转录调控[25]。碱性螺旋–环–螺旋(Basic helix-loop-helix protein, b HLH)家族成员Twist2对间质细胞系的发生和发育起转录调节作用,直接或间接机制发挥分子开关功能,从而激活或抑制靶基因,Twist2对骨骼发育存在影响[26]。Liu N等[27]通过在小鼠中表达Twist2转录因子的祖细胞的谱系追踪,发现了位于成年骨骼肌基底层之外的肌源系,并发现Twist2祖细胞在肌肉再生过程中对IIb/x型肌纤维有重要贡献。同时,2019年Albizua I等[28]对18-三体综合征患者的基因组表达谱分析后得出结论:SHOX2、TBX4、ALX3、ALX4和Twist1等关键转录因子在18-三体相关骨骼发育中起关键作用,他们的研究结果中报告了Twist家族的转录因子Twist1表达降低的小鼠表现出多指畸形。2010年,Koide等[29]对18-三体综合征和对照组的羊水上清提取总RNA,发现与35个基因与肾上腺发育有关的基因的显著下调,其中17个是焦点基因。

图5 B细胞开放性区域差异转录因子调控基因GO分析
本研究在单细胞染色质可及性的水平上揭示免疫细胞类群异常的转录因子,特别是经过实验证实下调表达4个的转录因子(TEAD1、TEAD2、TEAD4、Twist2)。TEAD (TEAD1、TEAD2、TEAD4)转录因子家族与成肌细胞分化有关,特别是TEAD1与心脏,心血管发育等有关,的下调表达可能会影响18-三体综合征患者心脏的正常的发育调控;Twist2是可能导致18-三体综合征的骨骼异常的转录因子。揭示18-三体综合征表型发生和发展的机制,仍有更远的路要走。
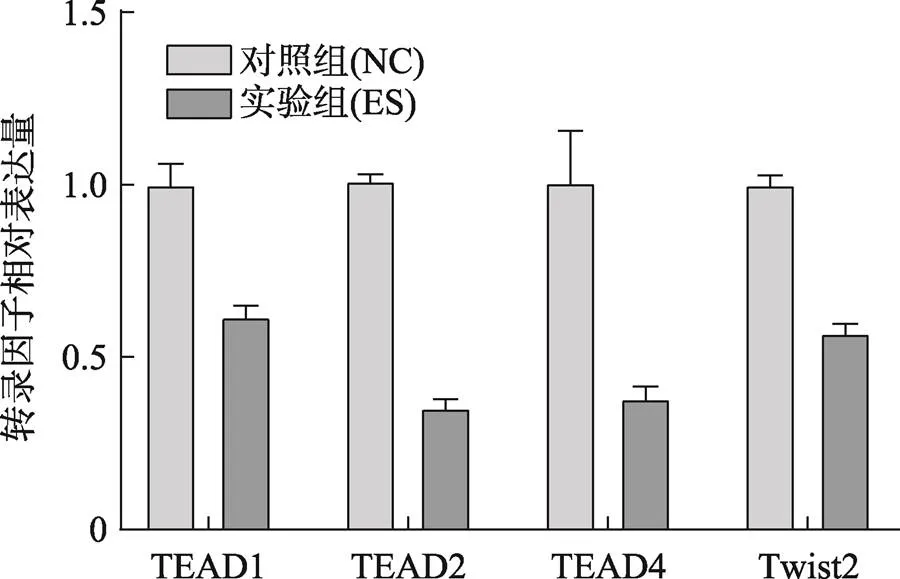
图6 脐带血单个核细胞转录因子qPCR相对表达量
附录:
附加材料详见电子版http://www.chinagene.cn。

附图1 Monocytes开放性区域差异转录因子调控基因GO分析
Suppl fig. 1 GO analysis of genes regulated by differential transcription factors in accessible chromatin region of monocytes
附图2 T细胞群开放性区域差异转录因子调控基因GO分析
Suppl fig. 2 GO analysis of genes regulated by differential transcription factors in accessible chromatin region of T cells

附图3 CD8+T细胞群开放性区域差异转录因子调控基因GO分析
Suppl fig. 3 GO analysis of genes regulated by differential transcription factors in accessible chromatin region of CD8+T cells

附图4 DC细胞群开放性区域差异转录因子调控基因GO分析
Suppl fig. 4 GO analysis of genes regulated by differential transcription factors in accessible chromatin region of DC

附图5 NK细胞群开放性区域差异转录因子调控基因GO分析
Suppl fig. 5 GO analysis of genes regulated by differential transcription factors in accessible chromatin region of NK
[1] Edwards JH, Harnden DG, Cameron AH, Crosse VM, Wolff OH. A new trisomic syndrome.,1960, 1(7128): 787–790.
[2] Smith DW, Patau K, Therman E, Inhorn SL. A new autosomal trisomy syndrome: multiple congenital anomalies caused by an extra chromosome.,1960, 57: 338–345.
[3] Cereda A, Carey JC. The trisomy 18 syndrome.,2012, 7: 81.
[4] Cavadino A, Morris JK. Revised estimates of the risk of fetal loss following a prenatal diagnosis of trisomy 13 or trisomy 18.,2017, 173(4): 953–958.
[5] Yamanaka M, Setoyama T, Igarashi Y, Kurosawa K, Itani Y, Hashimoto S, Saitoh K, Takei M, Hirabuki T. Pregnancy outcome of fetuses with trisomy 18 identified by prenatal sonography and chromosomal analysis in a perinatal center.,2006, 140(11): 1177–1182.
[6] Rasmussen SA, Wong LYC, Yang QH, May KM, Friedman JM. Population-based analyses of mortality in trisomy 13 and trisomy 18.,2003, 111(4 Pt 1): 777–784.
[7] Nelson KE, Rosella LC, Mahant S, Guttmann A. Survival and surgical interventions for children with trisomy 13 and 18.,2016, 316(4): 420–428.
[8] FitzPatrick DR, Ramsay J, McGill NI, Shade M, Carothers AD, Hastie ND. Transcriptome analysis of human autosomal trisomy.,2002, 11(26): 3249– 3256.
[9] Buenrostro JD, Wu BJ, Litzenburger UM, Ruff D, Gonzales ML, Snyder MP, Chang HY, Greenleaf WJ. Single-cell chromatin accessibility reveals principles of regulatory variation.,2015, 523(7561): 486–490.
[10] Schueler MG, Sullivan BA. Structural and functional dynamics of human centromeric chromatin.,2006, 7: 301–313.
[11] Hsiung CCS, Morrissey CS, Udugama M, Frank CL, Keller CA, Baek S, Giardine B, Crawford GE, Sung MH, Hardison RC, Blobel GA. Genome accessibility is widely preserved and locally modulated during mitosis.,2015, 25(2): 213–225.
[12] González J, Muñoz A, Martos G. Asymmetric latent semantic indexing for gene expression experiments visualization.,2016, 14(4): 1650023.
[13] Sandelin A, Alkema W, Engström P, Wasserman WW, Lenhard B. JASPAR: an open-access database for eukaryotic transcription factor binding profiles.,2004, 32(Database issue): D91–94.
[14] Schelker M, Feau S, Du JY, Ranu N, Klipp E, MacBeath G, Schoeberl B, Raue A. Estimation of immune cell content in tumour tissue using single-cell RNA-seq data.,2017, 8(1): 2032.
[15] Sinha D, Kumar A, Kumar H, Bandyopadhyay S, Sengupta D. dropClust: efficient clustering of ultra-large scRNA-seq data.,2018, 46(6): e36.
[16] Makrydimas G, Plachouras N, Thilaganathan B, Nicolaides KH. Abnormal immunological development in fetuses with trisomy 18.,1994, 14(4): 239–241.
[17] Lin KC, Park HW, Guan KL. Regulation of the Hippo pathway transcription factor TEAD.,2017, 42(11): 862–872.
[18] Chan P, Han X, Zheng BH, DeRan M, Yu JZ, Jarugumilli GK, Deng H, Pan DJ, Luo XL, Wu X. Autopalmitoylation of TEAD proteins regulates transcriptional output of the Hippo pathway.,2016, 12(4): 282–289.
[19] Noland CL, Gierke S, Schnier PD, Murray J, Sandoval WN, Sagolla M, Dey A, Hannoush RN, Fairbrother WJ, Cunningham CN. Palmitoylation of tead transcription factors is required for their stability and function in hippo pathway signaling.,2016, 24(1): 179–186.
[20] Akerberg BN, Gu F, VanDusen NJ, Zhang XR, Dong R, Li K, Zhang B, Zhou B, Sethi I, Ma Q, Wasson L, Wen T, Liu JH, Dong KZ, Conlon FL, Zhou JL, Yuan GC, Zhou PZ, Pu WT. A reference map of murine cardiac transcription factor chromatin occupancy identifies dynamic and conserved enhancers.,2019, 10(1): 4907.
[21] Joshi S, Davidson G, Le Gras S, Watanabe S, Braun T, Mengus G, Davidson I. TEAD transcription factors are required for normal primary myoblast differentiationand muscle regeneration.,2017, 13(2): e1006600.
[22] Wen T, Liu JH, He XQ, Dong KZ, Hu GQ, Yu LY, Yin Q, Osman I, Peng JT, Zheng ZQ, Xin HB, Fulton D, Du QS, Zhang W, Zhou JL. Transcription factor TEAD1 is essential for vascular development by promoting vascular smooth muscle differentiation.,2019, 26(12): 2790–2806.
[23] Osman I, He XQ, Liu JH, Dong KZ, Wen T, Zhang FZ, Yu LY, Hu GQ, Xin HB, Zhang W, Zhou JL. TEAD1 (TEA domain transcription factor 1) promotes smooth muscle cell proliferation through upregulating slc1a5 (solute carrier family 1 member 5)-mediated glutamine uptake.,2019, 124(9): 1309–1322.
[24] Liu RY, Lee J, Kim BS, Wang QL, Buxton SK, Balasubramanyam N, Kim JJ, Dong JR, Zhang AJ, Li SM, Gupte AA, Hamilton DJ, Martin JF, Rodney GG, Coarfa C, Wehrens XH, Yechoor VK, Moulik M. Tead1 is required for maintaining adult cardiomyocyte function, and its loss results in lethal dilated cardiomyopathy.,2017, 2(17): e93343.
[25] Franco HL, Casasnovas J, Rodríguez-Medina JR, Cadilla CL. Redundant or separate entities?--roles of Twist1 and Twist2 as molecular switches during gene transcription.,2011, 39(4): 1177–1186.
[26] Zhao CX, Ze Y. Biological function and molecular mechanism of Twist2., 2015, 37(1): 17–24.
[27] Liu N, Garry GA, Li S, Bezprozvannaya S, Sanchez-Ortiz E, Chen BB, Shelton JM, Jaichander P, Bassel-Duby R, Olson EN. A Twist2-dependent progenitor cell contributes to adult skeletal muscle.,2017, 19(3): 202–213.
[28] Albizua I, Chopra P, Sherman SL, Gambello MJ, Warren ST. Analysis of the genomic expression profile in trisomy 18: insight into possible genes involved in the associated phenotypes.,2020, 29(2): 238–247.
[29] Koide K, Slonim DK, Johnson KL, Tantravahi U, Cowan JM, Bianchi DW. Transcriptomic analysis of cell-free fetal RNA suggests a specific molecular phenotype in trisomy 18.,2011, 129(3): 295–305.
Analysis of transcription factors in accessible open chromatin in the 18-trisomy syndrome based on single cell ATAC sequencing technique
Xiaofen Qiu1,2,3, Dong’e Tang3, Haiyan Yu3, Qiuyan Liao3, Zhiyang Hu3, Jun Zhou3, Xin Zhao3, Huiyan He3, Zhuojian Liang3, Chengming Xu2, Ming Yang1,2, Yong Dai3
Trisomy 18 syndrome is one of the most common autosomal aneuploidy disorders. Little is known about the genetic regulation leading to the clinical phenotypes associated with the occurrence and development of trisomy 18 syndrome disorders (e.g., mental retardation, cardiac and renal abnormalities). To explore the regulatory factors that influence the phenotypes of the disease, this study used single-cell ATAC sequencing to analyze transcription factors in the accessibility chromatin regions of the single-nucleus cells of the cord blood from 18-trisomy syndrome and control subjects. A single-cell library constructed by capturing 11,611 cells identified seven major immune cell populations, and the results of cell number statistics suggested the presence of abnormalities in the immune system of 18-trisomy syndrome patients. Fourteen transcription factors (<0.05, |FC|>1.2) were identified by analyzed accessibility chromatin regions. The relative expression levels of four of these transcription factors (TEAD1, TEAD2, TEAD4, Twist2) were confirmed using real-time quantitative fluorescence PCR. In conjunction with information from the literature, this study suggests that these four transcription factors may be associated with abnormalities in cardiac and skeletal development in patients with the 18-trisomy syndrome, thereby providing candidate molecules for mechanistic studies on the occurrence and development of the 18-trisomy syndrome phenotypes.
trisomy 18; single nucleated cells; single-cell ATAC sequencing; transcriptional regulation; transcription factors
2020-11-08;
2020-12-24
广东省科技计划项目(编号:2017B020209001),深圳市科技计划项目(编号:GJHZ20180413181714541)广西重点实验室建设项目(编号:20-065-76)和桂林市科学研究与技术开发计划项目(编号:2016012702-1)资助[Supported by Science and Technology Planning Project of Guangdong Province, China (No. 2017B020209001), the Science and Technology Plan of Shenzhen (No. GJHZ20180413181714541), Guangxi Key Laboratory of Metabolic Diseases Research (No. 20-065-76), and the Scientific Research and Technology Development Planning Project of Guilin (No. 2016012702-1)]
邱晓芬,在读硕士研究生,专业方向:生物学。E-mail: byqxf123@163.com
戴勇,博士,主任医师,教授,研究方向:医学遗传和产前诊断。E-mail: daiyong22@aliyun.com 杨明,主任医师,学士,硕士生导师,研究方向:传染病学。E-mail: yangming181@yeah.net
10.16288/j.yczz.20-283
2021/1/13 13:51:18
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210112.1513.004.html
(责任编委: 吴旭东)

