新型冠状病毒SARS-CoV-2特异性可检测靶点的确认与重叠PCR合成
肖正午,肖 莉,传 军,李亚梅,彭 壮,林丽美,李 凯,戴爱国,庹勤慧*,廖端芳*
(1.湘产大宗药材品质评价湖南省重点实验室,湖南 长沙 410208;2.苏州大学附属第二医院,江苏 苏州 215004;3.人和未来生物科技长沙有限公司,湖南 长沙 410000;4.湖南中医药大学医学院呼吸疾病研究室,湖南 长沙 410000)
对病原体的基因检测是感染性疾病确诊的关键指标之一。根据病原体在机体内繁殖部位的不同,采集生物标本的方式相对应有所差异,包括采集各种体液(如血液、脑脊液、痰液、尿液)以及直接采集细胞(如咽喉拭子、宫颈刮片)等。一般情况下,由于病原体在感染人体后有较快速和较大量的繁殖,敏感性一般不是制约基因检测的关键因素,但对特定种类的病毒如SARS-CoV-2,由于其感染的部位特点,检测敏感性显得尤为重要。
2019年底发生的COVID-19大流行,其严重程度都远远超过了医学界的预期。观察发现SARS-CoV-2主要在下呼吸道繁殖,上呼吸道细胞内的病毒拷贝数相对较低[1-2]。因此,导致以咽喉拭子作为检测标本的SARS-CoV-2基因检测假阴性比例相对偏高[3]。而假阴性的真阳性个体,无论是对患者的治疗,还是对无症状感染者的追踪朔源,都是新冠疫情防治中需要克服的重要节点。近期,国际上一些国家和地区再度出现的聚集性感染,特别是一些不可溯源的病例,极有可能是由于现有核酸检测方法的敏感性不够(假阴性比例相对偏高),导致一些无症状感染者没能及时被发现所致。
目前临床上采用的SARS-CoV-2检测试剂盒,主要技术原理为二代测序,基于TaqMan探针的实时PCR,基于环介导等温扩增技术(loop-mediated isothermal amplification, LAMP)的实时PCR和基于Cas核酸酶的检测[4-7]。在检测靶点上,大部分选择1~2个靶点[8-10]。靶点的数量和探针的多少,一定程度上除决定检测的特异性外,更对检测的敏感性有直接的影响。本研究从生物信息学分析入手,并采用重叠PCR合成方法,选择了可用于SARS-CoV-2检测的8个新靶点。这些靶点在扩增引物和适宜于TaqMan探针检测两方面均呈现出很好的的SARS-CoV-2特异性。有望成为未来SARS-CoV-2高灵敏度检测方法及试剂盒开发的重要对象。
1 材料与方法
1.1 材料
本实验所有寡核苷酸包括引物、模板片段、TaqMan探针均由上海生工合成。PCR所用聚合酶购自上海生工公司的Taq Mix。
1.2 方法
1.2.1 SARS-CoV-2特异性序列比对采用如下策略
(1)选定代表序列;(2)提取近源序列;(3)从病毒序列数据库排除代表序列和近源序列,作为非近源序列;(4)利用blast局部比对将代表序列与近源序列进行比对;(5)根据比对结果提取非同源保守区域;(6)利用blast局部比对将代表序列与非近源序列进行比对;(7)根据比对结果提取同源保守区域;(8)综合排除非同源保守区域与同源保守区域,得到该物种的特异性保守区域;(9)选择合适的特异性保守区域作为模板,用来设计引物及探针。
1.2.2 SARS-CoV-2特异性引物设计采用如下策略
(1)选择refseq数据库中的新型冠状病毒序列作为代表序列;(2)选择其它新型冠状病毒株的完整序列作为近源序列;(3)从病毒序列数据库排除SARS-CoV-2相关序列,作为非近源序列;随后的6个步骤与SARS-CoV-2特异性序列比对步骤(4)~(9)相同。模板片段设计为侧翼含有相应扩增引物的特异性序列和中间包含至少可以设计一个TaqMan探针的特异性序列。其中引物序列长度不短于20个碱基,探针长度不短于23个碱基。
拟合成和测试的SARS-CoV-2靶点片段采用重叠PCR合成。各片段重叠碱基为18个或以上。根据选取的靶点长度,分别合成4~5个片段用于重叠PCR,重叠PCR采用一步法或多步法。重叠PCR合成的产物最后送测序验证。
2 结 果
2.1 8个SARS-CoV-2特异性模板序列和相应探针序列的确定
表1列出了8个SARS-CoV-2特异性模板序列,用于重叠PCR的寡核苷酸片段和可用于荧光定量PCR的TaqMan探针及用于荧光定量PCR的引物。从表1可见,这些模板片段长度在100~300碱基之间,是荧光定量PCR扩增元的适宜长度;片段侧翼含有连续18个以上的特异性序列;片段中含有至少一个可用于TaqMan探针设计、连续长度等于或大于23个碱基的SARS-CoV-2特异性序列。

表1 8个SARS-CoV-2特异性模板序列、引物和探针序列
2.2 8个SARS-CoV-2特异性模板片段的重叠PCR合成
模板序列5和6采用一步法合成,分别以两段模板片段为DNA模板各1 μL加入PCR反应体系,2×Taq PCR Master Mix 12.5 μL,补水至总体积25 μL。PCR反应条件如下:95 ℃预变性2 min,95 ℃ 10 s,55 ℃ 20 s,72 ℃ 30 s,共30个循环,72 ℃ 10 min。其PCR扩增结果见图1a。
模板序列1,2,3,4,7和8采用多步法合成。第一步分别以1号2号两段模板片段为DNA模板各1 μL加入PCR反应体系,2×Taq PCR Master Mix 12.5 μL,补齐水至总体积25 μL。PCR反应条件如下:95 ℃预变性2 min,95 ℃ 15 s,52 ℃ 25 s,72 ℃ 10 s,共35个循环,72 ℃ 10 min。将第一步PCR反应产物稀释10000倍作为第二步PCR的模板,并加入3号和4号模板片段各1 μL,2×Taq PCR Master Mix 12.5 μL,补齐水至总体积25 μL。PCR反应条件如下:95 ℃预变性2 min,95 ℃ 15 s,52 ℃ 25 s,72 ℃ 10 s,共35个循环,72 ℃ 10 min。以此类推,将第二步PCR反应产物稀释10 000倍作为第三步PCR的模板,最终扩增得到PCR产物序列,分步结果见图1b~d。8个SARS-CoV-2特异性模板序列最终产物电泳结果见图1e。
将8个SARS-CoV-2特异性模板序列送上海生工进行Sanger测序,测序结果显示均与预期结果相符(见图2)。
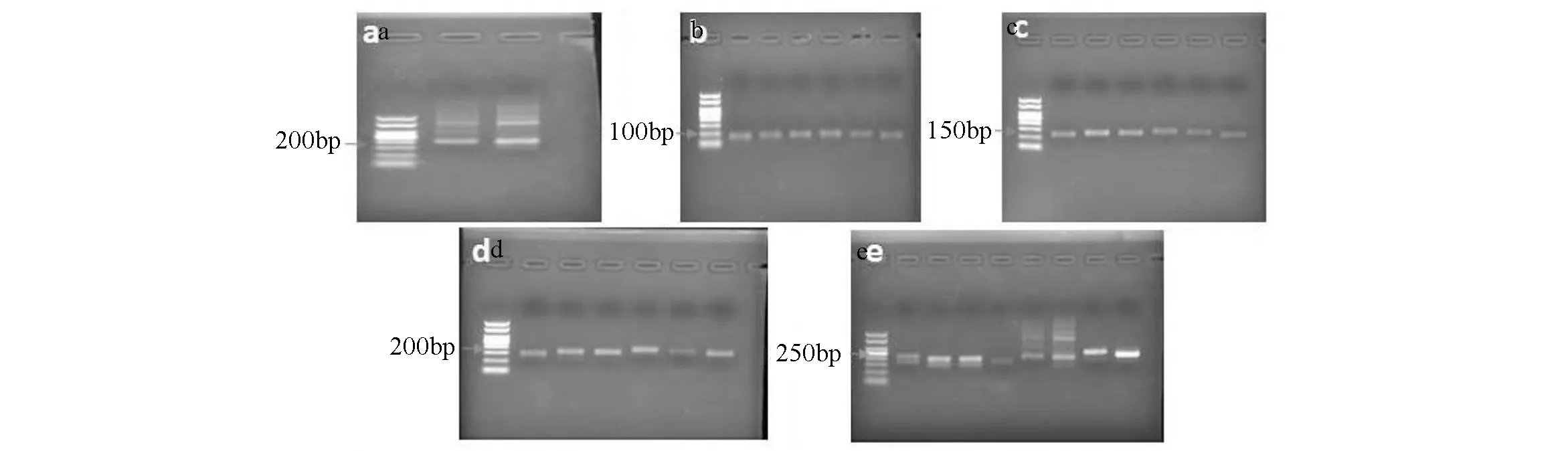
图1 8个SARS-CoV-2特异性模板片段重叠PCR合成电泳结果图a:PCR一步法合成模板5和6的结果电泳图;b:模板1,2,3,4,7,8分步法第一次连接电泳图,连接成功之后的片段约100bp左右;c:模板1,2,3,4,7,8分步法第二次连接图,连接成功之后的片段约为150bp左右;d:模板1,2,3,4,7,8分步法第三次连接图,连接成功之后的片段约为200bp左右;e:SARS-CoV-2特异性模板序列1-8最终产物电泳结果
图2 8个SARS-CoV-2特异性模板序列一代测序结果图a~h所示为8个SARS-CoV-2特异性模板序列中TaqMan探针的部分测序序列结果
2.3 SARS-CoV-2合成模板用于实时荧光PCR检测的可行性分析
重叠PCR合成的8个SARS-CoV-2模板序列,利用相应的TaqMan探针检测分析其用于荧光定量PCR的可能性。荧光定量PCR反应体系如下:2×Taq qPCR Master Mix 10 μL,SARS-CoV-2模板序列5 μL,探针工作液2 μL,补齐水至总体积20 μL。其中探针工作液包含正向、反向引物,和十分之一浓度的探针引物。PCR反应条件如下:95 ℃预变性2 min,95 ℃ 10 s,55 ℃ 20 s,72 ℃ 30 s,延伸步骤收集荧光信号,共40个循环。其PCR扩增结果见图3。
结果如图3所示,8个模板片段序列均能被相应的探针有效检测,在使用模板拷贝数为300拷贝时,其Ct值大都位于21~24之间。
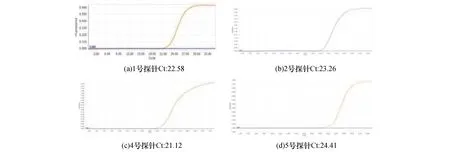
图3 SARS-CoV-2探针检测特异性模板的荧光定量PCR扩增曲线图a:1号探针的工作曲线;b:2探针的工作曲线;c:3号探针的工作曲线;d:4号探针的工作曲线
2.4 SARS-CoV-2合成模板拷贝数与探针量效关系
模板1和模板8进一步进行了不同拷贝数的量效关系检测。取起始拷贝数约为105cp/μL的SARS-CoV-2合成模板1 μL,依次按30倍、100倍、300倍、1 000倍、3 000倍、10 000倍等比稀释,分别加入TaqMan探针检测的荧光定量PCR反应体系中。qPCR扩增结果均表现为随着模板拷贝数增加,Ct值下降的良好关系(表2,图4)
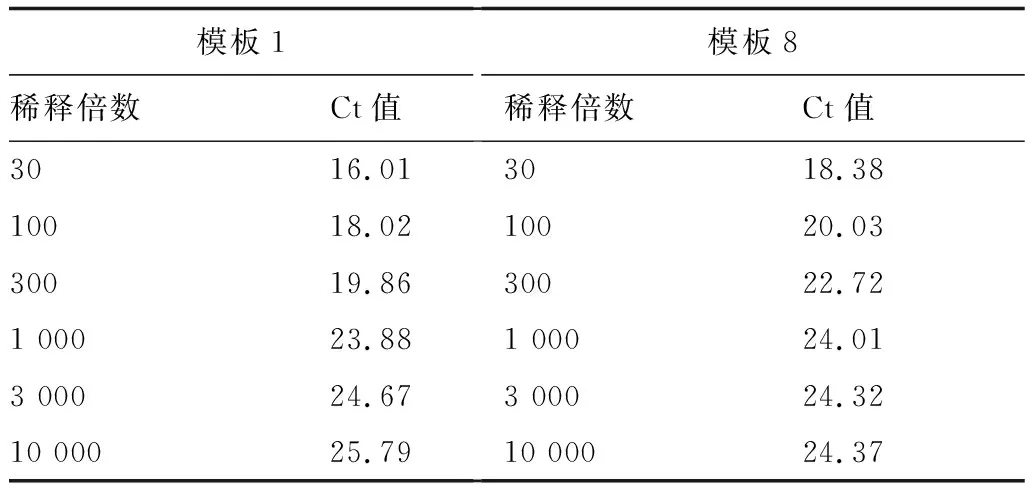
表2 SARS-CoV-2合成模板1和8拷贝数与探针qPCR扩增的Ct数值
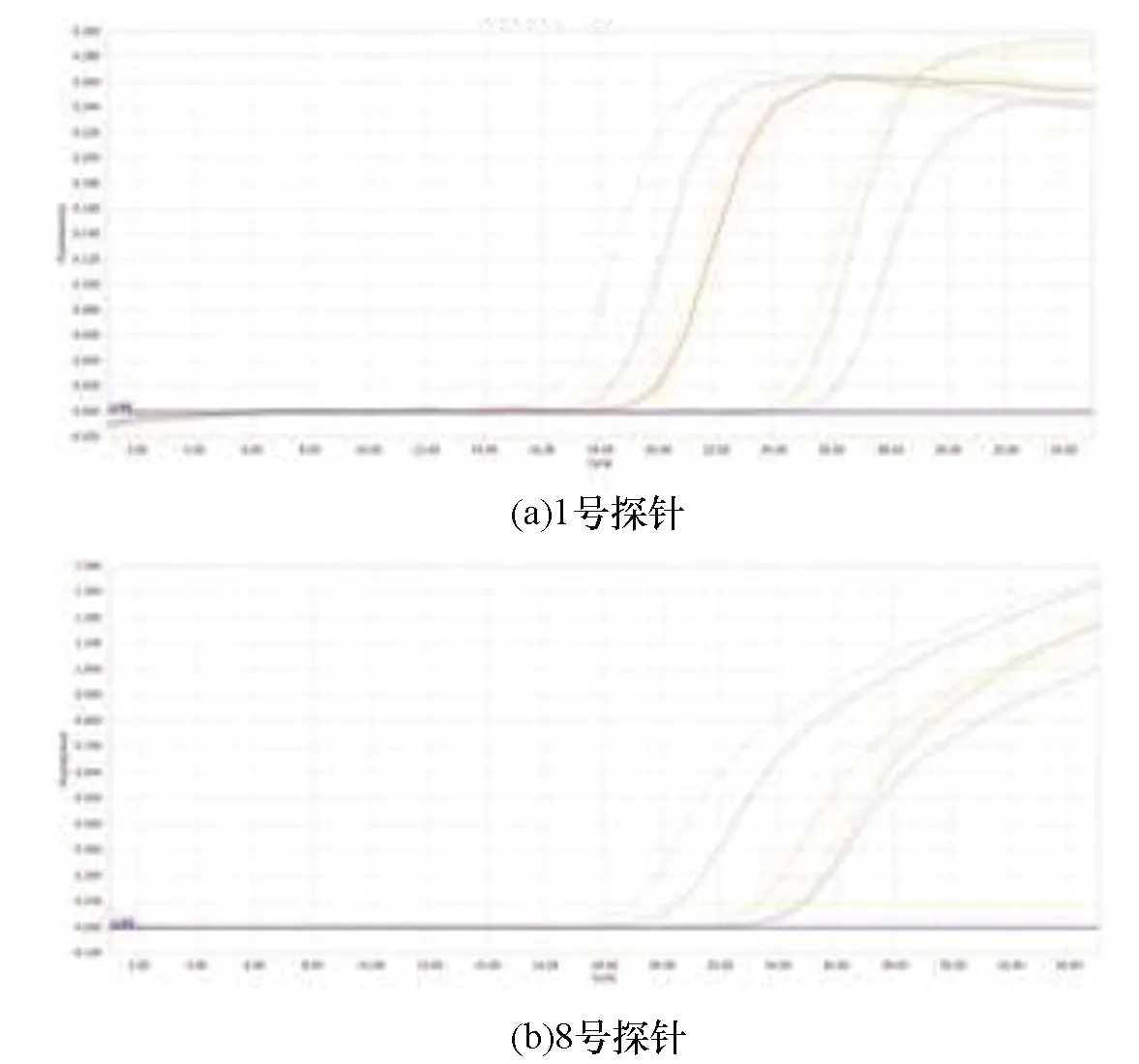
图4 SARS-CoV-2合成模板拷贝数与探针量效关系的荧光定量PCR扩增曲线图
3 讨 论
传染病的实验室诊断属于病因诊断的范畴。传统的病因诊断包括病原体的培养、形态学观察、抗原抗体检测等。基因检测为抗原检测,在特异性上较其它实验室检测手段具有一定优势。然而,基因检测优势的实现有赖于病原体靶点的选择,即靶点的特异性和靶点的数量。鉴于现有SARS-CoV-2基因检测试剂盒存在较高假阴性和低阳性率[3,8-10],本文针对该病毒的序列进行可检测靶点的研究,获得了8种可用于靶点的序列片段,并采用重叠PCR人工合成和通过设计TaqMan探针,验证了这8个序列片段作为实时PCR检测的可行性。
从表1可以看出,本文选择的8个靶点片段,在SARS-CoV-2基因组上分布相对较广,为其作为基因检测靶点时避免基因降解和基因局部二级结构导致假阴性提供了可能性。另外,相对广泛和互不连续的靶点序列,也为多靶点检测试剂盒的研发时呈现不同靶点单独形成扩增单元而不互相干扰提供了结构基础。本研究虽然只从一个靶点、一个探针的层面对相关靶点进行了其实用性的初步研究,但这些靶点在SARS-CoV-2基因组上相对分散的序列位置,使这些靶点可望成为多重实时PCR检测的良好候选对象。
本研究基于TaqMan探针确认和人工合成SARS-CoV-2 8个可用于基因检测的新靶点,并利用TaqMan探针验证了这些靶点的可行性。虽然这些靶点的片段长度相对较短,但其保守性相对较高,大部分均含有6个或以上连续20个碱基的序列片段,从而使这些片段同样可以作为LAMP原理的基因检测靶点。基于Cas酶的基因检测对靶点要求不高[7,11],可以预期这些靶点片段能完全用于开发基于Cas酶的基因检测。
综上所述,本文通过生物信息学比对,在现有SARS-CoV-2检测靶点之外,发现了多个具有可用于基于TaqMan原理的新靶点。在采用重叠PCR合成之后,利用TaqMan探针开展了一探针、一靶点的实时荧光PCR验证,确认了这些靶点作为SARS-CoV-2基因检测靶点的可行性。由于这些靶点同时还能作为基于Cas酶原理的检测和基于LAMP原理的检测,因而这些基因片段,为研发新的更高敏感性的SARS-CoV-2检测试剂盒提供了多种可供选择的靶点,尤其是对多重实时荧光PCR试剂盒的研发,价值尤为显著。

