山羊地方性鼻内肿瘤病毒ENTV/CH/GT/2015株全基因组cDNA文库的构建及生物信息学分析
林裕胜,江锦秀,张靖鹏,游 伟,胡奇林
(福建省农业科学院 畜牧兽医研究所,福建 福州 350013)
地方性鼻内肿瘤(dnzootic nasal tumor,ENT) 的病原是地方性鼻内肿瘤病毒(enzootic nasal tumor virus,ENTV),绵羊肺腺瘤(ovine pulmonary adenocarcinoma,OPA)的病原是绵羊肺腺瘤病毒(jaagsiekte sheep retrovirus,JSRV),两者都是以呼吸道鼻内黏膜分泌型上皮细胞发生肿瘤转化为特征的慢性、接触传染性疾病[1-2]。OPA源自呼吸道末梢的分泌性上皮细胞(Ⅱ型巨噬细胞和克拉拉细胞),而ENT源自羊鼻腔中鼻甲骨筛骨的分泌性上皮细胞。这两种病是制约养羊业健康发展的重要疫病,均给养羊业造成了较大的经济损失[2]。
ENTV基因组由线性单股正链RNA组成,大小约为7.5 kb,属于乙型反转录病毒科正反转录病毒亚科的β反转录病毒属,通过全基因序列分析发现,ENTV同JSRV及山羊和绵羊自身细胞基因组中的内源性反转录病毒(endogenous retroviruses,ERVs)高度同源[3]。ENTV基因组两端为非编码区LTR序列,中间编码区为典型的乙型反转录病毒结构即gag、pro、pol、env基因相互重叠。pro基因编码蛋白酶,主要负责病毒蛋白水解处理;pol基因编码逆转录酶和整合酶,是逆转录和整合基因组所必须的复制酶;其gag基因编码芯髓衣壳蛋白,直接与基因组RNA相互作用;env基因编码囊膜蛋白,包含表面蛋白(surface protein,SU)和跨膜蛋白(transmembrane protein,TM)[4-7]。除了澳大利亚和新西兰外,全世界养羊国家均有ENA发生的报道,在一些地区的流行率高达10%[8]。
我国对于该病的相关报道较少,由于缺乏有效体外培养适应细胞,且ENTV-2全基因序列仅有17株,其中14株为四川和陕西收集的病料中克隆所得,使得病毒生物学特性的研究滞后[8-9]。为了更好的了解ENTV-2的分子进化过程,本研究从福建省古田县南江黄羊养殖场采集疑似山羊ENT的鼻内肿瘤组织, 通过对PCR扩增产物克隆测序获得ENTV-2前病毒全基因组序列,并与其他逆转录病毒进行比较和分析,明确其生物信息,不仅丰富了ENTV-2全基因序列库,还为今后研究ENTV-2快速检测方法及致病机理提供研究素材。
1 材料与方法
1.1 病料来源采自福建古田患病南江黄羊养殖场的鼻内肿瘤组织样品,且经组织病理学检查确诊为腺瘤。
1.2 主要试剂DL5000 DNA Marker、Ex Taq Mix、pMD19-T 购自宝生物工程(大连)有限公司;反转录试剂盒购自Progerma公司;病毒DNA/RNA基因组提取试剂盒购自北京全式金生物技术股份有限公司;胶回收试剂盒购自OMEGA公司。
1.3 引物设计从NCBI中下载已公布的16株ENTV-2全基因序列,经生物学软件DNAStar比对分析后,应用Oligo 7.0 软件设计7对引物(表1),用于扩增ENTV全基因组序列。引物由福州白鲸生物科技有限公司合成。
1.4 ENTV/CH/GT/2015全基因组的分段扩增及克隆按照Trans EasyPure Viral DNA/RNA Kit试剂盒说明书提取ENTV总RNA,随后按Promega GoscriptTM Reverse Transcription Mix试剂盒说明书进行反转录,以转录后的cDNA为模板,通过PCR方法分段扩增ENTV全基因组序列。扩增体系为:Ex Taq Mix 10 μL,上、下游引物(10 μmol/L)各1 μL,cDNA 2 μL,水补足至20 μL,反应程序为:95℃ 5 min;95℃ 30 s,52~55℃ 30 s,72℃ 30 s,35个循环;72℃ 7 min。目的片段经胶回收后分别连接至pMD19-T载体上,并转化至基因工程菌DH5a,验证正确的阳性克隆进行扩增培养,随后将提取的阳性质粒送至福州白沙生物科技有限公司测序。

表1 ENTV-2引物序列信息
1.5 ENTV/CH/GT/2015株全基因序列分析将克隆测序结果利用生物信息学软件DNAStar中的seqMan进行拼接,获得ENTV/CH/GT/2015株的全基因组序列。利用DNAStar软件中的EditSeq程序对ENTV/CH/GT/2015株的全序列进行基因注释和分析,利用NCBI基因在线功能直接将注释好的全基因序列上传到GenBank库中,利用Protean程序对gag、pro、pol和env蛋白二级结构、亲水性和蛋白抗原表位进行预测。利用N-糖基化位点在线预测软件(http://www.cbs.dtu.dk/services/NetNGlyc)对编码区gag、pro、pol和env蛋白进行糖基化位点预测。
2 结果
2.1 ENTV/CH/GT/2015全基因分段扩增从病料组织中提取RNA,转录后获得cDNA作模板,利用设计的7对引物分别进行PCR扩增,分别得到相应大小的基因片段,均与预期结果相符(图1)。
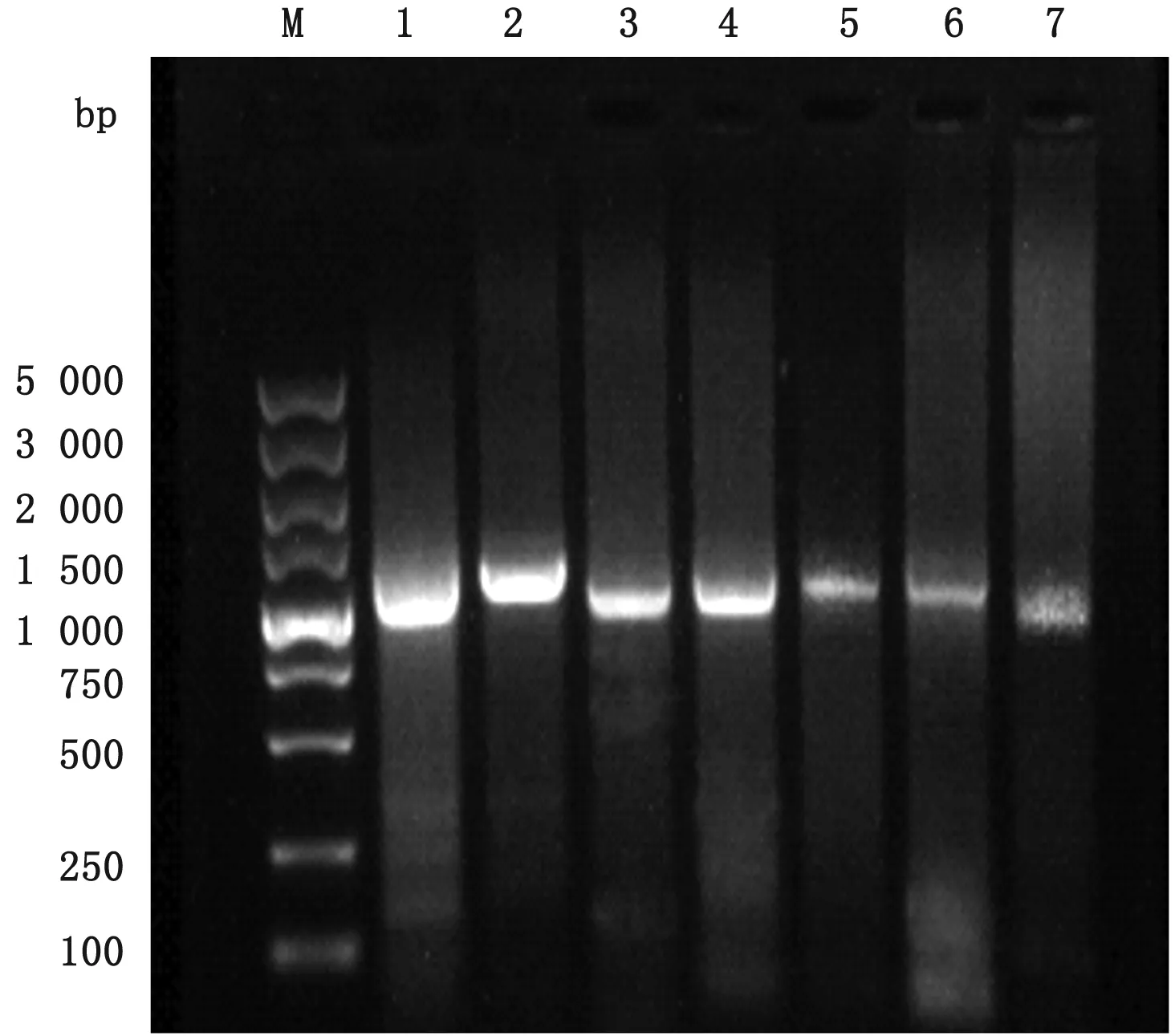
图1 ENTV/CH/GT/2015全基因组扩增结果 M.DL5000 DNA Marker;1~7.7对引物扩增结果
2.2 ENTV/CH/GT/2015全基因拼接及序列分析利用生物软件将ENTV/CH/GT/2015克隆测序后获得的7个基因片段进行拼接,拼接后的序列全长为7 506 bp,包含相互重叠的4个较长的ORF,分别为gag、pro、pol和env基因。通过NCBI基因在线功能将该序列上传至GenBank,获得的申请登录号为MK210250。通过与乙型反转录病毒属成员小鼠乳腺瘤病毒(mouse mammary tumor virus,MMTV)、马非猴病毒(mason-pfizer monkey virus,MPMV)、松鼠猴反转录病毒(squirrel monkey retrovirus,SMRV)和JRSV进行对比,确定ENTV/CH/GT/2015株的gag基因位于基因组中的265~2 124位碱基,全长1 860 bp,编码620个氨基酸,预测的相对分子质量为68 370,在516~529 aa位和543~556 aa为序列均为典型的CCHC型锌指结构。pro基因位于基因组中1 959~2 885位碱基,编码308个氨基酸,蛋白相对分子质量预测为33 790。pol基因位于基因组中的3 099~5 444位碱基,编码874个氨基酸。env基因位于基因组中的5 373~7 208位碱基,全长1 857 bp,编码1个多聚蛋白前体,由619个氨基酸残基组成。
2.3 ENTV/CH/GT/2015株全基因组序列比较分析应用生物学软件对GenBank中已公布的16株ENTV-2参考株基因组全序列、11条OPA(ENTV-1)和ENTV/CH/GT/2015株全基因序列进行比对分析。结果显示,本研究获得的ENTV/CH/GT/2015株基因组全序列与国外NC004994.2株和AY197548.2株相似性均为87.5%,与国内ENTV-2CHN8株相似性为97.7%,与已公布的11株ENTV-1相似性为88.1%~88.2%。
2.4 ENTV/CH/GT/2015株gag和env蛋白序列相似性分析将ENTV/CH/GT/2015株gag和env核苷酸序列推导氨基酸序列与其他参考株进行比较。ENTV/CH/GT/2015株gag蛋白序列与国内ENTV-2的相似性为88.6%~96.1%,与国外AY197548.2株和NC004994.2株的相似性均为88.9%,与ENTV-1的相似性为89.4%~89.7%,与JSRV的相似为89.1%~89.4%;ENTV/CH/GT/2015株 gag核苷酸推导的氨基酸序列与参考株相比,ENTV/CH/GT/2015株在124~126 aa插入GVE 3个氨基酸和229~232 aa插入APPP 4个氨基酸(图2)。env蛋白序列与国内 ENTV-2相似性为93.1%~97.7%,与国外AY197548.2株和NC004994.2株的相似性均为91.4%,与ENTV-1的相似性为86.6%~86.7%,与JSRV的相似性为89.7%~88.9%;ENTV/CH/GT/2015株env核苷酸推导的氨基酸序列与参考株相比,在590~593 aa处存在AESL 4个氨基酸缺失(图3)。
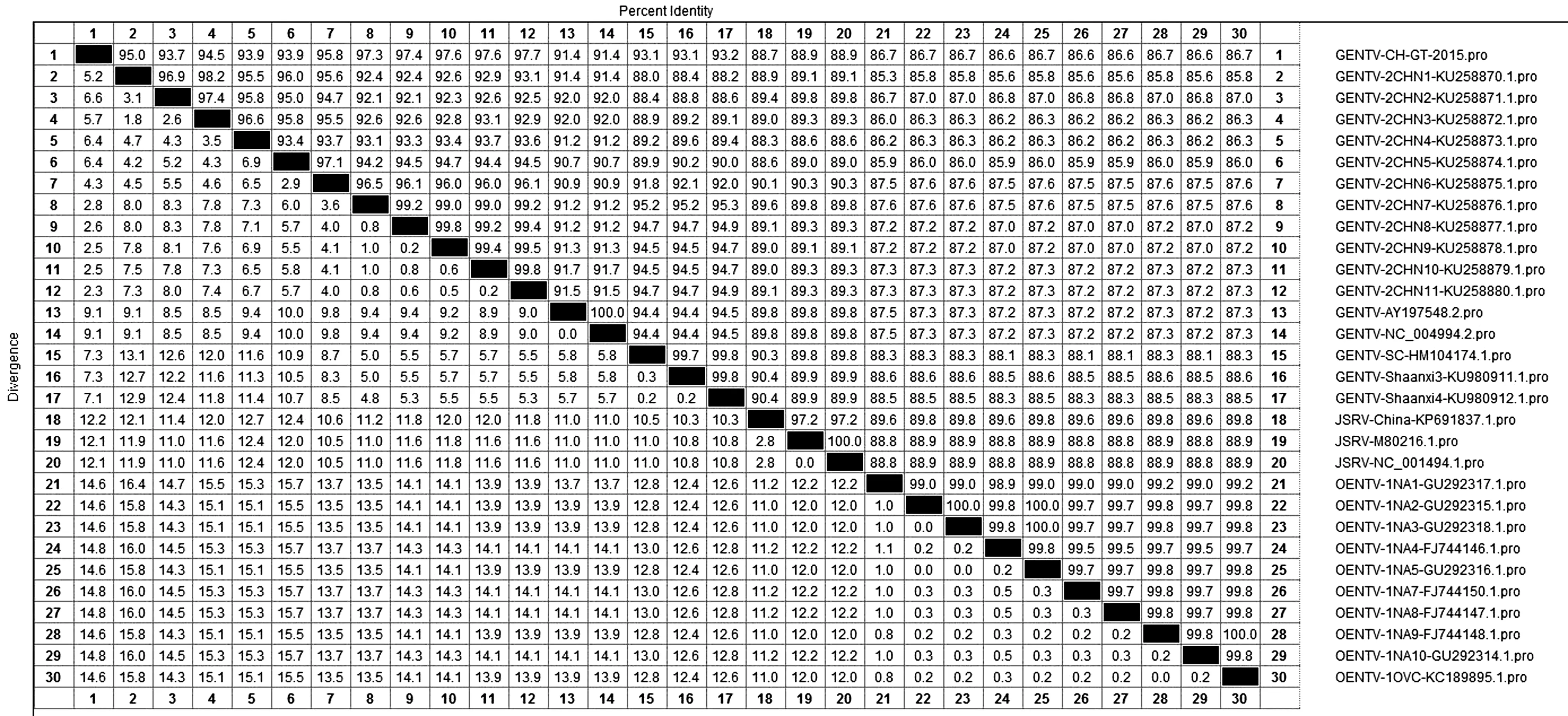
图2 3种乙型逆转录病毒gag基因核苷酸同源性比较

图3 3种乙型逆转录病毒env基因核苷酸同源性比较
2.5 ENTV/CH/GT/2015株gag、pro、pol和env蛋白结构分析利用Protean软件对ENTV/CH/GT/2015株gag、pro、pol和env蛋白结构进行分析,Garnier 等gag蛋白二级结构中α螺旋在整个肽段中分布较多也较均匀;β折叠在肽段N端和中部有少量分布;β转角均匀分布在在肽段中;无规则卷曲均匀分布在肽链上;亲水性分析结果显示,gag蛋白的亲水性较高,区域分布较为广泛(图4)。通过对ENTV/CH/GT/2015株和参考株gag蛋白抗原表位进行预测(图略),虽然ENTV/CH/GT/2015株 gag蛋白存在氨基酸的突变和插入,与其他参考株相比其抗原表位并未发现明显的差异。Garnier 等方法预测pro蛋白二级结构中α螺旋仅有1处存在肽段中;β折叠在肽段N端和中部有少量分布;β转角均匀分布在在肽段中;无规则卷曲均匀分布在肽链上;亲水性分析结果显示,pro蛋白的亲水性较低,主要在肽段的C端和中后部(图5);通过对参考株和ENTV/CH/GT/2015株 pro蛋白抗原表位进行预测(图略),结果显示虽然pro蛋白存在氨基酸的突变,与其他参考株相比其抗原表位并未发现明显的差异。Garnier 等方法预测pol蛋白二级结构中α螺旋分布在肽段C段和中后部;β折叠均匀分布于整段肽链中;β转角均匀分布在在肽段中;无规则卷曲均匀分布在肽链上;亲水性分析结果显示,pol蛋白的亲水性较高,区域分布较为均匀(图6);通过对参考株和ENTV/CH/GT/2015株 pol蛋白抗原表位进行预测(图略),虽然pol蛋白存在氨基酸突变,与其他参考株相比其抗原表位并未发现明显差异。Garnier 等方法预测env蛋白二级结构中α螺旋在整个肽段中分布较多也较均匀;β折叠在肽段N端和中部有少量分布;β转角均匀分布在在肽段中;无规则卷曲均匀分布在肽链上;亲水性分析结果显示,env蛋白的亲水性一般,主要分布在肽段两端(图7);通过对ENTV/CH/GT/2015株和参考株env蛋白抗原表位进行预测(图略),结果显示尽管ENTV/CH/GT/2015株env蛋白存在氨基酸的突变和缺失,与其他参考株相比其抗原表位并未发现明显的差异。
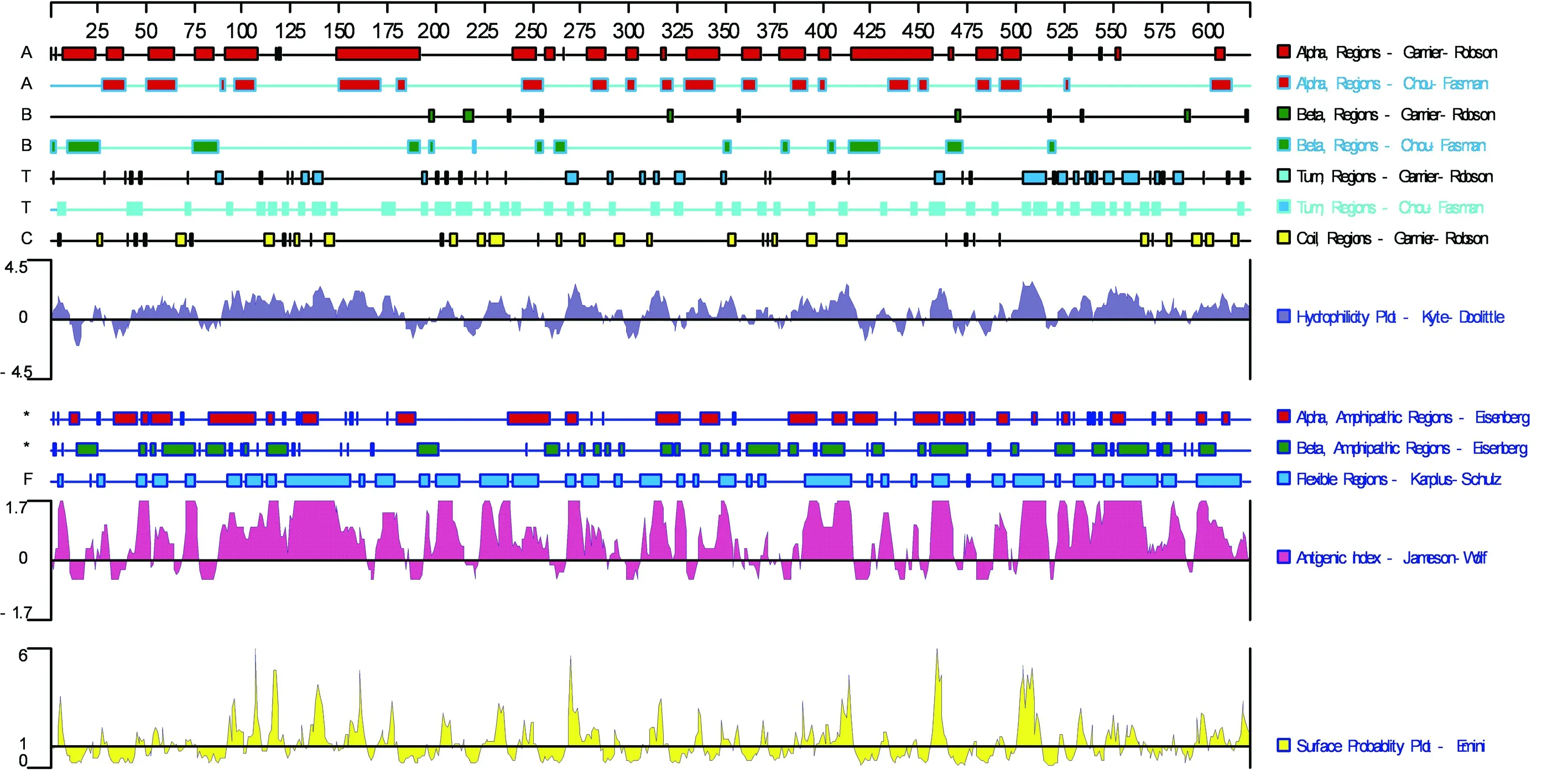
图4 gag蛋白二级结构、亲水性和抗原表位
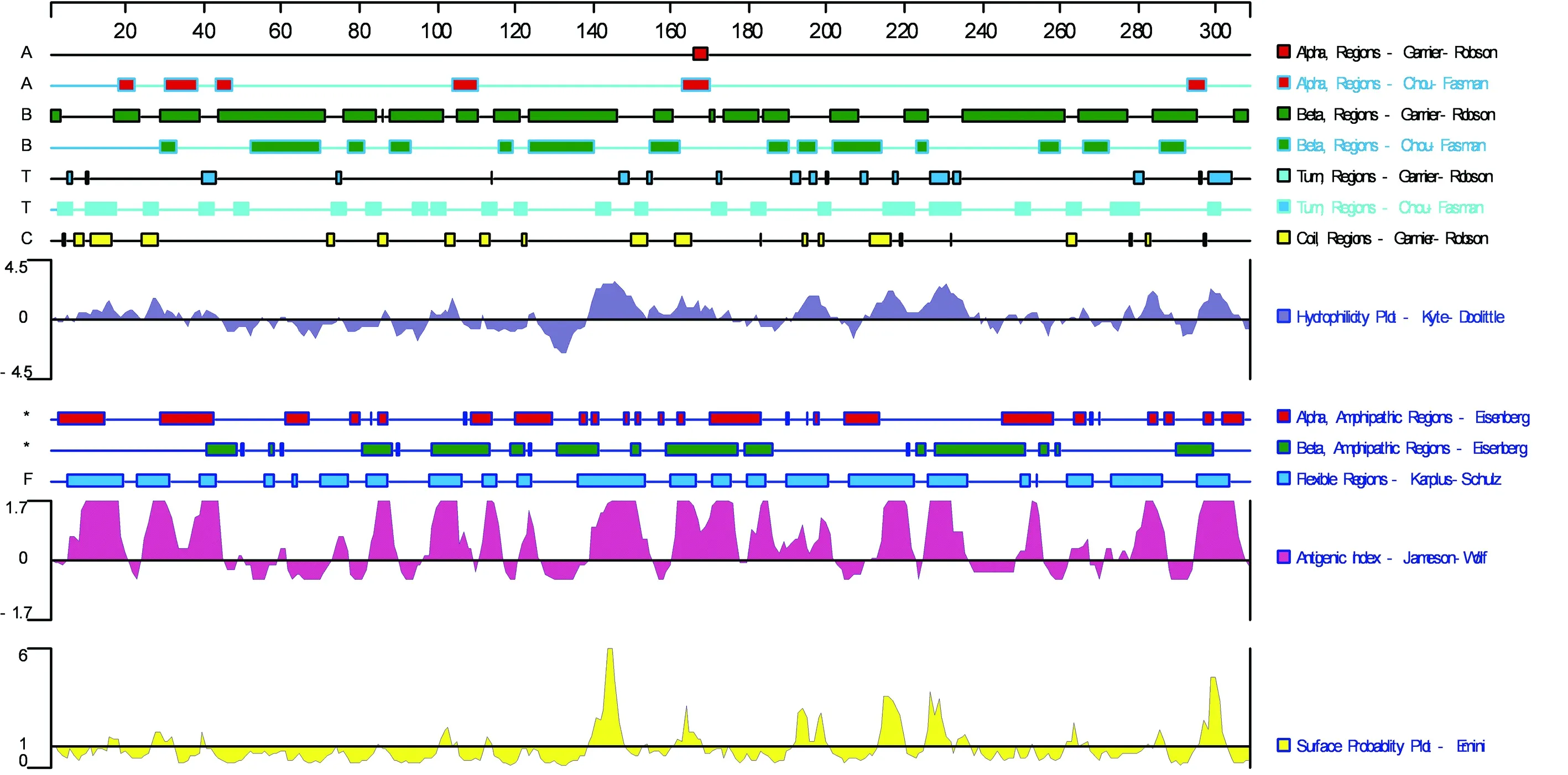
图5 pro蛋白二级结构、亲水性和抗原表位
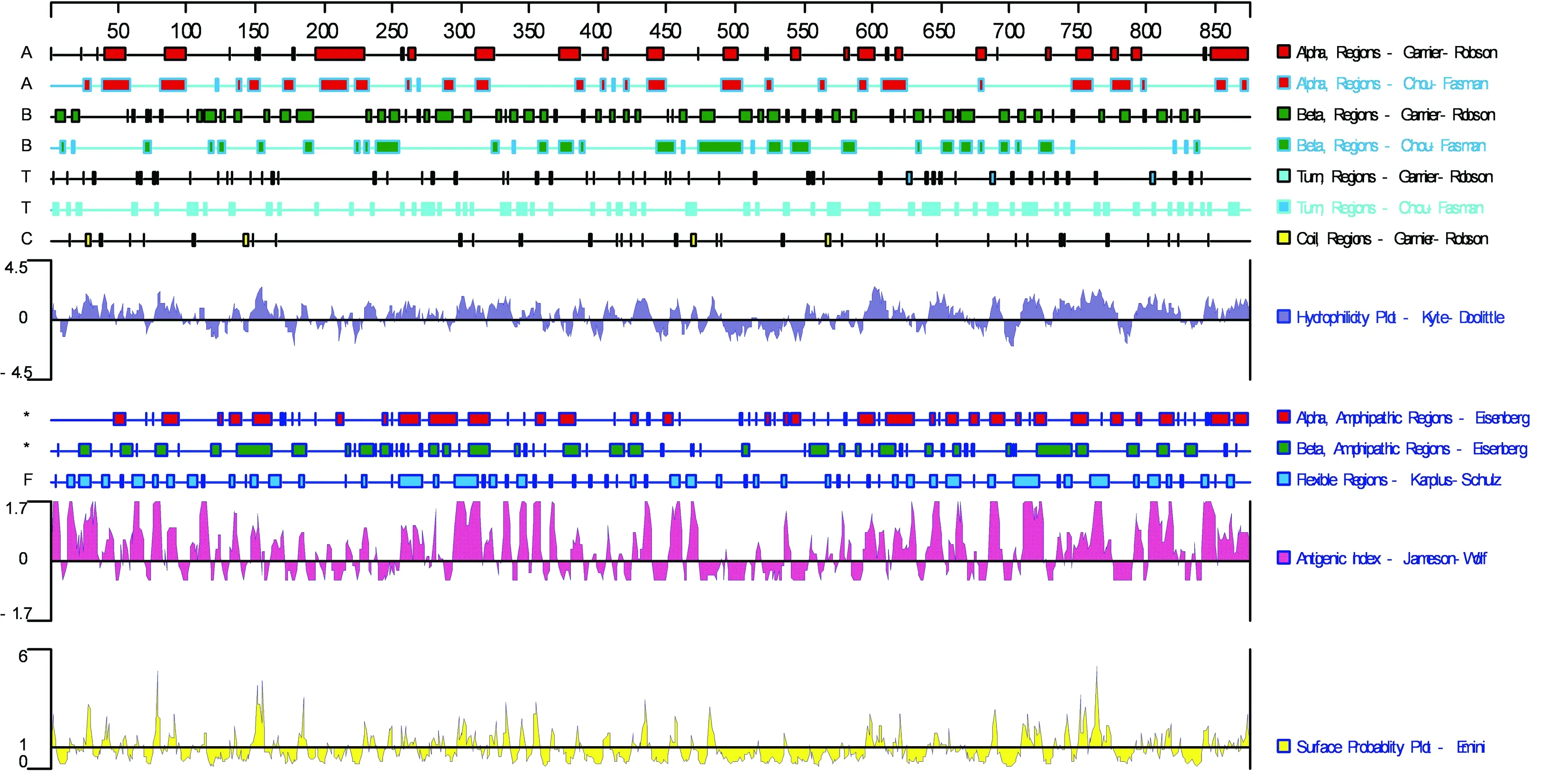
图6 pol蛋白二级结构、亲水性和抗原表位
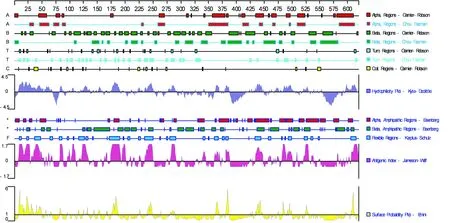
图7 env蛋白二级结构、亲水性和抗原表位
2.6 ENTV/CH/GT/2015 gag、pro、pol和env蛋白糖基化位点预测通过N-糖基化位点在线预测软件对ENTV/CH/GT/2015株的gag、pro、pol和env蛋白进行在线分析,确定潜在的糖基化位点的数目、位置和类型,软件预测结果显示,ENTV/CH/GT/2015株的gag蛋白可能潜在的糖基化位点数1个,在186(NLTV);pro蛋白可能潜在的糖基化位点数1个,在159(NVTE);pol蛋白可能潜在的糖基化位点数4个,分别为91(NETM)、416(NWSI)、470(NGTA)和576(NHTA);env蛋白可能潜在的糖基化位点数为6个,分别为115(NDTS)、134(NISF)、282(NSSG)、427(NVTK)486(NASD)和507(NVSL)。预测结果还提示4个序列可能不包含信号肽。
2.7 ENTV/CH/GT/2015株系统进化分析通过MegAlign软件中Clustal W方法比对构建遗传进化树。结果显示ENTV/CH/GT/2015株与国内ENTV-2CHN2株和ENTV-2CHN10株亲缘关系最近,与陕西株、四川株及2株国外AY197548.2株和NC004994.2株之间亲缘关系相对较远,由此推测ENTV/CH/GT/2015株是国内ENTV-2CHN2株和ENTV-2CHN10株变异毒株。此外,遗传进化系统上ENTV-1和ENTV-2分别处在进化树中不同的分支上,暗示两者差异性较大(图8,9)。
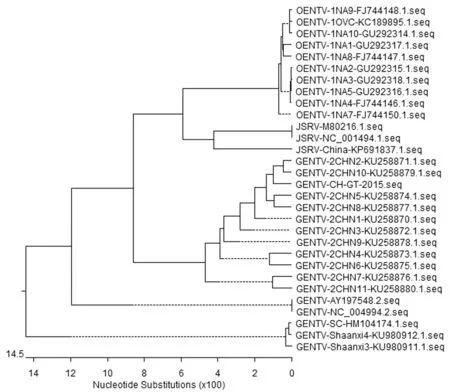
图8 基于gag基因构建的遗传进化树
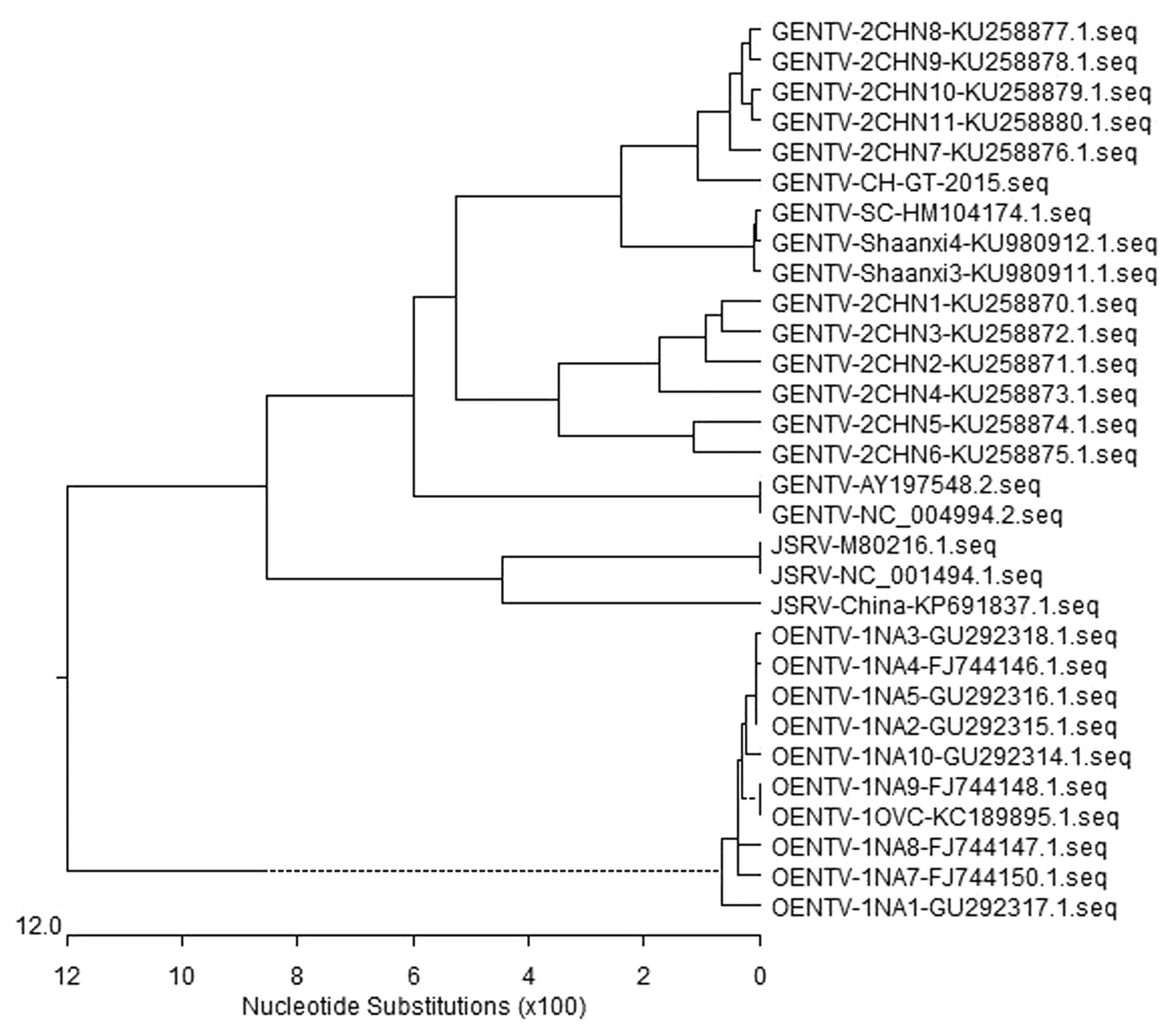
图9 基于env基因构建的遗传进化树
3 讨论
ENT是近年来出现在我国的动物源性鼻内肿瘤。临床上,感染山羊表现为出现大量的浆液性鼻腔分泌物,然后发生鼻塞,逐渐转变为呼吸困难、眼突出和颅骨变性,最终死于窒息。通过对福建省ENT的流行病学、临床症状和病理组织学特征进行研究发现,与国外和中国其他省份报道的ENT都极为相似[10-12]。关于山羊ENTV-2的研究报道较少,目前国内外上传到GenBank上的全基因组序列仅有17株,且有10株为同一人上传[2,8]。由于缺乏合适的体外培养系统,导致ENTV-2的研究难以深入,对于该病毒是否可以产生特异性的免疫应答至今争议不断[13-14]。
本研究从患病山羊鼻内组织中通过克隆测序获得1株山羊ENTV/CH/GT/2015全基因组序列,进一步丰富ENTV-2全基因组序列库,为国内外学者后续深入研究该病的分子流行病学特征及相关的致病机制奠定基础。然而本研究也存在一定的缺陷,无法获得纯化的病毒,导致获得的基因组序列2段并不完整。本研究获得的ENTV/CH/GT/2015株基因组全序列与国外NC004994.2和AY197548.2株相似性均为87.5%,与国内ENTV-2CHN8株相似性为97.7%,与已公布的11株ENTV-1相似性为88.1%~88.2%;与何亚鹏等[11]和冯迎春等[12]公布的ENTV-2全基因组序列相似较低,与WANG等[15]公布的ENTV-2全基因组序列ENTV-2CHN2株和ENTV-2CHN10株相似性较高,此外国外2株ENTV-2毒株与本研究中ENTV/CH/GT/2015株及国内发表毒株在基因序列差异较大,亲缘关系相对较远。由此推测ENTV/CH/GT/2015株是ENTV-2CHN2株和ENTV-2CHN10株的变异毒株。何亚鹏等[11]认为是由于地理环境的不同和该病毒对宿主品系的选择所造成的,本研究也证实了这一点。冯迎春等[12]报道ENTV-2与ENTV-1是2种不同的病毒,进化系统上ENTV-1和ENTV-2分别处在进化树中不同的分支上,暗示两者差异性较大;在基因水平上通过遗传进化树图谱结果也证实ENTV-2和ENTV-1确实是2种病毒。
ENTV/CH/GT/2015株gag蛋白序列与国内ENTV-2株的相似性为88.6%~96.1%,与国外AY197548.2株和NC004994.2株的相似性均为88.9%,与ENTV-1的相似性为89.4%~89.7%,与JSRV的相似性为89.1%~89.4%;ENTV/CH/GT/2015株gag核苷酸推导的氨基酸序列与参考株相比,ENTV/CH/GT/2015株在124~126 aa插入GVE 3个氨基酸和229~232aa插入APPP 4个氨基酸。这与ORTN等[1]报道gag基因在350~390位碱基处属于高变区基本一致,但在687~696位碱基处ENTV/CH/GT/2015株还插入了12个碱基。env蛋白序列与国内 ENTV-2相似性在93.1%~97.7%之间,与国外AY197548.2株和NC004994.2株的相似性均为91.4%,与ENTV-1的相似性为86.6%~86.7%,与JSRV的相似性为89.7%~88.9%;ENTV/CH/GT/2015株env核苷酸推导的氨基酸序列与参考株相比 在590~593 aa处存在AESL 4个氨基酸的缺失。
对ENTV/CH/GT/2015株gag、pro、pol和env蛋白结构进行分析发现gag、pro、pol和env蛋白二级结构中含有大量的α螺旋和β折叠。gag蛋白的亲水性较高,区域分布较为广泛; pro蛋白的亲水性较低,主要在肽段的C端和中后部; pol蛋白的亲水性较高,区域分布较为均匀;env蛋白的亲水性一般,主要分布在肽段两端;gag、pro、pol和env蛋白抗原预测结果显示,与其他参考株相比gag、pro、pol和env抗原表位均未发现明显的变化
通过N-糖基化位点在线预测软件对 ENTV/CH/GT/2015株的gag、pro、pol和env蛋白进行在线分析,确定潜在的糖基化位点的数目、位置和类型,软件预测结果显示,ENTV/CH/GT/2015株的gag蛋白可能潜在的糖基化位点数1个;pro蛋白可能潜在的糖基化位点数1个;pol蛋白可能潜在的糖基化位点数4个;env蛋白可能潜在的糖基化位点数为6个。糖基化位是否与蛋白的抗原性有关还需进一步的研究,预测结果还提示4个序列可能都不包含信号肽。
综上所述,本研究通过克隆获得山羊ENTV/CH/GT/2015株的全基因组序列,并对ENTV/CH/GT/2015株的全基因序列以及4个主要编码蛋白进行结构分析、糖基化位点预测和遗传进化分析,明确ENTV/CH/GT/2015株基因组生物信息学,为今后开展ENTV-2的相关研究奠定基础。

