太平洋真宽水蚤CYP4C和CYP44基因克隆及分子特征分析
(国家海洋设施养殖工程技术研究中心,浙江舟山 316022)
浮游动物是海洋生态环境中鱼类与其他物种的主要饵料,通过食物链联系初级生产者与更高层次的消费者[1],在海洋生态系统物质循环和能量流动过程中发挥重要作用[2]。浮游动物种类繁多,包括尾索动物、栉水母、甲壳动物和轮虫等。其中,桡足类是浮游动物群落中分布最广、种类最多的类群。桡足类的生命周期短、对环境变化敏感,可作为研究海洋环境变化和人类活动的指示物种[3]。太平洋真宽水蚤Eurytemora pacifica 是一种暖水性沿岸桡足类,是我国东海沿岸的季节性优势种[4],由于食物链中的特殊地位使其在海洋环境检测过程中有重要作用,因此对太平洋真宽水蚤功能基因的研究将有助于了解其生态学重要性及海洋环境变化。
细胞色素酶P450 (Cytochrome P450,CYP450)是以血红素作为辅因子的一类超家族,是重要的I 相代谢酶[5]。CYP450 酶系广泛存在生物体不同组织中,发挥不同的生物学作用,例如,参与脂质等内源性化合物的代谢过程[6-8];参与环境污染物和致癌物质的解毒反应[9-10]。CYP450 超家族基因已在动物、植物、真菌和细菌中被鉴定并广泛研究(http://drnelson.uthsc.edu/CytochromeP450.html)。另外,CYP450 超家族被认为是海洋污染的生物标志物[11]。多名学者研究指出在海洋脊椎动物多环芳烃代谢过程中CYP1 基因发挥重要作用[12-14];KASAI,et al[15]在海洋无脊椎动物中发现CYP4 家族参与其生物代谢和外部污染物的解毒反应;中国学者在多种海洋无脊椎动物中发现CYP4 基因参与污染物的解毒反应[12,16],如双齿围沙蚕Perinereis aibuhitensis、翡翠贻贝Perna viridis 等。但对桡足类中CYP450 家族基因研究较少,因此研究太平洋真宽水蚤CYP450 家族基因将有助于了解桡足类CYP450 家族分子特征,揭示其对环境污染物的解毒代谢机制。
本研究通过RACE 技术首次克隆太平洋真宽水蚤EpCYP4C 和EpCYP44 基因全长序列,并通过生物信息学技术进行序列分析。EpCYP4C 和EpCYP44 基因序列分析结果有助于桡足类CYP450 酶系的研究,为后续研究太平洋真宽水蚤对环境污染物的代谢机制和解毒机理奠定基础,同时为发现太平洋真宽水蚤的生物学功能提供理论基础。
1 材料与方法
1.1 实验动物
实验所用太平洋真宽水蚤(长约1.2~1.4 mm)来自中国浙江省舟山市长峙岛。太平洋真宽水蚤在盐度28、温度(20±0.5) ℃且充氧的水槽中暂养7 d,每天定时投喂小球藻。实验挑选生命力强且健康的太平洋真宽水蚤成体作为研究对象。
1.2 实验方法
1.2.1 总RNA 提取和反转录
本研究选取大约80 只太平洋真宽水蚤成体,用DEPC 水冲洗去除海水残存的杂质,根据Total RNA Kit II (OMEGA,USA)说明书[17]提取总RNA,并用DNase I (OMEGA,USA)处理。1%琼脂糖凝胶电泳检测RNA 完整性,并通过NanoDrop 2000 核酸蛋白检测仪测定其浓度。然后按照SuperScriptTMIII 反转录酶试剂盒(Invitrogen,USA)说明书,将5 μg 总RNA 反转录合成cDNA 第一链,并储存于-20 ℃。
1.2.2 太平洋真宽水蚤CYP4C 和CYP44 基因中间片段的扩增
从实验室太平洋真宽水蚤转录组中[17]获得CYP4C 和CYP44 基因部分序列,利用primer premier 5.0软件设计CYP4C 和CYP44 基因特异性引物(表1),以cDNA 第一链作为模板进行聚合酶链式(PCR)反应。20 μl PCR 反应体系:cDNA 0.4 μl,EasyTaqDNA Polymerase(全式金,中国)0.4 μl,EasyTaqbuffer 2 μL,上下游引物0.4 μL,10 mM dNTPs 0.4 μL,加DEPC 水至20 μL。PCR 反应程序为预变性(95 ℃,5 min);变性(95 ℃,30 s)、退火(CYP4C:52 ℃,30 s;CYP44:56 ℃,30 s)、延伸(72 ℃,40 s),共32 个循环;终延伸(72 ℃,7 min)。1%琼脂糖凝胶电泳检测PCR 产物,使用普通琼脂糖凝胶DNA 回收试剂盒(天根,中国)对其进行胶回收,并连接至T 载体,4 ℃过夜。取10 μL 连接产物克隆至DH5α 感受态细胞中(Vazyme,USA),通过蓝白斑筛选获得阳性克隆子,送至上海华大基因公司测序。
1.2.3 太平洋真宽水蚤CYP4C 和CYP44 基因5’-RACE 和3’-RACE 扩增

表1 RACE 引物信息Tab.1 RACE primer sequences information
1.2.4 太平洋真宽水蚤CYP4C 和CYP44 基因生物信息学分析
通过DNAMAN 软件拼接太平洋真宽水蚤CYP4C 和CYP44 基因中间序列、5’末端序列和3’末端序列,获得全长cDNA 序列,命名为EpCYP4C 和EpCYP44。利用MEGA7.0 软件[18]将EpCYP4C 和EpCYP44基因编码序列翻译成氨基酸序列,登录NCBI 网站用BLAST 分析EpCYP4C 和EpCYP44 氨基酸序列同源性。利用ExPAsy (https://web.expasy.org/protparam/)在线软件分析EpCYP4C 和EpCYP44 蛋白质理化性质;通过PROSITE 分析EpCYP4C 和EpCYP44 蛋白质功能位点。另外,利用SMART 在线软件(http://smart.embl-heidelberg.de/)预测结构功能域。通过DNAMAN 软件进行多序列同源性比较。根据相邻连接(Neighbor-joining,NJ)法[19]构建系统进化树。利用SOPMA 在线程序(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html) 预测蛋白质二级结构;通过SWISS-MODEL 软件(https://www.swissmodel.expasy.org/)模拟构建蛋白质三维结构。通过PSORT 程序(https://psort.hgc.jp/form.html)预测蛋白亚细胞定位。
1.3 序列来源
根据EpCYP4C 蛋白BLAST 结果下载其他无脊椎动物基因序列。分别是甲壳纲:近亲真宽水蚤(Eurytemora affinis,XM_023478114.1),加利福尼亚虎斑猛水蚤(Tigriopus kingsejongensis,KY249923.1),日本虎斑猛水蚤(Tigriopus japonicus,KF640012.1),凡纳滨对虾(Litopenaeus vannamei,GU969106.2),中国明对虾(Fenneropenaeus chinensis,GU218693.1),日本对虾(Marsupenaeus japonicus,KP234017.1);软甲纲:三疣梭子蟹(Portunus trituberculatus,JN835470.1),中华绒螯蟹(Eriocheir sinensis,KX812713.1);昆虫纲:寄生蜂(Diachasma alloeum,XM_015264016.1),阿里山潜蝇茧蜂(Fopius arisanus,XM_011302246.1),寄生木黄蜂(Orussus abietinus,XM_023432286.1),双果蝇(Drosophila bipectinata,XM_017245718.1),果蝇(Drosophila willistoni,XM_002073483.2),暗果蝇(Drosophila obscura,XM_022357779.1),黑腹果蝇(Drosophila melanogaster,NM_079859.4),波斯果蝇(Drosophila persimilis,XM_002020380.2),米兰达果蝇(Drosophila miranda,XM_017284500.1),双翅果蝇(Drosophila biarmipes,XM_017095903.1),麦茎蜂(Cephus cinctus,XM_015734568.2),铜绿蝇(Lucilia cuprina,XM_023441491.1),瓜实蝇(Bactrocera cucurbitae,XM_011186956.1),家蝇(Musca domestica,XM_005185973.3),桔小实蝇(Bactrocera dorsalis,XM_011216085.2),白蚁(Cryptotermes secundus,XM_023858360.1),烟粉虱(Bemisia tabaci,XM_019045778.1)。
根据EpCYP44 蛋白BLAST 比对下载其他无脊椎动物基因序列。分别是甲壳纲:近亲真宽水蚤(Eurytemora affinis,XM_023474320.1),加利福尼亚虎斑猛水蚤(Tigriopus kingsejongensis,KY249925.1),日本虎斑猛水蚤(Tigriopus japonicus,KF640014.1),矮小拟镖水蚤(Paracyclopina nana,KP899600.1);昆虫纲:白蚁(Cryptotermes secundus,XM_023847705.1),湿木白蚁(Zootermopsis nevadensis,XM_022074691.1),烟粉虱(Bemisia tabaci,XM_019047605.1),褐飞虱(Nilaparvata lugens,XM_022333163.1),西花蓟马(Frankliniella occidentalis,XM_026418937.1),温带臭虫(Cimex lectularius,XM_014404579.2);内口纲:锑对土壤跳虫(Folsomia candida,XM_022106671.1);曳鳃纲:尾曳鳃虫(Priapulus caudatus,XM_014807126.1);双壳纲:美洲牡蛎(Crassostrea virginica,XM_022442025.1),长牡蛎(Crassostrea gigas,XM_011426803.2),虾夷扇贝(Mizuhopecten yessoensis,XM_021492286.1);尾感器纲:隐杆线虫(Caenorhabditis briggsae,XM_002630688.1),秀丽隐杆线虫(Caenorhabditis elegans,NM_062651.3),普通隐杆线虫(Caenorhabditis remanei,XM_003109029.1);腹足纲:福寿螺(Pomacea canaliculata,XM_025252237.1),海蜗牛 (Aplysia californica,XM_005097356.2),光滑双脐螺(Biomphalaria glabrata,XM_013220438.1);无铰纲:鸭嘴海豆芽(Lingula anatina,XM_013564455.1)。
2 结果
2.1 太平洋真宽水蚤CYP4C 和CYP44 基因序列分析
根据DNAMAN 软件拼接获得太平洋真宽水蚤EpCYP4C 全长cDNA 序列2 032 bp (GenBank:MK327539),包括60bp 5’非翻译区,487 bp 3’非翻译区,3’末端具有多腺苷酸加尾信号(ATAAA)和poly(A)尾,开放阅读框(ORF) 1 485 bp,编码494 个氨基酸(图1A)。ExPAsy 预测EpCYP4C 基因分子式是C2526H3976N676O738S18,原子总数为7 934,分子量为56.2 kDa;理论等电点为6.34,其中带负电荷的氨基酸数量(Asp+Glu)是65,带正电荷的氨基酸数量(Arg+Lys)是61;富含亮氨酸(Leu,11.3%) (表2),不稳定指数40.34(>40),为不稳定蛋白质。PROSITE 在线软件预测EpCYP4C 蛋白具有CYP4 家族特征序列EVDTFMFEGHDTT (第295—307 位氨基酸),CYP450 蛋白特有的血红素结合区FxxGxxxCxG (FSAGPRNCI,第433—442 位氨基酸),C 螺旋保守区WxxxR (WGSRR,第121—125 位氨基酸) 和K 螺旋保守区ExxR(ESLR,第360—363 位氨基酸) (图1A)。经SMART 预测发现EpCYP4C 蛋白具有TM 结构域(第2—21 位氨基酸)和p450 结构域(第31—490 位氨基酸),未发现信号肽(图1A)。
太平洋真宽水蚤EpCYP44 基因全长1 752 bp (GenBank:MK327540),其中开放阅读框1 485 bp,编码494 个氨基酸,5’非翻译区85 bp,3’非翻译区182 bp,3’末端具有多腺苷酸加尾信号(AATAAA)和poly(A)尾(图1B)。EpCYP44 基因分子式是C2534H3953N675O732S15,原子总数是7909,分子量为56.067 kDa;带负电荷的氨基酸数量(Asp+Glu)是58,正电荷的氨基酸数量(Arg+Lys)是57,理论等电点为6.88;富含亮氨酸(Leu,11.5%)(表3),EpCYP44 是不稳定蛋白质(不稳定指数:43.80)。EpCYP44 蛋白具有血红素结合区保守序列FGHGTRMCAG (第434—443 位氨基酸),C 螺旋保守区WYKLR (第123—127 位氨基酸)和K 螺旋保守区ETFR (第360—363 位氨基酸)(图1B)。SMART 预测发现EpCYP44 蛋白具有CYP450 家族成员特有的保守结构域,p450 结构域(第24—490 位氨基酸),无信号肽(图1B)。
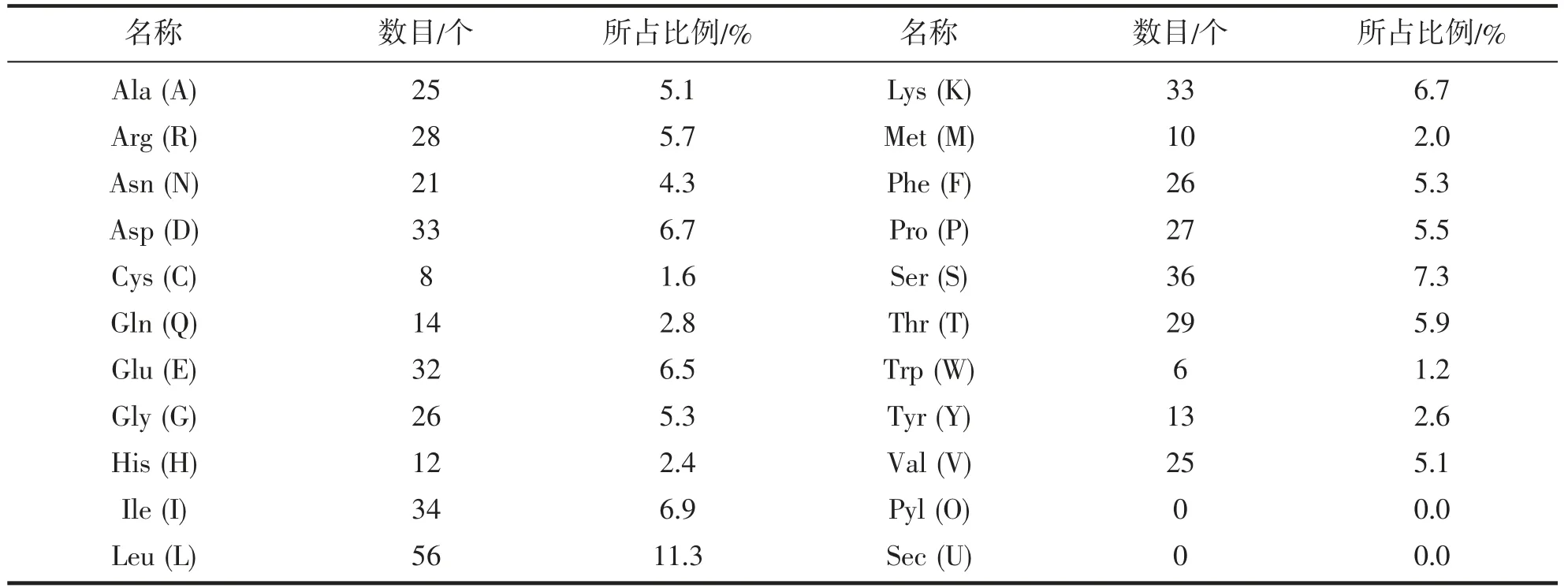
表2 EpCYP4C 氨基酸组成Tab.2 Amino acid composition of EpCYP4C
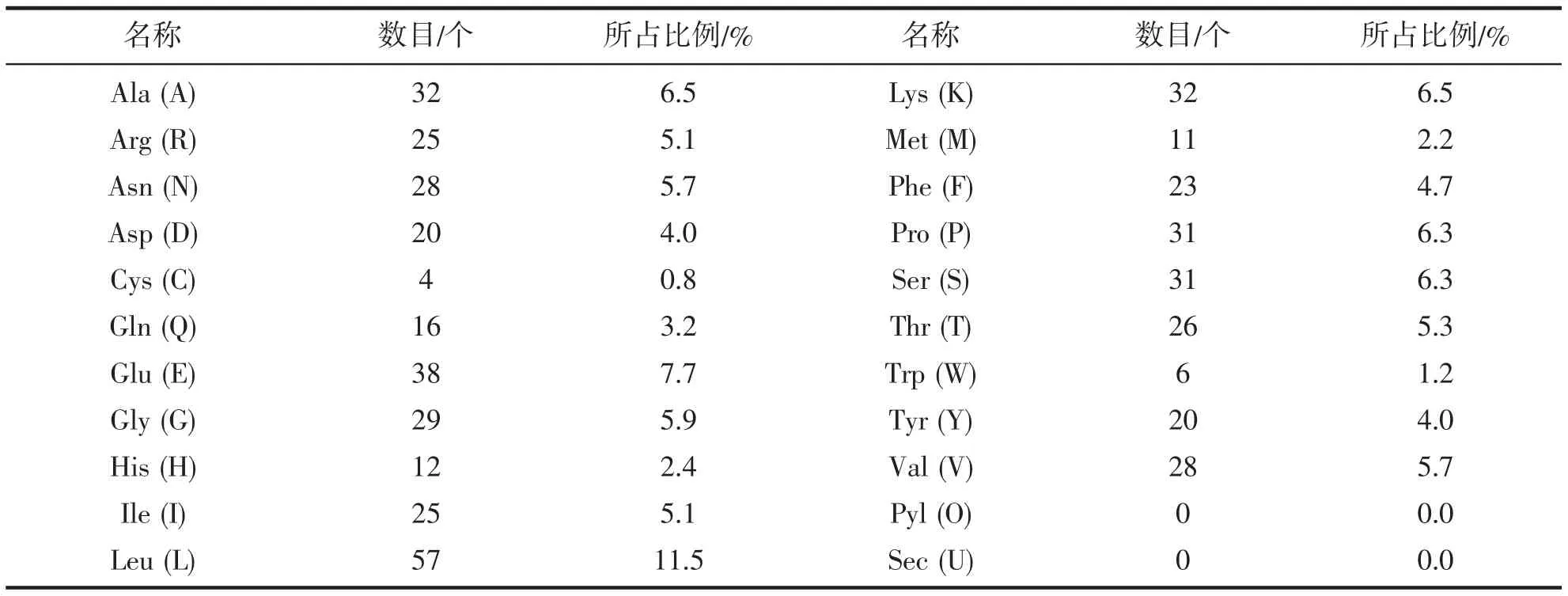
表3 EpCYP44 氨基酸组成Tab.3 Amino acid composition of EpCYP44
2.2 CYP4C 和CYP44 蛋白多序列比对
经BLAST 比对发现EpCYP4C 蛋白与近亲真宽水蚤CYP4C 蛋白同源性最高,高达81.38%。EpCYP4C蛋白与近亲真宽水蚤CYP4C、日本虎斑猛水蚤CYP4V22、加利福尼亚虎斑猛水蚤CYP4V22、中华绒螯蟹CYP4、黑腹果蝇CYP4C3、阿里山潜蝇茧蜂CYP4C3 和中国明对虾CYP4 的氨基酸序列通过DNAMAN 软件进行多序列比对。多序列比对结果发现TM 结构域在多个物种间保守性较差,但与近亲真宽水蚤氨基酸序列保守性较高;p450 结构域在多物种间的同源性较高;CYP4 家族特征序列和血红素结合区在进化上是高度保守的(图2A)。
BLAST 比对发现EpCYP44 蛋白与近亲真宽水蚤CYP44 氨基酸序列同源性最高(83.60%)。通过DNAMAN 软件对EpCYP44 蛋白与近亲真宽水蚤CYP44、日本虎斑猛水蚤CYP44D1、加利福尼亚虎斑猛水蚤CYP44D1、美洲牡蛎CYP44、隐杆线虫CYP44A1、烟粉虱CYP44 和白蚁CYP44 的氨基酸序列进行同源性比对。结果表明EpCYP44 蛋白p450 结构域在多个物种间同源性较高;其血红素结合区在进化上保守性较高(图2B)。

图1 EpCYP4C 和EpCYP44 核苷酸和氨基酸序列分析Fig.1 Nucleotide and amino acid sequences analysis of EpCYP4C and EpCYP44 gene

图2 不同物种间的蛋白多序列比对Fig.2 Multiple sequence alignment in different species
2.3 CYP4C 和CYP44 氨基酸序列系统进化树构建
收集来自不同物种的CYP4C 氨基酸序列,根据NJ 法构建系统进化树。系统进化树结果显示,太平洋真宽水蚤CYP4C 与甲壳纲、桡足亚纲物种(近亲真宽水蚤、日本虎斑猛水蚤和加利福尼亚虎斑猛水蚤)聚集成1 个单独的分支;昆虫纲物种CYP4C3 聚集成1 个分支;中国明对虾、凡纳滨对虾和日本对虾与软甲纲物种聚集成1 个分支(图3A)。结果分析表明太平洋真宽水蚤CYP4C 和近亲真宽水蚤亲缘关系更近,同源性更高;与昆虫纲物种亲缘关系较远(图3A)。
根据甲壳纲、昆虫纲、内口纲、曳鳃纲、尾感器纲、腹足纲、无铰纲和双壳纲八类物种CYP44 氨基酸序列构建系统进化树。结果分析发现太平洋真宽水蚤CYP44 先与桡足亚纲物种聚为1 个小分支,然后与内口纲、尾感器纲、昆虫纲和曳鳃纲四类物种聚为1 个分支,再与腹足纲物种聚为1 个大分支;双壳纲和无铰纲两类物种聚为1 个大分支(图3B)。结果说明太平洋真宽水蚤CYP44 与近亲真宽水蚤同源性更高,亲缘关系更近;与双壳纲物种和无铰纲的鸭嘴海豆芽亲缘关系较远(图3B)。
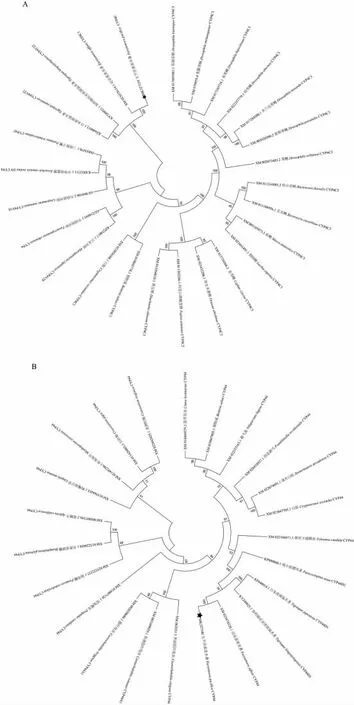
图3 不同物种氨基酸序列的系统进化树(NJ 树)Fig.3 Phylogenetic trees of amino acid sequences from various species (Neighbor-joining)
2.4 EpCYP4C 和EpCYP44 蛋白二级结构与三级结构预测
通过SOPMA 软件预测EpCYP4C 蛋白二级结构,结果显示,α 螺旋(h)由235 个氨基酸残基组成,占二级结构的47.57%;无规则卷曲(c)由181 个氨基酸残基组成,占二级结构的36.64%;由60 个氨基酸残基组成的β 折叠(e)和由18 个氨基酸残基组成的β 转角(t),分别占二级结构的12.15%和3.64% (图4A);这说明EpCYP4C 蛋白二级结构主要由α 螺旋和无规则卷曲组成。通过SWISS-MODEL 软件构建EpCYP4C 蛋白三维结构,结果显示其空间结构由23 个α 螺旋和8 个β 折叠组成(图4B)。
经SOPMA 预测发现EpCYP44 蛋白二级结构主要由α 螺旋和无规则卷曲组成。α 螺旋(h)由245 个氨基酸残基组成,占二级结构的49.60%;无规则卷曲(c)由177 个氨基酸残基组成,占二级结构的35.83%;β转角(t)和β 折叠(e)分别由23 和49 个氨基酸残基组成,β 转角占二级结构的4.66%,β 折叠占二级结构的9.92% (图4C)。EpCYP44 蛋白三级结构模型显示由20 个α 螺旋和11 个β 折叠构成(图4D)。
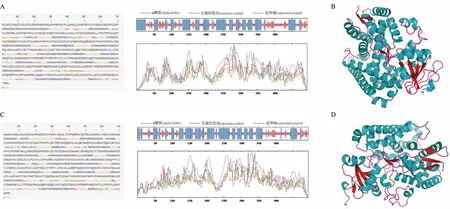
图4 EpCYP4C 和EpCYP44 蛋白二级结构与三级结构Fig.4 Secondary structure and three-dimensional structure of EpCYP44 and EpCYP44 protein
2.5 EpCYP4C 和EpCYP44 蛋白亚细胞定位
通过PSORT 程序预测EpCYP4C 和EpCYP44 蛋白亚细胞定位。EpCYP4C 蛋白亚细胞定位预测显示,分布在细胞质的可能性为39.10%,分布在细胞核的可能性为21.70%,分布在线粒体的可能性为17.40%,分布在内质网的可能性为8.70%,分布在过氧化物酶体、高尔基体和分泌系统囊泡的可能性均为4.30%(图5A)。另外,亚细胞定位预测发现EpCYP44 蛋白分布在细胞质和线粒体的可能性较大,分别为47.80%和34.80%,分布在细胞核的可能性为8.70%,分布在细胞骨架和分泌系统囊泡的可能性为4.30% (图5B)。
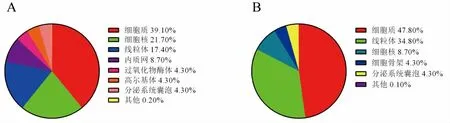
图5 EpCYP4C 和EpCYP44 蛋白亚细胞定位Fig.5 The subcellular localization of EpCYP4C and EpCYP44 protein
3 讨论
CYP450 超家族广泛存在于脊椎动物和无脊椎动物等多种生物中[20],参与动物体内多种药物的生物转化[21],在内源性和外源性化合物代谢过程中发挥重要作用[9],因此CYP450 酶家族被认为是检测环境的生物标志物。另外,CYP450 超家族成员众多,周君霞等学者指出CYP450 超家族包括36 个基因家族,主要是CYP1-CYP44 家族[22]。其中,CYP1-4 家族参与脊椎动物和无脊椎动物的I 相代谢解毒反应[23],且CYP4 家族是CYP450 酶系的重要家族,国内外多项研究[12,16,24]表明CYP4 家族参与生物解毒代谢过程,例如CYP4家族在哺乳动物内源性底物代谢过程中发挥功能作用[25-26];在昆虫中,CYP4 家族参与解毒代谢反应[27];CYP4 家族在节肢动物中参与蜕皮甾类生物的合成[28]。CYP450 酶系的代谢机制及药物应用等方面在不同物种中已被广泛研究[26-29]。
目前,CYP450 酶系在海洋无脊椎动物中也有相关研究,如2010 年发现翡翠贻贝在污染物多氯联苯暴露下,对性腺中CYP4 基因的表达有显著的诱导作用[16];在2017 年对海洋纤毛虫的研究中发现CYP450家族基因参与苯并芘和β-萘黄酮的解毒作用[30]。然而,桡足类作为鱼类等物种的主要饵料[31],海洋环境变化的指示物种[3],其CYP450 酶家族的分子特征和解毒机制还尚未明确。本研究通过RACE 技术首次克隆太平洋真宽水蚤EpCYP4C 和EpCYP44 全长cDNA 序列;序列分析发现EpCYP4C 和EpCYP44 蛋白具有CYP450 家族特有的血红素结合区,属于CYP450 酶系;而且EpCYP4C 蛋白具有CYP4 家族特有的保守序列,归类为CYP4 家族,具有TM 结构域,推测其可能是一种膜蛋白;亚细胞定位预测发现EpCYP4C 和EpCYP44 蛋白分布在细胞质和线粒体的可能性较大,这可能与其解毒代谢功能相关。本研究对太平洋真宽水蚤CYP4C 和CYP44 基因的研究既揭示了海洋桡足类CYP450 家族基因的存在,表明海洋桡足类CYP450 家族基因的多样性;也为后续研究太平洋真宽水蚤EpCYP4C 和EpCYP44 基因的本底表达及在污染物暴露下的表达奠定基础;更为研究海洋桡足类CYP450 超家族的分子特征提供参考。
——以“不同咖啡因饮料对水蚤心率的影响”为例

