一种可预测弥漫大B细胞淋巴瘤患者生存的新型6基因预测模型
王亮 周璇 梁晓杰 何颖芝
作者单位:100730 北京 1首都医科大学附属北京同仁医院血液科;2北京大数据精准医疗高精尖创新中心,北京航空航天大学&首都医科大学,北京同仁医院;510280 广州 3南方医科大学第二临床医学院,南方医科大学珠江医院血液科
弥漫大B细胞淋巴瘤(diffuse large B-cell lymphoma,DLBCL)为最常见的非霍奇金淋巴瘤亚型。随着利妥昔单抗时代的到来,DLBCL患者的治疗效果获得极大改善,但仍有部分患者复发,预后较差[1]。研究表明国际预后指数(international prognostic index,IPI)、改良国际预后指数(revised-IPI,R-IPI)及 NCCN-IPI等在预后评估中有较好的实用性[2-3]。近年来反映肿瘤炎症微环境的指标也被用于DLBCL的预后评估[4]。然而,对预后有重要影响的遗传学标记、细胞起源等因素并未纳入其中,这可能造成部分患者预后评估的偏差。随着基因芯片及测序技术的广泛应用,基因标记亦被广泛应用于肿瘤患者的风险分层[5]。本研究基于公开的数据集,构建可预测DLBCL患者生存的基因预测模型,并将其与其他预后评估指标相结合,以期能够更好地评估患者的预后,以进一步指导治疗方案的选择。
1 方法
1.1 数据来源及处理
分别从 Gene Expression Omnibus(GEO)和The Cancer Genome Atlas(TCGA)下载DLBCL患者的基因表达数据和临床数据,包括GSE10846、GSE32918、NCICCR数据集。将随访信息不全及随访时间≤0 d的样本剔除后,纳入GSE10846数据集 412例患者,GSE32918数据集172例患者,NCICCR数据集234例患者;剔除GSE32918数据集中未采用标准治疗的32例患者后,共786例患者符合标准纳入分析。GEO数据集的原始数据处理步骤如下:⑴使用limma包中的normalizeBetweenArrays函数对GEO样本进行组间矫正。⑵GSE10846、GSE32918数据集的探针分别使用GPL570、GPL8423平台文件进行注释。对于对应多个探针的基因,保留最大平均值。NCICCR数据集的原始数据处理步骤如下:⑴NCICCR数据集的IDs使用hg38参考基因组进行注释,对于对应多个IDs的基因,保留最大平均值。⑵使用limma包的voom函数对NCICCR原始数据进行标准化处理[6]。
1.2 模型的构建及验证
采用单因素Cox回归筛选GSE10846、GSE32918数据集中与DLBCL患者总生存期(OS)相关的基因[7]。风险比(HR)>1为危险基因,HR<1为保护基因,将P<0.01设定为基因与生存具有相关性的阈值标准。将两个数据集共有的危险基因及保护基因取交集,并与Lasso回归、Cox回归相结合,构建最终的基因风险预测模型[8]。
将预后基因的表达值与其回归系数加权后计算风险评分[9]。以各队列的中位风险评分作为分界点,将患者分为高风险组和低风险组。采用Kaplan-Meier法评估高风险组和低风险组DLBCL患者的生存率,组间比较采用Log-rank检验;采用多因素Cox回归分析及分层分析评估风险评分与OS的关系;采用受试者工作特征曲线(ROC曲线)的下面积评价该模型的预测价值并与其他临床特征进行比较[10];在多因素Cox回归分析的基础上构建列线图模型,根据模型中各影响因素对结局变量的贡献程度,给影响因素的每个取值水平进行赋值,再将各个评分相加得到总评分,计算个体结局事件的预测值,并评估列线图的预测能力[11];采用Pearson相关性检验识别与风险评分有关的基因(风险评分相关基因定义为r>0.4且P<0.001),并对这些基因进行GO与KEGG分析,探讨相关基因的生物学功能[12]。详细流程见图1。
采用R(版本3.5.2)软件进行数据分析。采用“glmnet”包行 Lasso 回归分析,“survival”包行单因素、多因素Cox回归分析及绘制生存曲线,“survival ROC”包绘制ROC曲线,“pheatmap”包绘制风险相关性热图,“clusterProfiler”包行GO及KEGG分析;根据多因素分析结果,用“rms”包绘制列线图及校准图。观察的主要结局是OS,定义为从确诊到任何原因死亡的时间或末次随访时间。
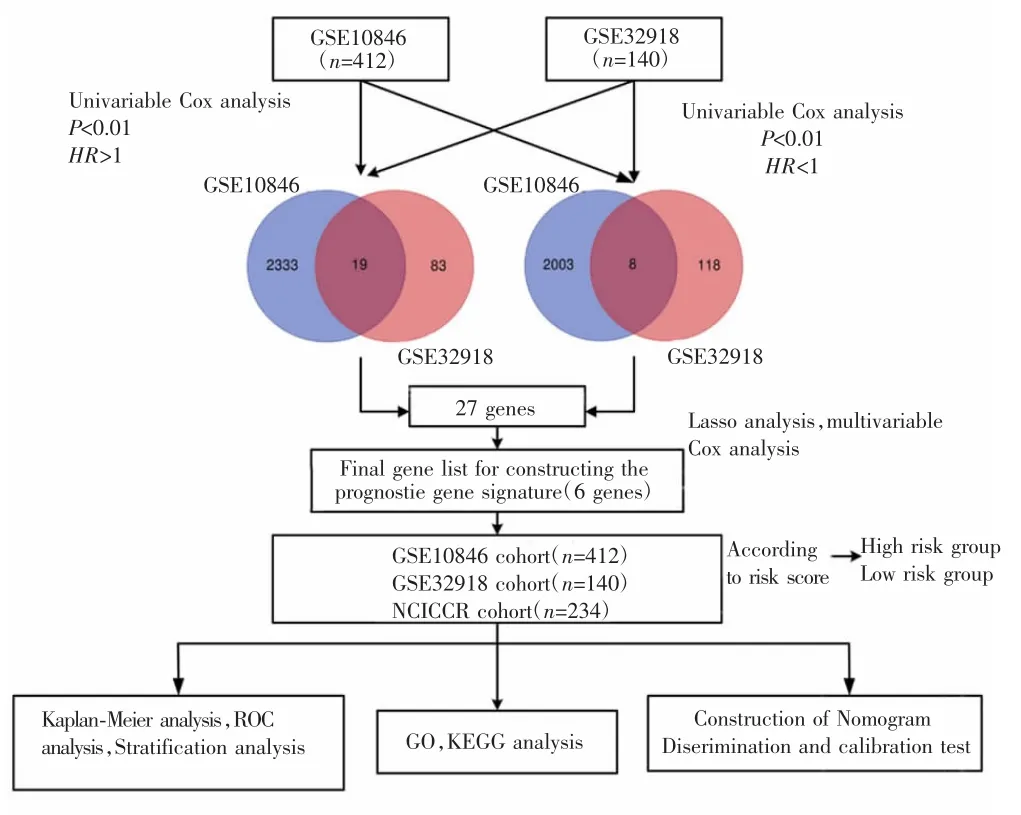
图1 流程图Fig.1 Study flow diagram
2 结果
2.1 基因预测模型的构建
对GSE10846、GSE32918数据集进行单因素Cox回归分析后,在P<0.01和HR<1筛选条件下,GSE10846和GSE32918数据集中分别有2 011个基因和126个基因被确定为候选保护基因。在P<0.01和HR>1筛选条件下,GSE10846数据集有 2 352个基因、GSE32918数据集有102个基因被确定为候选危险基因。对候选保护基因及候选危险基因取交集后共保留27个基因。以GSE10846数据集为训练集,通过Lasso回归对变量进行筛选,保留13个基因,见图2。通过多因素Cox回归识别出 6个基因(LNPEP、SNX20、GTPBP10、CALR、BDH1、C5orf30)作为 DLBCL 患者的预后标志物,见表1。
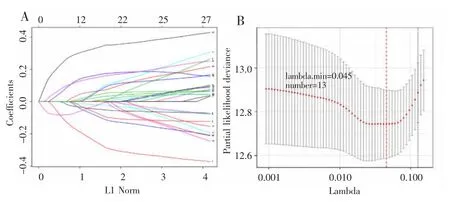
图2 Lasso回归确定模型所需基因数量Fig.2 Determination of gene numbers in the model through Lasso regression analysis
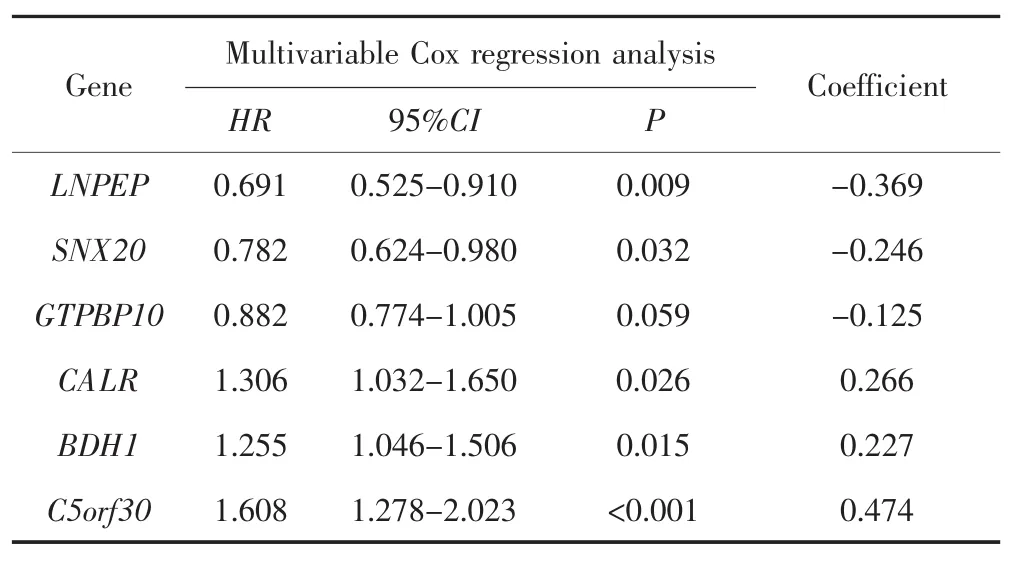
表1 基于GSE10846数据集构建的6基因风险模型Tab.1 Construction of 6-gene based risk model from GSE10846 dataset
2.2 基因预测模型的验证
将预后基因的表达值与其回归系数加权得出风险评分,根据风险评分的中位数将患者分为高风险组和低风险组。生存曲线结果显示,高风险组患者的OS短于低风险组。在训练集GSE10846中,6基因模型预测3年总生存率的AUC为0.722,在验证集GSE32918、NCICCR中分别为0.758、0.693。见图3。
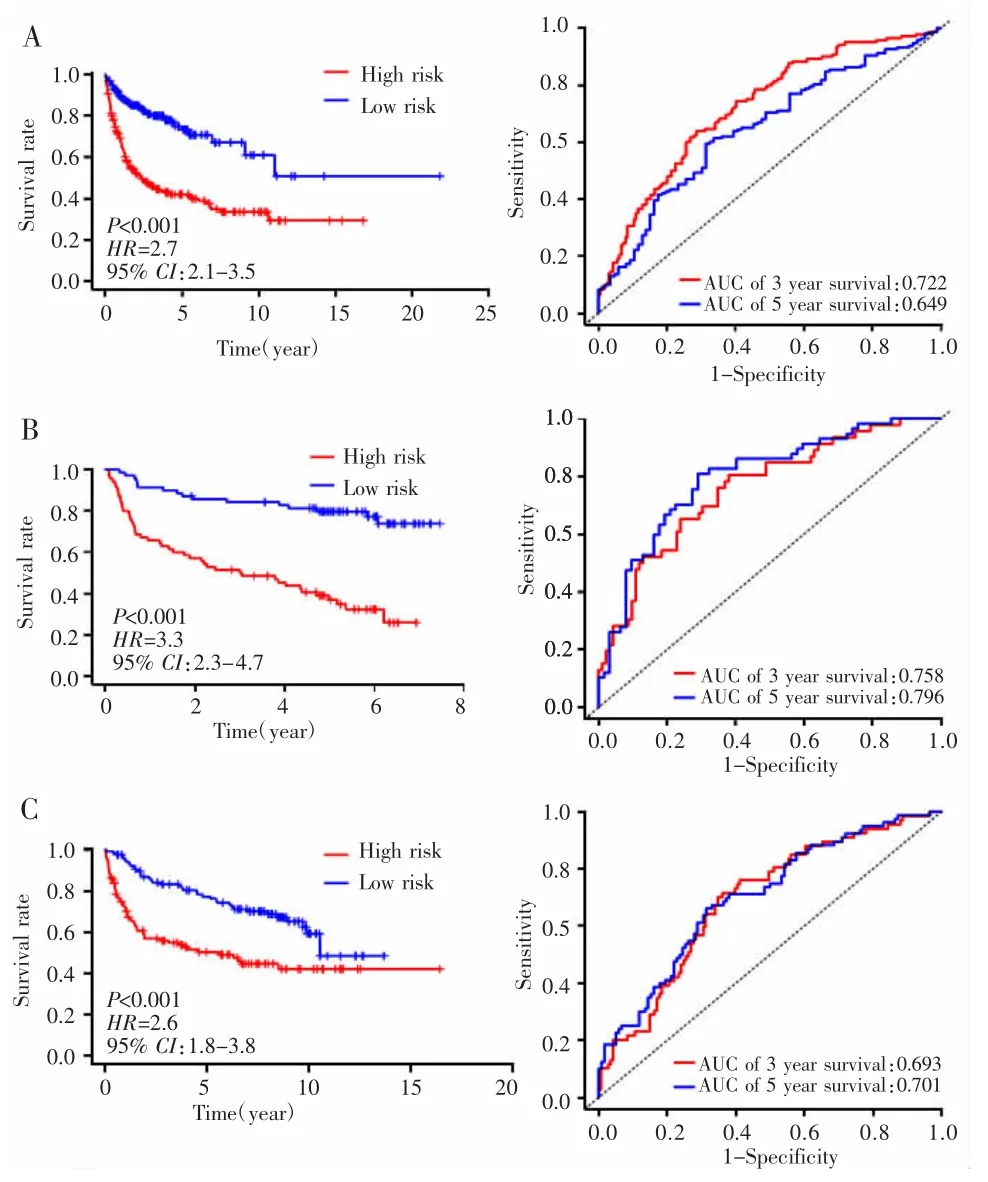
图3 训练集及验证集中6基因模型的Kaplan-Meier曲线和ROC曲线Fig.3 Kaplan-Meier and ROC curves for the 6-gene based model in the training and testing cohorts
在该基因模型与临床相关性热图中(去除临床信息不完整的患者后,共保留344例患者),发现保护基因LNPEP、SNX20、GTPBP10在低风险组中高表达,在高风险组中低表达。危险基因CALR、BDH1、C5orf30在低风险组中低表达,在高风险组中高表达;验证队列中亦观察到相似的结果。风险评分与分期、治疗方案、细胞来源、年龄及生存状态有关,见图4。ROC曲线结果显示,与其他临床指标相比,风险评分的AUC最高,且在验证集中AUC高于IPI评分(0.706 vs 0.674),见图5。
2.3 6基因风险评分是DLBCL患者的独立预后因素
多因素Cox回归分析结果显示,在3个独立的数据集中,6基因风险评分是预测患者总生存的独立预后因素,见表2。
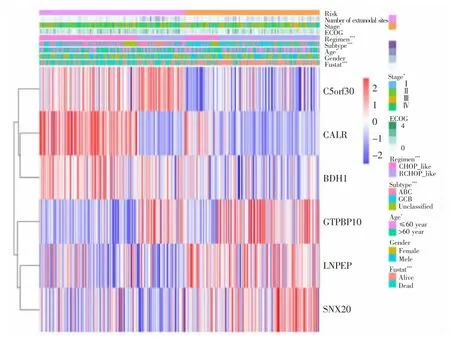
图4 GSE10846数据集的风险评分与临床特征的相关性热图Fig.4 Heat map of risk score and clinical relevance in the GSE10846 dataset
2.4 分层分析
对年龄、性别、分期和细胞起源、ECOG等临床特征进行分层分析,结果显示,高风险组患者OS均较低风险组短(均P<0.05),见图6。
2.5 列线图模型的构建及评估
在多因素Cox回归的基础上,将风险评分与临床因素相结合构建列线图模型。结果显示列线图模型在GSE10846数据集中的AUC为0.796,均高于任何单一因素,见图7。校准曲线显示,在列线图预测生存概率和实际观测的生存概率之间具有较好的一致性,见图8。
2.6 功能分析
Pearson相关性检验识别出与风险评分有关的1 123个基因(r>0.4且P<0.001),其中629个基因与风险评分呈正相关,494个基因与风险评分呈负相关。GO与KEGG分析结果显示,以上基因主要富集于DNA复制和修复、蛋白加工、细胞周期、病毒致癌机制相关等生物功能及通路上,见图9。
3 讨论
DLBCL作为高度异质性肿瘤,在治疗早期发现高危患者并及时调整治疗策略对延长生存期至关重要[13-14],然而现有的预后评分系统尽管具有很好的临床价值,但对部分患者仍不能发挥很好的识别作用。近年来,随着生物信息学的发展,DLBCL的基因表达谱也已被应用于预测肿瘤特征和预后。ZHOU等[15]研究确认了一组由17个lncRNA组成的可区别GCB和ABC亚型的生物标志物。然而,目前直接用于预测DLBCL患者总体生存率的基因预测模型的研究较少。
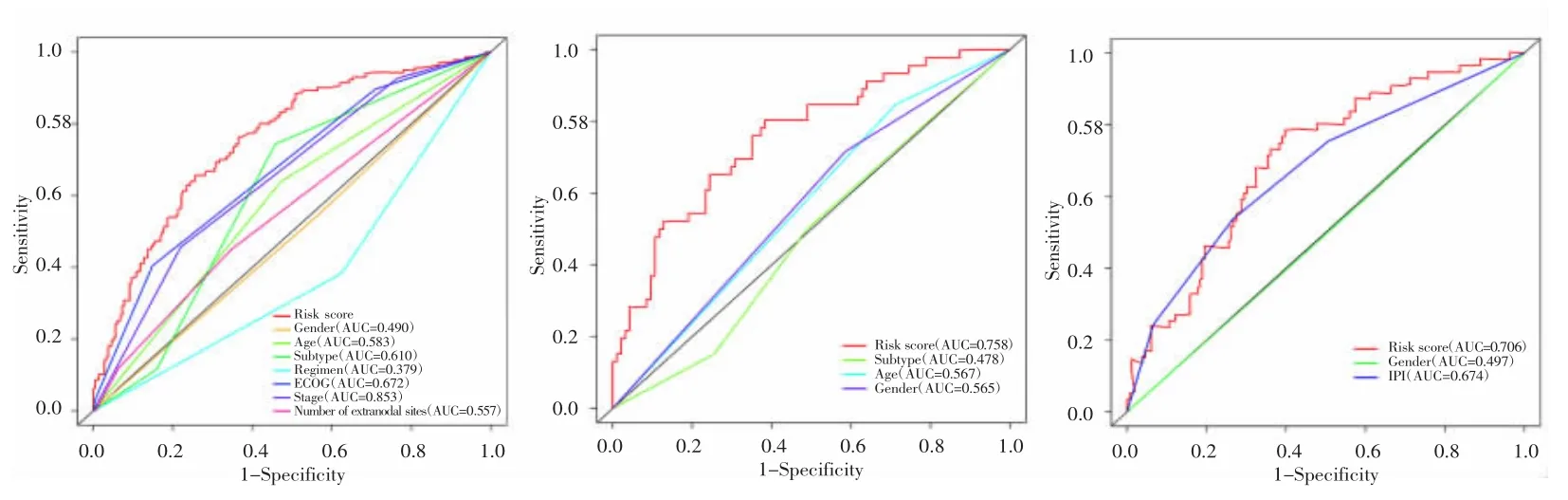
图5 多指标ROC曲线Fig.5 Multi-index ROC curves
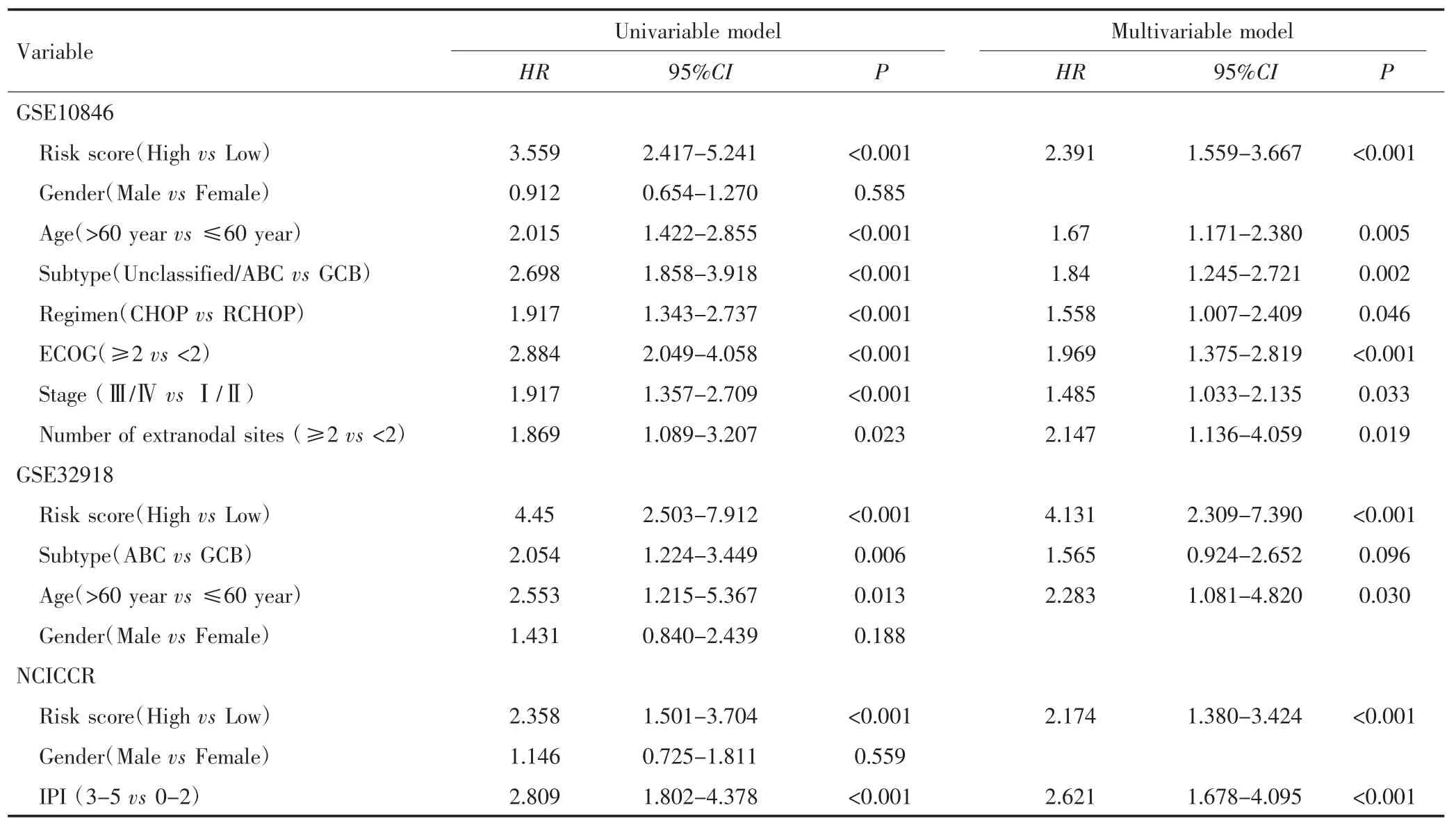
表2 影响DLBCL患者预后因素的单因素和多因素Cox回归分析Tab.2 Univariable and multivariable Cox regression analyses of prognostic factors affecting DLBCL patients
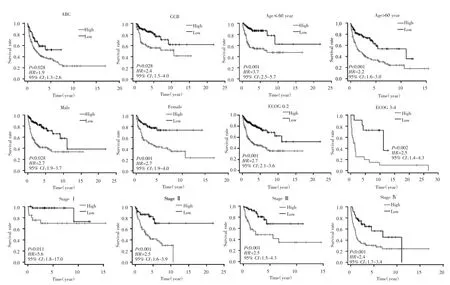
图6 不同细胞起源、年龄、性别、ECOG评分、分期患者的Kaplan-Meier生存曲线Fig.6 Kaplan-Meier survival curves for patients in different subgroups,stratified by cell origin,age,gender,ECOG scores and stage
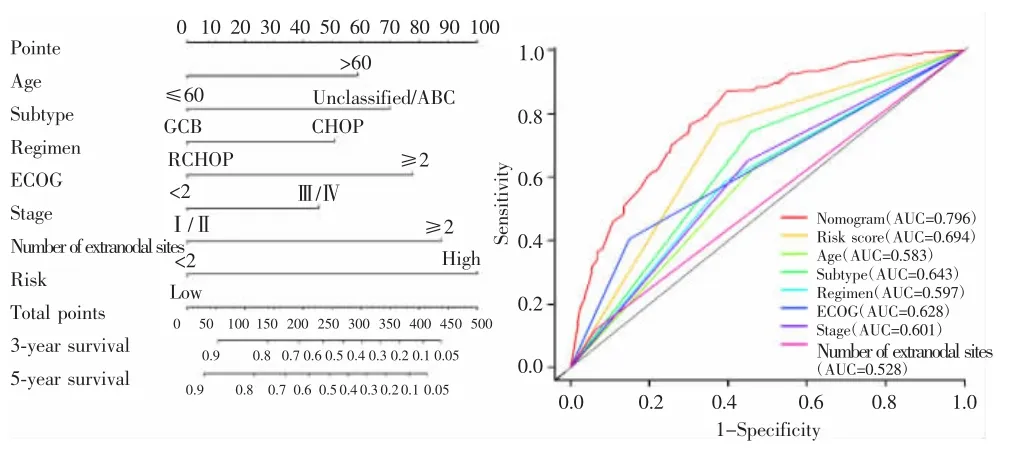
图7 预测DLBCL患者生存的列线图Fig.7 Nomogram of survival prediction in patients with DLBCL
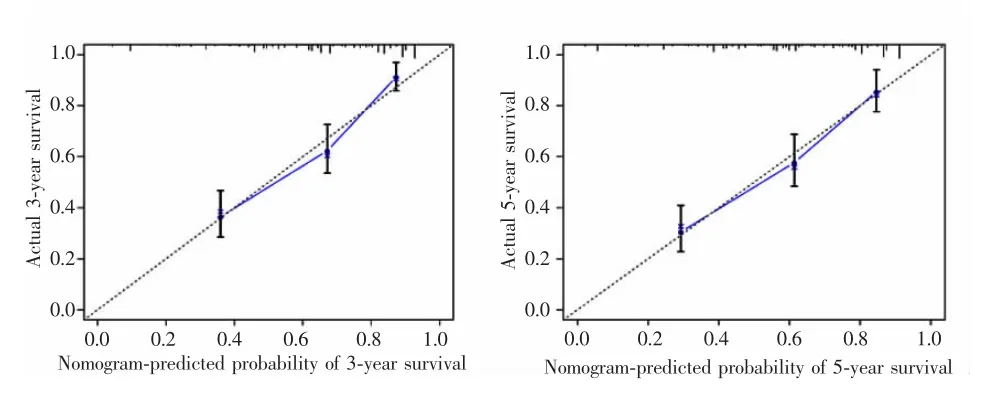
图8 训练队列患者3年和5年生存的校准曲线Fig.8 The calibration curves for predicting survival at 3 years and 5 years in the training cohort
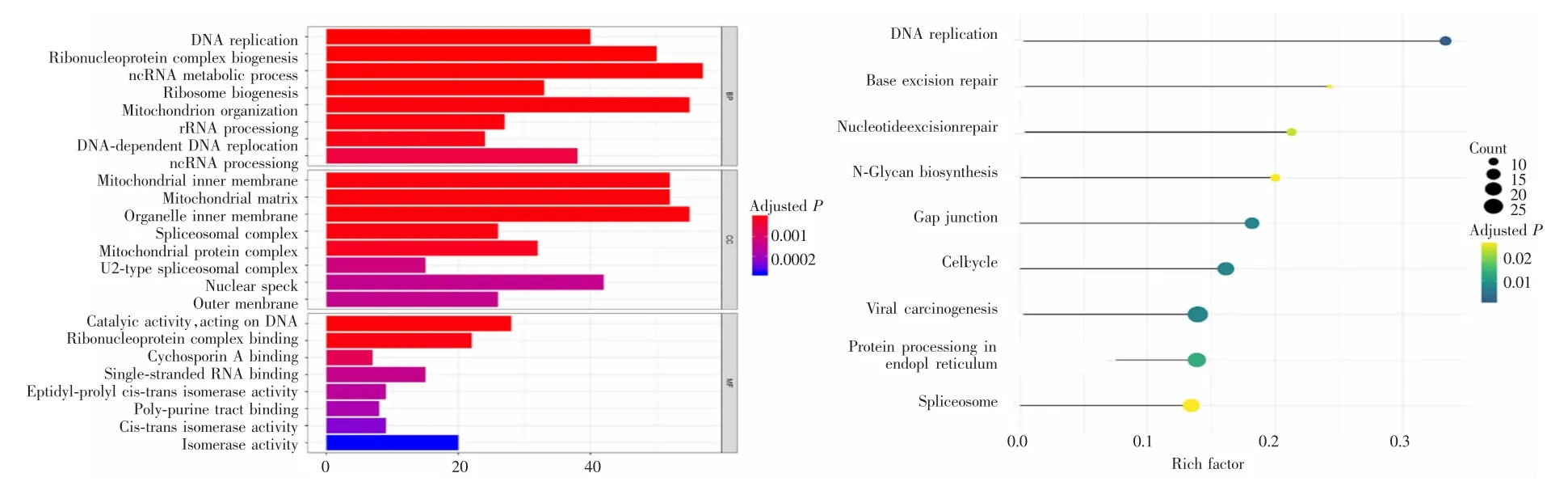
图9 与风险评分相关基因的GO及KEGG富集分析Fig.9 Analysis of GO and KEGG enrichment of genes related to risk score
本研究利用DLBCL患者的基因表达谱数据构建并验证了一种新的6基因预后模型。基因数据是来源于2个独立的数据集,较以单个数据集为基础所构建的预后模型更稳健。此外,本次构建的模型仅以6个基因为基础,可减少检测工作量及费用,并从分子生物学层面弥补了临床指标的不足。将其与年龄、亚型、治疗方案、ECOG、分期、结外部位数量等临床因素结合后发现能更准确地评估患者的预后,进一步识别出被遗漏的高危患者以及某些可能被高估的低危患者,同时列线图的AUC及校正曲线说明其具有较好的预测能力。但由于数据集缺乏完整的临床数据,尚不能完成外部验证,仍需在今后的临床工作中进一步验证其价值。
本研究通过多因素Cox回归识别出可作为DLBCL患者的预后标志物 6个基因为 LNPEP、SNX20、GTPBP10、CALR、BDH1、C5orf30,其影响患者预后的可能原因如下:研究表明生酮疗法通过减少葡萄糖供给,选择性切断肿瘤细胞的能量供应,并提高脂肪供能比例以抑制肿瘤细胞增殖,而BDH1正是酮体代谢的关键酶,其与BDH2基因相邻并相互调节,而BDH2被发现可作为抗凋亡因子参与急性白血病的发生、发展[16-17]。CALR基因编码的钙网蛋白是具有多种生物学功能的Ca2+结合蛋白,可参与肿瘤免疫逃逸、未折叠蛋白反应(unfolded protein response,UPR)和Ca2+信号传递,在人类肿瘤发生发展中发挥重要作用[18-19]。C5orf30被证实与类风湿性关节炎的组织破坏程度以及TNF、IL-1、IL-10等因子的表达相关,目前认为炎症因子与肿瘤发生发展有关,但暂无C5orf30与肿瘤的相关报道[20]。Genecards数据库检索结果表明LNPEP与胎盘滋养细胞肿瘤相关,其相关途径包括囊泡介导的转运和Ⅰ类MHC介导的抗原加工和呈递。GTPBP10在基本细胞过程,如蛋白质合成,核转运,膜运输和信号转导的调节中起关键作用[21]。本研究通过Pearson相关性检验识别出与风险评分相关的1 123个基因,发现这些基因主要富集在DNA复制和修复、蛋白加工、细胞周期、病毒致癌等生物功能及通路上,这从侧面反映出模型基因的功能作用。
本研究存在以下局限性:⑴用于构建模型的GSE10846及GSE32918数据集获得基因表达的测序平台不同,分别是Affymetrix Human Genome U133 Plus 2.0 Array及 Illumina HumanRef-8 WG-DASL v3.0,其探针数目及所对应的基因表达水平有一定区别,可能存在一定偏倚。⑵虽然本研究发现在验证队列中,风险评分模型的预测效能高于IPI评分,但由于各个数据集中包含的临床信息不完整,无法在所有队列中进行验证。⑶本研究缺少在病理组织标本中进行相关基因表达情况的检测。
综上所述,本研究成功构建了一个具有较好预测能力的6基因风险评分预测模型,与年龄、亚型、治疗方案、ECOG、分期、结外部位数量等临床因素构建的列线图模型,有助于更全面地评估患者的预后,各基因可能是预后预测的有效指标。

