互花米草NAC转录因子家族的鉴定与表达分析
王涛涛,杨勇,魏唯,林辰涛,马留银
研究报告
互花米草NAC转录因子家族的鉴定与表达分析
王涛涛1,2,杨勇1,2,魏唯2,林辰涛2,马留银2
1. 福建农林大学林学院,福州 350002 2. 福建农林大学基础林学与蛋白质组学研究中心,福州 350002
互花米草()作为一种海岸带盐生植物,高度耐盐胁迫,但因为缺少参考基因组,其耐盐的分子机制却尚未见报道。NAC家族蛋白是植物特有的转录因子,调控植物的生长发育和胁迫应答。为了鉴定互花米草NAC蛋白(SaNAC)并探究它们与互花米草生长发育及胁迫响应之间的关系,本研究以互花米草三代全长转录组数据为参考,通过与水稻()、拟南芥()和玉米()NAC蛋白序列进行比对,并结合保守功能域进一步筛选,最终找到62个SaNAC蛋白。从蛋白序列比对、进化、motif预测、同源性比较、亚细胞定位、组织表达以及非生物胁迫下的基因差异表达等方面分别对互花米草NAC家族成员进行分析,结果发现SaNAC蛋白均含有保守的NAM结构域,且在进化上与水稻NAC家族具有一定的相似性;SaNAC家族中的两个蛋白SaNAC9和SaNAC49在细胞核表达;另外,本研究还发现互花米草基因表达具有高度组织和胁迫应答差异性。这些结果表明互花米草NAC转录因子家族不仅具有保守的功能域,而且在调控互花米草的生长发育和非生物胁迫响应过程中具有重要的作用。
互花米草;NAC;转录因子;基因家族;基因表达
由于固定生长的特点,植物极度依赖其所在的生存环境,因此环境因素对植物生长发育有着深远的影响[1]。其中,土壤盐渍化是制约植物生产的主要环境压力之一,高盐胁迫诱使细胞膜解体、产生活性氧、削弱代谢过程、抑制光合作用和减弱营养吸收,进而延缓植物生长和发育、降低农林作物品质[2]。因此,研究植物高盐耐受性的调控机理具有十分重要的理论与实践价值。
转录因子是一类重要的调控子,一般通过结合在胁迫相关基因的顺式作用元件,参与植物对干旱、高盐、低温等非生物胁迫的响应[3]。NAC (NAM、ATAF1/2和CUC2)家族是植物特有的且最大的转录因子家族之一。NAC家族蛋白在N端拥有一个大约160个氨基酸残基大小的保守区域,该区域被进一步分为5个亚结构域(A~E)[4,5]。在植物中,NAC转录因子调控大量的生长过程,包括花形态建成[6]、根部发育[7]、叶片衰老[8]和种子萌发[9]等。同时NAC转录因子也参与植物各类胁迫应答过程[10],例如小麦() NAC家族因子TaNAC2TaNAC47和TaNAC67在促进自身耐多种胁迫压力过程中具有重要的作用[11~13],林木类植物刚毛柽柳() ThNAC13因子能促进柽柳和拟南芥()更加耐盐及渗透胁迫[14],表明NAC转录因子在促进农林物种耐胁迫改良方面均具有重要的利用价值。
随着高通量测序技术的发展,越来越多的植物NAC家族被发掘和鉴定,大大加速了NAC蛋白在调控植物生长发育和胁迫应答方面的研究。研究人员不仅在拟南芥和水稻()中发现117和151个NAC蛋白[15],在大豆()和乌拉尔图小麦()中也发现含有152和87个NAC蛋白[16,17]。同时,在一些林木植物如果梅()、白梨()和海岸松()中也分别识别出113、146和37个NAC因子[18~20]。分析NAC家族因子有助于探究植物在非生物胁迫下的应答机理,然而大多数植物因为缺少高质量的参考基因组,NAC家族还没有被系统地鉴定和研究。第三代转录组测序(Pacbio单分子测序)技术的诞生为很多缺少基因组的物种提供了可参考的全长转录组序列[21],在一定程度上解决了这些物种家族分析研究上的难题。
互花米草()作为耐盐性良好的盐生植物,自1979年引入我国以后,广泛分布于沿海地区的潮滩及河口湾地带,根系发达,茎秆粗壮,能够忍受恶劣的气候和环境,尤其是高盐胁迫。本研究以互花米草为对象,将全长转录组序列作为参考,通过与拟南芥、水稻和玉米NAC蛋白序列进行比对,并经过功能域分析和筛选,获得62个SaNAC蛋白,随后从序列比对、进化比较、蛋白定位、组织表达以及非生物胁迫下的基因差异表达等角度对SaNAC家族进行系统的分析,完成了NAC转录因子家族在互花米草转录组水平上的初步鉴定以及在生长发育和胁迫响应过程中的表达分析,为进一步探究互花米草NAC蛋白功能及遗传应用提供理论依据。
1 材料与方法
1.1 材料
互花米草种子和植株采自江苏省赣榆市(E119°16ʹ, N34°46ʹ),把种子浸泡在1.5%盐水中并且储存于4℃以备长期使用。幼苗种植:将互花米草种子放在含1/2 Hoaglang营养液的滤纸上萌发7 d,然后转移至含有营养液的生长盒中继续生长3周,期间每隔3 d更换一次营养液。盐处理:将互花米草幼苗分别培养在正常营养液以及含350、500和800 mmol/L NaCl的营养液中24 h,然后进行整株取样并立即置于液氮保存。干旱处理:将4周大的互花米草幼苗放在干燥的滤纸上,分别在第0 h、1 h、8 h和24 h取样并立即置于液氮保存。ABA处理:将4周大的互花米草幼苗放入含有100 μmol/L ABA[22]的营养液里培养,分别在第0 h、1 h、8 h和24 h取样并冻存。
1.2 互花米草NAC家族的鉴定
拟南芥、水稻以及玉米NAC家族蛋白序列从植物转录因子数据库上(http://planttfdb.cbi.pku.edu.cn/)下载,互花米草全长转录组数据来自于第三代Pacbio测序数据[23],通过ORF预测(http://bioinformatics. ysu.edu/tools/OrfPredictor.html)获得所有互花米草转录本对应的氨基酸序列。将拟南芥、水稻以及玉米的NAC蛋白序列分别与互花米草蛋白序列进行比对,值取小于−50,得到所有与NAC蛋白序列相似性较高的互花米草转录本序列号。根据序列号在互花米草转录组网站(http://plantpolya.org/SAPacBio/)下载对应的转录本序列并通过ORF预测得到对应的氨基酸序列。将所有可能的SaNAC蛋白序列放在NCBI (https://www.ncbi.nlm.nih.gov/cdd)中进行保守功能域预测,去除不含NAC功能域的蛋白,并得到最终的互花米草NAC家族蛋白。在ProtPaeam (https://web.expasy.org/protparam/)上进行SaNAC蛋白氨基酸数量、分子量和等电点的分析,同时在Protcomp 9.0 (http://linux1.softberry.com/berry.phtml? topic=protcomppl&group=programs&subgroup=proloc)上进行蛋白亚细胞定位预测。
1.3 序列比较、同源进化树分析和motif预测
利用DNAMAN软件进行复合序列比对,保守的结构域参照拟南芥和水稻的NAC家族进行划分[5]。利用MEGA7软件构建系统进化树,选择Neighbour- Joining方法,并设置自检次数为1000次。利用MEME (http://meme-suite.org/)进行motif预测,数量设置为10。
1.4 烟草瞬时表达
利用基因特异性引物(表1)从互花米草cDNA中扩增和基因全长CDS序列,并连接到植物表达载体pCambia3301上GFP标签的N端,随后将载体借助农杆菌AGL0瞬时转入烟草(L)叶片中。烟草浸染实验步骤:首先从转化目的载体的培养板上挑取单克隆菌落培养并进行PCR鉴定,然后取200 μL阳性菌液接入50 mL含有10 mmol/L MES、20 μmol/L 乙酰丁香酮以及相应抗生素的LB液体培养基中培养过夜。第二天,将菌液离心(4000 r/min,15 min),并用适量体积重悬液(10 mmol/L MES、150 μmol/L乙酰丁香酮、10 mmol/L MgCl2)将沉淀混匀至值为1.0~1.5之间,重悬的菌液放置室温2 h后用无菌注射器轻轻注射入新鲜烟草的叶片中,并对注射位置进行标记。将烟草放在暗室过夜培养后继续放置温室生长1~2 d。进行显微观察时,取小块注射位置的叶片浸泡在1 mg/L的DAPI染液中30 min,分别在倒置显微镜(型号Zeiss Axio Observer.A1)下观察SaNAC9、SaNAC49以及对照蛋白在GFP和DAPI通道下的荧光表达,找到同时表达的核定位信号后与明场图片进行重叠。
1.5 SaNAC基因家族表达模式分析与验证
1.5.1 RNA-seq数据分析
表达模式分析参考互花米草转录组测序结果[23],RNA-seq数据从Bioproject(PRJNA413596)下载。使用bowtie2 (v2.2.9)[24]将测序数据与互花米草全长转录组数据进行比对,然后对数据进行均一化处理,最后使用RSME[25]对处理后的数据进行计算,得到基因标准化后的表达量FPKM值。
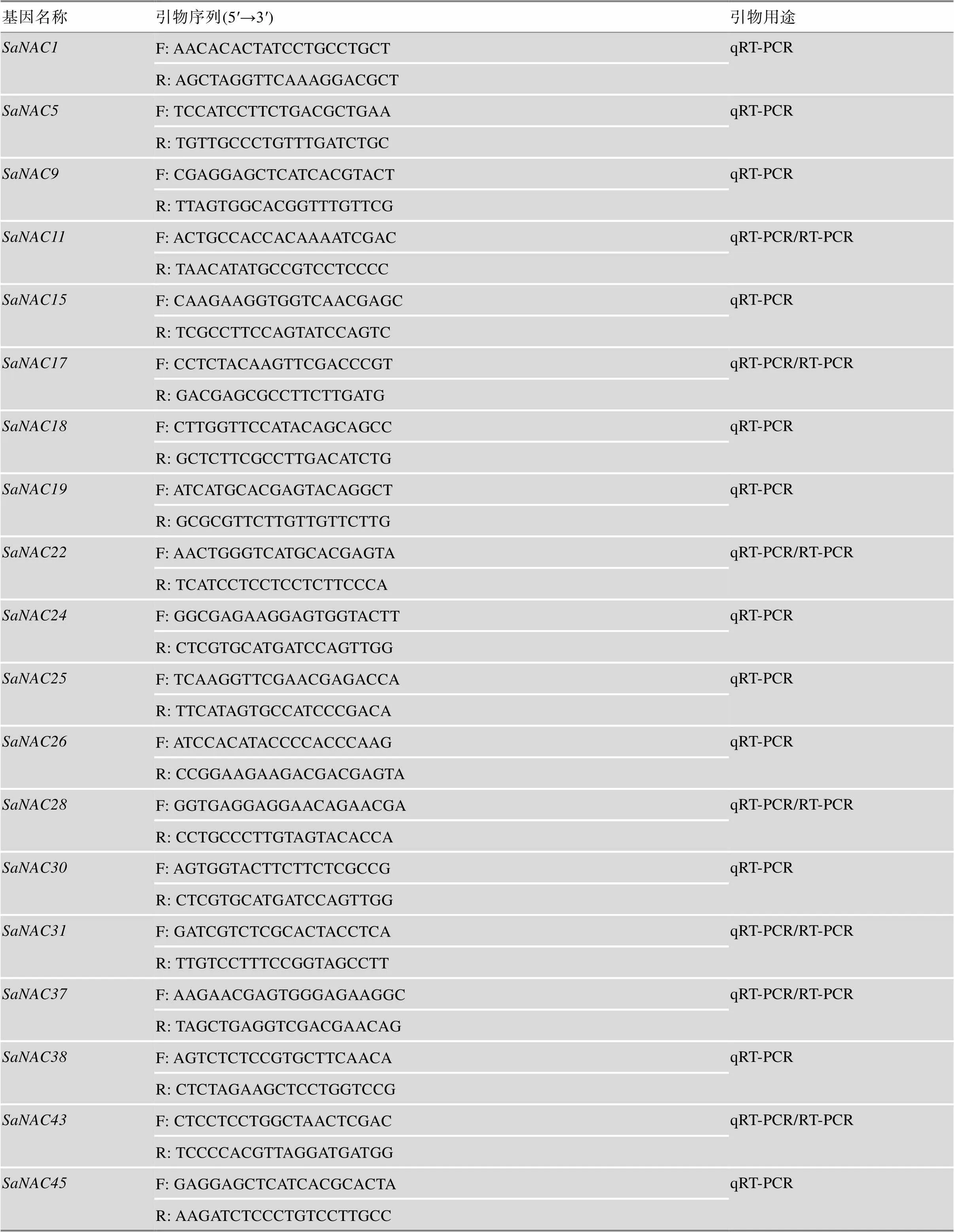
表1 本研究使用的引物序列
下划线部分代表载体构建接头序列。
1.5.2 RT-PCR验证
用试剂盒(广州美基生物公司,R4151-02)提取不同组织(叶、茎、根和地下茎)以及胁迫处理的互花米草幼苗总RNA,具体操作参照说明书进行,对RNA进行定性定量检测确保其质量满足后续实验。取1 μg RNA,利用MonScript™ RTIII All-in-One Mix反转录试剂盒(武汉莫纳生物公司,RN05004M)合成第一链cDNA,具体操作参照说明书进行,将cDNA的浓度稀释至5 ng/μL后备用。挑选、、、、、、、、、、和基因进行RT-PCR验证,引物序列如表1所示。扩增前先用内参基因对模板进行均一化,然后取合适体积的模板进行PCR。PCR反应体系:10 μL MonAmp™ 2× Taq Mix (武汉莫纳生物公司,MP05001),0.5 μL正向引物,0.5 μL反向引物,适当体积的模板cDNA,适量体积RNAse-free水,总体系20 μL。反应程序:94℃ 3 min;94℃ 30 s,55℃ 30 s,72℃ 30 s,35个循环;72℃ 5 min。
1.5.3 qRT-PCR验证
通过qRT-PCR验证、、、、、、、、、、、、、、和基因在互花米草叶、茎、地下茎和根部的表达量,同时验证、、、、、、、、、、和基因在ABA及干旱处理下的表达量。引物在Primer3 (http://primer3.ut.ee/)网站上设计,GC含量在40%~60%且值在55℃~65℃之间,序列如表1所示。每个qRT-PCR反应体系包含1 µL正反向引物(10 µmol/L)、1 µL cDNA模板、7 µL RNAse-free水和10 µL SYBRGreen Master Mix(莫纳生物,RN04002M)。反应程序为95℃ 5 min;95℃ 10 s,60℃ 10 s,72℃ 30 s,40个循环。样本之间的表达量差异使用-test进行计算,值小于0.01定义为具有显著性差异。
2 结果与分析
2.1 互花米草NAC转录因子家族鉴定
为了从转录组数据库中鉴定出含有功能域的NAC转录因子,本研究首先对复合序列比对产生的所有可能NAC蛋白进行保守功能域分析。CDD (Conserved Domain Database)预测结果显示,62个SaNAC蛋白在N端均含有NAM结构域(附图1)。NAM是NAC家族蛋白特有的结构域(NAM、ATAF1/2和CUC2)之一,也是NAC转录因子能够发挥结合功能的关键区域,预测结果表明62个SaNAC蛋白均是具有潜在NAC功能的转录因子。
根据三代全长转录组数据对所有互花米草基因的命名[23],按从小到大的顺序将互花米草NAC蛋白命名为SaNAC1~SaNAC62,然后根据氨基酸序列对所有SaNAC蛋白的基础特征包括氨基酸数量、蛋白分子重量、等电点以及蛋白定位进行预测(表2)。通过分析发现,SaNAC蛋白大小介于186~ 858个氨基酸之间,蛋白分子量则介于21.2~93.2 kDa之间,其中SaNAC6和SaNAC15分别对应最大和最小的蛋白。所有SaNAC蛋白的等电点在4.3~ 10.7之间,SaNAC47和SaNAC2蛋白分别具有最大和最小的等电点。蛋白定位预测结果显示大多数SaNAC蛋白定位在细胞核,少数蛋白定位在细胞外(表2)。
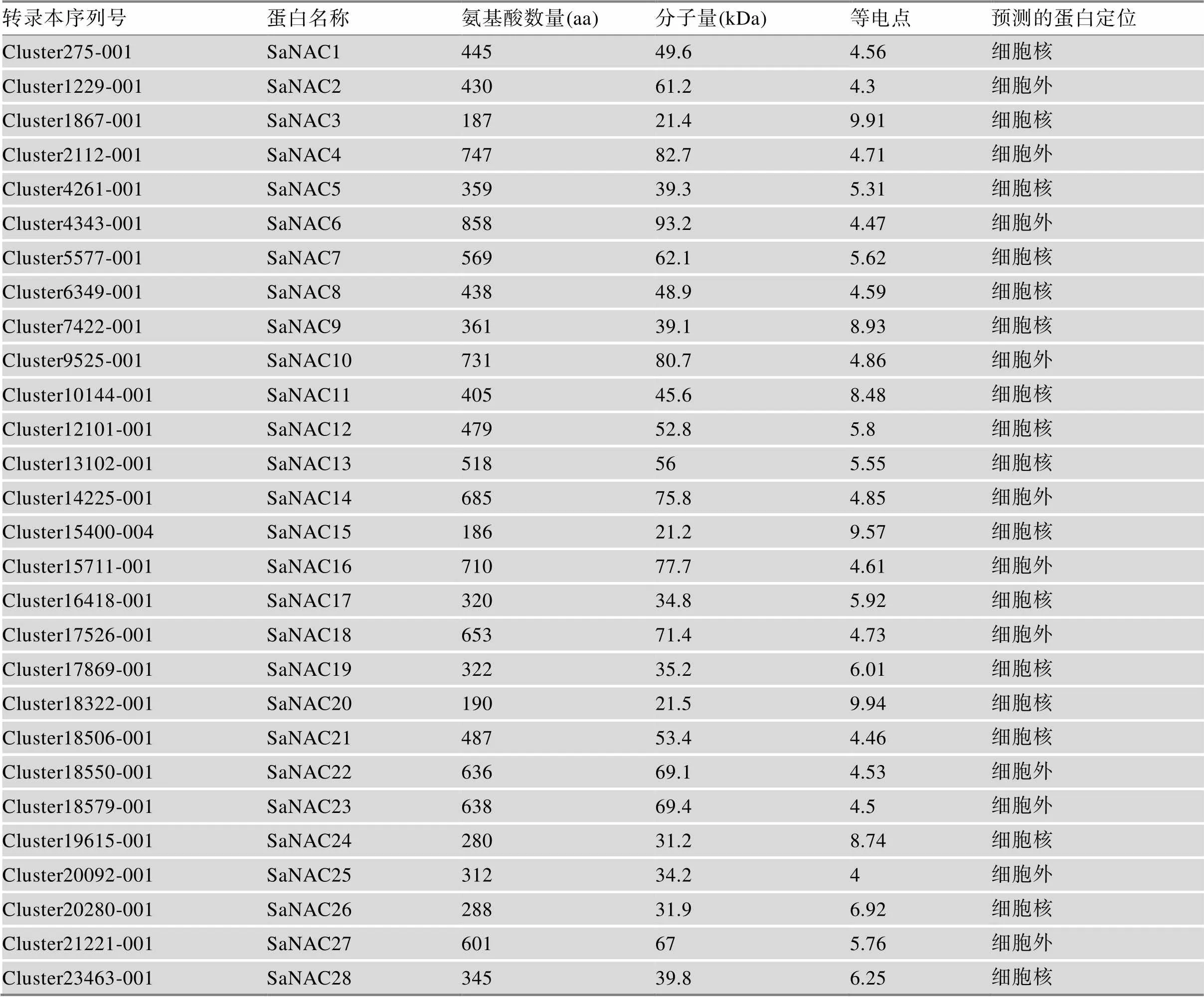
表2 互花米草NAC蛋白基本信息
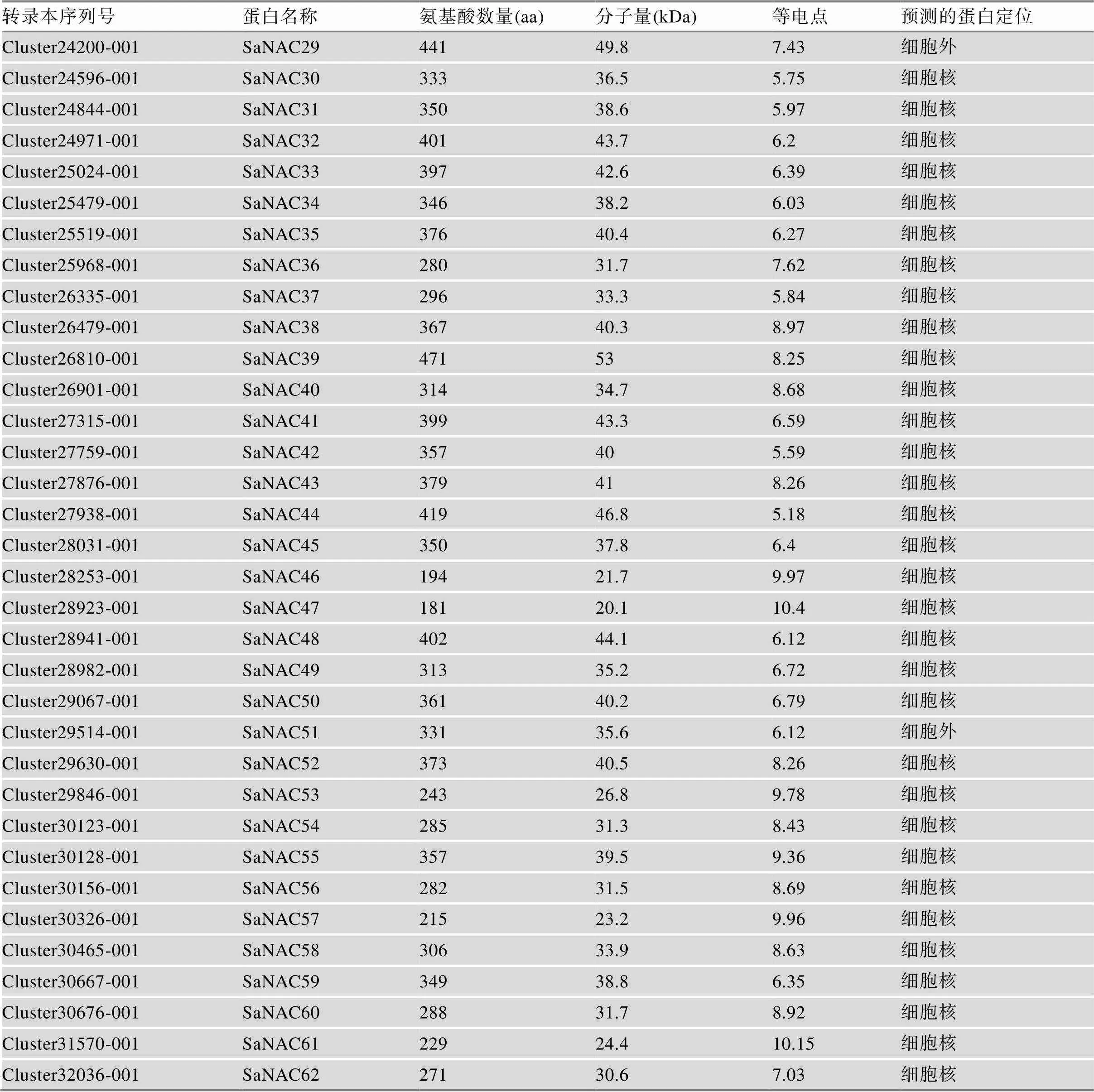
续表
2.2 蛋白序列比对及功能域分析
复合序列比对分析结果显示,所有的互花米草NAC蛋白在N端都具有保守的NAC结构域(图1),这与拟南芥和水稻的报道结果相一致[5]。除了少数基因具有不完整的NAC结构域外,大多数基因具有5个保守的亚结构域(A~E)。其中,蛋白SaNAC2、SaNAC3、SaNAC13和SaNAC60缺少A、B结构域;SaNAC58缺少B、C结构域;SaNAC25缺少A、B和C结构域;SaNAC46、47、51和53缺少E结构域。蛋白序列比对结果进一步证明了SaNAC家族蛋白在N端具有保守的NAM功能域(A~D),表明SaNAC转录因子可能与其他植物NAC蛋白功能相似。
为了研究互花米草NAC转录因子之间的进化关系,本研究利用62个SaNAC蛋白序列构建NJ进化树。通过进化树分析将互花米草NAC家族分成9个小组,分别命名为a~I (图2)。其中小组e中包含最多的SaNAC蛋白,有12个,小组b中的SaNAC蛋白最少,只有2个,分别是SaNAC4和SaNAC10。为了进一步探究NAC蛋白功能域之间的关系,对所有的SaNAC蛋白序列进行motif分析(MEME),通过预测最终获得10个保守的motif (motif 1~10) (图2)。分析motif与蛋白结构域,发现motif 3、4、1、7、2和5分别对应SaNAC转录因子N端5个保守的亚结构A~D,其中motif 2和motif 7共同组成结构域D。另外,SaNAC11、29、36、38、40和58蛋白缺少motif 1和4,但是都拥有motif 6。Motif 8则主要存在于小组d和h中,而motif 9则出现在一些SaNAC蛋白的C端。结合小组分类分析motif,发现小组g中的全部NAC蛋白均具有不完整的NAC亚结构,而小组a、b、d里的NAC蛋白亚结构最为完整,其他小组里都有个别基因缺少某些亚结构(图2)。
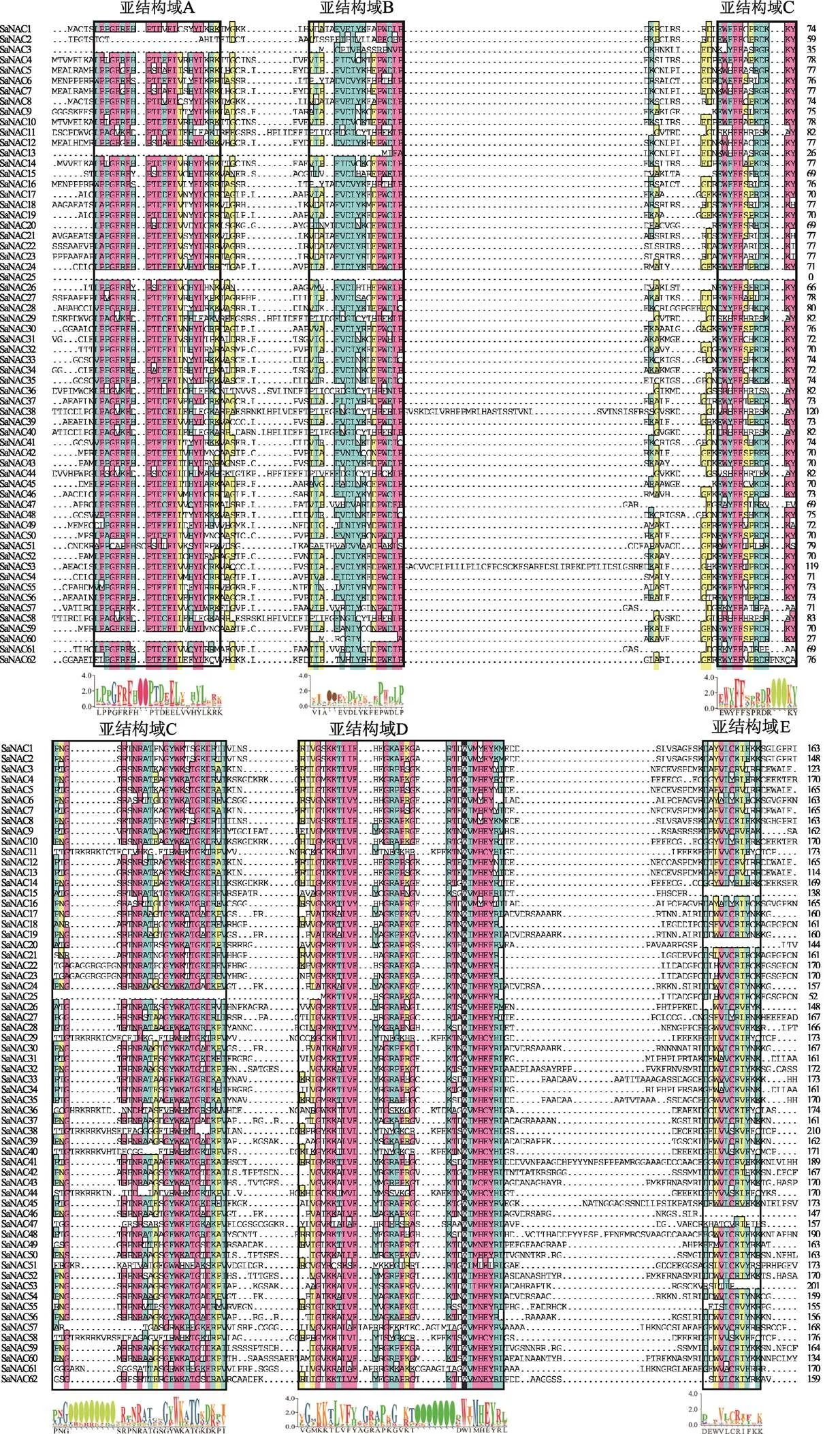
图1 互花米草NAC蛋白序列比对
黑色线框圈出的位置代表不同的亚结构域A~E,不同颜色代表序列之间的相似性程度(黑色>红色>绿色>黄色),线框下方的Logo代表对应亚结构域的保守序列。
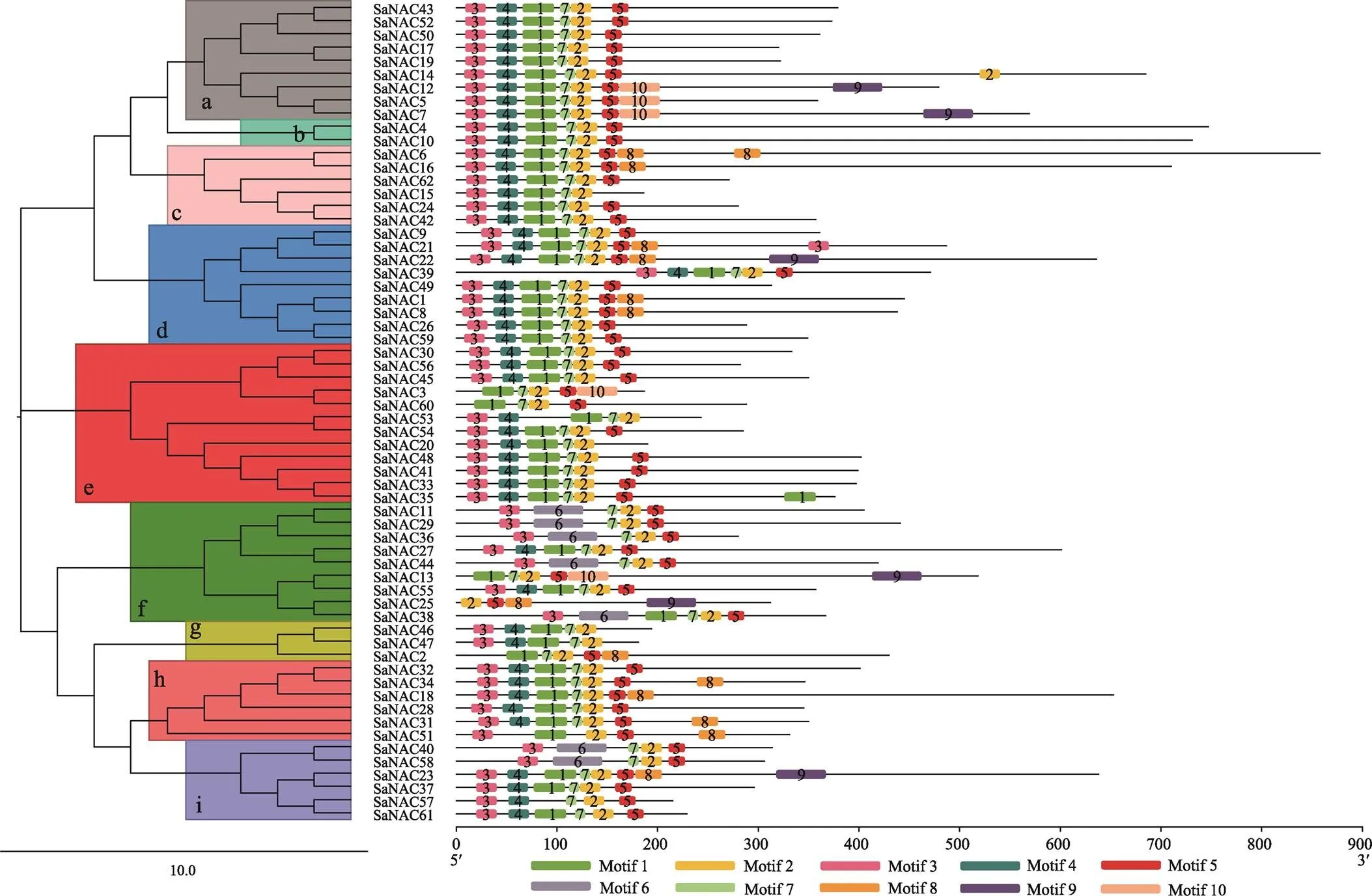
图2 互花米草NAC家族进化及MEME分析
左侧为SaNAC家族NJ进化树,a~i代表进化树不同的小组;右侧为SaNAC蛋白MEME预测结果,1~10代表不同的motif。
2.3 互花米草与水稻NAC家族进化分析
为了探究互花米草与水稻NAC家族成员之间的进化关系,本研究利用MEGA7软件构建互花米草与水稻NAC蛋白NJ进化树(图3)。结果显示,所有NAC蛋白被分为12个小组(Ⅰ~Ⅻ),其中小组Ⅰ中蛋白数量最多,包含11个SaNAC和18个OsNAC蛋白,小组Ⅲ中蛋白数量最少,不包含SaNAC蛋白。小组Ⅷ和Ⅺ中也只包含1个(SaNAC9)及2个(SaNAC36和SaNAC44) SaNAC蛋白,说明互花米草基因组在长期的进化过程中可能丢失了一些基因。但是,几乎所有小组都同时包含互花米草和水稻NAC蛋白,表明两个物种的NAC家族在进化水平上具有一定的相似性。
2.4 互花米草NAC蛋白亚细胞定位
蛋白定位预测结果表明大多数互花米草NAC转录因子定位在细胞核中(表1)。为了进一步验证SaNAC蛋白的表达定位,本研究将和基因全长CDS序列克隆到植物表达载体pCambia3301中,并利用烟草瞬时转化系统将目的载体和空载体对照同时转入烟草叶片表皮中。通过对SaNAC蛋白表达GFP信号的观察以及烟草叶片细胞核的染色,可以清楚的发现SaNAC9和SaNAC49蛋白在烟草表皮细胞的细胞核中表达(图4)。
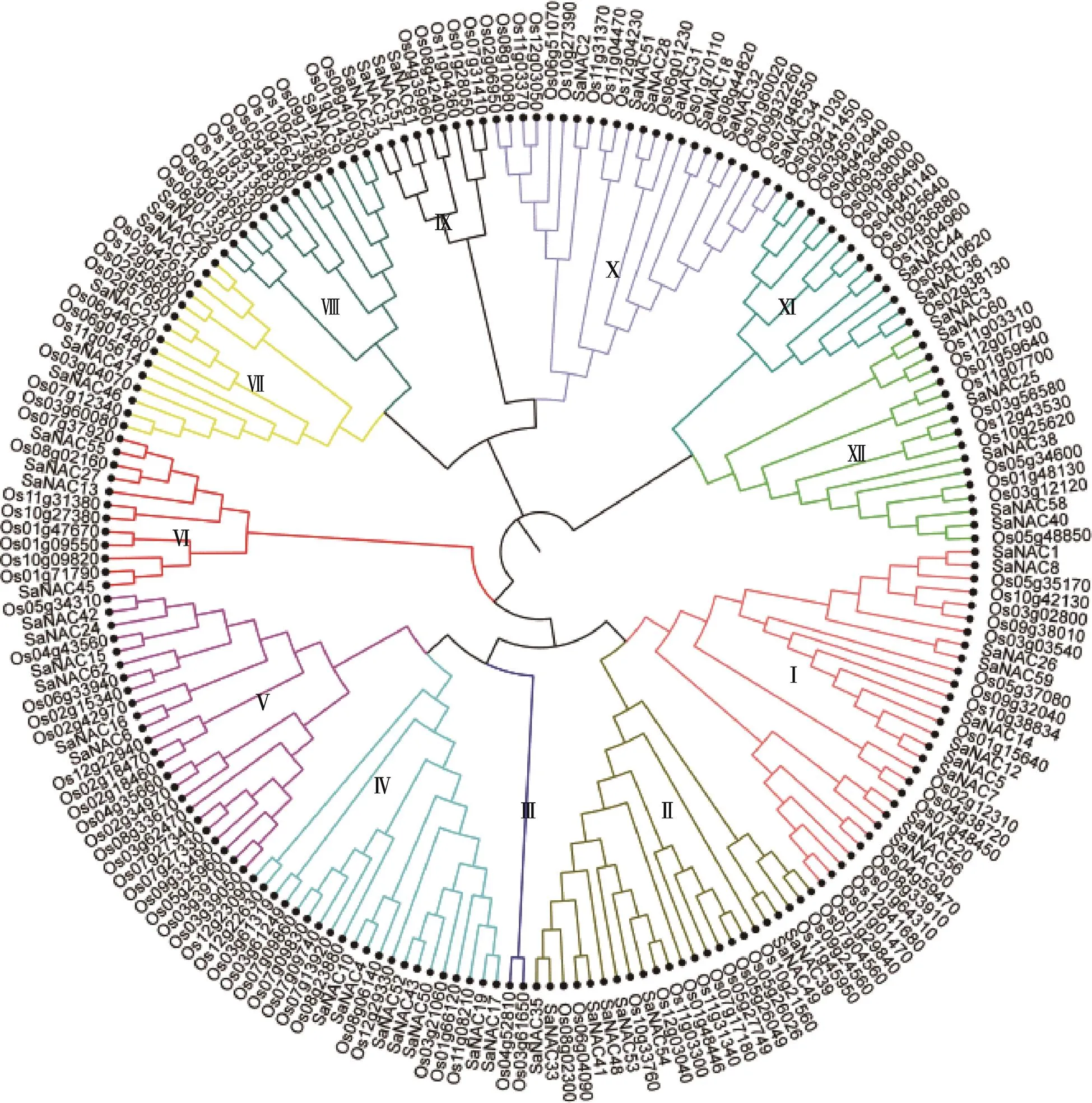
图3 互花米草与水稻NAC家族系统进化树
Ⅰ~Ⅻ分别代表进化树的不同小组。
2.5 互花米草NAC基因在不同组织中的表达特征
为了研究基因在互花米草不同组织中的表达情况,本研究随机挑选16个基因,并通过qRT-PCR检测它们在互花米草的叶、茎、根以及地下茎中的表达量。结果显示,这些基因在互花米草不同组织中的表达量各不相同,其中、和在叶片表达量最高,而和则在地下茎中有较高的表达量(图5)。另外,和在根部有较高的表达量,这些基因在茎中的表达量都相对较低(图5)。结果显示,基因的表达具有一定的组织差异性,表明基因可能与互花米草的生长发育有关。
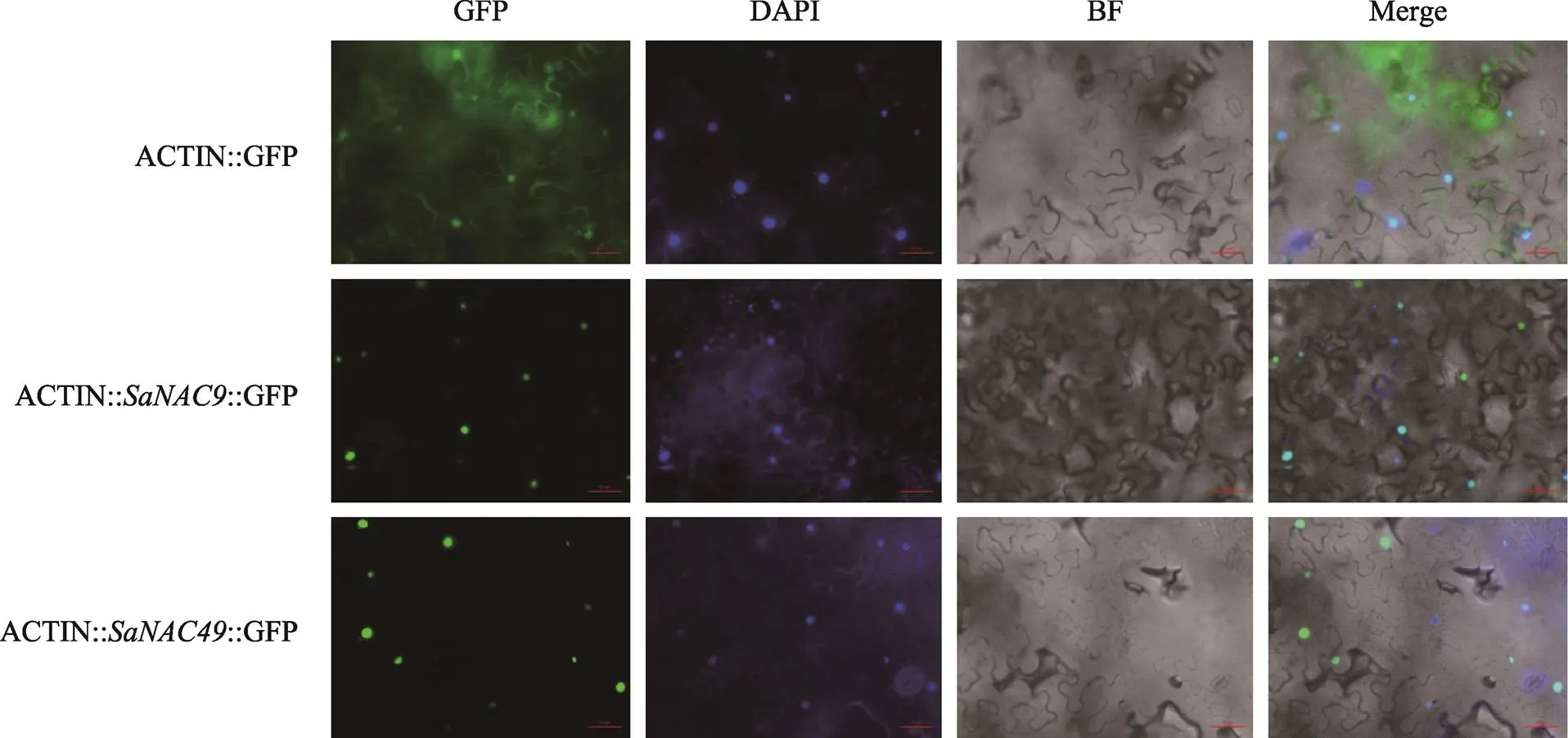
图4 互花米草NAC蛋白亚细胞定位
BF代表明场通道下的细胞,GFP和DAPI分别代表绿色荧光和细胞核信号,Merge代表BF、GFP和DAPI图像的重叠。所有图像在显微镜下放大倍数:20´。
2.6 互花米草NAC基因在盐胁迫下的表达特征
为了探究基因在盐胁迫下的表达特征,结合不同浓度(0、350、500以及800 mmol/L)NaCl处理下的互花米草RNA-seq测序数据[23],对所有基因的表达水平进行了系统分析。通过不同盐浓度处理下的表达热图可以看出,大部分基因在盐处理下发生了差异表达(图6A),说明盐胁迫可以调控基因的表达。其中和随着盐浓度的升高表达量持续降低(图6A),表明这些基因可能负向调控互花米草的盐胁迫响应。在800 mmol/L高盐处理时,与对照组相比,以及等25个基因表达量显著增加,基因的表达量上调最多,增加近60倍,表明高盐对基因具有显著调控作用。其中S以及等10个基因随着盐浓度的增加,表达量呈逐渐上升趋势(图6A)。另外,一些基因随着盐浓度的增加,表达量呈先升高后降低的趋势,例如以及等(图6A),表明这些基因虽然受盐胁迫的调控,但具有一定的浓度范围,一旦超过最适的浓度,则开始发生负向调控。为了证明RNA-seq数据分析的可信性,本研究挑选了、、、、、、、、、、和基因进行RT-PCR验证(图6B),实验结果与RNA-seq分析结果基本一致,进一步证明盐胁迫调控互花米草NAC家族基因的表达。
2.7 互花米草NAC基因在ABA处理下的表达特征
NAC家族除了调控植物响应盐胁迫,还参与多种胁迫的调控过程[12,13]。脱落酸(abscisic acid, ABA)信号通路已被确定为植物非生物胁迫反应的中枢调节因子,可以引起植物生理反应和基因的表达[26],为了探究互花米草基因在ABA胁迫下的表达变化,对12个随机的基因在ABA处理下的表达量进行检测。qRT-PCR结果显示,在ABA处理下,大部分检测的基因发生了表达量的改变(图7)。其中,及的表达随着ABA处理时间的增加逐渐降低,、、及则在ABA处理下呈现显著上调的趋势,这些基因中只有的表达水平没有受到ABA的影响(图7),表明基因的表达受到ABA的调控。
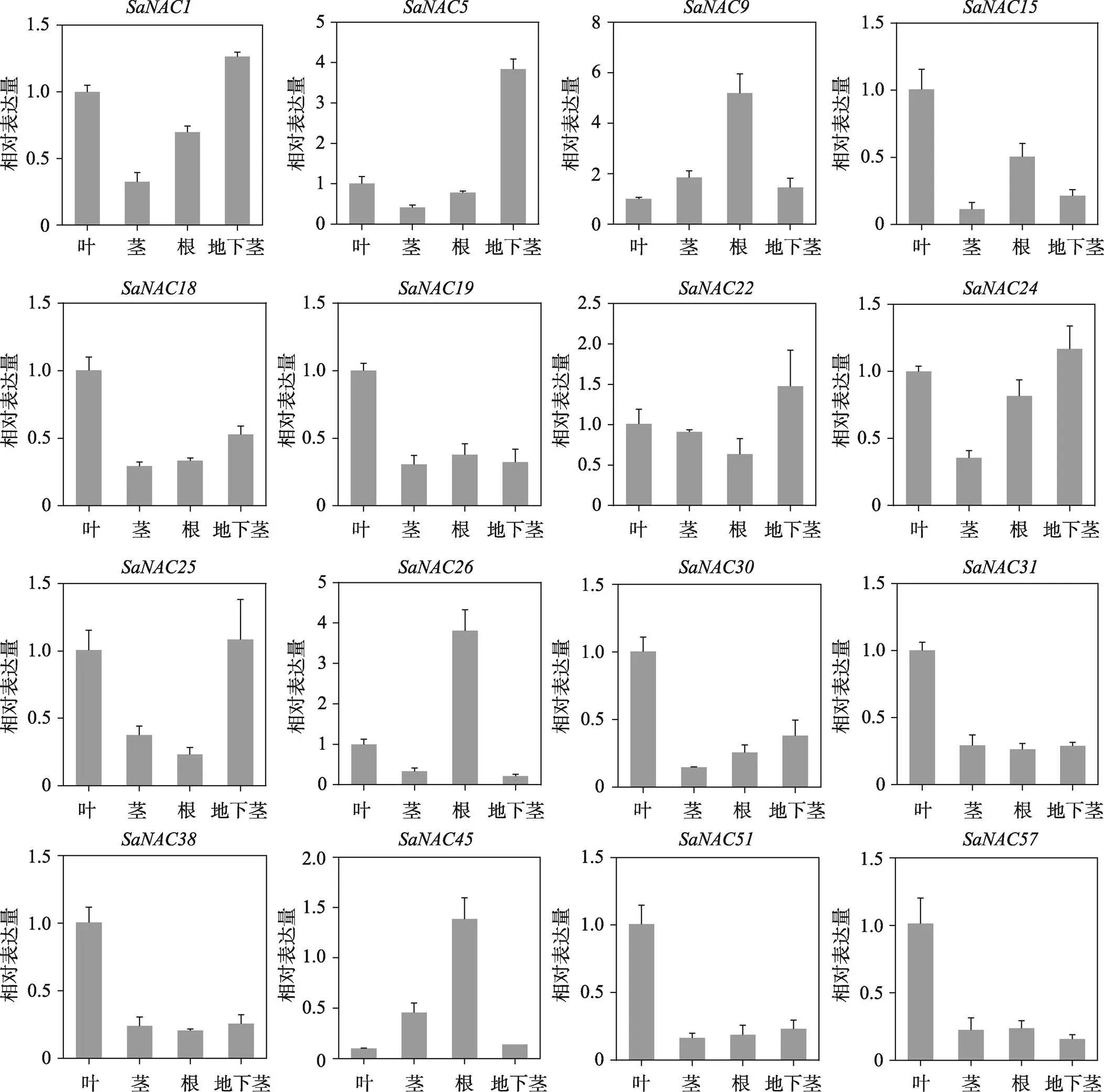
图5 16个SaNAC基因组织表达特征
每个样品取3个生物学重复,叶片的表达量被均一化成1。
2.8 互花米草NAC基因在干旱处理下的表达特征
与ABA胁迫类似,植物对干旱胁迫的应答同样是一个复杂的调控过程[27]。研究表明水稻ONAC066和ONAC095蛋白都参与干旱胁迫的调控[28,29],大豆NAC家族因子在抗旱过程中也具有重要作用[30],表明NAC蛋白是植物干旱胁迫响应过程中的重要调控子。本研究对12个基因在干旱胁迫下的基因表达进行qRT-PCR验证,结果显示,与ABA胁迫类似,干旱胁迫促进大多数基因发生差异表达(图8)。其中和在干旱处理时表达量均呈显著上调趋势,且除在干旱胁迫下表现出显著下调的趋势,其中和基因的表达量在1 h干旱处理时先降至最低,然后随着处理时间的增加开始逐渐增加(图8)。这些结果表明干旱胁迫同样可以调控基因的表达。外,其他基因的表达量都表现出先上升后下降的趋势。和
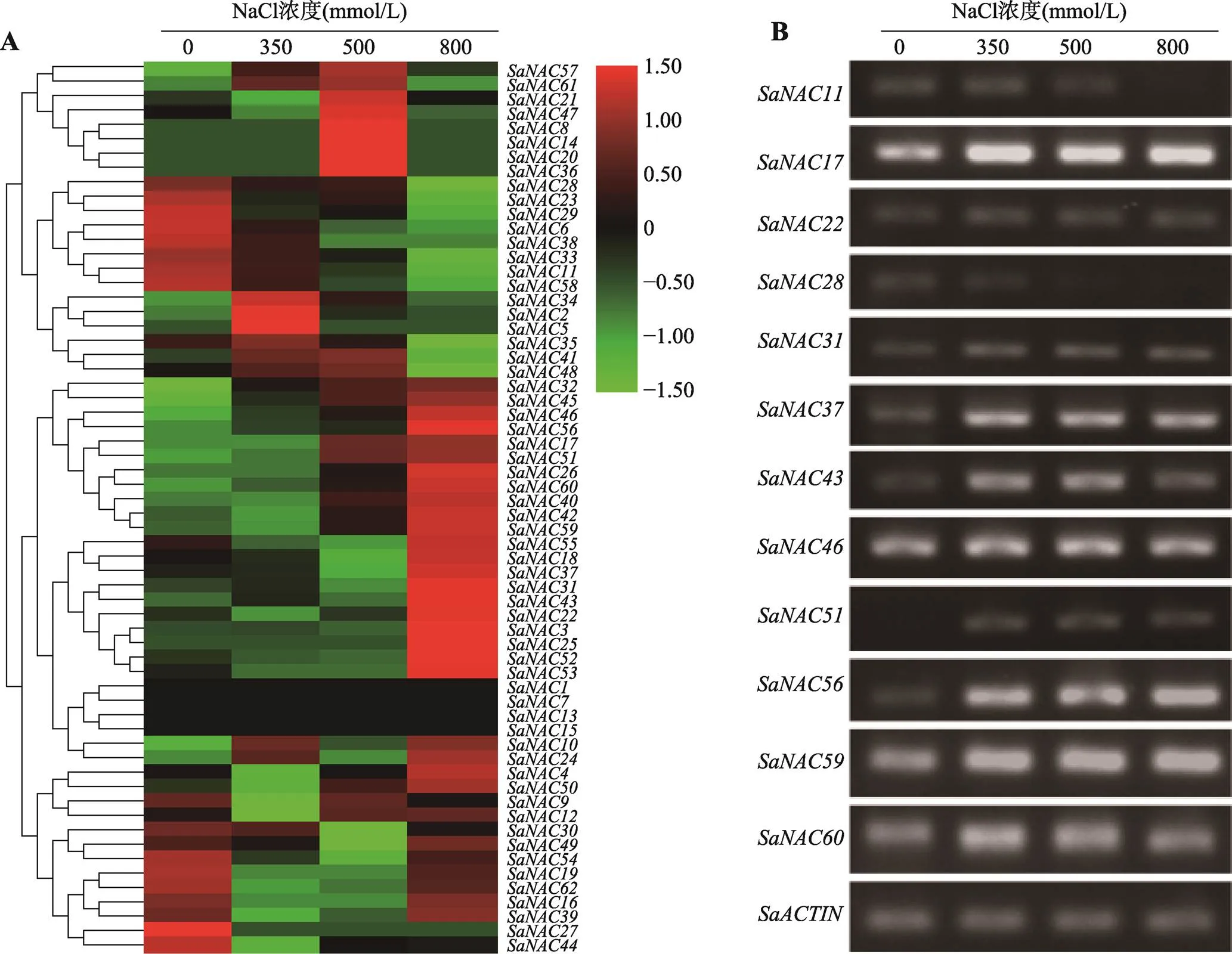
图6 SaNAC基因在不同盐浓度下的表达特征
A:不同浓度盐胁迫下所有基因的表达热图。表达量的值经过均一化后呈现在热图的右边,红色代表高表达,绿色代表低表达。B:12个盐胁迫应答基因的RT-PCR验证作为RT-PCR扩增均一化的内参基因。
3 讨论
互花米草作为高度耐盐的盐生植物,在沿海地带具有良好的生长优势。NAC家族蛋白具有保守的结构域和功能域,对植物的生长发育以及非生物胁迫应答具有重要的作用[16,18,20]。本研究从互花米草全长转录组数据中鉴定出62个完整的NAC转录因子,明显少于拟南芥和水稻基因组中的数目[5]。三代测序技术虽然可以得到较长的基因序列,但受到文库构建和机器读取不确定性的影响,相比基因组测序存在基因测序不全的缺点,因此互花米草NAC家族成员的完整性还有待进一步确定和完善。
蛋白序列比对结果显示SaNAC家族蛋白在N端含有基本的NAC结构域,除了SaNAC2、SaNAC3和SaNAC13等一些蛋白缺少个别亚结构域(A~E)外,绝大多数SaNAC蛋白都含有完整的NAC结构。NAC蛋白结构可以分为两个部分:N端保守的DNA结合区域(BD)和C端变化的转录激活区域(TR)[5],其中BD包含150~160个氨基酸残基,主要被分为A~E五个亚结构域,与DNA结合、同源或异源二聚体的形成以及核定位具有密切联系。保守功能域预测结果显示SaNAC蛋白均含有NAM功能域,NAM区域虽然只包含A~D共4个NAC亚结构域,但结构域E却是AtNAM蛋白一个重要的DNA结合区域[31],因此可以用NAM结构域来替代分析NAC结构域。同时,亚细胞定位实验也证明SaNAC9和SaNAC49在细胞核里表达,这些分析结果共同表明SaNAC蛋白可能发挥植物NAC转录因子的功能。

图7 ABA处理下12个SaNAC基因的表达特征
每个时间点的样本取3个生物学重复,处理0 h的样本表达量被均一化为1,星号代表ABA处理1 h、8 h或者24 h的样本与0 h样本之间具有差异。*表示<0.05,样本之间具有统计学差异;**表示<0.01,样本之间具有显著性差异;***表示<0.001,****表示<0.0001,均代表样本之间具有极显著差异。
在进化分组上共同位于某一小组的蛋白一般具有相似程度较高的序列,因此可能发挥相似的蛋白功能。互花米草与水稻同为单子叶植物,在结构上具有一定的相似之处,而且研究表明互花米草基因与水稻具有90%以上的相似度[32],因此分析它们NAC家族蛋白之间的进化保守性(图3),有助于更好的研究互花米草NAC蛋白的功能。系统进化树分析结果显示互花米草与水稻NAC蛋白相似程度较高,因此可以通过分析水稻NAC蛋白来预测互花米草NAC同源蛋白的功能。水稻ONAC095 (Os06g51070)被证明参与干旱和冷胁迫的响应,在干旱和ABA处理下,基因表达量增加,通过显性嵌合抑制因子抑制基因的表达后减少了水稻体内水分的丢失并且增加了脯氨酸和可溶性糖的含量,同时使一些干旱相关基因的表达发生上调,从而促进水稻抗干旱胁迫[29]。互花米草SaNAC2、SaNAC18、SaNAC28、SaNAC31、SaNAC32、SaNAC34及SaNAC51与OsNAC095蛋白共同位于X小组,研究发现、和基因在干旱处理下的表达量同样增加,因此猜测这些蛋白可能同样参与互花米草对干旱胁迫的调控。
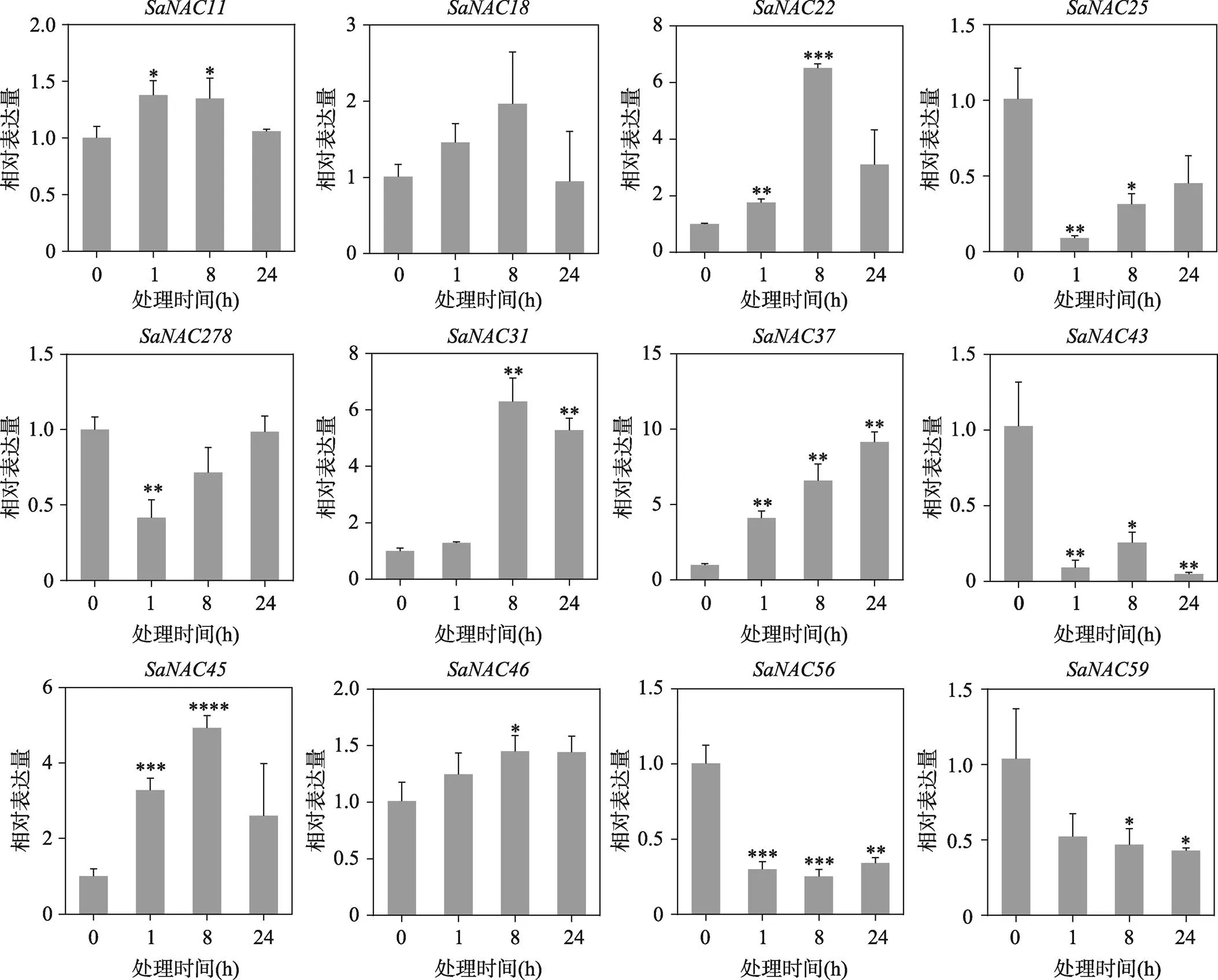
图8 干旱处理下12个SaNAC基因的表达特征
每个时间点的样本取3个生物学重复,处理0 h的样本表达量被均一化为1,星号代表干旱处理1 h、8 h或者24 h的样本与0 h样本之间具有显著性差异。*表示<0.05,样本之间具有统计学差异;**表示<0.01,样本之间具有显著性差异;***表示<0.001,****表示<0.0001,均代表样本之间具有极显著差异。
组织表达差异是基因选择性表达的结果,实验结果表明基因在互花米草叶、茎、根以及地下茎中的表达具有明显差异。基因在拟南芥里的同源基因编码一种转录激活因子,在干旱诱导的叶片衰老过程中,通过与ROS生物合成酶基因的启动子直接结合来促进ROS的产生[33]。互花米草能够生长在潮水涨落频繁的海岸带,主要原因之一是具有发达的地下根茎系统,地下茎的横向扩张也是互花米草繁殖的主要途径之一。本研究结果显示基因在互花米草地下茎中的表达量相对较高,同时在干旱处理下表达量也显著上调,因此猜测基因参与的干旱应答过程可能在互花米草地下茎中进行。在拟南芥的同源基因,参与调控拟南芥韧皮部薄壁转移细胞壁的生长[34],的同源基因,也影响拟南芥纤维次生细胞壁发育以及增加纤维细胞面积相关基因的表达[35],和基因在互花米草叶片中具有较高的表达,却在茎秆部分表达量较低,表明它们很可能同样参与调控互花米草器官的生长发育。另外,叶片中含有盐腺是互花米草耐盐的特点之一,根部吸收的盐大多可以由盐腺排出体外,因此叶片在互花米草的耐盐过程中也极为重要[36]。在研究红树林的盐腺时,发现相比叶肉组织,在富含盐腺的组织中红树林基因具有更高的表达量[37],说明NAC家族因子可能参与调控盐腺器官的发育。因此,大量基因在互花米草叶片中的高度表达,可能与盐腺的发育以及耐盐的调控有关。
为了系统的研究非生物胁迫对基因表达的调控,本研究同时分析了盐、ABA以及干旱胁迫对基因表达的影响。耐盐是互花米草最显著的特征,找出耐盐相关基因对研究其机理具有重要意义。非生物胁迫的控制机制依赖于大量胁迫相关基因的激活和调控[38],通过RNA-seq数据和qRT- PCR分析发现和基因同时受到高盐、ABA和干旱胁迫的调控,基因的表达量在3种胁迫下均显著增加,表明这些基因都是潜在的胁迫相关基因,在非生物胁迫的调控过程中可能扮演重要的角色。其中,的同源基因参与调控拟南芥对干旱胁迫的应答[33],的同源基因调控拟南芥细胞的衰老[39],而同源基因则可以负向调控ABA信号通路[40],充分显示基因在互花米草组织发育和对非生物胁迫应答方面的重要作用,同时也表明互花米草对非生物胁迫的应答是一个多基因复杂调控的过程。本研究丰富了互花米草NAC家族转录因子的信息,不仅为更好的研究互花米草生长发育和胁迫响应机制提供一定的参考,同时也为农林作物耐盐改良提供良好的基因资源。
附录:
附图1详见文章电子版www.chinagene.cn。
[1] Zhu JK. Abiotic stress signaling and responses in plants., 2016, 167(2): 313–324.
[2] Anumalla M, Roychowdhury R, Geda CK, Bharathkumar S, Goutam KD, Dev TSSM. Mechanism of stress signal transduction and involvement of stress inducible transcription factors and genes in response to abiotic stresses in plants., 2016, 7(8): 12754–12771.
[3] Khan SA, Li MZ, Wang SM, Yin HJ. Revisiting the role of plant transcription factors in the battle against abiotic stress., 2018, 19(6): E1634.
[4] Aida M, Ishida T, Fukaki H, Fujisawa H, Tasaka M. Genes involved in organ separation in Arabidopsis: an analysis of the cup-shaped cotyledon mutant., 1997, 9(6): 841–857.
[5] Ooka H, Satoh K, Doi K, Nagata T, Otomo Y, Murakami K, Matsubara K, Osato N, Kawai J, Carninci P, Hayashizaki Y, Suzuki K, Kojima K, Takahara Y, Yamamoto K, Kikuchi S. Comprehensive analysis of NAC family genes inand., 2003, 10(6): 239–247.
[6] Sablowski RW, Meyerowitz EM. A homolog of NO APICAL MERISTEM is an immediate target of the floral homeotic genes APETALA3/PISTILLATA., 1998, 92(1): 93–103.
[7] Wang TZ, Liu M, Zhao MG, Chen R, Zhang WH. Identification and characterization of long non-coding RNAs involved in osmotic and salt stress inusing genome-wide high-throughput sequencing., 2015, 15: 131.
[8] Kim HJ, Nam HG, Lim PO. Regulatory network of NAC transcription factors in leaf senescence., 2016, 33: 48–56.
[9] Park J, Kim YS, Kim SG, Jung JH, Woo JC, Park CM. Integration of auxin and salt signals by the NAC transcription factor NTM2 during seed germination in., 2011, 156(2): 537–549.
[10] Sun LJ, Li DY, Zhang HJ, Song FM. Functions of NAC transcription factors in biotic and abiotic stress responses in plants., 2012, 34(8): 993–1002.孙利军, 李大勇, 张慧娟, 宋凤鸣. NAC转录因子在植物抗病和抗非生物胁迫反应中的作用. 遗传, 2012, 34(8): 993–1002.
[11] Mao XG, Chen SS, Li A, Zhai CC, Jing RL. Novel NAC transcription factor TaNAC67 confers enhanced multi-abiotic stress tolerances in., 2014, 9(1): e84359.
[12] Mao XG, Zhang HY, Qian XY, Li A, Zhao GY, Jing RL. TaNAC2, a NAC-type wheat transcription factor conferring enhanced multiple abiotic stress tolerances in., 2012, 63(8): 2933–2946.
[13] Zhang LN, Zhang LC, Xia C, Zhao GY, Jia JZ, Kong XY. The novel wheat transcription factor TaNAC47 enhances multiple abiotic stress tolerances in transgenic plants., 2015, 6: 1174.
[14] Wang LQ, Li Z, Lu MZ, Wang YC. ThNAC13, a NAC transcription factor from, confers salt and osmotic stress tolerance to transgenic Tamarix and., 2017, 8: 635.
[15] Nuruzzaman M, Manimekalai R, Sharoni AM, Satoh K, Kondoh H, Ooka H, Kikuchi S. Genome-wide analysis of NAC transcription factor family in rice., 2010, 465(1–2): 30–44.
[16] Sun H, Hu ML, Li JY, Chen L, Li M, Zhang SQ, Zhang XL, Yang XY. Comprehensive analysis of NAC transcription factors uncovers their roles during fiber development and stress response in cotton., 2018, 18(1): 150.
[17] Ma JH, Tong DD, Zhang WL, Zhang DJ, Shao Y, Yang Y, Jiang L. Identification and analysis of the NAC transcription factor family in., 2016, 38(3): 243–253.马建辉, 仝豆豆, 张文利, 张黛静, 邵云, 杨云, 姜丽娜. 乌拉尔图小麦NAC转录因子的筛选与分析. 遗传, 2016, 38(3): 243–253.
[18] Gong X, Zhao LY, Song XF, Lin ZK, Gu BJ, Yan JX, Zhang SL, Tao ST, Huang XS. Genome-wide analyses and expression patterns under abiotic stress of NAC transcription factors in white pear ()., 2019, 19(1): 161.
[19] Pascual MB, Cánovas FM, Ávila C. The NAC transcription factor family in maritime pine (): molecular regulation of two genes involved in stress responses., 2015, 15: 254.
[20] Zhuo XK, Zheng TC, Zhang ZY, Zhang YC, Jiang LB, Ahmad S, Sun DL, Wang J, Cheng TR, Zhang QX. Genome-wide analysis of the NAC transcription factor gene family reveals differential expression patterns and cold-stress responses in the woody plant., 2018, 9(10): 494.
[21] Rhoads A, Au KF. PacBio sequencing and its applications., 2015, 13(5): 278– 289.
[22] Karan R, Subudhi PK. Overexpression of an adenosine diphosphate-ribosylation factor gene from the halophytic grass Spartina alterniflora confers salinity and drought tolerance in transgenic., 2014, 33(2): 373–384.
[23] Ye WB, Wang TT, Wei W, Lou ST, Lan FX, Zhu S, Li QZ, Ji GL, Lin CT, Wu XH, Ma LY. The full-length transcriptome of Spartina alterniflora reveals the complexity of high salt tolerance in monocotyledonous halophyte., 2020,DOI:10.1093/pcp/pcaa013.
[24] Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2., 2012, 9(4): 357–359.
[25] Li B, Dewey CN. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome., 2011, 12: 323.
[26] Finkelstein R. Abscisic acid synthesis and response., 2013, 11: e0166.
[27] Todaka D, Shinozaki K, Yamaguchi-Shinozaki K. Recent advances in the dissection of drought-stress regulatory networks and strategies for development of drought- tolerant transgenic rice plants., 2015, 6: 84.
[28] Yuan X, Wang H, Cai JT, Bi Y, Li DY, Song FM. Rice NAC transcription factor ONAC066 functions as a positive regulator of drought and oxidative stress response., 2019, 19(1): 278.
[29] Huang L, Hong YB, Zhang HJ, Li DY, Song FM. Rice NAC transcription factor ONAC095 plays opposite roles in drought and cold stress tolerance., 2016, 16(1): 203.
[30] Hussain RM, Ali M, Feng X, Li X. The essence of NAC gene family to the cultivation of drought-resistant soybean () cultivars., 2017, 17(1): 55.
[31] Duval M, Hsieh TF, Kim SY, Thomas TL. Molecular characterization of AtNAM: a member of the Arabidopsis NAC domain superfamily., 2002, 50(2): 237–248.
[32] Bedre R, Mangu VR, Srivastava S, Sanchez LE, Baisakh N. Transcriptome analysis of smooth cordgrass (), a monocot halophyte, reveals candidate genes involved in its adaptation to salinity., 2016, 17(1): 657.
[33] Lee S, Seo PJ, Lee HJ, Park CM. A NAC transcription factor NTL4 promotes reactive oxygen species production during drought-induced leaf senescence in., 2012, 70(5): 831–844.
[34] Wu YZ, Hou JX, Yu F, Nguyen STT, Mccurdy DW. Transcript profiling Identifies NAC-domain genes involved in regulating wall ingrowth deposition in phloem parenchyma transfer cells of., 2018, 9: 341.
[35] Hussey SG, Mizrachi E, Spokevicius AV, Bossinger G, Berger DK, Myburg AA. SND2, a NAC transcription factor gene, regulates genes involved in secondary cell wall development infibres and increases fibre cell area in Eucalyptus., 2011, 11: 173.
[36] Skelding AD, Winterbotham J. The structure and development of the hydathodes of, 1939, 38(1): 69–79.
[37] Jyothi-Prakash PA, Mohanty B, Wijaya E, Lim TM, Lin Q, Loh CS, Kumar PP. Identification of salt gland-associated genes and characterization of a dehydrin from the salt secretor mangrove., 2014, 14: 291.
[38] Haak DC, Fukao T, Grene R, Hua Z, Ivanov R, Perrella G, Li S. Multilevel regulation of abiotic stress responses in plants., 2017, 8: 1564.
[39] Takasaki H, Maruyama K, Takahashi F, Fujita M, Yoshida T, Nakashima K, Myouga F, Toyooka K, Yamaguchi- Shinozaki K, Shinozaki K. SNAC-As, stress-responsive NAC transcription factors, mediate ABA-inducible leaf senescence., 2015, 84(6): 1114–1123.
[40] Liu YC, Sun J, Wu YR.ATAF1 enhances the tolerance to salt stress and ABA in transgenic rice., 2016, 129(5): 955–962.
Identification and expression analyses of the NAC transcription factor family in
Taotao Wang1,2, Yong Yang1,2, Wei Wei2, Chentao Lin2, Liuyin Ma2
As a coastal halophytehas high salt tolerance. However, the mechanism at the molecular level has not been widely studied due to the absence of a reference genome. The proteins of NAC families are plant-specific transcription factors that regulate the growth, development and stress response in plants. To identify the NAC family and explore the relationship between NACproteins and the growth, development and stress response of, full-length transcriptome data ofby the third generation sequencing technology was used as reference sequences in this study to blast with the NAC protein sequences from,and. Finally, 62 SaNAC proteins were found inby deep analysis on conserved domains. Then we analyzed sequence alignment, evolution, motif prediction, homology comparison, subcellular localization, tissue and abiotic stress-induced gene differential expression profile on the NAC family members in. As a result, all SaNAC proteins were found containing a conserved NAM domain and having certain evolutionary similarity with rice; two family proteins, SaNAC9 and SaNAC49, were expressed in the nucleus; moreover,genes were identified to have distinct expressional profiles in different tissues and stress response ofThese results indicated the SaNAC transcription factor family not only had conserved functional domains but also played important role in the regulation of growth, development and abiotic stress response.
; NAC; transcription factor; gene family; gene expression
2019-08-27;
2019-12-26
福建农林大学林学高峰学科建设项目(编号:71201800725,71201800773)和福建农林大学优秀博士学位论文基金项目(编号:324-1122yb043)资助[Supported by Peak Subject Construction Project of Fujian Agricultural and Forestry University (Nos. 71201800725, 71201800773) and Scientific Research Foundation of the Graduate School of Fujian Agriculture and Forestry University (No. 324-1122yb043)]
王涛涛,博士研究生,研究方向:林木遗传学理论基础。E-mail: fjnlwtt@163.com
马留银,博士,副教授,博士生导师,研究方向:植物转录后调控。E-mail: lma223@163.com
10.16288/j.yczz.19-250
2020/2/21 11:21:17
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200220.1501.001.html
(责任编委: 赵方庆)

