脱发相关差异表达基因的生物信息学分析
向虹,阳小胡,艾亮霞,潘燕平,胡勇
研究报告
脱发相关差异表达基因的生物信息学分析
向虹,阳小胡,艾亮霞,潘燕平,胡勇
中国科学院深圳先进技术研究院,合成生物研究所,深圳 518055
利用生物信息学方法分析脱发相关差异表达基因,有望帮助了解脱发发生发展的分子机制。本研究从NCBI的子数据库GEO中选择基因表达谱GSE45512和GSE45513数据集,利用R语言limma工具包,筛选出两个物种斑秃样本与正常样本的共同显著差异表达基因。对这部分基因进行功能注释和蛋白互作网络分析,同时对全部差异表达基因进行基因集富集分析。结果发现,人头皮斑秃样本共筛选出225个差异表达基因;C3H/HeJ小鼠自发斑秃皮肤样本共筛选出337个差异表达基因;两个物种的共同显著差异表达基因有23个。GO功能富集分析和蛋白互作网络分析显示,这部分差异基因显著富集于免疫相关功能,并且彼此间存在蛋白互作关系。基因集富集分析显示两个物种的差异基因都能显著富集到趋化因子信号通路、细胞因子受体相互作用、金葡菌感染及抗原加工与呈递通路;而且人的下调差异基因不仅映射到了人类表型数据库的脱发表型,也映射到皮肤附属物病理相关表型。综上所述,本研究通过生物信息方法分析脱发皮肤组织与正常皮肤组织的差异表达基因,最终筛选出23个在人和小鼠中共同存在的显著差异表达基因;此外,分析发现脱发与免疫过程及皮肤附属物病变密切相关,这些结果为脱发的诊断和治疗提供了新思路。
脱发;斑秃;GEO;生物信息学
斑秃(alopecia areata, AA)是一种常见的脱发症状,AA患者一般表现为斑秃部位无疤痕、斑片状脱发,并且头皮附近的毛干变窄。AA作为一种自身免疫性疾病,具有高度变异的表型和基因型,是由遗传和环境因素共同决定的复杂遗传病,终生患病率约为2%[1]。尽管对AA患者和小鼠模型的遗传学研究表明AA是一种复杂的多基因疾病,然而毛囊免疫豁免功能的破坏也被认为是AA形成的重要诱因[2]。AA传统上被分为斑片状斑秃(alopecia patchy)、全秃(alopecia totalis, AT)和普秃(alopecia universalis, AU)[2]。AA症状最常发生在头皮,但也可能发生在眉毛和睫毛(AT患者)或体毛(AU患者)。基于疾病的持续时间和斑状秃发的程度,AA还可被分为隐秃(alopecia incognita)、蛇形斑秃(ophiasis)、马蹄形斑秃(sisaipho)和玛丽·安托瓦内特综合征(Marie Antoinette syndrome)[2]。
AA也是一种对生活质量有重大影响的疾病。AA患者表现出较差的健康相关生活质量(Health- related quality of life, HRQoL)评分,头皮受累程度越大,HRQoL评分越低[3]。由于AA的可见性、缺乏治疗和慢性病的性质,AA及其衍生症状可能会给患者造成严重的心理负担,更会导致包括高度焦虑和抑郁在内的心理疾病,尤其是青少年患者更易引发社交障碍[4,5]。此外,有调查分析显示,年轻的男性患者具有更高的心理压力和自杀风险[6]。因此,有效的治疗方法将会对AA患者的生活质量和身心健康产生深远和积极的影响。脱发影响着全球数千万人,但目前缺乏一种安全的、合理的和有针对性的用于治疗头发再生的方法。现代常用的治疗方法包括局部使用米诺地尔(2%和5%),口服非那雄胺和植发手术等。由于存在药物的副作用、停药后复发、新毛囊存活率和患者个体差异等问题,对AA的治疗富有挑战性,并且治疗效果并不令人满意。
生物信息学是生命科学领域前沿交叉学科,可为脱发或斑秃的形成揭示潜在的分子机制,从而帮助阐明和理解AA独特的病理生理学。使用生物信息学技术对高通量数据进行联合分析不仅为科学研究提供可行的思路和方案,也为该类疾病的治疗提供更有针对性和合理选择的疗法。本文通过生物信息学相关方法对Xing等[7]构建的人类AA头皮表达谱芯片和C3H/HeJ小鼠自发AA皮肤表达谱芯片数据进行生物信息学分析,以探讨影响AA在人和小鼠中保守的差异表达基因以及由脱发引起的一系列生理病理过程的改变,为AA分子机制的进一步研究提供生物信息学依据。
1 材料与方法
1.1 数据来源
在美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)的基因表达综合数据库(Gene Expression Omnibus, GEO)下载GEO数据集,包括GSE45512和GSE45513[7]。其中,GSE45512是基于GPL570 Affymetrix Human Genome U133 Plus 2.0 Array的人AA皮肤表达谱,含有5例AA患者人头皮样本和5例健康人头皮样本,而GSE45513是基于GPL1261 Affymetrix Mouse Genome 430 2.0 Array的C3H/HeJ小鼠自发性AA皮肤表达谱,含有3例自发小鼠自发AA皮肤样本和3例正常小鼠皮肤样本。
1.2 基因芯片的处理与差异表达基因的筛选
芯片的处理使用R语言“affy”包对GSE45512和GSE45513数据中的每个样本探针表达值进行背景校正归一化处理。通过“limma”包对实验组AA皮肤和对照组正常皮肤中每个表达值进行检验。GSE45512差异表达基因使用Bioconductor的“hgu133plus2.db”进行注释,GSE45513差异表达基因使用Bioconductor的“mouse4302.db”进行注释。差异表达基因(differently expressed genes, DEGs)的筛选标准为|log2FC|≥1 &value≤0.05。利用venn图获得两个数据集中共同出现的差异表达基因名称。利用“pheatmap”工具包对差异表达基因绘制热图。
1.3 DEGs的注释和富集分析
采用Gene Ontology Resource数据库[8],对前面筛选出的两物种中共有的DEGs进行GO (Gene Ontology)富集分析和KEGG (Kyoto Encyclopedia of Gene and Genome)信号通路分析。显著性富集的筛选标准为<0.01和FDR<0.05。分析的结果利用R语言“REVIGO”进行可视化呈现。对两物种中单独存在的差异表达基因使用DAVID数据库[9]进行GO注释分析、KEGG通路富集分析和组织表达分析。
1.4 蛋白互作网络的构建
将前面筛选的DEGs导入STRING (version 11.0)数据库,得到一个蛋白质相互作用网络(protein- protein interaction, PPI)图。利用软件参数将免疫相关功能蛋白标注出。
1.5 基因富集分析
基因集富集分析(gene set enrichment analysis, GSEA)使用WEB-based GEne SeT AnaLysis Toolkit数据库。对两个物种校正后的基因集进行富集分析,Redundancy reduction参数设定为Weighted set cover。
2 结果与分析
2.1 获得DEGs
对GSE45512和GSE45513两个数据集进行分析,分别获得差异倍数在2倍以上的显著上、下调差异表达基因。GSE45512人头皮AA样本共筛选出225个DEGs (图1A),其中上调基因79个,下调基因146个(图2A)。GSE45513 C3H/HeJ小鼠自发AA皮肤样本共筛选出337个DEGs (图1B),其中上调基因280个,下调基因57个(图2A)。两个物种共有的DEGs有23个(图2A),并对这23个在人和小鼠中相对保守的DEGs进行聚类热图分析(图2,B和C)。可见两类样本基因表达具有显著差异,上调基因主要包括、、和等19个,下调基因有、、和(图1和附表1)。
2.2 DEGs的注释富集分析和蛋白互作分析
DEGs的GO富集分析可分为生物学过程(biological process)、细胞组分(cellular component)和分子功能(molecular function) 3个方面。对于两物种共有的DEGs,GO富集分析图显示,生物学过程主要富集在免疫相关的一些过程,包括免疫反应(immune response),免疫效应过程(immune effector process),防御反应(defense response),T细胞活化(T cell activation)和细胞因子介导的信号通路(cytokine-mediated signaling pathway)等(图3A)。这部分DEGs的细胞组分条目较少,主要涉及一些细胞表面的组分包括质膜外侧面(external side of plasma membrane)、膜侧(side of membrane)、细胞表面(cell surface)、免疫突触(immunological synapse)、细胞外隙(extracellular space)和BMP受体复合物(BMP receptor complex) (图3A)。而分子功能主要富集在细胞因子活性(cytokine activity)、受体结合(receptor binding)和内肽酶活性(endopeptidase activity)等过程(图3A)。
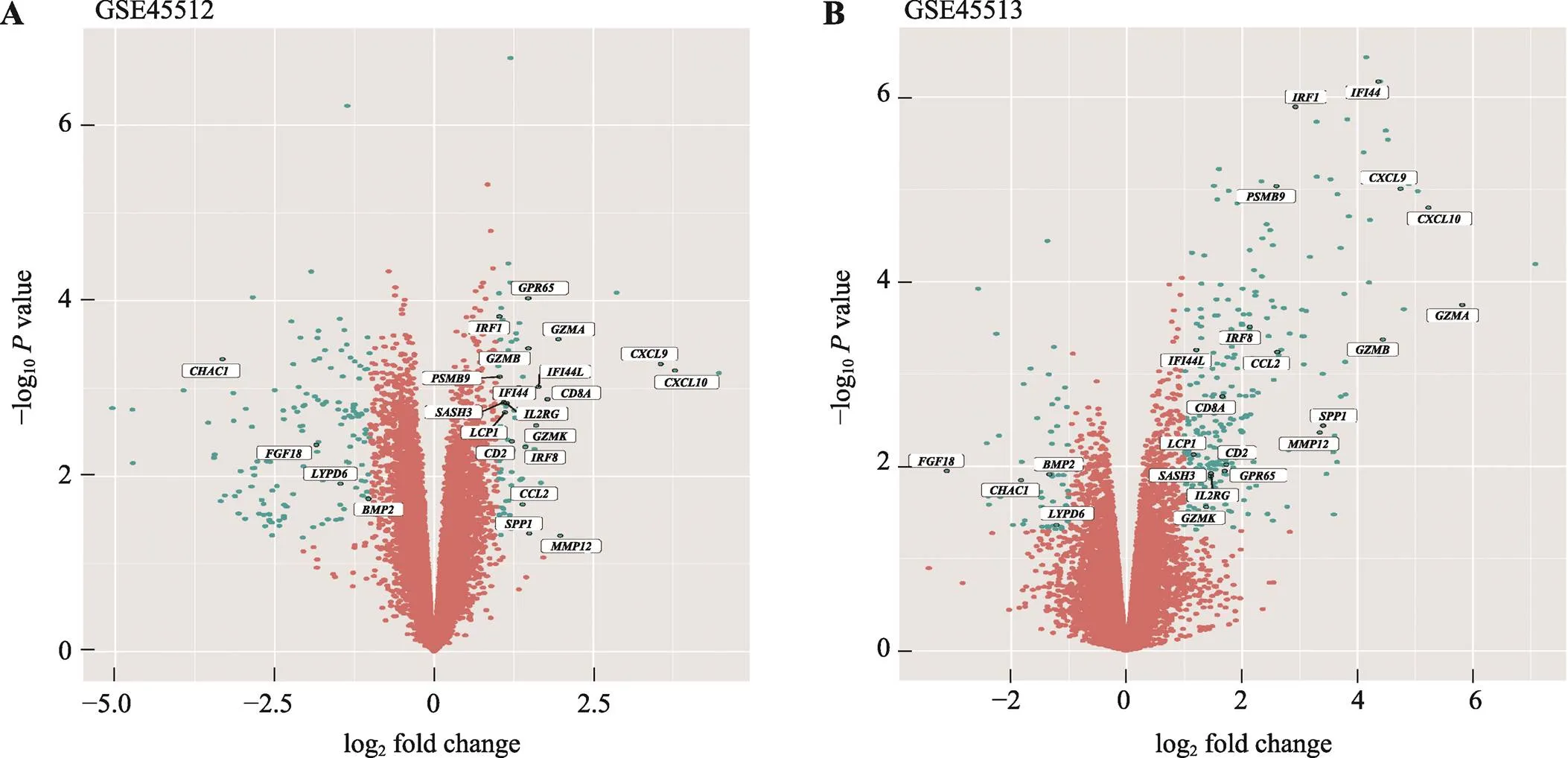
图1 差异表达基因火山图
A:GSE45512人头皮AA样本差异表达基因火山图;B:GSE45513 C3H/HeJ小鼠自发AA皮肤样本差异表达基因火山图。火山图横轴表示差异倍数,数值显示为log2fold change。纵轴表示值,数值显示为−log10value。深青色的点代表显著差异表达基因,玫瑰红色的点代表非显著差异表达基因。人和小鼠中共有的差异表达基因在图中显示基因名称。
对于两物种中单独存在的DEGs,GO富集分析显示,免疫相关的生物学过程仍然是两物种最主要的生物学过程,并且免疫反应(immune response)在人和小鼠中都出现了(附图1,A和D)。在细胞组分方面,小鼠的DEGs主要富集在膜(membrane)和细胞质(cytoplasm)范围较广的细胞组分,而人的DEGs主要富集在角蛋白纤维(keratin filament)专有的与皮肤附属物相关的组分。此外,胞外区(extracellular region)和细胞外隙(extracellular space)在人和小鼠中都出现了(附图1,A和D),这与前面提到的两物种共有DEGs富集在细胞表面的组分相关联。在分子功能方面,人的主要富集在结构分子功能相关的条目,小鼠的主要富集在结合功能相关的条目。在KEGG信号通路富集分析中,人和小鼠都出现了一个免疫相关的条目细胞因子受体相互作用(Cytokine-cytokine receptor interaction) (附图1,B和E)。然而,在组织富集方面,人的这些非共有DEGs主要富集在头皮、头发和发根,小鼠的主要富集在免疫器官中(胸腺和骨髓) (附图1,C和F)。
利用STRING数据库对在人和小鼠AA样本中共有的差异表达基因编码的蛋白分析发现,除了CHAC1、LYPD6和GPR65,其他20个蛋白之间都存在互作网络。结合之前的GO富集分析结果,发现处于该蛋白网络节点位置的蛋白都多与免疫相关(如:IRF1、CCL2、CXCL9、GZMA和GZMB等),如图3B所示标记出红色的蛋白表示参与免疫系统过程(GO:0002376, immune system process),标记出蓝色的蛋白表示参与免疫反应过程(GO:0006955, immune response)。说明AA症状在人和小鼠中都引起了一系列的免疫响应事件,如显著地增加了一些免疫相关基因和因子的表达。
2.3 DEGs的GSEA分析
对人头皮AA样本(GSE45512)的DEGs基因集和C3H/HeJ小鼠自发AA皮肤样本(GSE45513)的DEGs基因集分别进行信号通路富集分析后,发现上调或者下调基因都富集在一些信号通路上。如同共同出现在人和小鼠中的23个DEGs一样,两个物种的DEGs基因集也共同地富集在一些信号通路上(图4),包括:趋化因子信号通路(chemokine signaling pathway)、细胞因子受体相互作用(cytokine-cytokine receptor interaction)、金葡菌感染(infection)和抗原加工与呈递(antigen processing and presentation)。比较巧合的是,除了金葡菌感染(infection)这个条目外,其他3个条目与前面提到的23个DEGs一样,是与免疫学过程存在直接相关的。这也进一步表明了斑秃在人和小鼠中会触发一系列的免疫学事件。
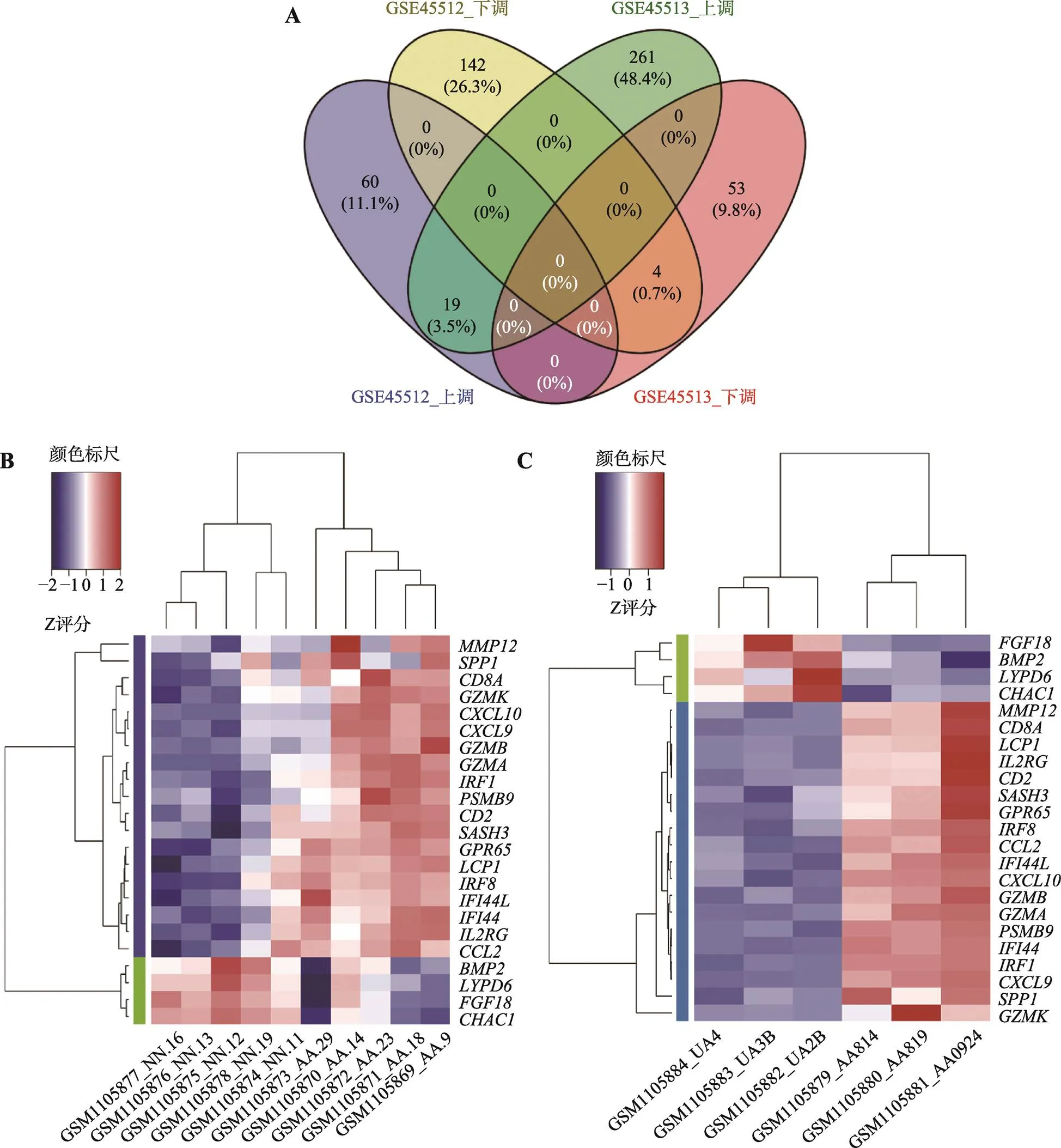
图2 人和小鼠AA样本中共同差异表达的基因
A:人和小鼠AA样本差异表达基因的韦恩图;B:人AA样本中共同差异表达基因的聚类热图;C:小鼠AA样本中共同差异表达基因的聚类热图。
将人头皮AA样本(GSE45512)的基因集富集到人类表型数据库发现,上调基因集富集的条目有自身免疫(autoimmunity)、反复细菌感染(recurrent bacterial infections)、中枢神经系统感染(CNS infection)、淋巴细胞形态异常(abnormal lymphocyte morphology)、血管炎(vasculitis)和咯血(hemoptysis)。下调基因集富集的条目有指甲营养不良(nail dystrophy)、发质异常(abnormality of hair texture)、脱发(alopecia)和睫毛稀疏或缺失(sparse or absent eyelashes)。有意思的是,上调的基因集仍然多富集在免疫、细菌感染和一些炎症表型,而下调的基因集都富集在脱发和皮肤附属物(指甲和睫毛)发育异常的表型上,这与本研究的样本表型相关,也与预期相符。
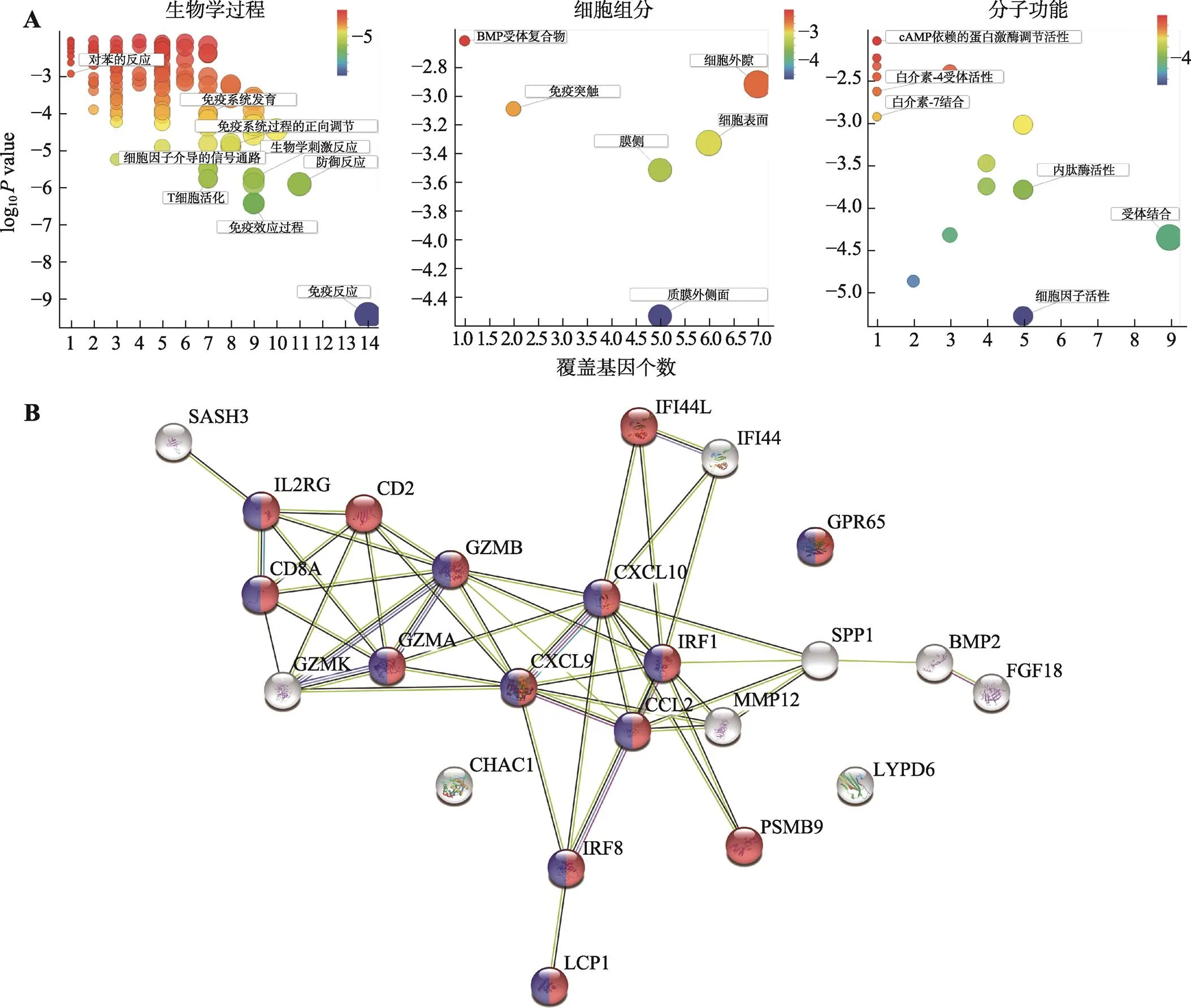
图3 差异表达基因的GO富集分析和蛋白互作分析
A:差异表达基因的GO富集分析,包括生物学过程、细胞组分和分子功能。纵坐标代表值,颜色变化从红色到蓝色代表值从高到低;横坐标代表包含的基因数,圆圈越大代表包含的基因数越多。B:差异表达基因的蛋白互作分析。红色代表参与免疫系统过程(GO:0002376),蓝色代表参与免疫反应过程(GO:0006955)。
3 讨论
脱发影响着全球数千万人。几十年来,对于脱发的治疗渐渐从广泛和非特异性疗法发展到现在更有针对性和合理选择的疗法。这需要通过临床医生、基础科学家和生物信息学家等多个领域之间的密切合作和交流来实现。关于脱发的治疗方法多种多样,包括植发手术[10]、低能量激光治疗[11]、光化学疗法(PUVA)[12]、针灸疗法[13]和药物治疗[14~16]等。然而,目前没有有效的疗法能医治所有受到脱发困扰的患者,且部分疗法除了具有较大的副作用外也会在停止治疗之后出现严重的复发情况,这主要与脱发独特的病理生理学和复杂的病因有关。因此,对于AA发生发展机制的研究特别重要,既可以帮助深入了解AA的发病机制,也有助于AA治疗药物的研发和治疗方法的开发。
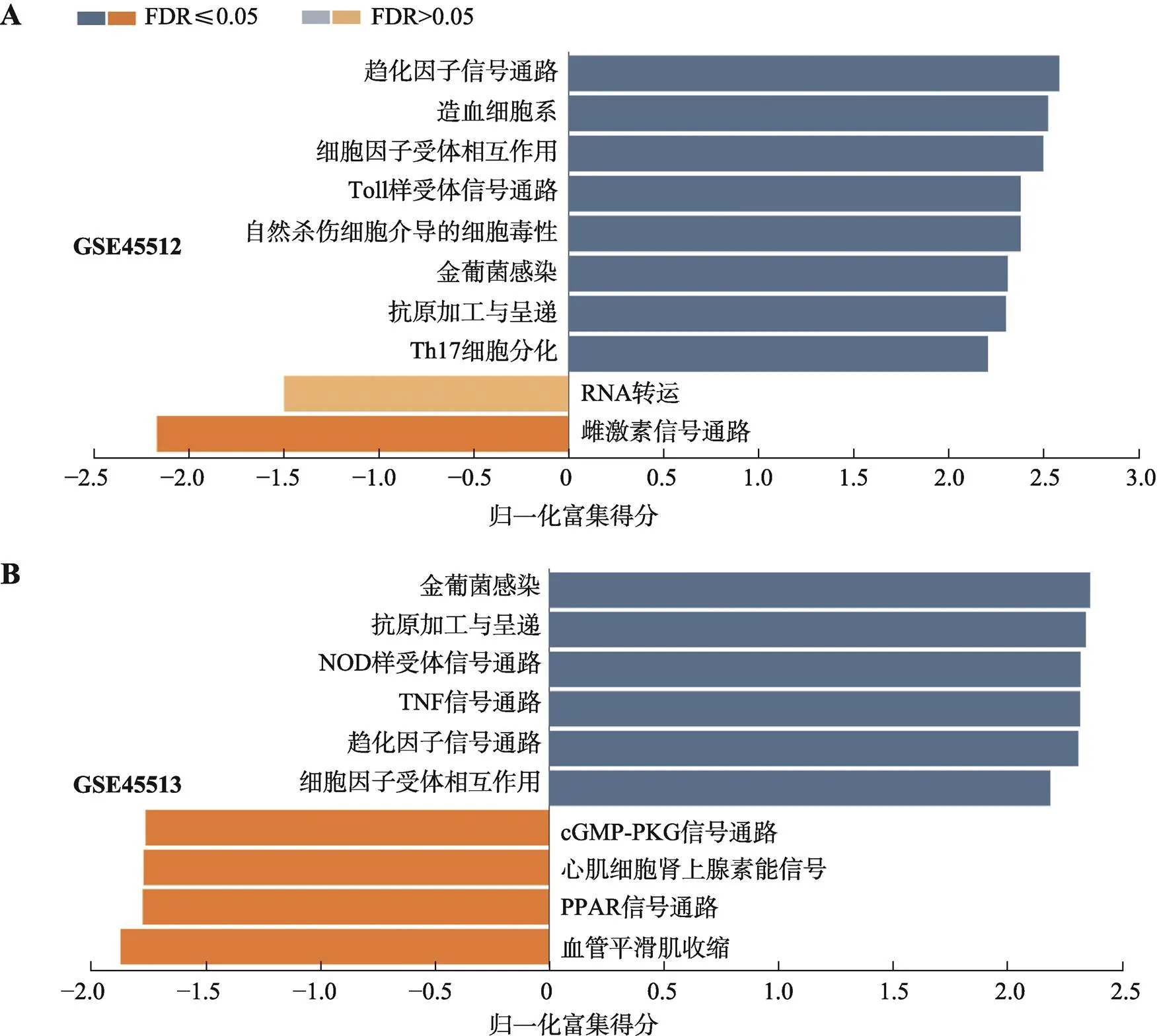
图4 参与信号通路的基因集富集分析
A:人斑秃样本(GSE45512)的基因集富集结果;B:小鼠斑秃样本(GSE45513)的基因集富集结果。
本研究通过生物信息学方法分析了人头皮AA样本和C3H/HeJ小鼠自发性AA皮肤样本,筛选出23个在两个物种AA样本与正常样本中共同显著差异表达基因。并且基于GO和STRING数据库对23个基因进行功能注释分析和蛋白互作网络分析。同时为了对所有数据集有一个全面的了解,进一步将校正后的差异表达基因集进行了基因集富集分析。除了人AA头皮样本数据,本研究也选用了C3H/HeJ小鼠自发性AA皮肤样本数据。C3H/HeJ小鼠自发脱发的发展过程和人的AA病理特征非常相似[17],因而能够作为一种良好、经济的模型用于研究脱发相关的疾病。本研究将两个物种的数据集联合分析,期望探究出在人和小鼠两个物种中相对保守的AA相关基因以及病理学和生理学过程。
人和小鼠中共有的DEGs有23个,比较有意思的是,其中部分基因也曾出现在AA相关的其他研究中。在一项关于AA的转录组测序研究中,也是差异表达基因,并参与细胞增殖的有丝分裂过程[18]。在毛囊休止期强表达,有研究表明,可以作为辐射诱导脱发的辐射保护剂[19]。DNA宏阵列分析显示与雄激素性脱发有关[20]。BMP信号参与调控头发的生长周期[21]。Anti-IL5能减少嗜酸性粒细胞的数量并导致脱发,加重红斑的严重程度,同时增加的表达[22]。利用免疫组化,在分级毛囊的球茎、小叶、峡部和漏斗部计数阳性细胞,发现AA小鼠中的阳性细胞显著高于对照组,而膳食维生素A可改变阳性细胞的数量和定位,从而影响AA的发展进程[23]。,作为T淋巴细胞的招募者,在AA患者血清中和表达水平明显升高,表明它们可能参与了T淋巴细胞募集到发炎组织的过程[24~26]。此外在C3H/HeJ小鼠自发形成AA过程中,和的表达水平逐渐增加[27]。另一项芯片和RT-PCR表达谱数据分析也显示和在AA组显著上调[28]。此外,通过阻断ifn诱导的趋化因子(和)可以预防AA的发生[29]。毛囊中能增加T细胞的趋化性,而抑制T细胞的趋化性可有效治疗AA[30]。上述已有研究报道表明了这些在人和小鼠中共有的差异表达基因不仅在本文中显示出显著的差异,而且在脱发相关的其他样本中也显示出类似的差异,并且机制研究显示部分基因参与AA发生发展进程。同时,后续也应关注那些挖掘出来尚未报道的DEGs,对它们深入细致的研究可能有助于丰富AA的病理学知识,进而帮助治疗AA病症。
GO富集分析显示两物种共有的23个基因主要富集在免疫反应(immune response),细胞因子介导的信号通路(cytokine-mediated signaling pathway)和T细胞活化(T cell activation)等一些免疫相关的过程。比较有意思的是,单独存在于两个物种的非共有DEGs的生物学过程也都富集在免疫相关的生物学过程中。而且,对这些非共有DEGs进行GO富集分析和KEGG通路分析发现,在人和小鼠中,除了都涉及免疫事件外,小鼠所涉及的生物学过程、细胞组分、分子功能和信号通路的范围更广泛,而上 述与人相关的内容则涉及的更具体甚至更直接,如人的某些条目直接显示与皮肤附属物相关(附图1, 头发生长周期(GO:0042633~hair cycle),上皮发育(GO:0008544~ epidermis development),毛囊发育(GO:0001942~hair follicle development),指甲发育(GO:0035878~nail development),角蛋白纤维(GO:0045095~keratin filament),上皮的结构组成(GO:0030280~structural constituent of epidermis))。尤其是对这些非共有基因进行组织富集分析,发现这些在人中的非共有基因直接富集在头皮和头发相关的组织(头皮(Scalp),头发(Hair)和发根(Hair root))。这些结果可能表明,虽然在两个物种中,斑秃都与免疫事件相关,然而,在小鼠中斑秃可能是一类全身性的疾病,而在人中,由于毛发分布的原因,斑秃更倾向于是一个局部发生的疾病。当然,这些结果也反应了样品来源的不同,一个来自人类AA患者头皮,另一个来自小鼠自发性AA皮肤。PPI网络节点位置的一些差异表达基因编码的蛋白也显示与免疫事件相关。除此之外,GSEA分析结果显示在人和小鼠中相对保守的信号通路也都集中在免疫相关的过程。这些结果表明斑秃能够引起了一系列的免疫响应相关事件。本研究结果与之前的报道相一致,AA是一类人类自身免疫性疾病,AA的发生被认为是毛囊的天然免疫功能崩溃导致的,包括上皮细胞中MHC I class异位表达和自身抗原呈递给自身反应性CD8+T细胞[31]。除了毛囊自身的免疫功能丧失,自身免疫会进一步破坏毛囊并引发一系列炎症反应[32]。近些年来,一类高效的免疫类药物JAK抑制剂(Janus kinase inhibitors)用于研究AA的治疗,具有比较好的疗效。在使用C3H/HeJ小鼠模型进行的临床前试验中,这些药物在局部和口服时均显示出预防和逆转AA的效果[7]。几个病例和个别报告已证明,不同类型斑秃患者在口服JAK抑制剂药物后,头发再生,这些药物包括托法替尼(tofacitinib)[33]、鲁索替尼(ruxolitinib)[34]和巴瑞克替尼(baricitinib)[35]。目前,美国FDA批准了几种JAK抑制剂用于治疗其他疾病,如ruxolitinib (JAK1/2抑制剂)用于治疗骨髓纤维化,tofacitinib (pan-JAK抑制剂)用于治疗类风湿性关节炎。JAK抑制剂等免疫类药物也提供了一种新的用于研究脱发治疗的策略,不过也应该意识到任何新药潜在副作用。
据报道,在2%~44%的AA患者中有指甲受累,并且这种情况在儿童(>40%)中比成人(<20%)更常见,此外,越是严重的斑秃患者也越容易出现在指甲受累[36,37]。指甲属于皮肤附属物,尽管GSEA(图5)显示上调基因集多富集在免疫相关的表型,然而下调基因集富集的条目都在皮肤附属物受损相关的表型,如指甲营养不良(nail dystrophy)、发质异常(abnormality of hair texture)、脱发(alopecia)和睫毛稀疏或缺失(sparse or absent eyelashes)。说明AA患者除了引起一些自身免疫相关的症状外,也会伴随出现自身皮肤附属物(如指甲、睫毛和眉毛等)生长异常的症状。
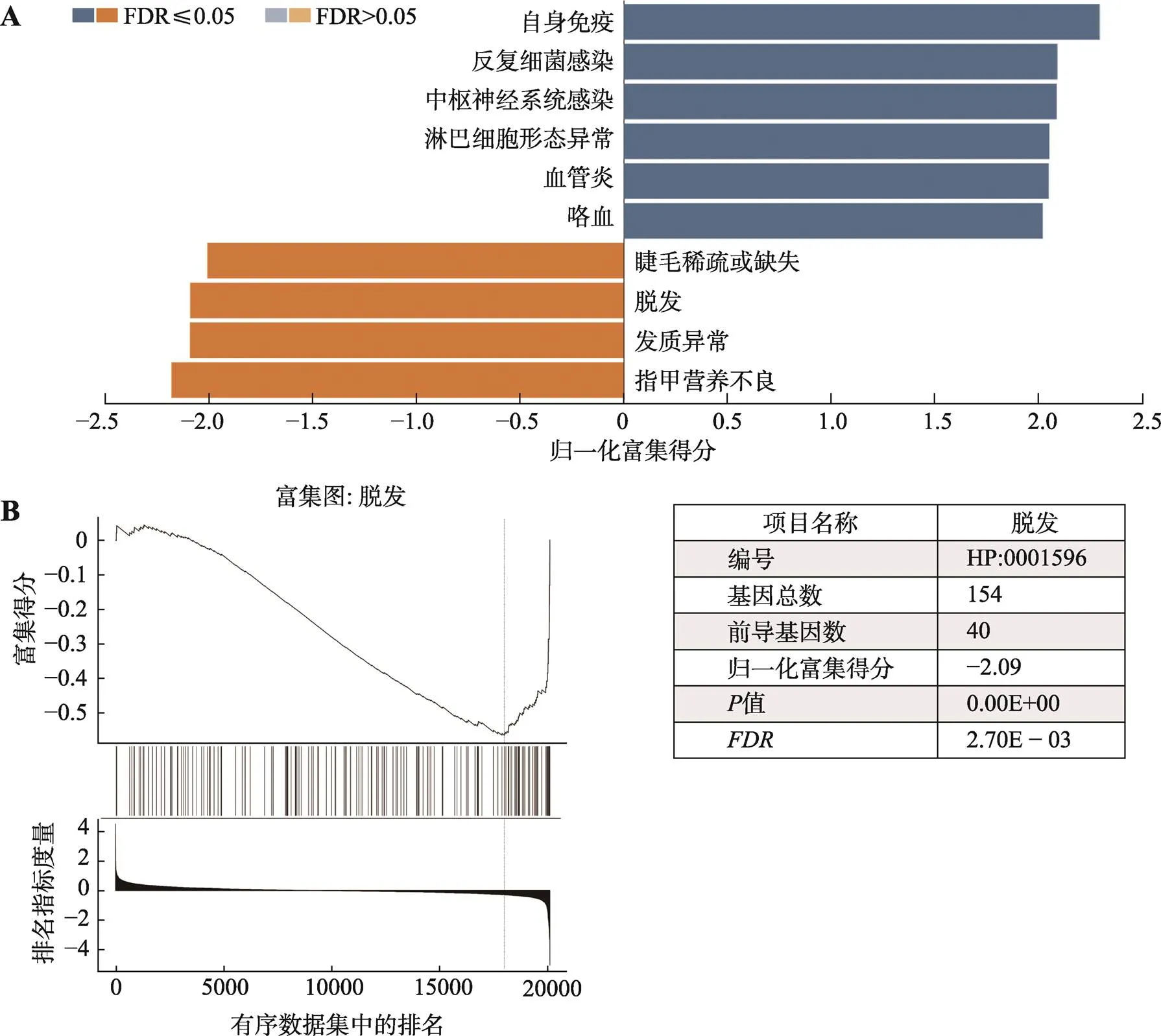
图5 涉及人类表型的基因集富集分析
A:人斑秃样本(GSE45512)基因集在人类表型上的富集结果;B:人斑秃样本基因集在秃头症中富集的结果。
高通量分析技术的发展和测序成本的降低产生了大量与脱发相关的研究数据。本研究通过利用NCBI公共数据库中的数据和生物信息学分析工具整合分析了两个物种的AA数据,从基因层面分析了AA相关的分子机制,并获得了一些可信的结果,为脱发发生发展的机制和治疗提供有价值的参考信息。本文挖掘的23个在人和小鼠AA样本中共有的DEGs,其中部分已在AA相关的研究报道中得到证实([22],[23],[29],[29],[38],[39],[40],IL2RG[41],[19,42]和[21,42]),然而,目前关于AA病症发生发展的机制以及AA与免疫成因的机理还有待完善。基于现有研究,对尚未报道这部分DEGs进行深入的分析和研究,有望对未来治疗斑秃提供了新的药物靶点。
附录:
附图1和附表1详见文章电子版www.chinagene.cn。
[1] Safavi KH, Muller SA, Suman VJ, Moshell AN, Melton LJ. Incidence of alopecia areata in olmsted county, minnesota, 1975 through 1989., 1995, 70(7): 628–633.
[2] Pratt CH, King LE Jr, Messenger AG, Christiano AM, Sundberg JP. Alopecia areata., 2017, 3: 17011.
[3] Liu LY, King BA, Craiglow BG. Health-related quality of life (HRQoL) among patients with alopecia areata (AA): A systematic review., 2016, 75(4): 806–812.e3.
[4] Hunt N, McHale S. The psychological impact of alopecia., 2005, 331(7522): 951–953.
[5] Singam V, Patel KR, Lee HH, Rastogi S, Silverberg JI. Association of alopecia areata with hospitalization for mental health disorders in US adults., 2019, 80(3): 792–794.
[6] Rencz F, Gulácsi L, Péntek M, Wikonkál N, Baji P, Brodszky V. Alopecia areata and health-related quality of life: a systematic review and meta-analysis.. 2016, 175(3): 561–571.
[7] Xing LZ, Dai ZP, Jabbari A, Cerise JE, Higgins CA, Gong WJ, de Jong A, Harel S, DeStefano GM, Rothman L, Singh P, Petukhova L, Mackay-Wiggan J, Christiano AM, Clynes R. Alopecia areata is driven by cytotoxic T lymphocytes and is reversed by JAK inhibition., 2014, 20(9): 1043–1049.
[8] The Gene Ontology Consortium. The Gene Ontology Resource: 20 years and still GOing strong., 2019, 47(D1): D330–d338.
[9] Huang da W, Sherman BT, Lempicki RA. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists., 2009, 37(1): 1–13.
[10] Chen HA, Pan JY, Chiang CH, Jhang AH, Ho WT. New idea for hair transplantation to preserve more donor hair follicles., 2019, 128: 83–85.
[11] Afifi L, Maranda EL, Zarei M, Delcanto GM, Falto- Aizpurua L, Kluijfhout WP, Jimenez JJ. Low-level laser therapy as a treatment for androgenetic alopecia., 2017, 49(1): 27–39.
[12] Tekin NS, Tekin IO, Cinar S, Altinyazar HC, Koca R, Esturk E. The PUVA-turban as an alternative treatment of alopecia associated with chronic graft versus host disease., 2005, 53(5): 902–903.
[13] Lee HW, Jun JH, Lee JA, Lim HJ, Lim HS, Lee MS. Acupuncture for treating alopecia areata: a protocol of systematic review of randomised clinical trials., 2015, 5(10): e008841.
[14] Suchonwanit P, Srisuwanwattana P, Chalermroj N, Khunkhet S. A randomized, double-blind controlled study of the efficacy and safety of topical solution of 0.25% finasteride admixed with 3% minoxidil vs. 3% minoxidil solution in the treatment of male androgenetic alopecia., 2018, 32(12): 2257–2263.
[15] Durdu M, Özcan D, Baba M, Seçkin D. Efficacy and safety of diphenylcyclopropenone alone or in combination with anthralin in the treatment of chronic extensive alopecia areata: a retrospective case series., 2015, 72(4): 640–650.
[16] Rodrigues BL, Montalvão SAL, Cancela RBB, Silva FAR, Urban A, Huber SC, Júnior JLRC, Lana JFSD, Annichinno- Bizzacchi JM. Treatment of male pattern alopecia with platelet-rich plasma: A double-blind controlled study with analysis of platelet number and growth factor levels., 2019, 80(3): 694–700.
[17] Sundberg JP, Boggess D, Montagutelli X, Hogan ME, King LE Jr. C3H/HeJ mouse model for alopecia areata., 1995, 104(5 Suppl): 16s–17s.
[18] Subramanya RD, Coda AB, Sinha AA. Transcriptional profiling in alopecia areata defines immune and cell cycle control related genes within disease-specific signatures., 2010, 96(3): 146–153.
[19] Kawano M, Umeda S, Yasuda T, Fujita M, Ishikawa A, Imamura T, Imai T, Nakayama F. FGF18 signaling in the hair cycle resting phase determines radioresistance of hair follicles by arresting hair cycling., 2016, 1(3): 170–181.
[20] Midorikawa T, Chikazawa T, Yoshino T, Takada K, Arase S. Different gene expression profile observed in dermal papilla cells related to androgenic alopecia by DNA macroarray analysis., 2004, 36(1): 25–32.
[21] Plikus MV, Widelitz RB, Maxson R, Chuong CM. Analyses of regenerative wave patterns in adult hair follicle populations reveal macro-environmental regulation of stem cell activity., 2009, 53(5–6): 857–868.
[22] Renninger ML, Seymour RE, Whiteley LO, Sundberg JP, Hogenesch H. Anti-IL5 decreases the number of eosinophils but not the severity of dermatitis in Sharpin-deficient mice., 2010, 19(3): 252–258.
[23] Duncan FJ, Silva KA, Johnson CJ, King BL, Szatkiewicz JP, Kamdar SP, Ong DE, Napoli JL, Wang J, King LE Jr, Whiting DA, McElwee KJ, Sundberg JP, Everts HB. Endogenous retinoids in the pathogenesis of alopecia areata., 2013, 133(2): 334–343.
[24] Zainodini N, Hassanshahi G, Arababadi MK, Khorramdelazad H, Mirzaei A. Differential expression of CXCL1, CXCL9, CXCL10 and CXCL12 chemokines in alopecia areata., 2013, 10(1): 40–46.
[25] Maouia A, Sormani L, Youssef M, Helal AN, Kassab A, Passeron T. Differential expression of CXCL9, CXCL10, and IFN-γ in vitiligo and alopecia areata patients., 2017, 30(2): 259–261.
[26] Bilgic O, Sivrikaya A, Unlu A, Altinyazar HC. Serum cytokine and chemokine profiles in patients with alopecia areata., 2016, 27(3): 260–263.
[27] McPhee CG, Duncan FJ, Silva KA, King LE Jr, Hogenesch H, Roopenian DC, Everts HB, Sundberg JP. Increased expression of Cxcr3 and its ligands, Cxcl9 and Cxcl10, during the development of alopecia areata in the mouse., 2012, 132(6): 1736–1738.
[28] Suárez-Fariñas M, Ungar B, Noda S, Shroff A, Mansouri Y, Fuentes-Duculan J, Czernik A, Zheng X, Estrada YD, Xu H, Peng X, Shemer A, Krueger JG, Lebwohl MG, Guttman-Yassky E. Alopecia areata profiling shows TH1, TH2, and IL-23 cytokine activation without parallel TH17/ TH22 skewing., 2015, 136(5): 1277–1287.
[29] Dai Z, Xing L, Cerise J, Wang EH, Jabbari A, de Jong A, Petukhova L, Christiano AM, Clynes R. CXCR3 blockade inhibits T Cell migration into the skin and prevents development of alopecia areata., 2016, 197(4): 1089–1099.
[30] Ito T, Hashizume H, Shimauchi T, Funakoshi A, Ito N, Fukamizu H, Takigawa M, Tokura Y. CXCL10 produced from hair follicles induces Th1 and Tc1 cell infiltration in the acute phase of alopecia areata followed by sustained Tc1 accumulation in the chronic phase., 2013, 69(2): 140–147.
[31] Gilhar A, Schrum AG, Etzioni A, Waldmann H, Paus R. Alopecia areata: Animal models illuminate autoimmune pathogenesis and novel immunotherapeutic strategies., 2016, 15(7): 726–735.
[32] Strazzulla LC, Wang EHC, Avila L, Lo Sicco K, Brinster N, Christiano AM, Shapiro J. Alopecia areata: Disease characteristics, clinical evaluation, and new perspectives on pathogenesis., 2018, 78(1): 1–12.
[33] Liu LY, Craiglow BG, Dai F, King BA. Tofacitinib for the treatment of severe alopecia areata and variants: A study of 90 patients., 2017, 76(1): 22–28.
[34] Mackay-Wiggan J, Jabbari A, Nguyen N, Cerise JE, Clark C, Ulerio G, Furniss M, Vaughan R, Christiano AM, Clynes R. Oral ruxolitinib induces hair regrowth in patients with moderate-to-severe alopecia areata., 2016, 1(15): e89790.
[35] Jabbari A, Dai Z, Xing L, Cerise JE, Ramot Y, Berkun Y, Sanchez GA, Goldbach-Mansky R, Christiano AM, Clynes R, Zlotogorski A. Reversal of alopecia areata following treatment with the JAK1/2 inhibitor baricitinib., 2015, 2(4): 351–355.
[36] Chelidze K, Lipner SR. Nail changes in alopecia areata: an update and review., 2018, 57(7): 776–783.
[37] Kasumagic-Halilovic E, Prohic A. Nail changes in alopecia areata: frequency and clinical presentation., 2009, 23(2): 240–241.
[38] Hamed FN, McDonagh AJG, Almaghrabi S, Bakri Y, Messenger AG, Tazi-Ahnini R. Epigallocatechin-3 gallate inhibits STAT-1/JAK2/IRF-1/HLA-DR/HLA-B and reduces CD8 MKG2D lymphocytes of alopecia areata patients., 2018, 15(12).
[39] Shi JX, Peng P, Liu WX, Mi P, Xing C, Ning GZ, Feng SQ. Bioinformatics analysis of genes associated with the patchy-type alopecia areata: CD2 may be a new therapeutic target., 2019, 1–7.
[40] Nagao K, Kobayashi T, Moro K, Ohyama M, Adachi T, Kitashima DY, Ueha S, Horiuchi K, Tanizaki H, Kabashima K, Kubo A, Cho YH, Clausen BE, Matsushima K, Suematsu M, Furtado GC, Lira SA, Farber JM, Udey MC, Amagai M. Stress-induced production of chemokines by hair follicles regulates the trafficking of dendritic cells in skin., 2012, 13(8): 744–752.
[41] Ibusuki A, Nishikawa T, Hiraki T, Okano T, Imai K, Kanegane H, Ohnishi H, Kato Z, Fujii K, Tanimoto A, Kawano Y, Kanekura T. Prominent dermal Langerhans cells in an Omenn syndrome patient with a novel mutation in the IL2RG gene., 2019, 46(11): 1019–1023.
[42] Jiang W, Fan YX, Qiao X, Zhang YJ, Liu ZH, Zhao YH, Wang RJ, Wang ZX, Zhang WG, Su R, Li JQ. The transcriptome research progresses of skin hair follicle development., 2015, 37(6): 528–534.江玮, 范一星, 乔贤, 张燕军, 刘志红, 赵艳红, 王瑞军, 王志新, 张文广, 苏蕊, 李金泉. 皮肤毛囊发育的转录组研究进展. 遗传, 2015, 37(6): 528–534.
Bioinformatics analysis of differentially expressed genes on alopecia
Hong Xiang, Xiaohu Yang, Liangxia Ai, Yanping Pan, Yong Hu
The molecular mechanism of alopecia areata (AA) is still elusive and here we utilized bioinformatics methods to analyze AA-related differentially expressed genes. In this study, GSE45512 and GSE45513 were downloaded from the NCBI sub-database Gene Expression Omnibus (GEO). The gene expressions of AA and normal samples were analyzed using the R package limma, which showed significant differences between AA and normal samples in two species. These genes were subject to functional annotation and protein interaction networks. At the same time, gene set enrichment analysis was conducted for all differentially expressed genes. The study revealed that a total of 225 differentially expressed genes were screened from human AA samples, and a total of 337 differentially expressed genes were screened from spontaneous AA skin samples in C3H/HeJ mice. There are 23 differentially expressed genes in the two species. GO and protein interaction network analysis shown gene enrichment in immune-related functions, and these proteins interact with each other. Gene set enrichment analysis showed that differential genes from both species were significantly enriched to chemokine signaling pathways, cytokine-cytokine receptor interactions, staphylococcus aureus infection, and antigen processing and presentation. Moreover, the human down-regulated differential gene not only maps to the alopecia in human phenotype ontology, but also maps to the pathologically relevant phenotype of the skin appendage. In brief, 23 significant differentially expressed genes were screened out coexisting in AA human and mouse by bioinformatics methods. In addition, the result demonstrated that AA is closely related to the immune process and skin appendage lesions. These results provide new ideas for the diagnosis and treatment of AA.
hair loss; alopecia areata; GEO; bioinformatics
2019-10-02;
2019-12-13
深圳市科技创新委员会基础科学研究基金(编号:JCYJ20180507182250795),中国博士后科学基金(编号:2019M663173)和深圳孔雀团队项目(编号:KQTD20170331160605510)资助[Supported by the Shenzhen Science and Technology Innovation Committee Basic Science Research Grant (No. JCYJ20180507182250795), China Postdoctoral Science Foundation Grand (No. 2019M663173) and the Shenzhen Peacock Team Project (No. KQTD20170331160605510)]
向虹,博士,研究方向:基因治疗。E-mail: hong.xiang@siat.ac.cn
胡勇,博士,副教授,研究方向:基因治疗。E-mail: yong.hu@siat.ac.cn
10.16288/j.yczz.19-214
2019/12/20 9:24:49
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20191218.1712.006.html
(责任编委: 赵方庆)

