一株红霉素耐药百日咳鲍特菌的完整基因组分析
于丹 栗东芳 袁林 冯欣 史伟 姚开虎★
百日咳鲍特菌(Bordetella pertussis,B.pertussis)又称为百日咳杆菌,是百日咳的病原菌。20世纪40年代开始推广接种百日咳疫苗后,百日咳发病率明显下降。但20世纪末,一些高疫苗接种率国家陆续报道百日咳发病率再度升高,称为百日咳再现(pertussis re-emerge),引起全球广泛关注[1]。百日咳再现涉及多方面的原因,其中百日咳鲍特菌的适应性变化是重要原因之一,包括对疫苗免疫选择压力和抗生素选择压力的适应性变化[1]。从不同国家报道数据看,尽管1994年第一例红霉素耐药百日咳鲍特菌出现在美国,但至今只有中国内地报道了红霉素耐药百日咳鲍特菌的广泛流行[2]。前期,本课题组研究结果提示红霉素耐药百日咳鲍特菌大多具有相同的抗原基因型,均为ptxA1/ptxP1/prn1型,与之前的研究结果相一致[3],提示耐药菌株具有相似遗传背景。为进一步明确耐药菌株的基因组特征,本研究对红霉素耐药株P2013109 进行了完整基因组测序,并与我国疫苗株CS 等已公开的完整基因组数据比较,为疫苗研究等提供参考。
1 材料与方法
1.1 菌株
菌株P2013109 为本实验室从百日咳患儿鼻咽拭子标本中分离。菌株的特点:药物敏感性检测显示该菌株红霉素最低抑菌浓度(Minimum inhibitory concentration,MIC)>256 mg/L,红霉素KB 纸片(15 μg/片)扩散法检测药物敏感性未见抑菌环,前期研究确定其疫苗相关抗原基因型为ptxA1/ptxC1/ptxP1/prn1/fim2-1/fim3-1/tcfA2[3]。
1.2 试剂/仪器
硅胶模型TM 基因组DNA 提取试剂盒(北京赛百盛公司),碳琼脂培养基(CM0119,英国OXOID 公司),培养箱,4℃冰箱,-80℃冰箱,离心机。
1.3 细菌培养以及基因组DNA 提取
冻存菌株标本接种于含10%羊血的碳琼脂培养基。培养基置于温度为35℃的培养箱中孵育3天后观察菌落特征和纯度,转种一次。刮取琼脂上的菌落,采用试剂盒提取细菌基因组DNA。
1.4 基因组测序和组装
将P2013109 的基因组DNA 委托上海美吉生物医药公司分别利用二代Illumina Hiseq 2000 平台和三代Pacbio 平台进行全基因组测序。采用SOAPdenovo v2.04 软件对二代测序数据进行初步组装;利用blasR 将三代测序数据比对到初步组装结果上,并对其进行矫正与纠错;然后利用纠正后的三代测序数据进行组装,使用的软件为Celera Assembler8.0。最后再次利用二代测序数据进行校验,同时进行局部内洞填充和碱基校正。
1.5 基因组注释
对组装得到的基因组序列,利用Glimmer 3.02软件进行开放阅读框(open reading frame,ORF)预测,并与数据库比对获得蛋白功能注释。利用Barrnap 0.4.2 和tRNAscan-SE v1.3.1 软件对基因组中包含的rRNA 和tRNA 进行预测。
1.6 比较基因组分析
从NCBI 下载已完成全基因组测序的15 株百日咳杆菌及其注释信息(见下表1),包括中国百日咳疫苗株CS[4]、该菌种最早完成测序的Tohama I菌株[5],以及欧美发达国家已经发表的当前流行的ptxP3型菌株的基因组序列[6-7]。利用MUMmer3.23 软件将其他基因组序列与参考基因组序列Tohama I 进行比对,得到每个基因组的单核苷酸多态性(SNPs)信息,去除落在重复序列区域的SNP 后,将在所有样本中都出现的SNP 位点来构建系统发育树,采用TreeBeST 软件中的最大似然法,迭代1 000 次。用blast v2.5.0 比对软件将其他菌株比对到菌株P2013109 上,得到每株菌在P2013109 的比对上的区域,然后将所有菌的比对结果用circos 软件做图。
2 结果
2.1 基因组组装与基因组预测
对测序得到的百日咳杆菌P2013109 二代和三代数据进行质控和组装后,得到一个完整的基因组序列,总长度4 126 010 bp,GC 含量为67.72%,预测P2013109 的基因共有4 085 个,基因的总长度为3 571 551 bp,占全基因组的86.6%,平均基因长度为874 bp。基因区GC 含量为68.4%,基因间区GC 含量为62.8%。共找到61 个tRNA 和3 组rRNA。P2013109 基因组序列已经提交到NCBI 的Genbank 数据库,其登录号为CP038790。与其他15 株菌的基因组信息见下表1。
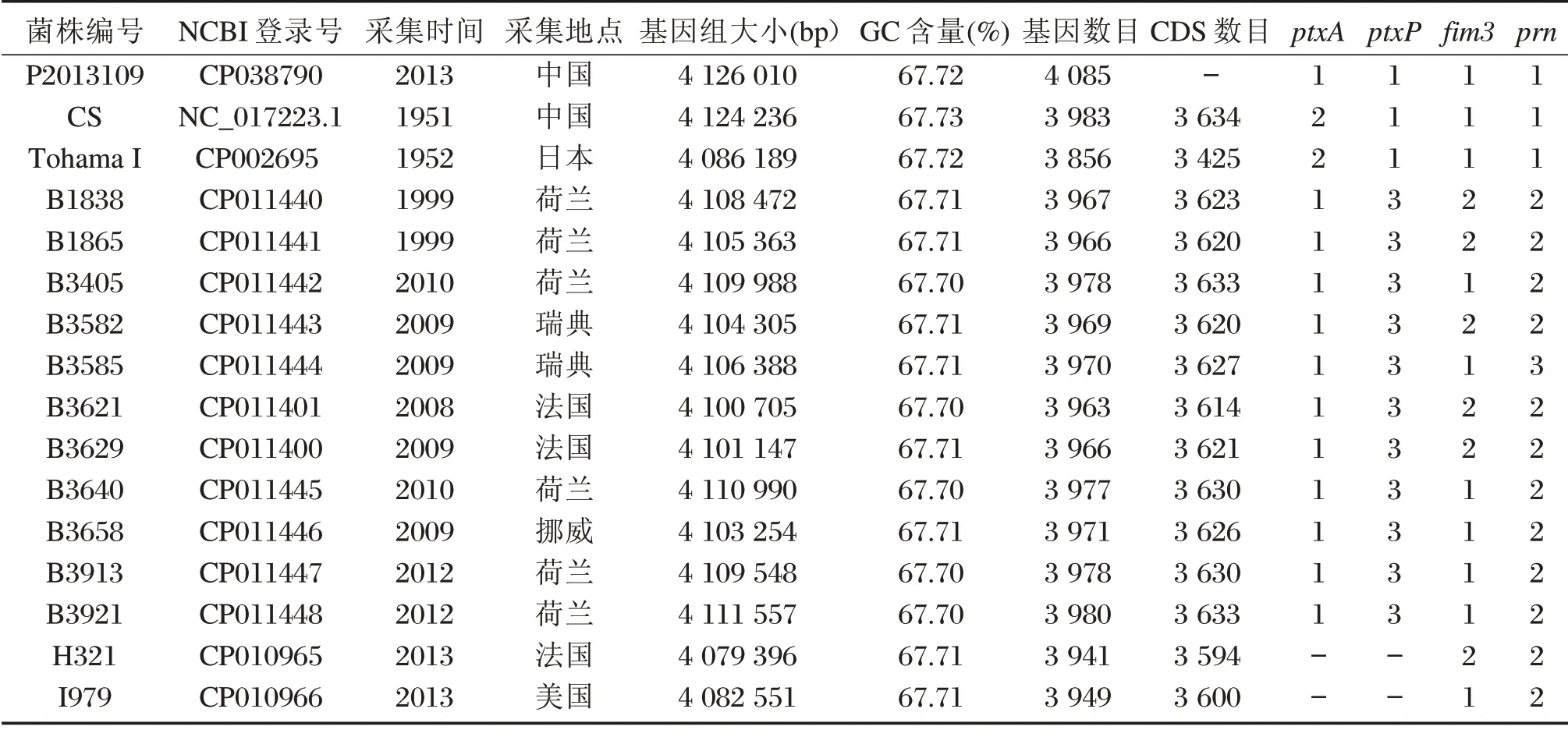
表1 百日咳杆菌P2013109 与其他15 个菌株基因组的基本特征Table 1 The genome characteristics of B.pertussis P2013109 and the others
2.2 COG 聚类和KEGG 代谢通路分析
通过与String 数据库比对,在P2013109 基因组上,共有3 388 个CDS 获得COG 和NOG 功能注释,根据注释结果可以将这些功能归为4 大类,23小类。如表2 所示。在这些注释结果中,与代谢相关的功能最多,主要以氨基酸、无机离子、脂类和碳水化合物的转运和代谢为主;其次是细胞过程与信号传导类的功能,主要以细胞壁/膜/包体的生物合成、翻译后修饰等为主;在信息储存和处理类的功能中,以转录和翻译为主。
通过与KEGG 数据库比对,在P2013109 基因组中共找到代谢通路相关的基因2 160 个。共注释上41 个类型,归属6 个大类,即细胞过程、环境信息处理、遗传信息处理、人类疾病,代谢和有机体系统相关的功能。
2.4 比较基因组分析
2.4.1 抗原基因型和耐药基因型确定
比对百日咳鲍特菌P2013109 抗原基因ptxA、ptxP、fim3和prn的核苷酸序列,得到P2013109 的基因组的抗原基因型结果,与前期抗原基因型测序结果一致。其他15 株菌通过搜集文献和比对结果相结合的方式得到这些抗原基因型的分析结果,见表1 所示。细菌染色体23s rRNA 基因有3个拷贝,每个拷贝中均具有A2047G 变异。
2.4.2 全基因组比对
将其他所有菌株与P2013109 比对结果如下图1 所示。与P2013109 相比,其他基因组主要有3 个比较大的缺失区域,R1 约4kb,编码转座酶;R2 是菌株H321 和I979 缺失的一个大约27 kb 的区域,是编码百日咳毒素和IV 型分泌系统的区域;R3 区域主要包含R3-1 和R3-2 两个子区域,除了CS 和Tohama I 菌株外,其他菌株均缺失。R3-1 主要包含BP1948-1951、BP1953 和BP1954 等6 个基因,前4个基因涉及支链氨基酸转运底物结合蛋白、转运系统渗透酶、APC 转运体ATP 结合蛋白;BP1953 和BP1954,分别编码氧化还原酶和单氧酶。R3-2 区域也包含4 个基因,BP1959 是IS1663 的转座酶基因,BP1961 编码一种细胞色素flavocytochrome,bfrI和BP1965 分别与铁载体受体和跨膜蛋白有关。
2.4.3 系统发育分析
将这16 株菌与广东报道的一株百日咳全基因序列一起做系统发育分析,共得到326 个SNPs。基于这些SNPs 构建系统发育树如图2 所示,由图可知,这16 株菌可以分成3 个型别,TohamaⅠ和CS 这两个疫苗株作为I 型,分别于1951年和1952年分离,与其他2 个型别有76 个SNP 的差异(23.3%),此外,TohamaⅠ与其他所有菌株比较呈现54 个SNPs(16.5%)。P2013109 自成一个分支,并且与其他所有菌株相比,呈现25 个SNPs(7.7%)。其他在2000年之后采集自国外的菌株自成一个型别,与另外两个型别有21 个SNP 的差异(6.4%),其中这14 株菌又可以分为两个亚型,主要区别是Ⅲa 亚型的fim3基因是2 型,而Ⅲb 型菌株是1 型。
分支旁的数字为自展1 000 次的置信值;标尺代表7%的序列差异。系统发育树分析时纳入了广东分离株BP_GD2017,见参考文献[8]。
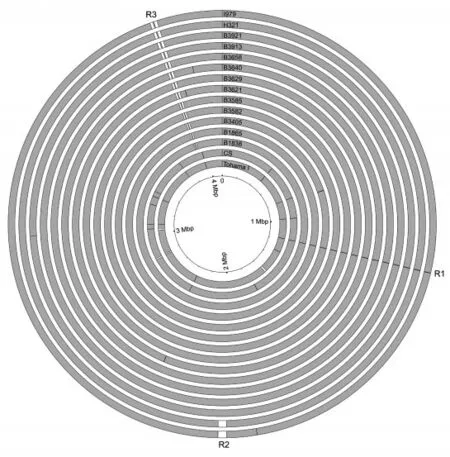
图1 P2013109 与其他15 株菌的全基因组比较结果Figure 1 The whole genome comparison analysis of B.pertussis P2013109 and the other 15 genomes

图2 百日咳杆菌P2013109 与其他百日咳杆菌的的全基因组进化树Figure 2 Genome-wide phylogenetic tree of Genome-wide phylogenetic tree of Bordetella pertussis P2013109 and the other genomes(including the BP_GD2017 isolated from Gangdong)

表2 百日咳杆菌P2013109 基因组的COG 功能分类Table 2 Gene distribution based on COG and NOG functional classification of B.pertussis P2013109
3 讨论
本研究显示百日咳鲍特菌P2013109 株基因组4,126,010 bp,与已经报道的百日咳鲍特菌全基因组[9-10]大小相当。该红霉素耐药株表型明确,全基因组序列也表明,细菌染色体23S rRNA 基因有3个拷贝,3 个拷贝中均具有A2047G 变异。23S rRNA 是核糖体RNA,核糖体是细胞内蛋白质合成的场所,红霉素可以通过与位于23S rRNA 区域的肽链形成的部位(肽酰转移酶区域)结合进而抑制蛋白质合成。23S rRNA 突变是目前百日咳鲍特菌耐药的主要机制[11],基因型与表型相互应证。与P2013109 株全基因组比较,其他菌株主要有3 个缺失区,尤其R1 区涉及转座酶。这些基因区在耐药变异过程中是否发挥作用,需要深入研究。基于完整基因组SNPs 构建系统发育树发现,纳入分析的菌株明确分为3 支。分离于20世纪50年代的百日咳流行菌株CS 株[4]和Tohama I 株形成独立的进化分支,与当前流行菌株在进化分支上存在较大差异。
近期,广东李柏生等[8]对广东分离到的1 株百日咳鲍特菌BP-GD2017 进行全基因组分析报道,该菌株的基因组草图序列数据纳入基因组比较分析发现,该菌株处于国外菌株分支Ⅲ中(见图3),它们的抗原基因型也完全相同,为ptxA1/ptxP3。P2013109 独属一支,抗原基因型为ptxA1/ptxP1。课题组前期检测了99 株百日咳鲍特菌的疫苗相关基因型,ptxA1/ptxP1占91.9%(91/99)[3]。这些数据明确提示长期疫苗使用之后,百日咳致病菌株百日咳毒素相关基因型发生了明显变化,与既往研究报告[12-13]相符。提示近年来流行的百日咳菌株的基因改变除了疫苗压力造成相关抗原性发生适应进化,也有可能在进化分支上出现改变。本课题组前期研究表明,临床分离菌株耐药株抗原基因型均为ptxA1/ptxP1,而基因型ptxA1/ptxP3的分离株全部对红霉素敏感,这提示国内外临床分离菌株耐药性的差异,与流行菌株的遗传背景不同相关。
当前百日咳疫苗生产用菌株基本上都是分离自上个世纪。研究已经发现,百日咳疫苗长期使用以后,临床分离菌株的抗原基因型与疫苗株明显不同[12-13]。尤其在无细胞疫苗使用国家,一些毒力抗原缺失的菌株,如缺失PRN、FIM 和PTX 的菌株逐渐被发现,甚至出现流行的情况,菌株疫苗抗原的变异和缺失等适应性变化可能是百日咳再现的重要因素[14]。但课题组在前期研究中没有发现国内百日咳鲍特菌分离株有抗原缺失,与疫苗株比较主要是抗原基因变异[3]。本研究从基因组序列证明P2013109 有抗原基因变异,无缺失,同时与疫苗株比较遗传进化分支明显不同。近期,Xu 等[15]分析了71 株中国分离的ptxP1菌株(其中包括46 株红霉素耐药株)的基因组重测序结果,基于2 744 个SNPs 位点构建遗传进化树,结果发现耐药株只出现在3 个遗传分支中,这些耐药株只分离自中国内地,包括香港、台湾在内的其他地区和国家公开的菌株基因组,均没有处于同一遗传支的分离株。
本研究第一次呈现了耐红霉素百日咳鲍特菌ptxP1型菌株的完整基因组序列,序列分析明确显示其在遗传学关系上与疫苗株和国外当前主要流行的ptxP3型菌株遗传关系较远,属于特有的一个遗传分支。已有基因组重测序研究提示还需要对其他耐药菌株进行完整基因组的构建和研究,才能反映红霉素耐药鲍特菌基因组的全貌,为国内将来新型百日咳疫苗的研发提供基础信息。
致谢:广东省疾病预防控制中心李柏生提供其课题组发表的1 株百日咳鲍特菌分离株的全基因组序列数据。

