合浦珠母贝丝氨酸蛋白酶抑制因子基因pfser1克隆与表达
陈 爽,梁 健,张荣庆,2,3
(1.蛋白质科学教育部重点实验室,清华大学生命科学学院,北京 100084;2.浙江省应用酶学重点实验室,浙江 嘉兴 314006;3.嘉兴大学生物化学科学与工程学院,浙江 嘉兴 314006)
合浦珠母贝(Pinctada fucata)隶属于软体动物门(Mollusca),瓣鳃纲(Bivalvia),珍珠贝目(Pterioida),珠母贝属(Pinctada),主要分布于日本沿海和我国南海,是生产海水珍珠的主要贝类。海水珍珠养殖为我国一项重要产业,但在珍珠培育过程中,珍珠贝高密度、集约化、工厂化养殖已出现多种疫病防治难题[1-2]。为提高合浦珠母贝产珠质量和产量,迫切需要对贝类天然免疫中应答过程机理开展深入研究。
丝氨酸蛋白酶家族是一类重要的蛋白水解酶,广泛参与调整机体消化、受精、胚胎发育、血液凝结、补体系统、细胞免疫和体液免疫等生理和病理过程[3-5]。丝氨酸蛋白酶抑制因子(Serine protease inhibitor,SPI)是对丝氨酸蛋白酶家族有抑制活性的一类蛋白质,广泛存在于动物、植物、微生物体,一般通过与蛋白酶相互结合,形成稳定复合体来抑制丝氨酸蛋白酶活性[6]。无脊椎动物缺少获得性免疫系统,天然免疫应答是主要免疫系统[7]。在天然免疫过程中,丝氨酸蛋白酶及其抑制因子起重要作用,包括信号转导、级联放大、特异性免疫防御机制激活、抗菌肽合成等[8]。根据结构和功能,SPI分为Serpin、Pacifastin、Kunitz、Kazal 和α2-巨球蛋白等74 个家族[9]。Serpin 家族的丝氨酸蛋白酶抑制因子有较为保守的抑制结构域,即C 端反应中心环(Reactive center loop,RCL),此结构域可与丝氨酸蛋白酶形成serpin-蛋白酶复合体,导致蛋白酶失活[10]。20 世纪90 年代末,从果蝇、疟蚊等无脊椎动物中先后分离和鉴定出多种参与免疫反应的SPI,并在血细胞等免疫器官中检测到表达,验证了其参与免疫级联反应激活的过程[11]。近年来,一些贝类中也发现了丝氨酸蛋白酶抑制因子,如太平洋牡蛎(Crassostrea gigas)、东方牡蛎(Crassostrea virginica)等,其在贝类天然免疫中的作用已得到初步验证[12-14]。但迄今未在合浦珠母贝体内发现这类丝氨酸蛋白酶抑制因子。
近年来在骨免疫学研究中发现,成骨与免疫之间有重要的调节关系[15]。在贝类中,贝壳和珍珠的生长与哺乳动物骨骼生长机制类似,均为重要的生物矿化过程。在自然环境中,贝壳在外源砂砾入侵体内环境时会通过天然免疫和生物矿化形成珍珠,但生物矿化与天然免疫之间的关系尚未阐明。虽已证明在天然免疫中发挥重要作用的赖氨酸基质蛋白在合浦珠母贝天然免疫过程中表达量升高[16],但关于在天然免疫过程中发挥重要作用的SPI 是否会参与生物矿化过程仍需进一步研究。
本研究通过RACE技术在合浦珠母贝中克隆得一个Serpin 家族基因(命名为pfser1)的全长序列,检测该基因在合浦珠母贝不同组织中的表达,以及在贝体感染大肠杆菌(Escherichia coli)MG1655后的表达变化,并通过合浦珠母贝贝壳损伤修复实验,研究该基因在合浦珠母贝天然免疫中的作用,探究该基因与生物矿化间的关系,为探索合浦珠母贝天然免疫与生物矿化之间的关系,研究合浦珠母贝天然免疫机理奠定理论基础。
1 材料与方法
1.1 材料
实验用合浦珠母贝购自广东省湛江市海滨养殖场,空运至北京。在实验室水族箱中用人工海水(速得牌海水素,盐度30)暂养1 周后,取生长状态良好、活力较强的贝进行实验。
大肠杆菌MG1655,取自清华大学海洋生物学实验室,将菌种接种至LB 培养基中,于37 ℃、200 r/min 条件下震荡培养10~ 12 h。将菌液转移至离心管中,以3 000g、4 ℃条件离心10 min。用4 ℃下预冷的磷酸盐缓冲液(PBS,pH 7.4)重悬菌体,再次离心。重复两次后,用PBS 重悬,稀释至OD600值为1(1×108~ 2×108CFU/mL)后,用于实验。
1.2 方法
1.2.1 合浦珠母贝总RNA 提取 取状态良好的合浦珠母贝3 只,剖取外套膜组织,用TRIzol 法提取其总RNA,用Nanodrop 2000TM(ThermoFisher,美国)检测RNA 浓度与质量。
1.2.2 RACE 实验克隆基因全长 通过对合浦珠母贝外套膜转录组数据分析,得到1 个有丝氨酸蛋白酶抑制因子同源序列的基因片段。使用SMARTerTMRACE cDNA Amplification kit (TAKARA) 对总RNA 进行反转录,得到RACE 实验模板。根据基因片段序列信息设计RACE 实验5′ 引物和3′ 引物,随后分别进行5′-RACE 和3′-RACE,将得到的结果拼接。最后,用验证引物cSer1F、cSer1R 对结果进行确认,获得pfser1的全长基因序列。本实验使用的引物见表1。
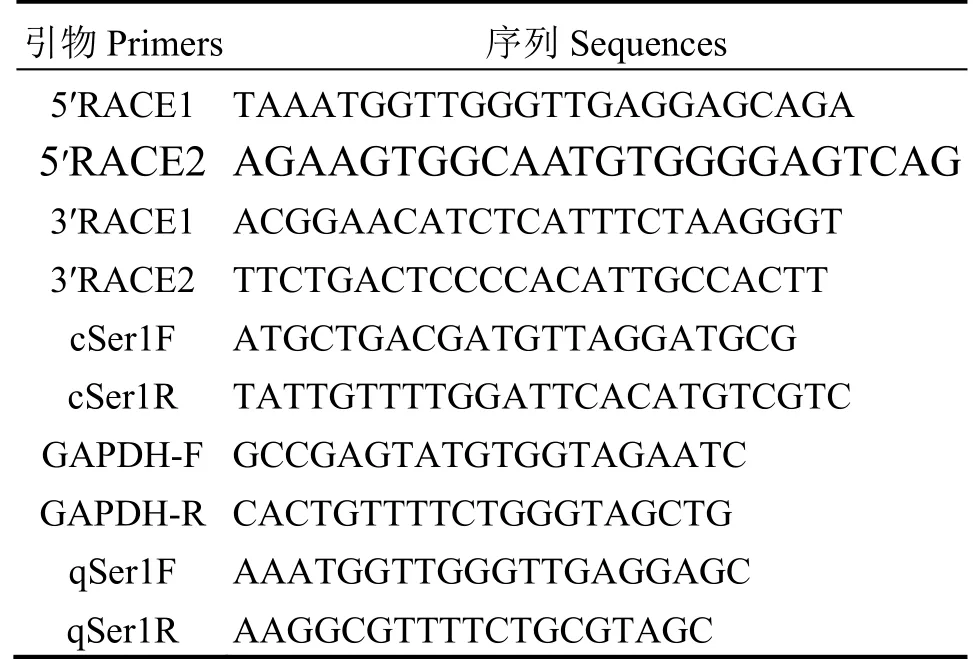
表1 PCR 引物序列Table 1 Primer sequences used in the cloning and Realtime PCR
1.2.3 生物信息学分析 基因序列的比对,同源性分析在NCBI 网站 (https://www.ncbi.nlm.nih.gov/)上 进 行;利 用 ExPASy (http://web.expasy.org/protparam/) 在线分析理化性质;通过SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/) 预测信号肽;利用SMART (Small Modular Architecture Research Tool,http://smart.embl-heidelberg.de/) 在线分析结构域;利用MEGA5.2 软件构建系统进化树。
1.2.4 组织分布检测 取合浦珠母贝的足、外套膜中央膜、鳃、闭壳肌、生殖腺、内脏团、外套膜边缘膜、血液8 个组织分别提取RNA,检测8 种组织中pfser1基因的相对表达量。具体操:提取RNA,使用SYBR®PrimeScript®RT-PCR Kit (TAKARA)对RNA 进行反转录,使用ABI 实时荧光定量PCR仪 (StepOnePlusTM,Life Technologies) 进行检测。选择GAPDH作为内参基因[17],用2-ΔΔCt法[18]计算基因相对表达量。实验中使用的引物见表 1(GAPDH-F,GAPDH-R,qSer1F,qSer1R)。
1.2.5 细菌吞噬实验 取24 只合浦珠母贝分成两组,分别向闭壳肌和外套膜间液中注射100 μL 大肠杆菌MG1655 菌液。在0、3、6、9 h 时分别从两组中各取合浦珠母贝3 只,取其外套膜边缘膜,提取RNA,检测不同时间点pfser1基因的相对表达量。具体步骤同1.2.4。
1.2.6 贝壳损伤修复实验 取24 只生长状况良好、大小相近的珍珠贝,分成8 组,每组3 只。在其边缘剪一深约3 cm、破坏贝壳珍珠层的缺口,放回人工海水中培养。在0、6、12、24、36、48、72、96 h 后收集合浦珠母贝外套边膜样品,提取RNA,检测不同时间点pfser1基因的相对表达量。具体步骤同1.2.4。
1.3 统计学分析
采用SPSS 统计软件对实验结果进行统计分析。其中,使用t-test 检测实验结果差异的显著性,α=0.05。
2 结果
2.1 合浦珠母贝pfser1 基因的 cDNA 的序列分析
通过RACE 技术获得pfser1基因全长序列(图1),该基因全长为1 240 bp,5′非编码区长73 bp,3′非编码区长132 bp,开放阅读框长度1 035 bp,编码344 个氨基酸。在线预测结果显示,pfser1基因编码的蛋白理论分子质量为38.5 ku,理论等电点5.52,1~ 322 位氨基酸属于SERPIN 结构域,且在pfser1末端有一个保守的反应中心环(reactive center loop,RCL),故确定pfser1是Serpin 型丝氨酸蛋白酶抑制剂家族成员(图2)。信号肽分析未显示pfser1 蛋白有信号肽序列。进化树分析显示该基因与牡蛎中蛋白酶抑制因子(leukocyte elastase inhibitor)相似性较高(图 3)。
2.2 pfser1 基因在不同组织中的表达分布
足、外套膜中央膜、鳃、闭壳肌、生殖腺、内脏团、外套膜边缘膜和血液中pfser1基因表达情况见图4。图4 显示,pfser1在外套膜边缘膜表达量最高,大约是足中表达量的4 倍;鳃组织次之,约为足中表达量的2 倍;在血细胞中表达量最少,约为足中pfser1基因表达量的1/20。

图1 pfser1 基因的核酸序列及编码的氨基酸序列Fig.1 Nucleotide and amino acids sequences of pfser1 gene from Pinctada fucata

图2 SMART 软件预测的pfser1 蛋白质功能结构域Fig.2 Structure of pfser1 protein analyzed by SMART

图3 N-J 法构建pfser1 系统进化树Fig.3 Phylogenetic tree of pfser1 by neighbour-joining method
2.3 细菌吞噬实验
向合浦珠母贝外套膜间液中注射大肠杆菌MG1655 后,外套膜边缘膜中的pfser1基因表达量急剧升高,在3 h 达到最高,约为对照组的58 倍,之后逐渐降低,在6 h 时是对照组的17 倍,到9 h时是对照组的3 倍(图5)。向合浦珠母贝闭壳肌中注射大肠杆菌MG1655 后,外套膜边缘膜中pfser1基因表达量逐渐升高,在3 h 时是对照组的18 倍,在6 h 附近达到峰值(约是对照组的32 倍),而后迅速降低,在9 h 已降至对照组的4 倍左右(图5)。

图5 外套膜间液和闭壳肌感染MG1655 后pfser1 基因表达量变化Fig.5 Expression of pfser1 after adductor and muscle injected by MG1655
2.4 贝壳修复过程中pfser1 基因的表达变化
将合浦珠母贝贝壳进行损伤处理后,会刺激合浦珠母贝进行生物矿化以修复贝壳。在贝壳损伤修补的过程中,pfser1基因表达量先上升,后下降至正常水平以下。在贝壳损伤约6 h 时,pfser1基因表达量达最大值,是正常水平的2 倍,在12 h 以后,pfser1基因的表达量均相较贝壳结构未损伤时更低,在48 h 检测到pfser1表达的最低值,约是正常水平的0.2%(图6)。

图6 pfser1 基因在贝壳损伤修复实验中的表达量变化Fig.6 Expression of pfser1 in the shell notching test
3 讨论
据估计,在哺乳动物体内蛋白酶抑制因子占血浆蛋白总量的10%,其中丝氨酸蛋白酶抑制因子的Serpin 家族占有很大比例[19]。当脊椎动物和无脊椎动物受到微生物感染后,其体内的丝氨酸蛋白酶可快速激活机体的免疫系统产生相应的免疫效应,杀灭入侵的病原菌。通常这个反应过程受丝氨酸蛋白酶抑制因子Serpin 家族的调节。研究发现,Serpin可参与无脊椎动物很多重要生理过程,如鲎(Tachypleus tridentatus)的血淋巴凝集反应和节肢动物的天然免疫反应[20-21]。当机体损伤或者受到病原微生物感染时,一些甲壳动物和昆虫体内的丝氨酸蛋白酶可通过激活联级反应过程和酚氧化酶途径对机体产生保护作用。
在细菌吞噬实验中,大肠杆菌MG1655 被注射到合浦珠母贝体内后,其浓度持续降低,一段时间后被彻底清除,说明MG1655 会引发合浦珠母贝的天然免疫反应[22]。在本研究中,大肠杆菌MG1655感染合浦珠母贝后,pfser1基因在外套膜边缘膜中被诱导表达,表明其参与了合浦珠母贝的天然免疫过程。在哺乳动物和节肢动物的研究中,天然免疫系统启动后,体内一些通路产生激活反应,并产生一些效应分子。而机体内蛋白酶的释放必须精确控制,所产生的过多蛋白酶对机体自身造成损害,这时需丝氨酸蛋白酶抑制因子来调控,确保蛋白酶维持正常水平[23-25]。Serpin 型蛋白多以自杀性底物的方式参与到蛋白酶的调解反应中,并以一种不可逆方式抑制特定的靶蛋白[26]。根据实验中的pfser1基因的表达特征,判断在合浦珠母贝受到大肠杆菌MG1655 刺激后可迅速开启天然免疫过程,在3 h内启动天然免疫的联级反应。随后丝氨酸蛋白酶抑制因子会得到大量表达,以调控体内天然免疫的精细过程,避免自身机体受到伤害。在6 h 及之后,丝氨酸蛋白酶抑制因子基因表达量降低,但相对0 h的对照组仍有较高表达量,这表明合浦珠母贝体内在天然免疫过程启动后,较长时间都会提高pfser1基因的表达量,可能结合降低体内因抗菌产生的蛋白酶。外套膜间液位于珠母贝贝壳和外套膜之间,在贝壳形成中起重要作用,研究发现间液蛋白在珠母贝贝壳形成及珍珠层与棱柱层的相互转化中起着关键作用,且在体外对CaCO3的成核及沉积起双重作用[26]。本研究中,在间液受到大肠杆菌MG1655刺激后,外套膜边缘膜分泌的丝氨酸蛋白酶抑制因子的表达变化,较闭壳肌受到大肠杆菌MG1655 刺激下,反应更为强烈,对细菌的刺激有更快的响应机制。
软体动物在贝壳损伤后有自我修复能力。贝壳受到损伤后,软体动物可启动一些途径,实现贝壳的再生过程,这个过程和贝壳的形成过程类似,在有机基质的指导下,碳酸钙晶体可在损伤的部位有序沉淀,形成新的贝壳[28]。本研究中,检测到合浦珠母贝丝氨酸蛋白酶抑制因子基因在贝壳损伤修复过程中表达发生了变化,表达量先高后低。相较于细菌感染实验,pfser1基因表达虽检测到升高,但升高幅度小得多。在48~ 96 h 实验组内,pfser1的表达量持续低于对照组,且在48 h 组出现最低值,约是正常表达量的0.2%。结合丝氨酸蛋白酶抑制因子的功能特征推测,这可能是合浦珠母贝生物矿化过程中,前期需要抑制特定的蛋白酶,而在后期(尤其在48 h 左右)需要分泌过量的特定蛋白酶时参与生物矿化过程,因此48 h 组丝氨酸蛋白酶抑制因子基因的表达显著降低。基于这些结果,认为丝氨酸蛋白酶抑制因子pfser1基因也参与了合浦珠母贝的生物矿化相关的调节过程。
4 结论
本研究克隆得合浦珠母贝丝氨酸蛋白酶抑制因子pfser1基因,研究其序列表达特征,以及其在细菌吞噬实验和贝壳损伤修复实验中的表达情况。结果表明,其参与了合浦珠母贝天然免疫过程,并与生物矿化过程有一定联系。本研究为探索合浦珠母贝天然免疫机理,以及天然免疫与生物矿化之间的关系奠定了基础。

