如何挖掘GEPIA数据库中研究数据并生成分析结果表达图
闫小妮,田国祥,潘振宇,杨津,柳青青,吕军,4
随着信息时代的发展,大数据战略思维被逐渐熟知,大数据源于数据的获取积累、数据的汇聚共享及数据的挖掘利用,基于大数据技术的挖掘和分析在生物医学研究和临床诊治中发挥了重大作用。大数据分析的运用促进了癌症基因组学研究的发展,从本质上来看,癌症的原因是细胞内部的基因差异表达所引起的一种基因疾病。近年来多基因诊断及风险评估模型被美国临床肿瘤学会(ASCO)、美国国家综合癌症网络(NCCN)等多个权威诊疗指南所采用,并开展了临床应用[1]。当前受到广泛关注的精准医疗主要依赖于基因测序技术,如何高效处理和分析海量病人的基因测序数据,从中获取个性化的癌变驱动信息成为实现肿瘤精准诊疗的关键和难点问题。
随着众多公共数据库的建立和开放,越来越多的研究者可以接触到测序数据。GEPIA(Gene Expression Profiling Interactive Analysis),即基因表达谱数据动态分析,是一个新开发的用于癌症和正常基因表达谱分析和交互分析的web服务器,填补了癌症基因组学大数据信息缺口,帮助临床科研爱好者更高效的利用公共数据资源。本文通过介绍GEPIA数据库,让GEPIA网页式的交互界面使用更简单,促进研究领域的数据挖掘、科学讨论和治疗的发现。
1 GEPIA简介
GEPIA官方网站:http://gepia.cancer-pku.cn/index.html,首页如图1。该网站由北大张泽民教授团队开发[2]。GEPIA使用的RNA-Seq数据集基于UCSC Xena项目(http://xena.ucsc.edu),该项目由标准管道计算,分析来自TCGA和GTEx项目的9736个肿瘤和8587个正常样本的RNA测序表达数据。TCGA在33种癌症类型中产生了9736个肿瘤样本,而这个项目只提供了726个正常样本。肿瘤与正常数据之间的不平衡会导致各种鉴别分析的效率低下。因此GEPIA还整合了GTEx的数据,GTEx项目产生了8000个正常样本的RNA测序数据。同时,UCSC Xena项目使用标准管道重新计算了TCGA和GTEx原始RNA-Seq数据,这使得两个数据集兼容。因此,可以将TCGA和GTEx数据进行整合,进行非常全面的表达分析。TCGA和GTEx的表达量数据都是在同一个pipeline下重新算出来的,可以直接进行比较。从架构上来讲,GEPIA用MySQL创建数据库,主题分析流程由R和PerL完成,用php实现网页式交互展示。GEPIA提供了关键的交互式分析,包括肿瘤/正常差异表达谱分析、剖面绘制、根据肿瘤类型或病理分期进行分析、患者生存分析、相似基因检测、相关性分析和降维分析等分析模块,同时还提供快速定制功能。基因表达通常由人体图和柱图点图来表示,如图2,人体基因表达图即在人体中肿瘤和正常样本的中位表达图;点图和柱图即所有肿瘤样本和配对正常组织的基因表达谱(点图:每个点代表样本的表达式;柱图:柱的高度代表某一肿瘤类型或正常组织的中位表达)。基因表达谱GEPIA生成多个癌症类型和成对的正常样本的基因表达谱点图,每个点代表一个不同的肿瘤或正常样本。

图1 网站首页界面

图2 基因表达图
2 检索研究资源
从搜索栏可以看出,GEPIA网站的分析主要有三个版块:单基因分析、肿瘤类型分析、多基因分析。
2.1 单基因分析(Single Gene Analysis)即基因在不同肿瘤中的概况,和大部分数据库一样,输入基因名的时候会出现下拉菜单,方便准确定位基因名。在输入的基因名称字段处,输入一个基因符号或基因ID并点击“GoPIA”按钮即可搜索感兴趣的基因。也可以从网页右上角的GoPIA进入,然后从Quick Search处输入,进行检索。我们以基因CCT6A为示例检索,两种方法都会跳转到同样的界面下如图3。检索出来的页面中有不同的标签,可以针对检索的基因进行各种分析:表达差异分析、生成表达DIY(基因表达谱、盒线图表达式、病理阶段表达图、多个基因比较)、生存分析(生存图、多数差异生存基因)、相似的基因检测、相关分析、主成分分析。
2.2 肿瘤类型分析(Cancer Type Analysis)对于一种癌症类型,GEPIA提供了这些分析:差异基因分析(允许用户在给定数据集上应用定制的统计方法和阈值,动态获取差异表达的基因及其染色体分布)和最重要的生存基因(即给定一种癌症类型,搜索与患者生存最相关的基因)。

图3 GoPIA首页界面
2.3 多基因分析(Multiple Gene Analysis)即多个基因比较,这个特性提供了基于给定基因列表的表达矩阵图。该函数为给定的TCGA和/或GTEx表达数据集提供两两配对的基因表达相关性分析。给定一个基因列表,GEPIA还可以使用定制的TCGA和/或GTEx表达数据进行主成分分析(PCA)。
3 相关分析结果表达
3.1 差异表达分析允许用户对给定数据集应用自定义统计方法和阈值,动态获取差异表达的基因及其染色体分布。对于每一种癌症类型,我们选择对给定基因表达水平最高的10%的肿瘤样本。为了进行比较,我们选择相同数量的对同一基因表达水平最高的正常样本。我们根据表达水平对肿瘤和正常样本进行排序,并将肿瘤样本在前50%排名列表中的百分比作为百分比值进行计算。表达数据首先进行log2(TPM+1)的缩放,log2FC定义为中位(肿瘤)-中位(正常)。默认情况下,表达的基因是那些通过以下阈值的基因:log2FC>1,百分比>0.9。方差分析和LIMMA方法认为,|log2FC|值较高,q值低于预先设定阈值的基因为差异表达基因。在前10个选项中,log2FC值较高、百分比值高于阈值的基因被认为是过表达基因。点击差异基因列表按钮:GEPIA将根据输入参数生成差异表达基因列表[默认情况下,按照log2FC降序排列],如图4。点击Plot按钮:GEPIA将生成一个染色体分布图,染色体上过表达的基因用红线表示,过表达的基因用绿色表示(图5)。

图4 差异基因列表
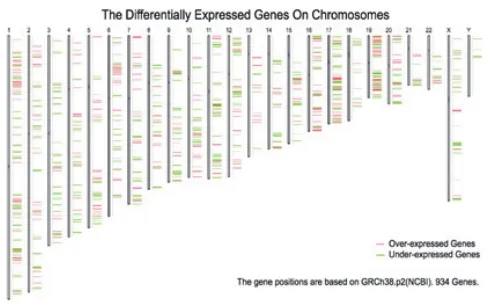
图5 染色体分布图
3.2 DIY表达GEPIA根据选定的数据集和统计方法,根据肿瘤类型或病理分期绘制给定基因的表达谱。GEPIA生成多个癌症类型和成对的正常样本的基因表达谱点图,每个点代表一个不同的肿瘤或正常样本。在方差分析和LIMMA选项中,|log2FC|值高于预设阈值,q值低于预设阈值的基因被认为是差异表达基因。在前10个选项中,log2FC值较高、百分比值高于阈值的基因被认为是过表达基因。因此,只有过表达的基因才会出现在列表和染色体图中(见图6)。

图6 基因表达谱点图
GEPIA可生成带有抖动的Box plot来比较几种癌症类型的表达(为了获得最佳的视觉效果,我们推荐1-4种癌症类型)。数据集选择/数据集:在数据集选择字段中选择感兴趣的癌症类型,然后单击add在数据集字段中构建数据集列表。手动输入用逗号分隔的癌症类型(如ACC、BRCA、BLCA)也是可以的。图形的x轴将遵循数据集的顺序。肿瘤颜色参数可设置肿瘤数据集的方框颜色。法线颜色参数可设置法线数据集的框色。Log Scale可选择使用线性还是log2(TPM+1)转换的表达式数据进行绘图。抖动大小参数可设置框的抖动大小。差异阈值:|log2FC|截止设置自定义折改阈值,P值截止设置自定义P值阈值。匹配正态数据可选择TCGA正态+ GTEx正态,或仅选择TCGA正态进行差分分析和绘图。本文的差异分析基于所选数据集(TCGA肿瘤 vs. TCGA正常+GTEx正常或TCGA肿瘤 vs. TCGA正常)。差异分析方法为单因素方差分析,以疾病状态(肿瘤或正常)为变量计算差异表达:基因表达~疾病状态将表达数据先转化为log2(TPM+1)进行差异分析,将log2FC定义为中位(肿瘤)-中位(正常)。|log2FC|值较高,q值低于预设阈值的基因被认为是差异表达基因。点击Plot按钮:GEPIA将显示一个基于用户自定义输入参数的基因表达框图(图7)。
3.3 病理分期剖面图在分期图中绘制了病理分期的基因表达情况。GEPIA可根据选定的数据集和统计方法,按照癌症类型或病理分期绘制给定基因的表达谱[3]。自定义参数设置:在数据集选择字段中选择感兴趣的一种或多种癌症类型,然后单击add在数据集字段中构建数据集列表,或手动输入用逗号分隔的癌症类型(如COAD、READ);在Log Scale栏选择使用线性还是log2(TPM+1)转换的表达式数据进行绘图;在使用大分期栏选择病理大分期或亚分期作图;在情节色彩栏设置小提琴图的颜色。差异基因表达分析方法为单因素方差分析,以病理分期为变量计算差异表达:基因表达~病理分期将表达数据首先转化为log2(TPM+1)进行差异分析。点击Plot按钮:GEPIA将根据用户自定义输入生成一个基因表达阶段图,如图8。如果点击code还可以给出代码。
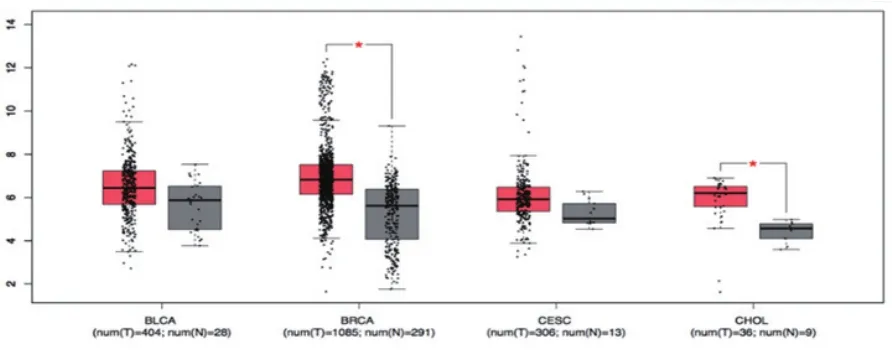
图7 匹配TCGA常规数据和GTEx数据的箱线图
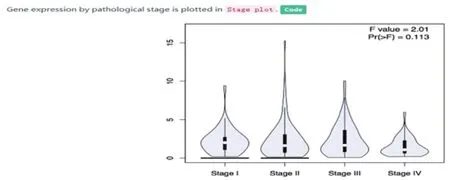
图8 根据患者病理分期生成表达小提琴图
用户可以通过箱线图比较一个基因在多种癌症中的表达情况,也可以通过矩阵图比较多个基因在多种基因比较中的表达情况。多基因表达比较这一特征提供了基于给定基因列表的表达矩阵图。每个方块中的颜色密度表示给定组织中某个基因的中值表达值,将所有块中的最大中值标准化。同一肿瘤或正常组织中的不同基因可以在一个图中进行比较。在参数设置页面输入感兴趣的基因列表,用逗号分隔的基因(如ERBB2、EGFR)也可以手工输入。在数据集字段中选择感兴趣的癌症类型,然后单击add或all在组织顺序字段中构建数据集列表。Log Scale栏选择使用线性还是log2(TPM+1)转换的表达式数据进行绘图。匹配正常数据可以仅选取TCGA肿瘤、TCGA肿瘤+TCGA正常+GTEx正常或TCGA肿瘤+TCGA正常绘制。点击Plot按钮:GEPIA将根据用户自定义输入参数生成一个表达式矩阵图,如图9。
3.4 生存分析在生存分析中,可以识别出与患者生存关系最显著的基因,GEPIA根据基因表达进行总生存(OS)或无病生存(DFS,也称为无复发生存和RFS)分析[4]。GEPIA使用Log-rank检验,可以调整队列阈值,并使用基因对。生存图中还可以包含cox比例危险比和95%置信区间信息。可以搜索与患者生存最相关的基因。自定义参数设置在基因归一化栏可设置基因场中用于归一化的基因;在方法栏可选择OS或DFS生存方法;轴向单位可选择绘制月或日单位;在数据集选择字段中选择感兴趣的一种或多种癌症类型,然后单击add在数据集字段中构建数据集列表,或者手工输入用逗号分隔的癌症类型(如COAD、READ)。颜色反转栏选择是否反转默认颜色;分组截止选择合适的表达阈值进行高表达组和低表达组的分割;Cutoff-High(%)即表达水平高于此阈值的样本为高表达队列;Cutoff-Low(%)即表达水平低于此阈值的样本为低表达队列,如图10。点击Plot按钮:GEPIA将根据用户自定义的输入参数生成一个生存图,如图11。

图9 矩阵图

图10 生成分析图参数设置界面
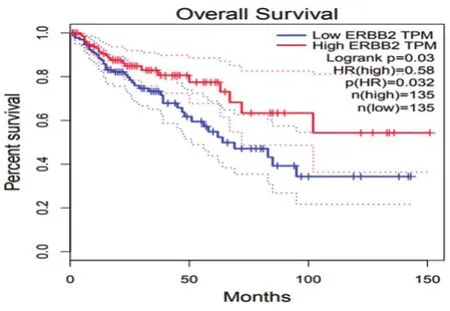
图11 生存曲线图
3.5 最重要的生存基因数据集选择利用公共数据库挖掘肿瘤发生过程的关键基因,为后续的功能验证奠定基础。在公共数据库中挖掘肿瘤关键基因能为研究者提供生物信息学信息,能减少研究范围。自定义选择一种感兴趣的癌症类型,再选择OS或DFS生存方法,分组截止选择合适的表达阈值进行高表达组和低表达组的分割。点击列表按钮:GEPIA将生成一份给定癌症类型的100个最重要生存基因列表,如图12。
3.6 相似基因检测此功能识别具有相似表达模式的输入基因和选定数据集的基因列表。点击列表按钮:GEPIA会生成一个由Pearson相关系数(PCC)排序的具有相似表达模式的基因列表,如图13。
3.7 GEPIA还提供一组给定的TCGA和/或GTEx表达数据的两两相关基因分析相关性分析函数利用Pearson、Spearman和Kendall等方法,对给定的TCGA和/或GTEx表达数据进行两两相关的基因表达分析。一个基因可以被另一个基因归一化。GEPIA使用非对数比例尺进行计算,使用对数比例尺轴进行可视化。先输入感兴趣的基因A,【x轴】输入感兴趣的基因B,【对于y轴】基因归一化设置用于归一化基因A和基因b的基因。相关系数栏选择设置计算相关系数的方法。在TCGA Tumor、TCGA Normal或GTEx字段中选择感兴趣的cancer types,点击add在Used Expression Datasets字段中构建数据集列表。也可以手工输入逗号分隔的癌症类型(如COAD肿瘤、READ肿瘤)。相关分析基于数据集列表。点击Plot按钮:GEPIA会生成一个相关分析结果的散点图,如图14。
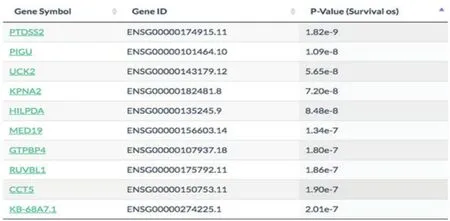
图12 最重要的生存基因
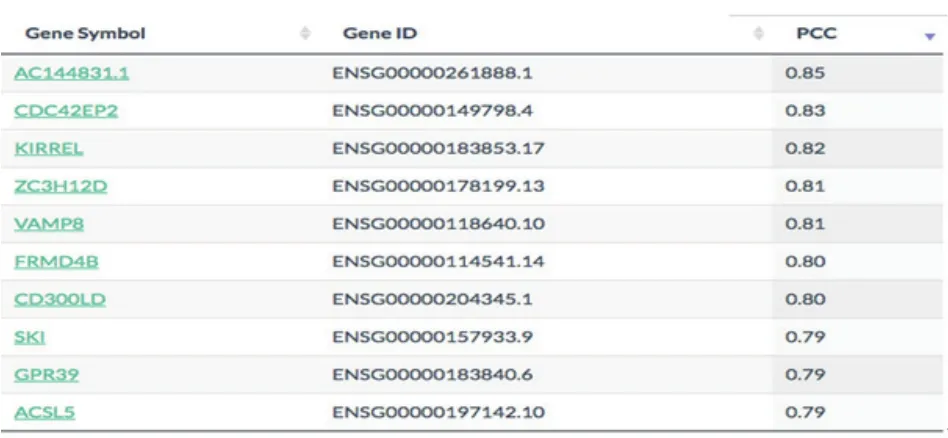
图13 相似基因检测

图14 相关性分析
3.8 GEPIA同时在PCA中提供多种基因和癌症类型的主成分分析并通过2D或3D图显示结果。对于给定的基因列表,GEPIA使用定制的TCGA和/或GTEx表达数据进行主成分分析(PCA)。首先,GEPIA给出了前三个主成分(PC)的3D图,并为每个PC解释的方差生成条形图。其次,GEPIA基于用户指定的pc呈现2D或3D图形。第一步点击列表按钮:GEPIA将根据相关分析结果生成3D散点图和方差分布,点击右上角的相机图标,可以将3D图形保存为.png文件。第二步点击Plot 2D按钮:GEPIA会生成一个相关分析结果的2D散点图,如图15。

图15 主成分分析2D、3D显示结果图
4 分析结果下载及编辑
结果下载可以通过单击结果旁边的按钮下载PDF和SVG,下载的PDF和SVG图形可以由adobeillustrator编辑。(Youtube面向全球用户,Tencent面向中国用户)。
5 总结
目前,生命科学和医学已进入大数据驱动的颠覆性变革时代。基因表达谱在生物学、医学和药物研发等多个领域发挥着重要作用[5]。GEPIA是一个由国人开发的公共数据库,通过使用GEPIA数据库,实验生物学家可以轻松地探索TCGA和GTEx数据集,找到问题的答案,并测试他们的假设。在差异分析和表达谱中,用户很容易发现差异表达的基因。随着基因检测的应用,逐渐改变了以往免疫组化为主的肿瘤预后评估及治疗方案选择的模式,并对肿瘤更为准确地分型,对预后评估及治疗具有更为重要的指导意义[6]。在GEPIA的所有功能中,最受欢迎的三大功能是:生存分析、相关性分析和盒式图绘制。
本文通过介绍GEPIA数据库的架构及数据提取、分析方法,旨在帮助对基因公共数据感兴趣的研究者了解GEPIA数据库、熟悉网站架构,高效便捷地应用该数据库资料进行科学研究,减少科研工作者在数据获取、分析制图方面所花费的时间和精力,提供高效、便利、明晰的数据资料获取途径,从而减轻科研工作者压力,提高工作效率。

