胃癌化疗抵抗基因PMP22下游的生物信息学机制
汪圣毅,张永红,闫亚飞,李旭升,程 彦,李永翔
化疗(chemotherapy,CTx)可控制非治愈因素,改善无法手术切除胃癌患者的预后[1],可降低循环肿瘤细胞数,降低淋巴结、局部和腹膜的复发,附加药物可逆转免疫细胞的功能,抑制胃癌细胞的增殖迁移[2]。但CTx亦使癌细胞发生适应性改变,产生耐受或抵抗,与癌细胞基因表达和相关通路的变化有关。外周髓磷脂蛋白22(peripheral myelin protein 22,PMP22)与CTx抵抗、肿瘤发生有关[3],PMP22表达干预可致下游基因表达变化[4],其上调与胃癌细胞抵抗顺铂的作用有关[5],但其下游机制尚不明确。生物信息学方法可揭示疾病基因模块和网络,应用广泛[6]。该研究采用生物信息学方法,比较短发夹核糖核酸敲减和未敲减PMP22的下游差异表达基因(defferentially expressed genes, DEGs),分析其相关通路,为胃癌CTx抵抗的分子机制研究提供参考。
1 材料与方法
1.1 细胞、试剂、平台基因表达数据库(gene expression omnibus,GEO)GSE(基因表达数据库系列,GEO series)94714胃癌细胞系MGC-803、慢病毒载体(plasmid lentiviral vector,pLKO.1)购自中科院细胞所。PMP22的短发夹核糖核酸(short hairpin RNA,shRNA)共2个,shRNA1: CCAAACTCAAACC AAACCAAA,shRNA2:CGGTGTCATCTATGTGATCT T,Lipofectamine 3000(美国Invitrogen公司)将含有shRNA1、shRNA2的慢病毒转染至293T细胞扩增,转染筛选MGC-803,美国安捷伦公司芯片平台测试基因表达谱[5]。
1.2 表达谱数据PMP22敲减和未敲减组的差异基因表达谱,取自美国国立生物技术信息中心(national center for biotechnology information,NCBI)的GEO(https://www.ncbi.nlm.nih.gov/geo),由Cai et al[5]提交,样本为:1:数据库样本(GEO sample, GSM)GSM2481329;2:GSM2481330;3:GSM2481331;4:GSM2481332;5:GSM2481333;6:GSM2481334,1~3为A组,PMP22_shRNA2敲减PMP22基因的表达,4~6为B组,未抑制PMP22基因的表达。
1.3 差异基因数据库R语言(GEO to R, GEO2R)(https://www.ncbi.nlm.nih.gov/geo/geo2r/?acc=GSE94714)定义A、B组别,美国国家标准学会(American national standards institute,ANSI)格式保存所有差异基因至.txt文件,导入Excel 2016筛选,筛选条件:① 校正P<0.01;② 倍数变化(fold change,FC)值≥10或≤-10,计算logFC:log2(A/B)=log2(A)-log2(B),分别为±3.321 928。
1.4 富集和通路分析差异基因于注释、可视化和综合发现数据库(database for annotation, visualization and integrated discovery,DAVID)网站(https://david.ncifcrf.gov/),分析基因本体(gene ontology,GO)的生物过程(biological process,BP)、分子功能(molecular function,MF)、细胞组分(cellular component,CC),分析京都基因和基因组百科全书(kyotoencyclopedia of genes and genomes,KEGG)的通路。选BP、MF、CC中P值最小的前6位,绘制复合条图。KEGG结果绘制泡泡图。
1.5 蛋白质相互作用DAVID转换基因名为官方基因标识,文本正则表达式截取基因名称,导入互作基因检索工具(search tool for the retrieval of interacting genes,STRING)数据库(http://string-db.org/),Homo Sapiens获蛋白质-蛋白质相互作用(protein-protein interaction,PPI)的网络。数据导入Cytoscape 3.6.1,分子复合检测(molecular complex detection,MCODE)插件行模块分析,单一模块中的基因导入DAVID 6.8行通路分析。
1.6 关键基因筛选PPI数据导入Cytoscape 3.6.1,CytoHubba插件筛选关键基因,用最大集团中心性(maximal clique centrality,MCC)的拓扑算法。
1.7 癌症药物敏感性基因组学数据库验证logFC绝对值由高到低排序,前10位的基因和MCC算法获得的关键基因,导入癌症药物敏感性基因组学(genomics of drug sensitivity in cancer,GDSC)数据库,分析与胃癌CTx敏感性的关联性。
1.8 统计学处理差异表达基因(DEGs)为GEO中R语言limma包完成分析,数据矩阵线性拟合,本雅米尼·霍赫贝格错误发现率(false discovery rate,FDR)校正。
2 结果
2.1 样本数据特征数据归一化的中位数均为5.4,均数为6.21,中位数位于同一水平,标准化特征良好,见图1。
图1 样本基因表达数据的归一化结果
1:GSM2481329;2:GSM2481330;3:GSM2481331;4:GSM2481332;5:GSM2481333;6:GSM2481334
2.2 差异基因GEO2R结果获34 183个与GEO ID对应的序列。AdjustedP<0.01,logFC=3.321 928,筛选到368个对应的ID,删除无基因库登录号(gene bank accession number,GB_ACC)的30行。GB_ACC转换基因名,获315个DAVID IDs,Excel查重删除1个重复,vlookup函数将基因名对应至差异筛选表,找回logFC值,countif函数计算获上调基因283个,下调31个。logFC绝对值最大的前10位见表1。
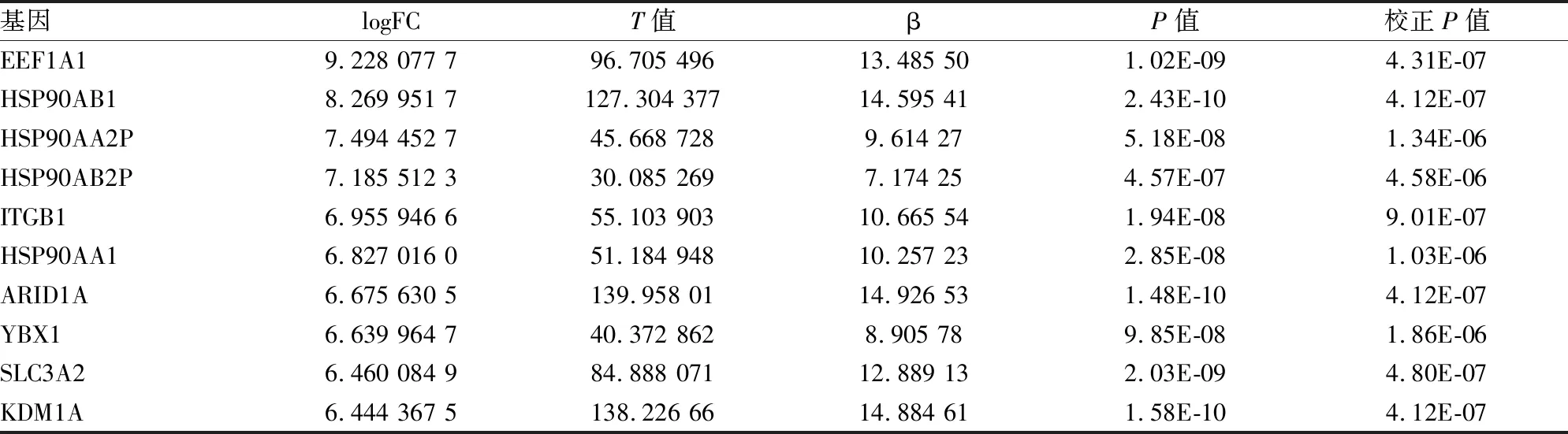
表1 logFC绝对值前10位的差异表达基因
EEF1A1:真核翻译延长因子1α1;HSP:热休克蛋白;ITGB1:整合素亚单位β1;SLC3A2:印记基因家族3成员2;KDM1A:赖氨酸脱乙基酶1A
2.3 GO和KEGG分析富集到BP、MF、CC的基因分别有271、277、283个,为注释基因的86.3%、88.2%、90.1%,140个基因(44.6%)参与KEGG通路。BP、MF、CC中P值最小的前6位,换算成-log10(P值)为纵坐标,横坐标各分类的基因计数为右侧y轴绘图,见图2。CC中富集在核、膜、核质、细胞外外泌体、细胞黏着连接、蛋白复合体等的基因差异显著,见图2A;BP中参与细胞-细胞黏附过程的基因P值最低,参与基因数为19,见图2B;MF中P值最低的为多聚腺嘌呤核糖核苷酸结合,参与基因数目最多的为蛋白质结合,见图2C。富集前5位的通路为细胞周期、氨基酸的生物合成、无翅型整合位点家族(wingless-type integration site family, Wnt)、转化生长因子β、半胱氨酸和蛋氨酸代谢通路,见图2D。
2.4 蛋白质相互作用PPI数据导入Cytoscape,MCODE聚类,节点连接度(degree)值=2,节点(node)评分值=0.2,k-分(core)=2,最大深度(max.depth)=100,获11个模块,评分前4的模块4个,评分分别为9、7、5、4.909,Node数目分别为9、7、7、12,边(Edge)数目分别为36、21、15、27,见图3。单个模块中的基因行通路分析,主要与剪接体、泛素介导的蛋白水解共2个KEGG(P<0.01)和3个反应组(REACTOME)(P<0.05)通路关联,见表2。
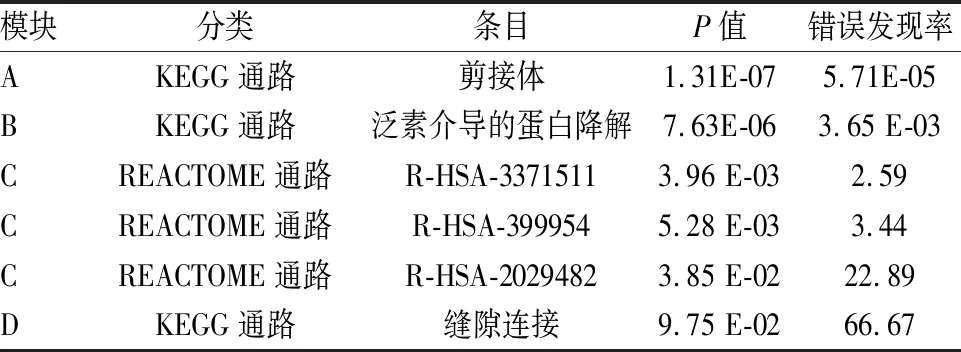
表2 PPI网络模块中的基因富集通路
R-HSA:反应组人类通路标识码
2.5 PPI中的关键基因MCC算法计算前10个关键基因为:肽基脯氨酰异构酶E(peptidylprolyl isomerase E,PPIE)、不均一核核糖核蛋白A1(heterogeneous nuclear ribonucleoprotein A1,HNRNPA1)、Y盒结合蛋白1(Y-box binding protein 1,YBX1)、不均一核核糖核蛋白K(heterogeneous nuclear ribonucleoprotein K,HNRNPK)、不均一核核糖核蛋白A2B1(heterogeneous nuclear ribonucleoprotein A2/B1,HNRNPA2B1)、死亡盒解旋酶9(DExH-Box helicase 9,DHX9)、U6小核糖核酸相关Sm样蛋白(U6 snRNA-associated sm-like protein,LSM4)、小核核糖核蛋白多肽N(small nuclear ribonucleoprotein polypeptide N,SNRPN)、多聚谷氨酰胺结合蛋白1(polyglutamine binding protein 1,PQBP1)、热休克蛋白90α家族A类成员1(heat shock protein 90 alpha family class a member 1,HSP90AA1),评分分别为41 970、40 395、40 372、40 352、40 346、40 341、40 324、40 324、40 321、2 336,颜色排序见图4。
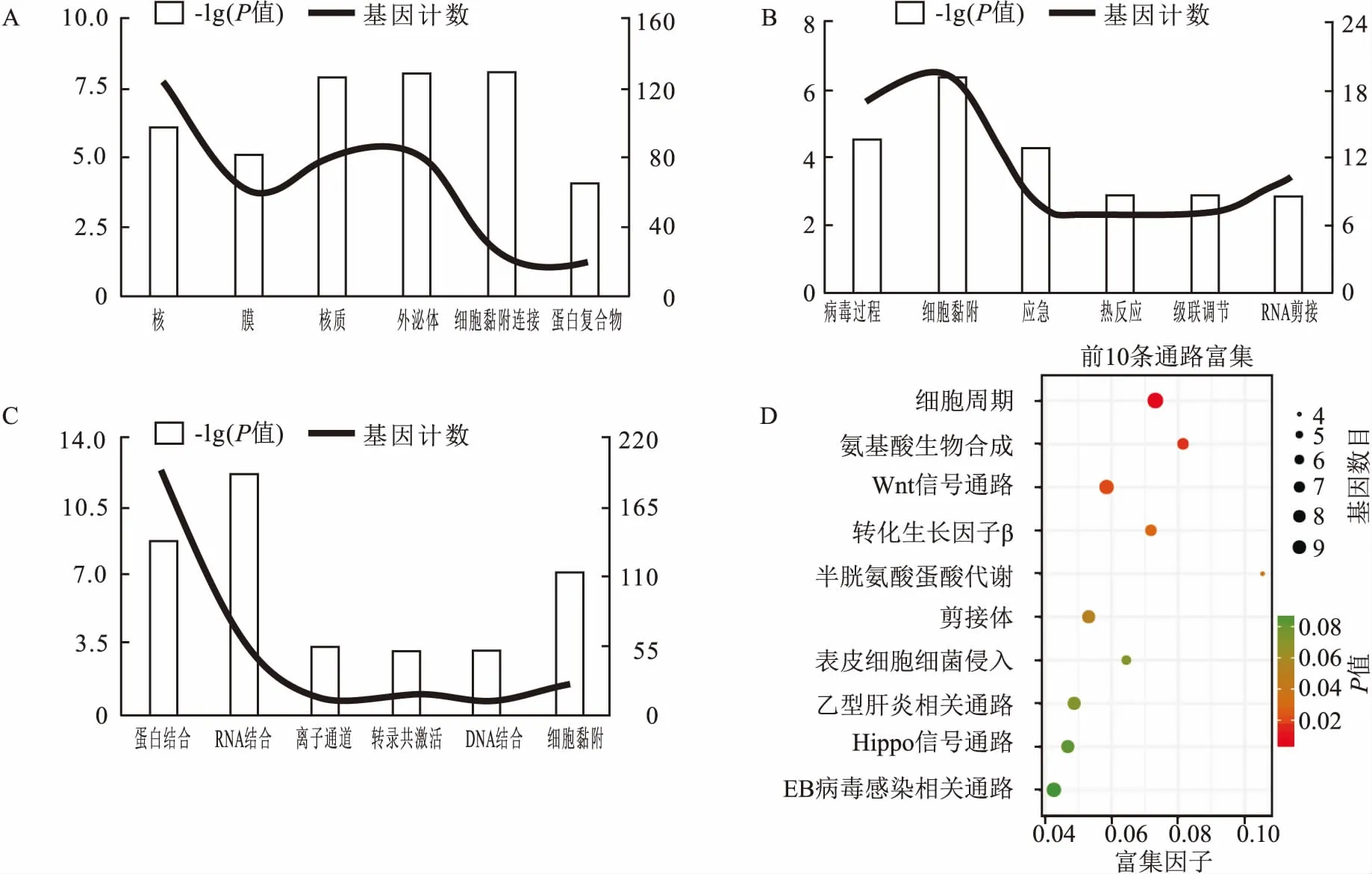
图2 基因本体(GO)分析
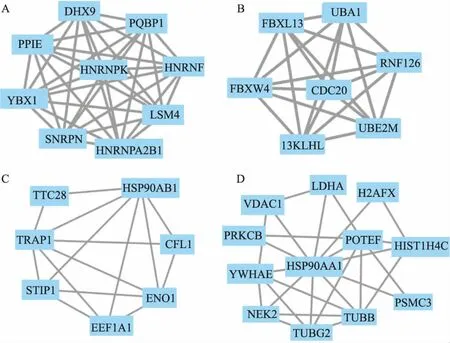
图3 PPI网络的聚类模块
A:评分第1的9节点网络结构;B:评分第2的7节点网络结构;C:评分第3的7节点网络结构;D:评分第4的12节点网络结构
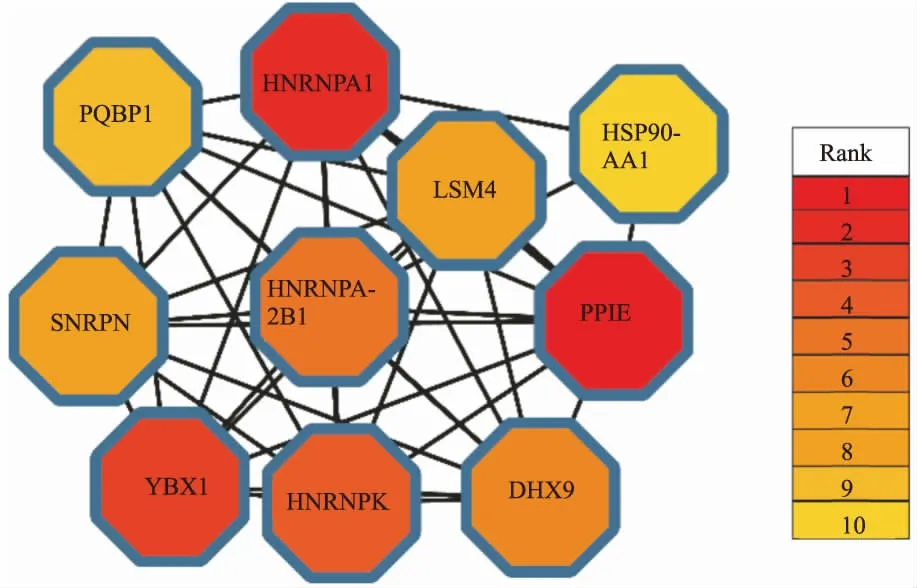
图4 MCC算法的关键基因及其相互作用关系
2.6 差异和关键基因与药物敏感性的关系GDSC分析显示,富含腺嘌呤胸腺嘧啶蛋白质1A交互域(AT-rich interactive domain-containing protein 1A,ARID1A)基因突变与胃腺癌对CTx药物的敏感性有关,顺铂作用ARID1A突变的胃癌细胞,半抑制浓度(half maximal inhibitory concentration,IC50)值升高(P=0.050 016);卵巢癌、结直肠腺癌ARID1A基因突变时,顺铂作用的IC50值降低,P值分别为0.005、0.047,见图5。DHX9基因突变与多种癌细胞的药物敏感性有关,但未见与胃腺癌药物敏感性的关联。其他top 10差异基因和MCC筛选的关键基因尚无GDSC数据库资料。
3 讨论
PMP22下游基因主要富集在细胞核、膜、核质、胞外外泌体、黏着连接、蛋白复合体等成分,参与细胞-细胞黏附过程、多聚腺嘌呤核糖核苷酸结合、蛋白质结合,参与细胞周期、氨基酸生物合成、Wnt和转化生长因子β、半胱氨酸蛋氨酸代谢等通路。PPI网络模块主要通过剪接体、泛素介导的蛋白水解、热休克因子1活化、轴突导向因子3A p21活化激酶依赖性轴突排斥、肌动蛋白动力调节形成吞噬杯、缝隙连接等通路发挥作用,与PPIE、ARID1A等有关。
PMP22敲减后ARID1A上调,顺铂敏感性增强,与ARID1A阳性表达胃癌患者无病生存率提高[7]的结果一致。GDSC分析见胃癌细胞顺铂敏感性与ARID1A突变的关联P值略大于0.05,可能与突变组的样本量少有关。DHX9突变与胃腺癌以外的其他多种癌细胞的顺铂敏感性有关。Top10差异基因、关键基因中的其他基因,GDSC未见记录,可能是PMP22下游尚未明确的新基因。
GO和KEGG分析显示,外泌体、细胞黏着粘附、蛋白质结合功能均与PMP22相关CTx敏感性有关。外泌体传递miR-155至敏感细胞介导乳腺癌的CTx抵抗[8]。外泌体合成分泌抑制使去势抵抗的前列腺癌细胞的CTx敏感性增强。细胞黏着连接因E-cadherin丢失而崩解,肿瘤细胞会避开失巢凋亡,CTx抵抗性增强[9]。细胞黏附生物过程的基因P值最小,表明与CTx抵抗显著关联。细胞间黏附分子-1中和抗体减少Jurkat细胞与间充质干细胞的黏附,减少细胞间线粒体转运,使Jurkat细胞的CTx敏感性增强[10]。分子功能中的蛋白质结合功能,通过结合互作调节CTx敏感性。分化抗原133与磷脂酰肌醇3激酶 (phosphatidylinositide 3-kinases,PI3K)PI3K-p85的直接作用可活化PI3K/PKB(蛋白激酶B)通路,导致胃癌细胞的多种药物抵抗[11]。通路方面,胃癌细胞CTx抵抗与Wnt通路的关联性已有报告[12],与本文的KEGG分析结果相似,其余未见文献报道的通路,如泛素化降解与胃癌CTx抵抗的关系,是未来研究的方向。
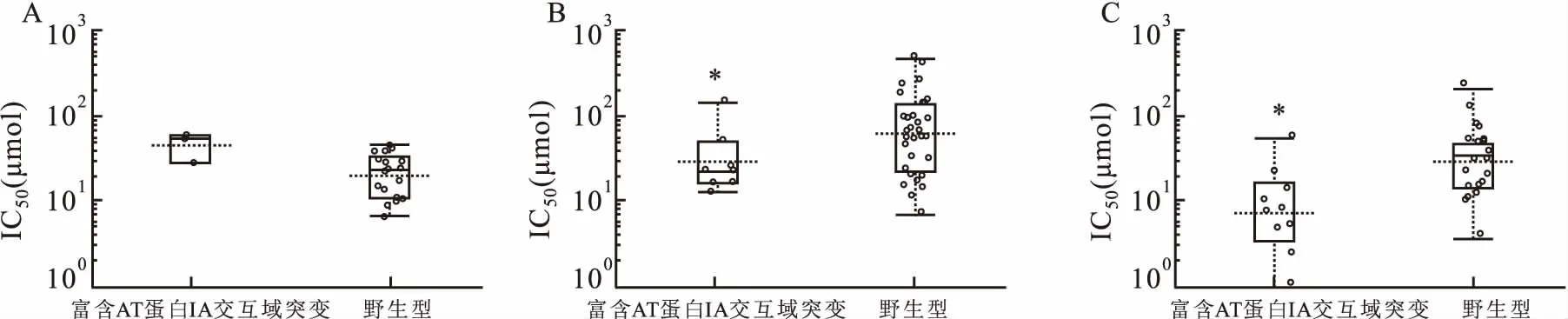
图5 ARID1A基因突变与顺铂敏感性的关系
PPI网络以系统视角揭示CTx抵抗的机制,本研究构建的PPI网络中提取4个亚模块,获10个关键基因,其中的DHX9、应激诱导磷蛋白1(STIP1)基因突变与CTx抵抗或敏感性关联,GDSC得到验证,尚无验证的可能是CTx抵抗相关新基因。PPI模块涉及的剪接体、泛素介导蛋白水解通路与骨肉瘤[13]、结直肠癌的发生发展相关联[14],睡美人转座子突变研究[15]显示,泛素介导蛋白水解和细胞黏着连接也是胃癌中的失控通路。
本研究显示了PMP22下游参与CTx抵抗的网络成员,作为胃癌CTx抵抗的基因标志,为CTx抵抗精准治疗的靶点研究提供了参考。

