长链非编码RNA与mRNA在胰腺癌中的差异表达及其预后价值
魏 超,张 晓*,高 杰
(东南大学公共卫生学院,江苏 南京 210009)
胰腺癌(pancreatic cancer,PC)是一种恶性消化系统肿瘤,全球每年因患胰腺癌而死亡的患者超过200万例[1-2],在我国胰腺癌的发病率也位居恶性肿瘤中的第10位,死亡率高居第6位,且每年均呈上升趋势[3],其发病隐匿、早期诊断困难、易发生转移、预后较差[4]。因此寻找更有效的临床指标对胰腺癌患者的诊断和治疗显得非常重要。
长链非编码RNA(long non-coding RNA,lncRNA)是指一组核苷酸数量大于200的RNA,缺乏蛋白质编码潜力[5]。研究发现lncRNA可以通过促进或抑制癌症的发展进而在诊断和治疗肿瘤的过程中发挥作用[6-11]。
近年来,生物信息学的快速发展为诊断和挖掘疾病治疗靶点提供了一个新的方向。因此,本文试图通过使用生物信息学方法,从基因芯片表达汇编(gene expression omnibus,GEO)数据库和癌症基因组图谱(the cancer genome atlas,TCGA)数据库中下载获得胰腺癌基因相关数据,对其进行分析整合,从而为诊断和治疗胰腺癌提供新的治疗靶点和分子标志物。
1 材料与方法
1.1 数据材料
本研究在美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)的GEO数据库中检索胰腺癌芯片数据。选用样本量较多的GSE15471、GSE16515、GSE71989数据集下载并进行后续分析。其中GSE15471包括39例胰腺癌和39例癌旁正常标本,GSE16515包括37例胰腺癌和36例癌旁正常标本,GSE71989包括13例胰腺癌和8例癌旁正常标本。
1.2 筛选差异表达基因和lncRNA
通过Gencode数据库V15(http://www.gencodegenes.org/releases/current.htm l)进行lncRNA注释,利用blast程序,通过U133PLUS 2.0技术将lncRNA数据库与mRNA测序数据进行比对,获得lncRNA;运用GEO数据库中自带的GEO 2R在线分析软件分析GSE15471、GSE16515、GSE71989中胰腺癌与正常组织的差异表达基因和 lncRNA,筛选标准为|log2FC|>2且 P<0.05。选取在3个数据芯片中的交集mRNA和lncRNA。
1.3 差异表达基因功能及通路富集分析
利用生物学信息注释数据库DAVID(https://david.ncifcrf.gov/)对差异基因进行基因本体(gene ontology,GO)生物学过程富集分析和京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路富集分析,富集标准为P<0.05。
1.4 差异表达基因信号转导网络构建
基于KEGG中Pathway的基因调控关系,解构数据库,在全KEGG-Pathway数据库的范围内筛选某个蛋白的上游或下游蛋白,从而得到数据库中任何一个基因的表达产物和其他基因表达产物的相互作用关系,并通过Cytoscape 2.8.2进行图标绘制。
1.5 lncRNA-mRNA-net共表达网络构建
取3个数据集GSE15471、GSE16515、GSE71989中共表达关系的交集,即将在3个数据集中均存在的lncRNA-mRNA共表达关系对纳入网络。通过计算lncRNA与mRNA的皮尔森相关系数r,选取相关系数的绝对值(|r|)≥0.85,且P<0.05的lncRNA-mRNA对,构建lncRNA-mRNA共表达网络,并通过网络作图软件Cytoscape 2.8.2绘制网络图。
1.6 lncRNA-mRNA-pathway网络构建
在KEGG数据库中将lncRNA与它对应的mRNA,以及参与重要通路的基因和通路名称的关系对,导入Cytoscape 2.8.2软件中进行可视化,构建信号通路的调节网络。
1.7 蛋白质相互作用网络(PPI)构建
对寻找到的lncRNA-mRNA调控网络中的编码蛋白基因,通过STRING软件(https://string-db.org/)寻找基因对应蛋白质之间的相互作用关系。设置最小相互调控作用预测得分(minimum required interaction score)>0.4,得到基因产物蛋白之间的调控关系,然后构建蛋白调控网络。
1.8 生存分析
从TCGA数据库中(https://tcga-data.nci.nih.gov/)提取177例胰腺癌患者生存数据,使用R语言“Survival”软件包,对筛选出的差异mRNA、lncRNA和胰腺癌患者生存时间进行相关性分析,检验方法为Log-rank χ2检验,以α=0.05为检验水准。
2 结果
2.1 差异基因筛选
根据图1可以发现,根据筛选条件,在芯片GSE15471中得到差异mRNA 3 864个,其中上调基因1 509个,下调基因2 345个;差异lncRNA 1 873个,其中上调基因197个,下调基因1 676个。在芯片GSE16515中得到差异mRNA 3 019个,其中上调基因1 020个,下调基因1 999个;差异lncRNA 1 330个,其中上调基因98个,下调基因1 232个。在芯片GSE71989中得到差异mRNA 3 631个,其中上调基因1 479个,下调基因2 152个;差异lncRNA 1 625个,其中上调基因208个,下调基因1 417个。对3个芯片筛选出的差异mRNA、lncRNA取交集并通过韦恩图(图2)展现,得到与胰腺癌相关可信度高的差异mRNA 1 147个、lncRNA 336个。
图1 胰腺癌中差异lncRNA和m RNA的热图
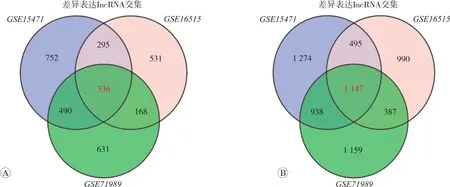
图2 胰腺癌中异常表达的差异lncRNA和mRNA的韦恩图
2.2 差异表达基因功能及通路富集分析
通过DAVID对上述得到的1 147个差异mRNA基因进行GO分析发现,上调的差异mRNA基因主要参与细胞外基质组织、细胞黏附、细胞因子介导的信号通路、胶原分解等过程,下调的差异mRNA基因主要参与信号转导、G蛋白偶联受体信号通路、蛋白水解、细胞锌离子稳态等过程。KEGG分析发现,上调差异mRNA基因主要涉及肿瘤通路、人类乳头瘤病毒感染、细胞外基质受体相互作用、蛋白质消化吸收等通路,下调差异mRNA基因涉及钙信号通路、代谢通路、神经活性配体-受体相互作用、胰液分泌等通路。排名前20位的差异mRNA基因显著性功能和通路如图3所示。
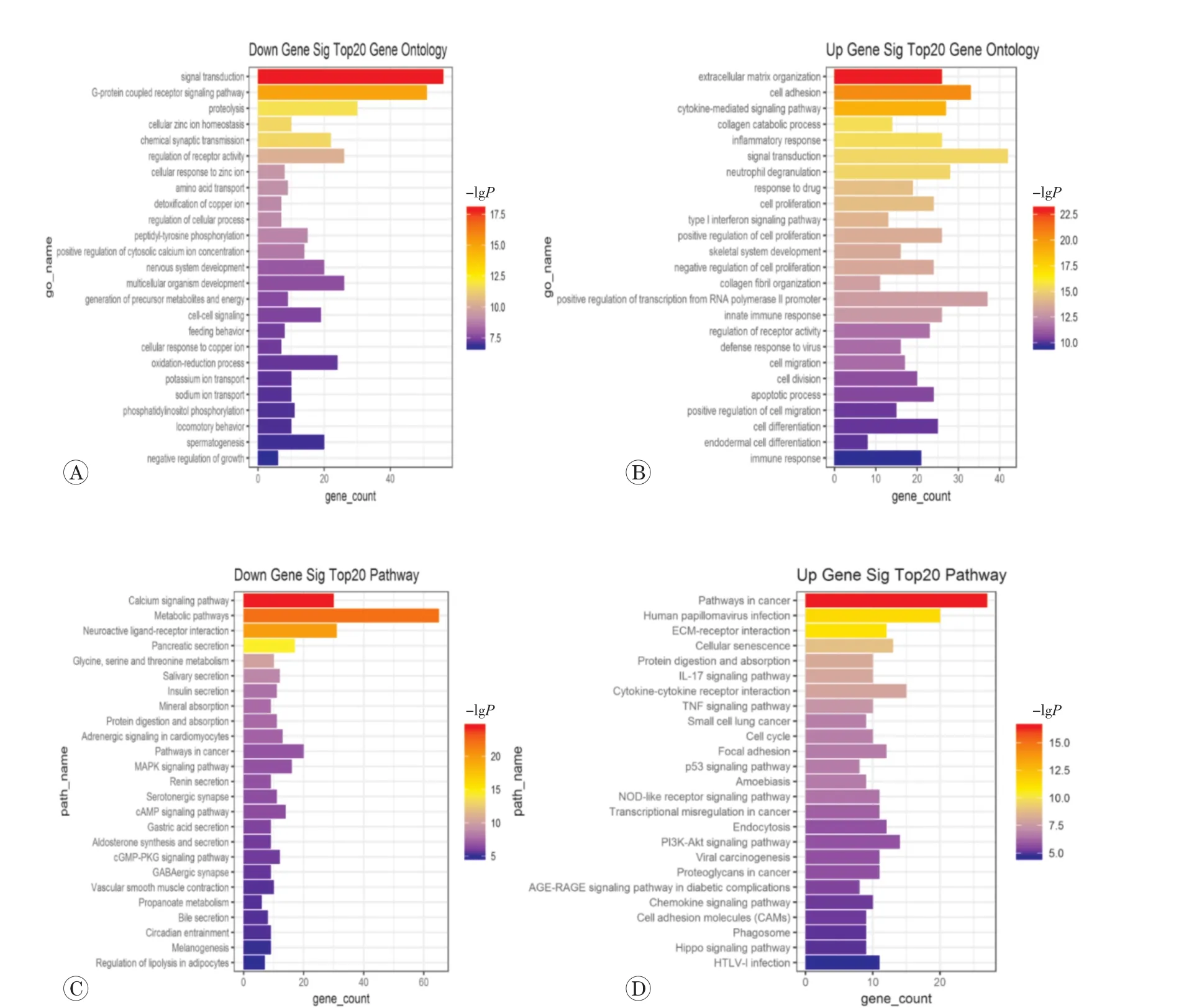
图3 差异表达基因GO分析和KEGG分析结果
2.3 差异表达基因信号转导网络分析
差异表达基因信号转导网中共有62个上调mRNA和79个下调mRNA,上调mRNA中调控数量前3位的是 GNA15(13 个)、SMAD3(9 个)、ITGA2(9 个)、STAT1(7个)、SDC1(7个)、CXCR4(7个),下调mRNA中调控数量前3位的是GNAS(13个)、PLCB1(11个)、CALML5(10个),见图4。
2.4 lncRNA-m RNA共表达网络分析
通过lncRNA-mRNA共表达网络发现有7个lncRNA与61个mRNA具有高度的共表达关系。NONHSAT166626.1、ENST00000536141.1与NONHSAT 138174.2居于lncRNA-mRNA共表达网络的核心位置,与众多mRNA均具有共表达关系。见图5。
2.5 lncRNA-m RNA-pathway调控网络分析
调控网络包括3个上调lncRNA和11个下调lncRNA,13个上调mRNA和16个下调mRNA及46条通路,其中调控数量前3位的lncRNA是NONHSAT166626.1(10个)、ENST00000536141.1(7个)、NONHSAT138174.2(6个),调控网络中的mRNA前5位分别是 HLA-F(12个)、HLA-G(12个)、FN1(11个)、COL1A1(10个)、COL1A2(10个),排名前3位的通路是蛋白质消化吸收(8个)、人类乳头瘤病毒感染(6个)、代谢途径(5个)。见图6。
2.6 差异m RNA及其蛋白质相互作用网络分析
在差异mRNA间的蛋白质相互作用网络中COL1A1、COL3A1、COL5A2居于重要节点,具体见图7。
2.7 生存分析
对TCGA数据集中胰腺癌的生存分析发现,12个lncRNA与胰腺癌的生存预后密切相关。它们的生存曲线见图8,其中基因ATP1A1-AS1、CBR3-AS1、CTD-3080P12.3、 FAM66D、 FAM87A、 FLJ38576、 LINC 00476、LINC00574、LINC01554、PYY2高表达的胰腺癌患者生存时间延长,而基因LINC00857、OVOL1-AS1的高表达会使胰腺癌患者的生存时间缩短(P<0.05)。
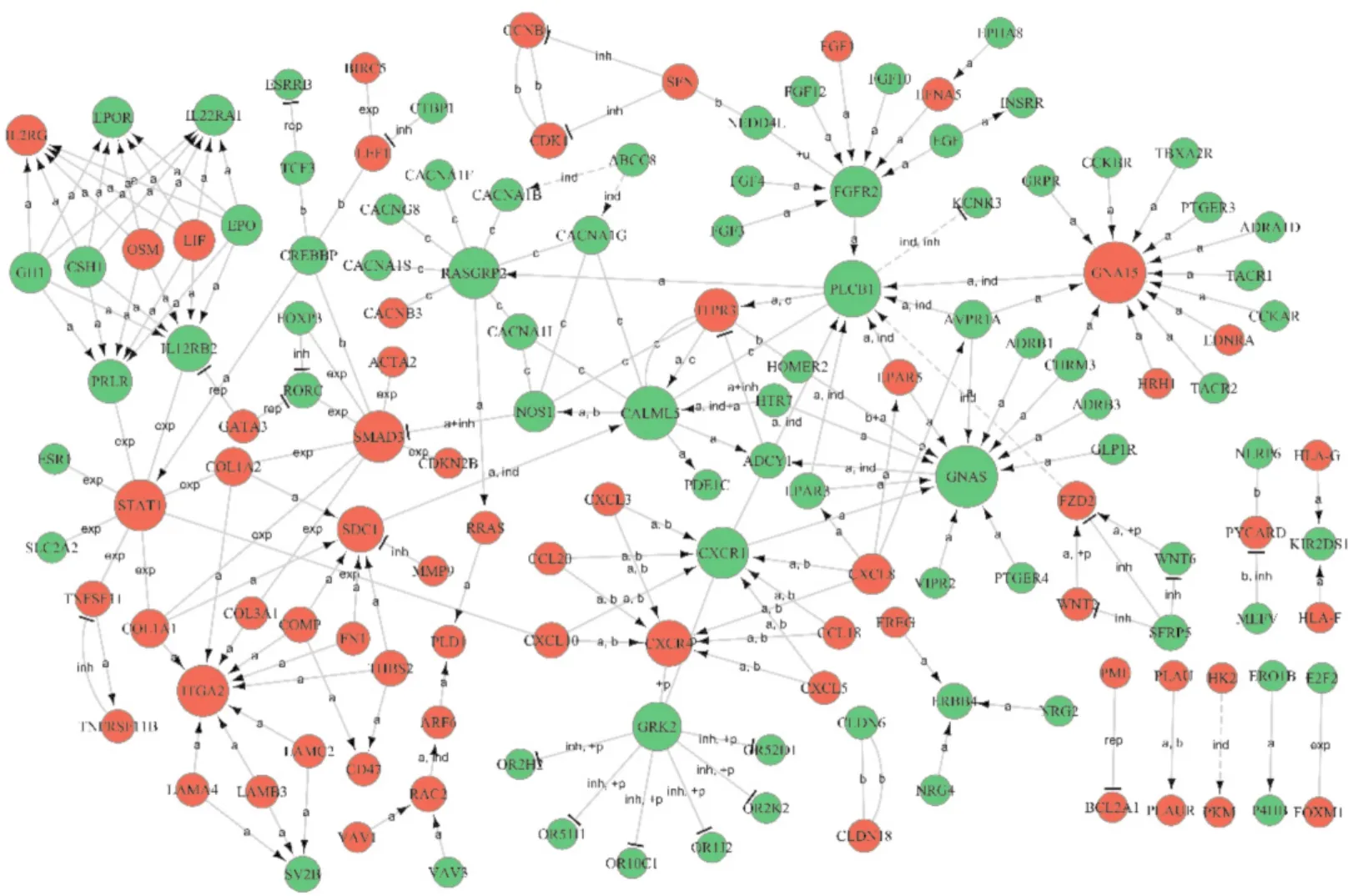
图4 差异表达基因信号转导网络
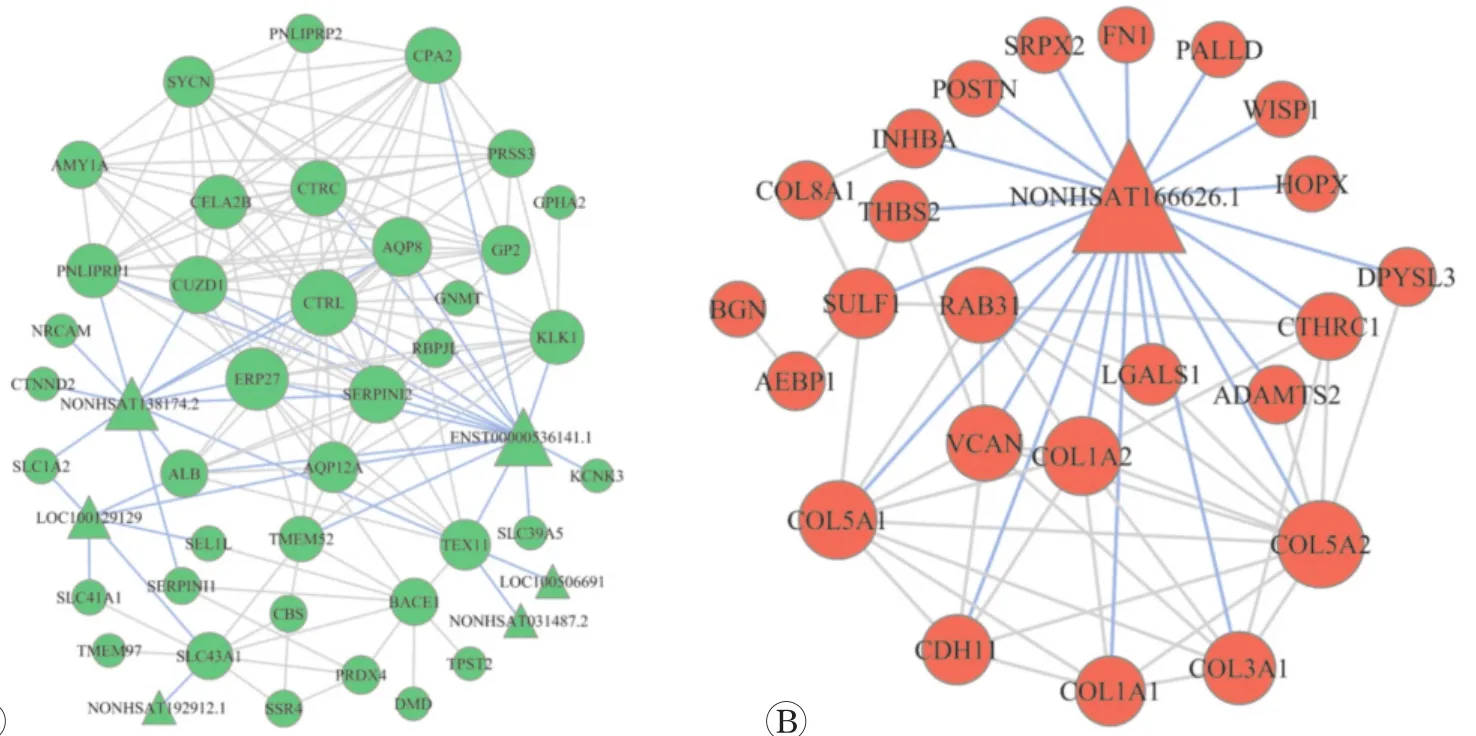
图5 lncRNA-m RNA共表达网络
3 讨论
胰腺癌是一种恶性程度较高的消化系统肿瘤,其病死率位居恶性肿瘤死亡的第4位。目前手术治疗仍是胰腺癌常规治疗手段,胰腺癌手术患者如能早期诊断并进行手术治疗,其5年生存率可达24%[12]。但由于其起病隐匿、侵袭性强、进展迅速且预后较差,所以目前胰腺癌治愈性切除率仅为5%[13],其5年整体生存率低于6%[14-15]。而近年来随着分子医学的迅速发展,生物信息学为诊断疾病和挖掘疾病治疗靶点提供了一项新的技术手段[16]。
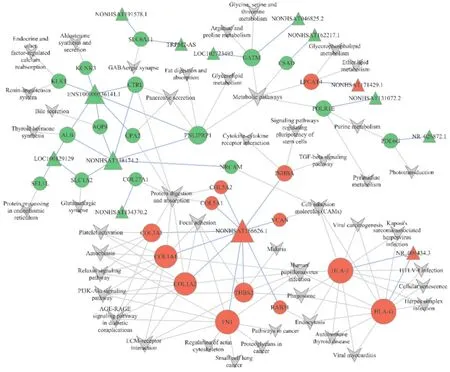
图6 lncRNA-m RNA-pathway调控网络
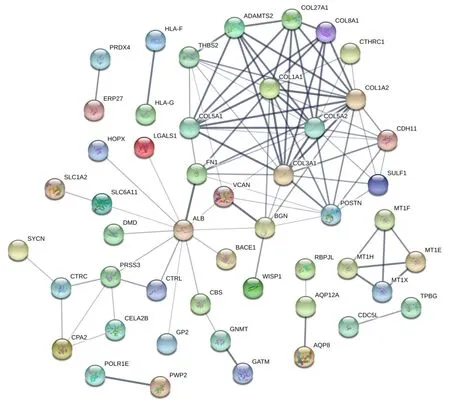
图7差异m RNA及其蛋白质相互作用网络
因此,本文运用生物信息学技术,通过对GEO数据库和TCGA数据库中的胰腺癌相关数据进行分析,筛选出胰腺癌相关可信度高的差异mRNA 1 147个、lncRNA 336个,对mRNA进行功能富集分析发现上调的差异基因主要参与细胞外基质组织、细胞黏附、细胞因子介导的信号通路、胶原分解等过程,下调的差异基因主要参与信号转导、G蛋白偶联受体信号通路、蛋白水解、细胞锌离子稳态等过程。信号通路分析发现,上调差异基因主要涉及癌症通路、人类乳头瘤病毒感染、细胞外基质受体相互作用、蛋白质消化吸收等通路,下调差异基因涉及钙信号通路、代谢通路、神经活性配体-受体相互作用、胰液分泌等通路。之后通过构建信号转导网络及蛋白质相互作用网络表明差异基因对胰腺癌的发生发展有重要影响。并通过lncRNA-mRNA共表达网络找到7个与mRNA具有高度的共表达关系lncRNA,通过lncRNA-mRNA-pathway网络发现了14个对mRNA及通路具有重要调控价值的lncRNA。最后通过与TCGA数据库信息结合进行生存分析发现有12个lncRNA与胰腺癌的预后相关,分别是 ATP1A1-AS1、CBR3-AS1、CTD-3080P12.3、 FAM66D、 FAM87A、 FLJ38576、 LINC 00476、 LINC00574、 LINC01554、 PYY2、 LINC 00857、OVOL1-AS1。
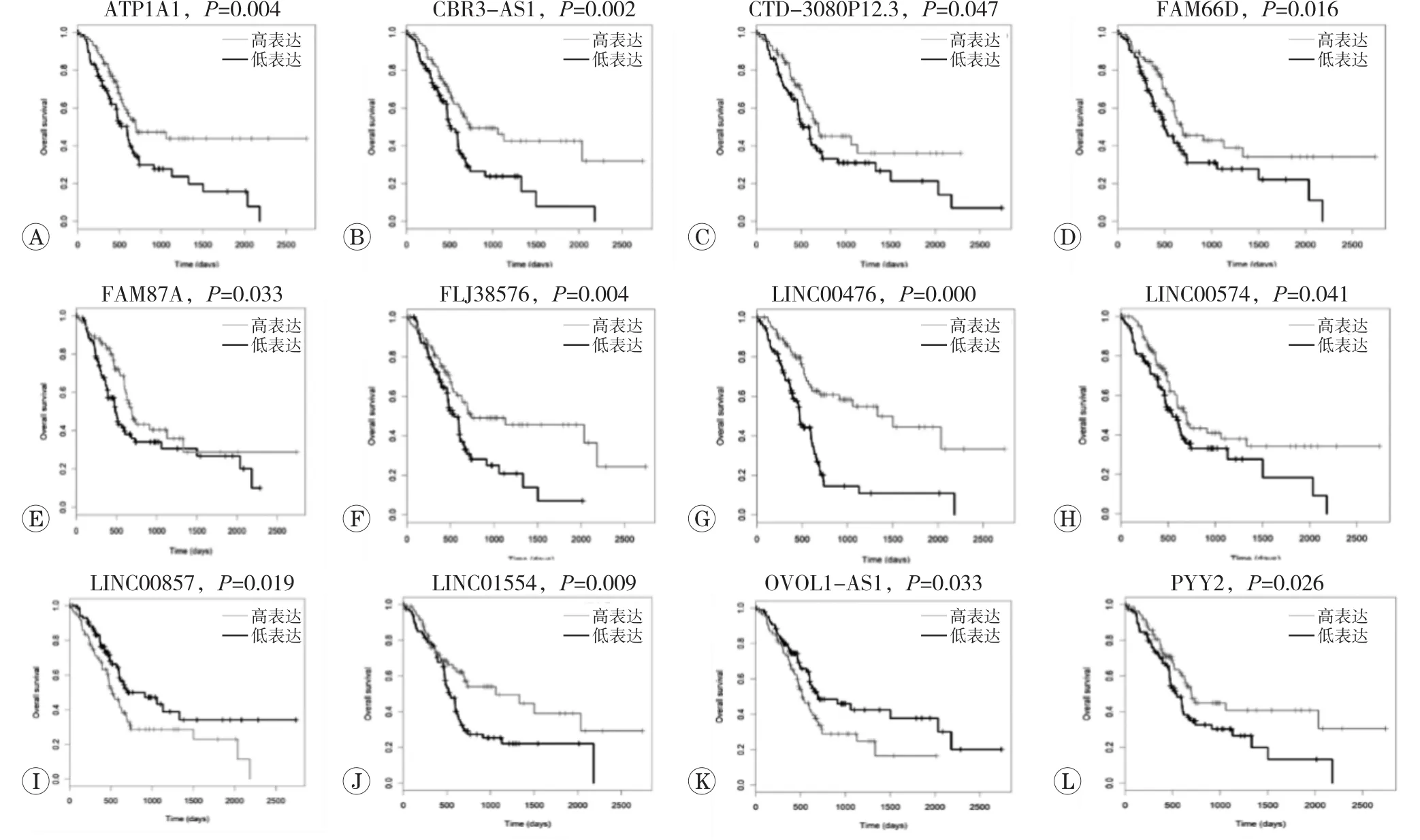
图8 lncRNA生存分析结果
以往的研究发现这些lncRNA对其他疾病也有着显著的影响。ATP1A1-AS1基因作为Na/K-ATPaseα1的中度负调控因子,可以调节人肾细胞中Na/KATPase相关信号通路[17];研究发现CBR3-AS1对骨肉瘤细胞增殖、迁移、侵袭和凋亡具有致癌作用,是骨肉瘤患者独立的不良预后影响因素[18];FAM66D在促性腺激素腺瘤的分子调控中起着至关重要的作用[19];LINC00476可以在某种程度上揭示尼古丁依赖治疗靶点的生物学机制和发展[20];LINC00574和LINC01554分别对乳腺癌、食管癌的预后有着显著影响[21-22];PYY2主要是在睾丸和前列腺中差异表达[23];LINC 00857通过细胞周期调控介导肿瘤进展,进而影响肺腺癌的诊断和预后[24]。除此以外,本研究还发现了其他几个影响胰腺癌预后的lncRNA,但这些lncRNA对疾病的作用及其机制大多不明,还需要更加深入的研究加以证实。
之前有学者对芯片GSE15471、GSE16515、GSE 71989进行研究[25-27],但以往基于芯片的研究大多局限于通过对胰腺癌的mRNA进行生物信息学分析,并通过生存分析筛选显著的mRNA;而针对lncRNA对胰腺癌影响的研究更多是集中在分析单个lncRNA对于胰腺癌的影响[28-30]。而本文利用GEO数据库和TCGA数据库筛选出了相关可信度高的差异mRNA 1 147个、lncRNA 336个,不仅比较系统的分析了mRNA和lncRNA对胰腺癌的影响,更通过构建lncRNA-mRNA共表达网络分析了lncRNA与mRNA的相互作用关系,找到7个与mRNA具有高度共表达关系的lncRNA,展现了lncRNA的功能和调控机制[31-32],通过lncRNA-mRNA-pathway网络发现了14个lncRNA,确定了3个核心lncRNA,揭示了其对mRNA及通路具有重要调控价值[33]。
综上所述,本文通过生物信息学方法研究了lncRNA和mRNA对胰腺癌的发生发展所产生的作用,并通过生存分析发现12个lncRNA和若干个mRNA会对胰腺癌的预后产生影响。这些lncRNA可能会成为新的胰腺癌治疗靶点和分子标志物,用以指导胰腺癌的靶向治疗和预后判断。

