甘蓝型油菜光敏色素互作因子4 (BnaPIF4)基因克隆和功能分析
冯 韬 官春云
甘蓝型油菜光敏色素互作因子4 (BnaPIF4)基因克隆和功能分析
冯 韬 官春云*
湖南农业大学农学院 / 国家油料改良中心湖南分中心, 湖南 长沙 410128
光敏色素互作因子4 (phytochrome interacting factor 4, PIF4)是光信号途径关键转录因子, PIF4与BZR互作介导光信号与油菜素内酯信号互作, 参与植物光响应。本文在甘蓝型油菜湘油15号中克隆到2个基因, 分别定位于A03号染色体和C03号染色体, 命名为和, 全长编码序列(coding sequence, CDS)、全长mRNA和全长基因分别为1242 bp和1245 bp、1701 bp和1731 bp、2527 bp和2665 bp, 各自编码413和414个氨基酸。具有7个外显子和6个内含子,具有8个外显子和7个内含子, 与测序品种中双11号相比,基因第1内含子存在单碱基插入突变, 第4和第6内含子存在缺失突变, 且具有更长的3'-UTR, 两基因其他序列在湘油15号和中双11号之间无差别。和基因编码蛋白具有典型的植物bHLH结构域, 亚细胞定位于细胞核, 是典型的植物PIF4蛋白。多序列比对和进化分析表明, BnaPIF4蛋白与白菜、拟南芥、亚麻芥等PIF4蛋白高度同源。PIF4蛋白进化关系与物种进化关系一致, 近缘物种中的PIF4蛋白在进化树中高度聚类, 大量植物中可见PIF4蛋白重复且低等植物中分化程度明显低于高等植物中, 表明PIF4蛋白是一个晚期进化事件且可能存在功能冗余。酵母杂交实验表明BnaPIF4与BnaBZR蛋白存在互作, 但BnaPIF4不能与基因的启动子互作, 表明与在蛋白水平而非转录水平发生互作。湘油15号中和基因表达规律一致,基因主要表达于油菜茎表皮、未成熟角果和叶中, 在花和根中表达较低, 且其表达随油菜生育进程逐渐降低。
甘蓝型油菜; 光敏色素互作因子4; 基因克隆; 基因互作; 基因表达; 生物信息学分析
低光寡照影响甘蓝型油菜结实, 是造成油菜减产的重要原因之一[1], 增强油菜对低光胁迫的抗性是生产上面临的重要议题。低光如何影响油菜产量尚无明确的机制研究, 光信号通过何种转化最终影响油菜产量形成尚不明确。有研究显示施用外源油菜素内酯可以增强番茄[2]、水稻[3]等多种作物光合效率、增强弱光抗性、增加产量, 过表达油菜素内酯合成基因增强甘蓝型油菜非生物胁迫抗性和产量[4], 表明光信号途径与油菜素内酯信号途径的互作可能是光最终影响油菜产量的原因之一。在拟南芥和水稻中均证实光敏色素互作因子4 (phytochrome interacting factor 4, PIF4)与芸薹素唑抗性因子(brassinazole-resistant, BZR/BES)互作介导光信号途径与油菜素内酯信号途径的信号转导[5], 对甘蓝型油菜基因及其与互作的相关研究是阐明弱光胁迫对油菜产量影响的关键问题之一, 作者前期工作中已克隆了甘蓝型油菜基因,本文主要关注甘蓝型油菜基因相关研究。
PIFs属于bHLH转录因子家族第15亚家族, 具有典型的bHLH结构域可自结合形成同源二聚体或与含有HLH结构域的其他蛋白形成异源二聚体参与光、温、激素等响应的下游基因表达调控[6]。PIF4是PIFs家族中重要成员, 是植物光信号途径中的核心转录因子之一, 最早发现其负调控光敏色素B信号参与拟南芥光质响应过程[7]。后续研究发现PIF4不仅参与光质响应, 同样对光强和温度响应[8], PIF4与光敏色素B互作调控光子总量影响的拟南芥气孔发育[9], 调控光敏色素合成介导高温胁迫下蓝光响应[10], 对生长素、赤霉素、油菜素内酯等多种植物激素信号调控或互作。PIF4在高温胁迫下, 通过调控上游基因转录影响生长素合成[11], 通过与DELLA互作影响赤霉素的合成与功能发挥[12], 油菜素内酯与PIF4则存在双向关系, 一方面PIF4通过与BZR/BES互作影响油菜素内酯信号的转导, 另一方面油菜素内酯依赖的磷酸化影响基因转录, 调控PIF4水平[13]。弱光胁迫下, 菊花基因表达出现先升高后趋稳缓降的规律, 乙烯、赤霉素和生长素等激素水平同时发生变动, 表明PIF4可能参与植物对弱光胁迫的响应[14]。
甘蓝型油菜为异源多倍体, 基因组结构复杂, 测序完成较晚[15], 因此相关基因功能研究滞后, 目前对甘蓝型油菜中的光信号转导, 以及与其他激素途径之间的互作尚不明确, 信号途径中关键基因的解析不足, 导致相关基因在育种中的应用受限。
本文通过甘蓝型油菜基因组数据库中基因序列设计引物, 从甘蓝型油菜品种湘油15号中克隆获得2个基因的编码序列(coding sequence, CDS)、全长mRNA和全长基因序列, 染色体步移证实一个位于A03染色体, 一个位于C03染色体。
1 材料与方法
1.1 试验材料
甘蓝型油菜(L.)双低品系湘油15号由国家油料改良中心湖南分中心提供。
1.2 RNA提取与cDNA合成
湘油15号种子在22℃、光周期为16 h/8 h条件下培养至2片真叶, 取幼苗50~100 mg。加液氮充分研磨后, 使用TRIzol RNA提取试剂盒(TransGen生物技术有限公司), 按说明书所示方法提取总RNA, 以此RNA为模板, 使用Easy Script First-Strand cDNA Synthesis SuperMix试剂盒(TransGen生物技术有限公司)按说明书所示方法合成第1链cDNA。
1.3 DNA提取
取1.2所述湘油15号叶片50~100 mg, 液氮研磨后加入1 mL CTAB-PVP buffer, 提取总DNA备用于目标基因全长序列和启动子序列的克隆。
1.4 BnaPIF4基因的克隆及序列分析
从BRAD数据库(http://brassicadb.org/brad/)甘蓝型油菜全转录组中提取的转录本序列。分析比对, 设计含起始密码子的5'端引物和含有终止密码子的3'端引物(湖南擎科生物技术有限公司合成), 用于CDS克隆; 根据CDS测序比对结果, 检索全长mRNA和全长基因并设计克隆引物。CDS克隆引物为: BnaPIF4-F: 5'-ATGGAACACCAAGGTTGGAG-3', BnaPIF4-R: 5'-CTAACGGGGACCGTCGGT-3';边界序列一致, mRNA克隆与全长基因克隆共用引物对为: dPIF4-F: 5'-TCTGTCTGTACCAAAAGAAG-3', dPIF4-R: 5'-TCTGGACGATTATGAAAAGC-3'; 分别以1.2所述cDNA和1.3所述DNA为模板进行PCR扩增。PCR体系含50 ng μL–1模板1 μL、10 mmol L–1dNTPs 0.5 μL、2 μmol L–1BnaPIF4-F/mPIF4-F 0.75 μL、2 μmol L–1BnaPIF4-R/mPIF4-R 0.75 μL、10×反应缓冲液1 μL、GXL高保真DNA聚合酶0.5 μL和ddH2O 15.5 μL。扩增程序为预变性95℃ 5 min; 95℃50 s, 58℃45 s, 72℃ 3 min, 35个循环; 72℃后延伸10 min。PCR产物经1.5%琼脂糖凝胶电泳, 用凝胶成像系统(Bio-Rad)检测和记录结果。将PCR产物回收后与pMD18-T载体(TransGen生物技术有限公司)连接, 转化大肠杆菌感受态细胞Trans1-T1 (北京天根生化有限公司), 筛选阳性克隆进行基因测序, 通过BRAD数据库(http://brassicadb.org/brad/)对获得的克隆序列进行染色体步移分析, 随后使用DNAman对克隆获得的基因拷贝进行序列比对。
1.5 BnaPIF4蛋白生物信息学分析
1.5.1 BnaPIF4_A03和BnaPIF4_C03氨基酸序列特征与二级结构预测 通过ANTHEPROT 6.2软件分析来源于甘蓝型油菜的和基因所编码的蛋白质。统计编码蛋白的氨基酸残基组分、分析等电点、特异性识别序列和亲疏水特征并使用Gariner模型进行二级结构预测。使用wolf-pSORT (https://www.genscript.com/tools/ wolf-psort)和Plant-mPLoc (http://www.csbio.sjtu.edu. cn/cgi-bin/PlantmPLoc.cgi)进行亚细胞定位预测。
1.5.2 BnaPIF4蛋白同源结构域分析 使用DNAman对BnaPIF4蛋白进行序列比对分析; 同时使用NCBI (http://blast.ncbi.nlm.nih.gov/)分别对不同的BnaPIF4蛋白序列进行保守结构域分析; 使用SMART (http://smart.embl-heidelberg.de/)、MOTIF (http://www.genome.jp/tools/motif/)以及InterPro (http://www.ebi.ac.uk/interpro/)分析BnaPIF4包含的特殊结构域。
1.5.3 PIF4蛋白聚类分析 通过NCBI蛋白数据库(http://www.ncbi.nlm.nih.gov/protein), String数据库(http://string.embl.de/), PDB数据库(http://www. pdb.org/pdb/home/home.do)检索来源于不同物种的PIF4蛋白序列, 并使用MEGA6.0对来源不同的PIF4相关蛋白进行聚类分析。
1.6 BnaPIF4互作因子酵母杂交分析
使用、和载体(本实验室保存),氨基酸合成缺陷酵母进行酵母单双杂交实验, 分析BnaPIF4与BnaBZR1/BnaBES1互作模式。、和基因的CDS分别亚克隆到载体上;各拷贝CDS亚克隆到载体; 从湘油15号gDNA中克隆、和基因启动子(起始密码子上游1 kb序列), 随后将各启动子序列亚克隆到pHIS2载体上; 随后分别将不同组合的AD与BD质粒共转化酵母并接种到添加浓度10 mmol L-13-AT的三缺培养基进行筛选, 用于验证BnaPIF4蛋白与BnaBZR1BnaBES1/2蛋白以及启动子之间的互作关系。
1.7 甘蓝型油菜中BnaPIF4_A03和BnaPIF4_ C03基因时空表达分析
以1.2方法提取甘蓝型油菜湘油15号不同时期根、茎、叶、花和角果皮总RNA, 使用宝生物工程(大连)有限公司PrimeScript RTreagent Kit with gDNA Eraser (Perfect Real-time)试剂盒进行反转录, 获得cDNA第1链作为qRT-PCR模板。分析1.4中克隆获得的片段, 根据5'-UTR序列特征设计两基因分型定量引物如下: PIF4A03-F: 5'-AAGTAAA GAACCTTACCCATCT-3', PIF4A03-R: 5'-TAAAGTT CCACGCCCAAA-3'; PIF4C03-F: 5'-AATCATTATCT CCCTCGCTGTA-3', PIF4C03-R: 5'-TGAAGTTCCA CGCCCA AC-3'。以作为内参基因, 内参引物为BnUBC21-F: 5'-CCTCTGCAGCCTCCTCAA GT-3', BnUBC21-R: 5'-TATCTCCCCTGTCTTGAAA TGC-3', 用于和时空表达分析, 每样品进行3次重复。在ABI 7500荧光定量PCR系统上运行qRT-PCR。PCR体系包含1 μL cDNA、10 μL 2×FastStart Universal SYBR GreenMaster with ROX、10 μmol L–1正向引物和反向引物各0.5 μL、8 μL ddH2O。PCR程序为95℃ 10 min; 95℃ 15 s, 60℃ 15 s, 72℃ 32 s, 35个循环。反应完成后进行95℃ 20 s, 60℃ 20 s, 95℃ 20 s, 59℃ 20 s绘制熔解曲线检测扩增产物的正确性和引物二聚体。
2 结果与分析
2.1 BnaPIF4基因克隆
以湘油15号cDNA为模板, 克隆到2个CDS序列(图1-A), 长度分别为1242 bp和1245 bp, 对应的克隆到2个基因全长mRNA (图1-B)分别为1701 bp和1731 bp, 以湘油15号gDNA为模板克隆得到对应基因的全长序列(图1-C)为2527 bp和2665 bp, 分别定位于A03和C03染色体, 命名为和。通过BRAD数据库比对, 确认2个CDS、mRNA和基因全长DNA序列的完整性, 对BRAD数据库检索未发现更多的基因拷贝, 表明和克隆完整。序列比对显示和基因的CDS相似度98.15%, mRNA相似度92.82%, 基因全长相似度94.64%, 表明和基因高度同源, 且基因序列变异主要发生在UTR区域和内含子区域。仔细分析和基因各序列, 并绘制基因结构示意图(图2)。
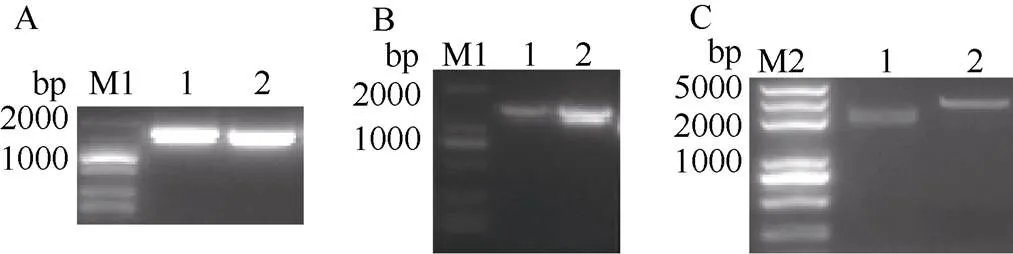
图1 BnaPIF4_A03和BnaPIF4_C03基因克隆
A: 编码序列; B: mRNA; C: 基因全长: M1: 2K DNA 标记; M2: 2K plus DNA标记; 1:: 2:。
A: CDS; B: mRNA; C: Full length gene; M1: 2K DNA marker; M2: 2K plus DNA marker; 1:; 2:.
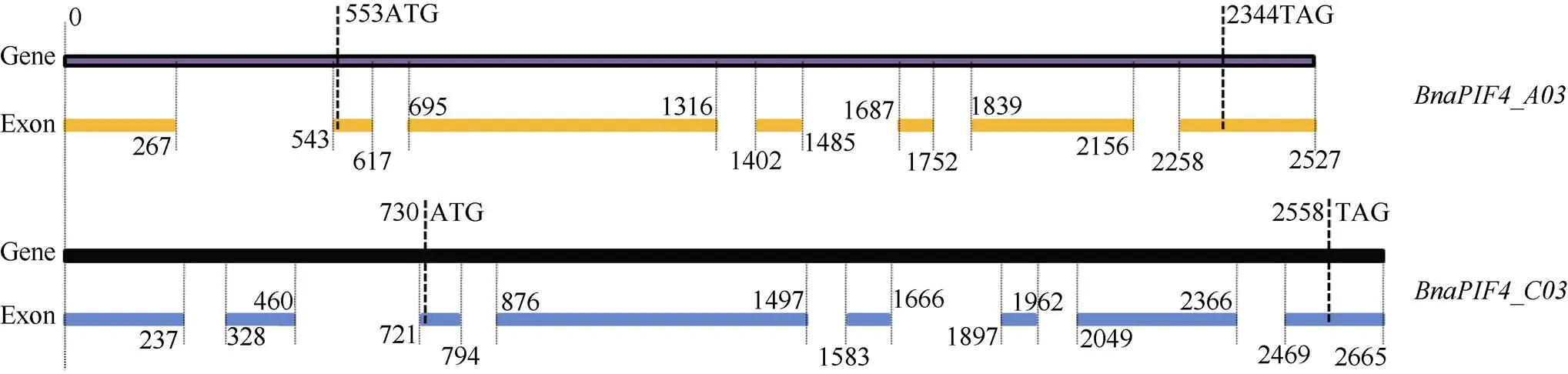
图2 BnaPIF4_A03和BnaPIF4_C03基因结构
序列分析显示,基因序列与测序品种中双11号中一致, 存在8个外显子和7个内含子, CDS序列位于第3~8外显子;基因具有7个外显子和6个内含子, 但序列与测序品种中双11号存在差异, 主要表现为第1内含子上352 bp位置存在单T碱基插入突变, 第4内含子1511 bp位置缺失AT碱基, 1582 bp位置缺失14碱基的富A/T序列, 第六内含子2228 bp处缺失ATTT, 且第7外显子3'端相对中双11号长91 bp。和基因间差异主要在5'端的外显子与内含子上,的5'端相对缺少C03拷贝具有的第2外显子, 造成其5'-UTR比C03拷贝更短, 二者CDS序列相似度极高, 且与测序品种中双11号中对应拷贝无序列差别。
2.2 BnaPIF4生物信息学分析
基因各自编码长度413和414氨基酸残基的蛋白, 分别命名为BnaPIF4_A03和BnaPIF4_C03, 2个BnaPIF4蛋白的氨基酸组成、摩尔质量和等电点如表1。
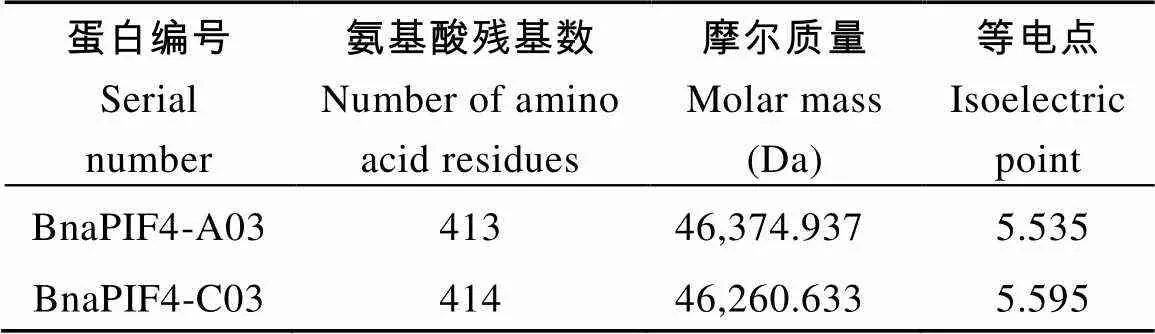
表1 BnaPIF4_A03和BnaPIF4_C03蛋白信息汇总表
亲疏水性分析显示, BnaPIF4蛋白主要表现疏水性, 在C端约280残基位置存在一个跨膜结构。Wolf-pSORT与Plant-mPLoc亚细胞定位预测显示, BnaPIF4_A03和BnaPIF4_C03均定位于细胞核, 这与PIF4蛋白作为转录因子的功能一致。
BnaPIF4_A03和BnaPIF4_C03的二级结构预测结果如图3, BnaPIF4_A03有23%α螺旋, 21%β折叠, 26%β转角, 30%无规卷曲; BnaPIF4_C03有25%α螺旋, 20%β折叠, 25%β转角和30%无规卷曲。
通过ANTHEPROT分析了BnaPIF4_A03和BnaPIF4_C03蛋白具有的序列特异性识别位点(表2)。

图3 BnaPIF4_A03和BnaPIF4_C03蛋白二级结构
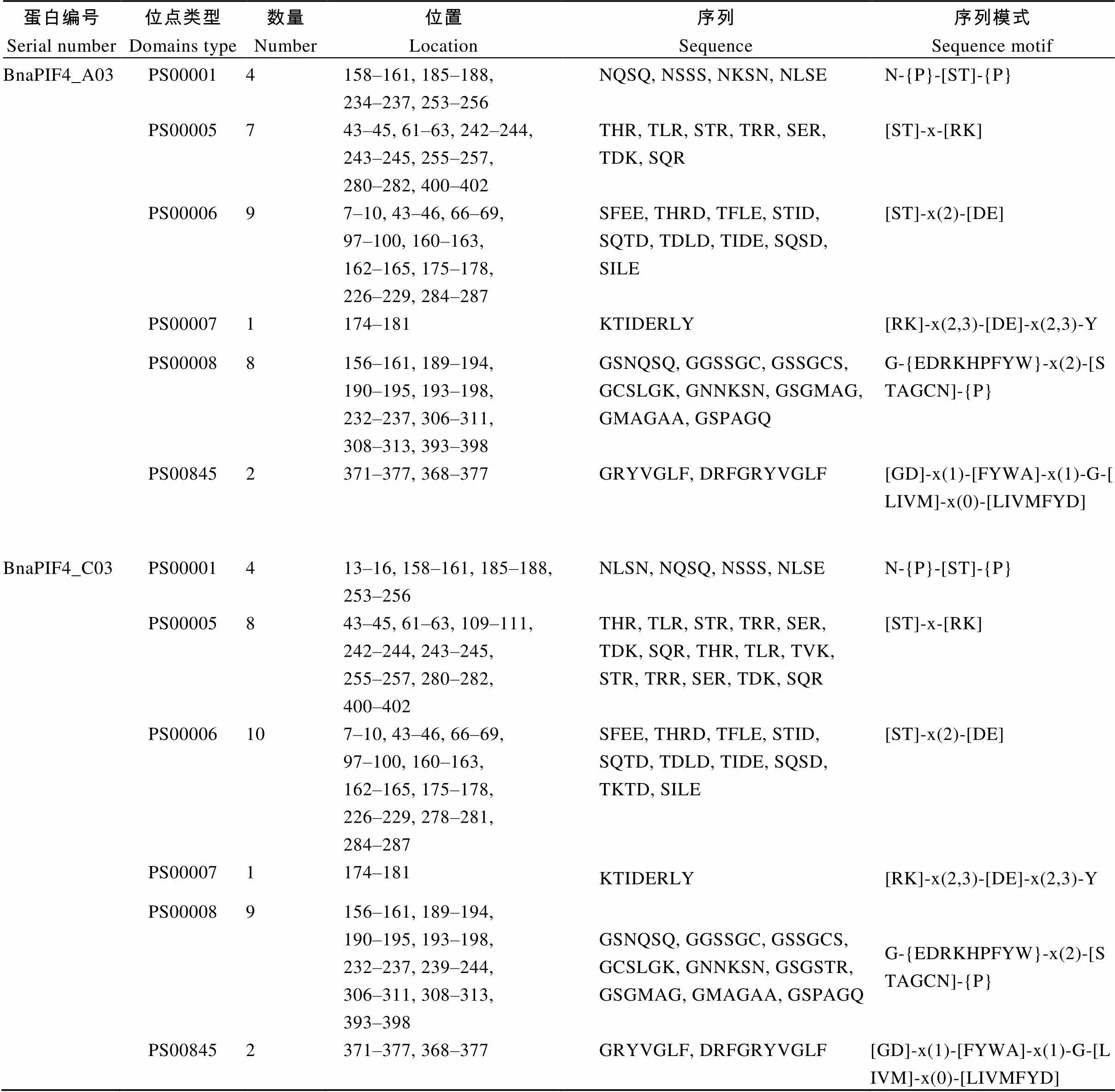
表2 BnaPIF4_A03和BnaPIF4_C03蛋白特殊识别位点序列总表
PS00001: 糖基化位点; PS00005: 蛋白激酶C磷酸化位点; PS00006: 酪蛋白激酶2磷酸化位点; PS00007: 酪氨酸激酶磷酸化位点; PS00008: 豆蔻酰化位点; PS00016: 细胞附着序列; PS00845: CAP-Gly信号域。
PS00001: N-glycosylation site; PS00005: protein kinase C phosphorylation site; PS00006: casein kinase II phosphorylation site; PS00007: tyrosine kinase phosphorylation site; PS00008: N-myristoylation site; PS00016: cell attachment sequence; PS00845: CAP-Gly domain signature.
BnaPIF4_A03和BnaPIF4_C03具有仅含微弱差别的特异性识别序列, BnaPIF4_A03相较BnaPIF4_ C03缺少109~111的蛋白激酶C磷酸化位点、278~ 281的酪蛋白磷酸化位点和239~244的酰化位点。整体来看BnaPIF4_A03和BnaPIF4_C03的特异性识别位点基本一致, 主要是多种激酶的磷酸化修饰位点、小分子结合位点和酰化等蛋白修饰位点, 多数与转录因子功能相关, 这与PIF4蛋白是bHLH家族成员的结论一致。
使用SMART和MOTIF分析结构域(图4)发现, 2个BnaPIF4蛋白具有基本相同的结构域且主要集中于C端, 且多为具有核酸结合功能的结构域, 表现转录因子特性。在第250残基到第310残基之间具有多个重叠的bHLH结构域, 亲疏水性分析发现的跨膜结构域在第280残基左右, 正位于bHLH结构域中段, 这与PIF4作为典型的bHLH家族成员的结论一致。

图4 BnaPIF4氨基酸序列比对
通过不同数据库, 共获得来源于41种不同植物的127条PIF4蛋白序列, 使用MEGA6.0进行聚类分析, 绘制进化树(图5)。
PIF4蛋白聚类表现出与植物亲缘关系的高度一致性, 大部分植物中均存在PIF4蛋白的高拷贝现象, 且各拷贝之间的相似性高, 基本上处于同一进化分支。PIF4蛋白大体上按植物科属分布聚类, 如豆科的花生、绿豆、大豆等多拷贝PIF4蛋白高度聚类(图5右上), 十字花科的甘蓝型油菜、白菜、拟南芥、亚麻芥等多拷贝PIF4蛋白高度聚类, 但禾本科植物中PIF4蛋白分化比较极端, 水稻、狗尾草、玉米等禾本科植物中PIF4蛋白具有较远的分支距离, 且同一物种中不同的PIF4拷贝也表现出较高的分化水平, 如水稻不同亚种、不同拷贝的PIF4蛋白被聚类到完全隔离的分支上(图5左上)。另外, PIF4蛋白表现出木本植物分化程度低于草本植物的规律, 如梨、李、榴莲、可可等木本植物处于不同的科属但PIF4蛋白分支距离相对较小, 且与卷柏中PIF4具有较近的分支距离, 而禾本科的水稻、玉米、狗尾草等, 茄科的番茄、马铃薯、烟草等草本植物PIF4蛋白均表现相对较高的分化程度。表明PIF4蛋白在进化过程中相对保守, 其分化发生在进化较晚的阶段, 且分化过程剧烈, 表现出低等植物中PIF4高度一致, 高等植物中PIF4分化程度显著提高, 在进化过程中PIF4蛋白大量发生重复, 表现为大量植物中存在多个PIF4蛋白拷贝。

图5 不同植物PIF4蛋白的进化树分析
: 拟南芥;subspLyrata: 深山南芥;: 花生;: 曼花生;: 二穗短柄草;L.: 甘蓝型油菜;L.: 白菜;: 亚麻荠;: 辣椒;: 碎米荠;: 克莱门柚;: 榴莲;: 油棕;: 盐芥;: 大豆;: 苜蓿;: 桑;: 林烟草;: 绒毛状烟草;Group: 水稻(粳);: 中国梅;: 狭叶羽扇豆;: 芝麻;: 番茄;: 小番茄;: 高粱;: 胡杨;: 欧洲大叶杨;: 桃;: 鸭梨;: 卷柏;: 狗尾草;: 醉蝶花;: 可可;: 红豆;: 绿豆;var.: 赤小豆;: 马铃薯;: 酿酒葡萄;: 玉米;: 枣。
2.3 BnaPIF4和BnaBZR/BES互作
使用酵母双杂检测了BnaPIF4与BnaBZR1, BnaBES1/2蛋白互作, 酵母单杂交检测了BnaPIF4与,基因启动子之间的互作(图6)。从酵母杂交实验结果可见, BnaPIF4_A03与BnaPIF4_C03互作模式一致, 均可以与BnaBZR1, BnaBES1/2蛋白互作, 但不能结合、基因的启动子,和也不相互与启动子结合, 显示BnaPIF4主要通过蛋白层面而非转录层面的互作参与甘蓝型油菜的生物学调控。BnaPIF4可能通过HLH结构域结合BnaBZR/BES蛋白, 从而影响后者磷酸化, 调控磷酸化修饰水平介导后者的核质穿梭, 调控与下游靶基因启动子的结合, 共同调控下游基因表达。
2.4 甘蓝型油菜中BnaPIF4时空表达特征
为进一步了解在甘蓝型油菜中的功能, 对甘蓝型油菜不同时期, 基于5'-UTR差异对不同组织中表达进行分析。
由图7-A与图7-B比较可见, 湘油15号中具有相同的表达模式, 基因表达均表现明显的组织差异和一定的生长发育时间差异。基因主要表达于油菜的绿色组织, 茎表皮、未成熟角果和叶中表达量较高, 在花和根中表达较低, 叶中表达水平最高, 其次为茎表皮和未成熟的角果, 这些位置是植物感受光信号刺激、发生光合作用的主要场所, 会聚集大量光敏色素, PIF4作为光信号的负调控因子, 可能受到光敏色素浓度的影响, 改变表达水平以对光信号做出响应。在生育进程中,基因表达量差异不明显, 整体表现为苗期和薹期表达量稍高, 随生育进程有缓慢降低的趋势, 这可能是由于随油菜生长发育, 光敏色素积累降低, 造成表达做出调整, 也可能与油菜激素水平有关系, 油菜快速生长的苗期和薹期生长素、油菜素内酯等激素均保持较高水平, 可能通过激素与光信号互作, 影响表达。表达与光信号之间存在着反馈调节, 一方面光信号受到的负调控, 另一方面外源光变化也能诱导表达变化, 这表明在油菜光信号途径中具有重要作用。
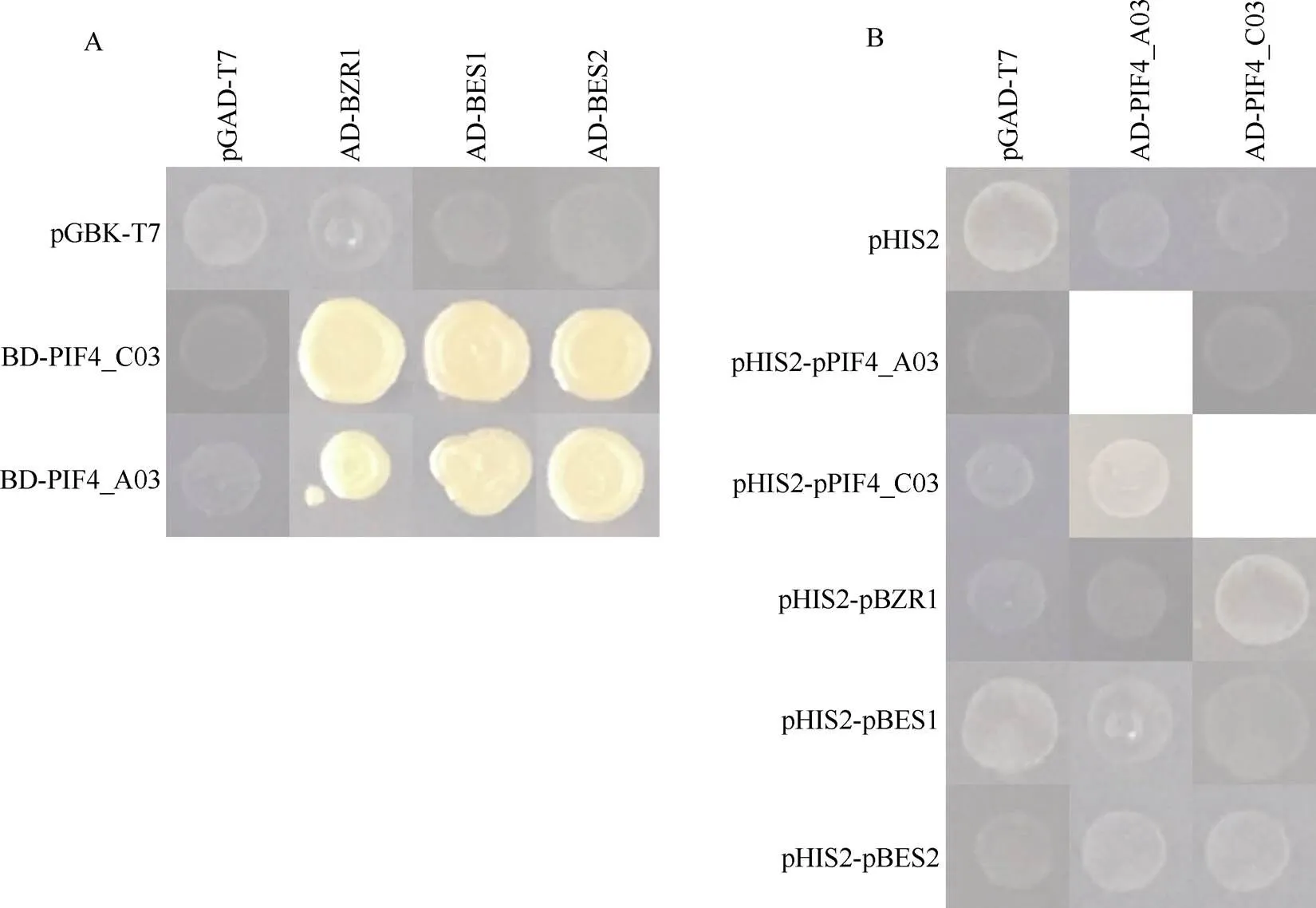
图6 BnaPIF4和BnaBZR/BES互作
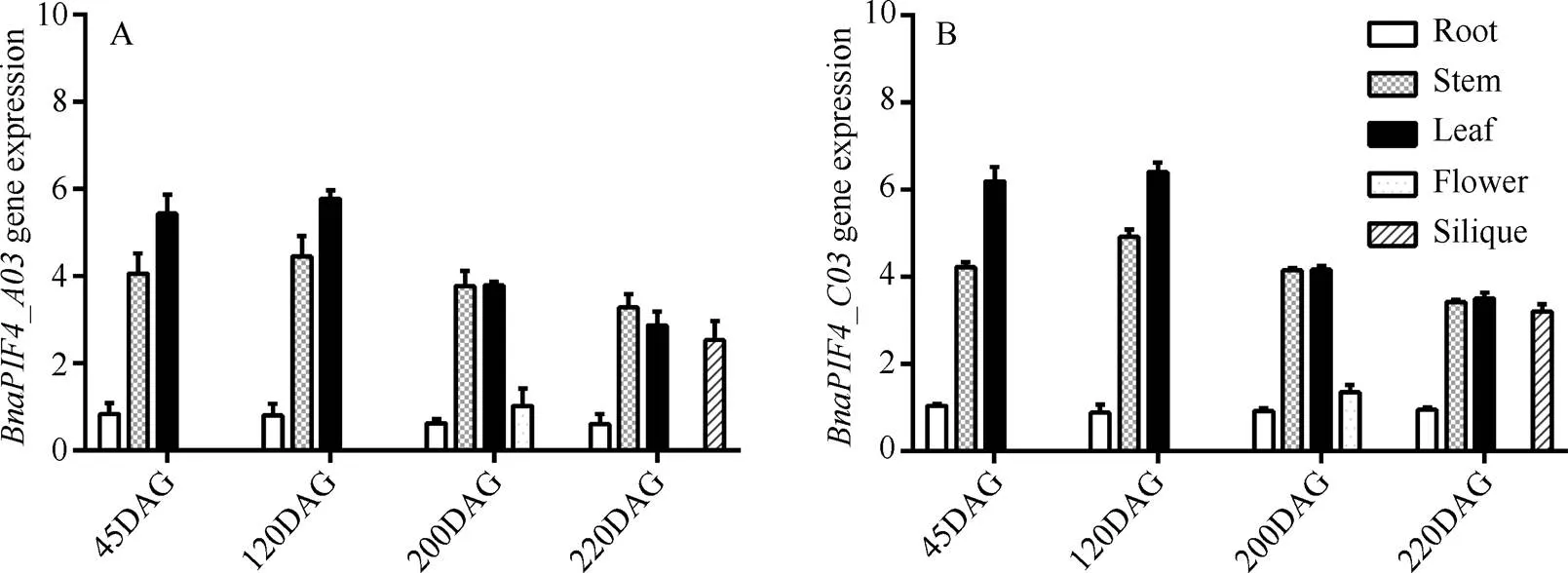
图7 不同发育时期的湘油15号根、茎、叶、花和角果中BnaPIF4_A03和BnaPIF4_C03的表达量
DAG表示种子萌发后天数。DAG means days after seed germination.
3 讨论
从甘蓝型油菜湘油15号中克隆获得2个基因拷贝, 其中拷贝与测序品种中双11号对应拷贝存在内含子和3'-UTR序列差异, CDS序列一致。和基因结构基本一致, 基因、全长mRNA和CDS序列相似度均超过90%, 表明基因高度保守。BnaPIF4_A03和BnaPIF4_C03蛋白具有典型的植物bHLH结构域, 预测亚细胞定位于细胞核, 具有多重转录因子功能的序列结合元件, 存在大量磷酸化和酰化功能位点, 表明克隆获得的基因属于植物基因家族, 这与拟南芥、水稻等植物[16]中结构和功能一致。
对植物PIF4蛋白的聚类分析显示其在进化上具有较高的保守性, 且蛋白进化关系与物种进化关系密切, 近缘物种中的PIF4蛋白高度保守, 但高等植物相对低等植物中PIF4蛋白明显高分化, 表明PIF4蛋白分化可能是一个较晚但相对剧烈的进化事件, 这与植物bHLH家族进化保守性基本一致[17]。NCBI数据库中共收录了6条甘蓝型油菜及其类似序列, 2个位于A染色体组, 4个位于C染色体组, 这表明家族序列在甘蓝型油菜亚基因组间分布可能不平衡, 但6个及类似序列保守性较高, 这显示了甘蓝型油菜A、C亚基因组在进化关系上具有较高的同源性但存在各自独立的进化过程, 也间接证实“禹氏三角”中白菜与甘蓝的近缘关系[18]。
植物PIF4蛋白参与植物开花、病原免疫、非生物胁迫抗性等生物学过程, 是光信号途径中重要的转录因子之一。在高温诱导下拟南芥中PIF4可直接激活FT的表达, 正调控拟南芥的开花,突变体在持续高温条件下表现小株型和晚花表型, 随着温度的升高, PIF4负调控植物免疫反应、促进拟南芥的生长发育[19]。PIFs转录因子家族是整合光信息与植物激素信号的桥梁[20], 其中PIF4介导光信号和多种植物激素信号的互作[21], PIF4与BZR/BES复合体结合介导光信号与油菜素内酯信号之间的转导。本文通过酵母杂交证实甘蓝型油菜中BnaPIF4和BnaBZR/BES基于蛋白结合而非转录层面的互作介导光信号和油菜素内酯信号转导, 但有研究显示PIF4[22]和BZR1[23]均能对油菜素内酯生物合成产生调控, 表明PIF4对油菜素内酯生物合成的反馈通路不同于PIF4通过与BZR/BES结合介导光信号与油菜素内酯信号互作的通路, 对油菜素内酯合成的反馈可能有更多生理功能。同时本文在PIF4蛋白进化分析中发现植物中大量存在PIF4蛋白重复, 也表明PIF4功能冗余或者具有除介导光信号之外的功能, 尚待进一步研究。
湘油15号中和的表达规律一致, 这与基因的保守性相统一, 2个基因拷贝CDS、mRNA、全长基因和启动子序列均具有高度相似性, 可能具有共同的上游调控因子, 对植物内、外环境因素可能具有相同的响应模式。和序列差异主要发生在5'端的内含子和5'-UTR上, 二者可能存在转录和翻译稳定性上的差异。甘蓝型油菜中基因的表达调控规律、蛋白翻译稳定性、翻译后修饰过程、基因功能等有待进一步研究。
4 结论
从甘蓝型油菜湘油15号中克隆到2个基因, 分别定位于A03和C03染色体, 分别命名为和, 全长编码序列(coding sequence, CDS)、全长mRNA和全长基因分别为1242 bp和1245 bp、1701 bp和1731 bp、2527 bp和2665 bp, 各自编码413和414个氨基酸。2拷贝差异序列集中于5'端UTR和内含子, 与测序品种中双11号中对应拷贝存在内含子序列差异。BnaPIF4_A03和BnaPIF4_C03具有典型bHLH结构域, 与白菜、拟南芥等十字花科植物PIF4蛋白高度相似, 植物中PIF4高度保守。甘蓝型油菜中PIF4与BZR/BES存在蛋白层面的互作关系。分析了和基因的时空表达。
[1] 卢坤, 申鸽子, 梁颖, 符明联, 贺斌, 铁琳梅, 张烨, 彭柳, 李加纳. 适合不同产量的环境下油菜高收获指数的产量构成因素分析. 作物学报, 2017, 43: 82–96.Lu K, Shen G Z, Liang Y, Fu M L, He B, Tie L M, Zhang Y, Peng L, Li J N. Analysis of yield components with high harvest index inunder environments fitting different yield levels., 2017, 43: 82–96 (in Chinese with English abstract).
[2] Irfan M, Alam J, Ahmad I, Ali I, Gul H. Effects of exogenous and foliar applications of brassinosteroid (BRs) and salt stress on the growth, yield and physiological parameters of(Mill.)., 2017, 4: 88–101.
[3] Thussagunpanit J, Jutamanee K, Kaveeta L, Chaiarree W, Pankean P, Homvisasevongsa S, Suksamrarn A. Comparative effects of brassinosteroid and brassinosteroid mimic on improving photosynthesis, lipid peroxidation, and rice seed set under heat stress., 2015, 34: 320–331.
[4] Sahni S, Prasad B D, Liu Q, Grbic V, Sharpe A, Singh S P, Krishna P. Overexpression of the brassinosteroid biosynthetic gene DWF4 insimultaneously increases seed yield and stress tolerance., 2016, 6: 28298.
[5] Oh E, Zhu J Y, Wang Z Y. Interaction between BZR1 and PIF4 integrates brassinosteroid and environmental responses., 2012, 14: 802–809.
[6] 杨剑飞, 王宇, 杨琳, 李玉花. 光敏色素互作因子 PIFs 是整合多种信号调控植物生长发育的核心元件. 植物生理学报, 2014, 50: 1109–1118. Yang J F, Wang Y, Yang L, Li Y H. Phytochrome-interacting factors integrate multiple signals to control plant growth and development., 2014, 50: 1109–1118 (in Chinese with English abstract).
[7] Huq E, Quail P H. PIF4, a phytochrome-interacting bHLH factor, functions as a negative regulator of phytochrome B signaling in., 2002, 21: 2441–2450.
[8] Castillon A, Shen H, Huq E. Phytochrome interacting factors: central players in phytochrome-mediated light signaling networks., 2007, 12: 514–521.
[9] Casson S A, Franklin K A, Gray J E, Grierson C S, Whitelam G C, Hetherington A M. Phytochrome B and PIF4 regulate stomatal development in response to light quantity., 2009, 19: 229–234.
[10] Koini M A, Alvey L, Allen T, Tilley C A, Harberd N P, Whitelam G C, Franklin K A. High temperature-mediated adaptations inplant architecture require the bHLH transcription factor PIF4., 2009, 19: 408–413.
[11] Franklin K A, Lee S H, Patel D, Kumar S V, Spartz A K, Gu C, Wigge P A. Phytochrome-interacting factor 4 (PIF4) regulates auxin biosynthesis at high temperature., 2011, 108: 20231–20235.
[12] Xu H, Liu Q, Yao T, Fu X. Shedding light on integrative GA signaling., 2014, 21: 89–95.
[13] Bernardo–García S, Lucas M, Martínez C, Espinosa-Ruiz A, Davière J M, Prat S. BR-dependent phosphorylation modulates PIF4 transcriptional activity and shapes diurnal hypocotyl growth., 2014, 28: 1681–1694.
[14] 韩霜, 陈素梅, 蒋甲福, 房伟民, 管志勇, 陈发棣. 弱光下菊花‘清露’的激素水平及相关基因表达. 中国农业科学, 2015, 48: 324–333. Han S, Chen S M, Jiang J F, Fang W M, Guan Z Y, Chen F T. Hormone levels and gene expression analysis of chrysanthemum cultivar ‘puma sunny’ under low light intensity., 2015, 48: 324–333 (in Chinese with English abstract).
[15] Chalhoub B, Denoeud F, Liu S, Parkin I A, Tang H, Wang X, Corréa M. Early allopolyploid evolution in the post- Neolithicoilseed genome., 2014, 345: 950–953.
[16] Carretero-Paulet L, Galstyan A, Roig-Villanova I, Martínez Gacía J F, Bilbao-Castro J R, Robertson D L. Genome-wide classification and evolutionary analysis of the bHLH family of transcription factors in, poplar, rice, moss, and algae., 2010, 153: 1398–1412.
[17] Feller A, Machemer K, Braun E L, Grotewold E. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors., 2011, 66: 94–116.
[18] Surhone L M, Timpledon M T, Marseken S F. Rapeseed. Germany: Betascript Publishing, 2010. pp 6–8.
[19] Kumar S V, Lucyshyn D, Jaeger K E, Alós E, Alvey E, Harberd N P, Wigge P A. Transcription factor PIF4 controls the thermosensory activation of flowering., 2012, 484: 242–245.
[20] Lucas M, Prat S. PIFs get BR right: PHYTOCHROME INTERACTING FACTORs as integrators of light and hormonal signals., 2014, 202: 1126–1141.
[21] Choi H, Oh E. PIF4 integrates multiple environmental and hormonal signals for plant growth regulation in., 2016, 39: 587–593.
[22] Wei Z, Yuan T, Tarkowská D, Kim J, Nam H G, Novák O, Li J. Brassinosteroid biosynthesis is modulated via a transcription factor cascade of COG1, PIF4 and PIF5., 2017, 174: 1260–1273.
[23] Wang Z Y, Nakano T, Gendron J, He J, Chen M, Vafeados D, Chory J. Nuclear-localized BZR1 mediates brassinosteroid- induced growth and feedback suppression of brassinosteroid biosynthesis., 2002, 2: 505–513.
Cloning and characterization of phytochrome interacting factor 4 (BnaPIF4) gene fromL.
FENG Tao and GUAN Chun-Yun*
College of Agronomy, Hunan Agricultural University / National Oilseed Crops Improvement Center in Hunan, Changsha 410128, Hunan, China
Phytochrome interacting factor 4 (PIF4) is a key transcription factor in light signaling pathway of plants, PIF4 interacts with Brassinazole-resistant (BZR) to mediate the interaction between light signal and brassinosteroid signal and participates in plant photoresponse. In this study, two novelgene were isolated fromL. cv. Xiangyou 15, they were identified on chromosomes A03 and C03 and encoding 413 and 414 amino acids, respectively, named asand, their coding sequence (CDS), full-length mRNA and full-length gene were 1242 bp and 1245 bp, 1701 bp and 1731 bp, 2527 bp and 2665 bp, respectively.andhad seven and eight exons, six and seven introns, respectively. Compared with the sequenced Zhongshuang 11,gene had a single base insertion mutation in the first intron, a deletion mutation in the fourth and sixth introns, and a longer 3'-UTR. Other sequences of the two genes did not differ between Xiangyou 15 and Zhongshuang 11. Theandgene-encoded proteins had a typical plant bHLH domain and were subcellularly localized in the nucleus. They are typical plant PIF4 proteins. Multiple sequence alignment and phylogenetic analysis showed that the BnaPIF4 protein was highly homologous to the PIF4 protein of,, and. The evolutionary relationship of PIF4 protein was consistent with that of species, and the PIF4 proteins in the closely related species are highly clustered in the phylogenetic tree. PIF4 protein repeats were observed in a large number of plants and the degree of differentiation of PIF4 was lower in lower plants than in higher plants. It indicates that PIF4 protein differentiation is a late evolutionary event and there may be functional redundancy in PIF4 protein. Yeast hybridization experiments showed that there were interactions between BnaPIF4 and BnaBZR proteins, but BnaPIF4 could not interact with the promoter ofgene, indicating thatinteracts withat the protein level but not at the transcription level. The genes expression patterns ofandin Xiangyou 15 were consistent.gene was mainly expressed in the green tissue ofL., with higher expression levels in stem epidermis, immature pods and leaves, and lower expression levels in flowers and roots, and the gene expression level ofgradually decreased in the development process ofL.
L; phytochrome interacting factor 4; gene clone; interaction of gene; gene expression; bionformatic analysis
2018-06-21;
2018-08-20;
2018-09-25.
10.3724/SP.J.1006.2019.84085
官春云, E-mail: guancy2011@aliyun.com
E-mail: 812298771@qq.com
本研究由国家重点基础研究发展计划(973计划)项目(2015CB150206)资助。
This study was supported by the National Basic Research Program (973 Program) (2015CB150206).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20180920.1555.002.html

