茶树鸟苷酸交换因子CsRopGEF1和CsRopGEF3基因的克隆及表达特性
赵 悦, 申加枝, 马媛春, 文 博, 周 琳,2, 朱旭君, 王玉花, 房婉萍,①
(1. 南京农业大学茶叶科学研究所, 江苏 南京 210095; 2. 上海市农业科学院林木果树研究所, 上海 201106)
茶树〔Camelliasinensis(Linn.) O. Ktze.〕是重要的叶用经济作物,在农业经济中有重要地位。在自然的生态系统中,茶树常受到气象灾害和病虫害的威胁,导致其产量严重损失。相对于其他园艺作物,从茶树中发掘的基因多涉及次生代谢[1-2]和茶叶品质[3]等方面,而对生长发育调控基因以及生物或非生物抗性基因方面的研究[4-6]还处于基础研究阶段。研究茶树的抗逆机制、鉴定抗性基因,进而通过分子育种技术培育优良的茶树抗逆品种具有重要意义。
Rop蛋白是植物特有的一类小G蛋白,在植物的生长发育和信号传导过程中起重要作用[7-12]。其中,鸟苷酸交换因子RopGEF能够催化Rop蛋白由GDP结合形式转化为GTP结合形式,从而活化Rop蛋白使之发挥相关功能[13-15]。相关研究结果表明:拟南芥〔Arabidopsisthaliana(Linn.) Heynh.〕Rop蛋白是对脱落酸(ABA)特异的负调控因子,并且通过RopGEF基因参与ABA信号转导[16-17];此外,外源ABA可抑制拟南芥AtRopGEF基因的表达,进而调控Rop蛋白活性[18];拟南芥RopGEF基因家族还参与了干旱、盐和低温等非生物胁迫应答,且呈现出不同的表达模式[19]。目前,对于RopGEF基因功能的研究仅限于拟南芥和水稻(OryzasativaLinn.)等模式植物[20-22]。
作者以茶树品种‘龙井长叶’(‘Longjingchangye’)为实验材料,克隆CsRopGEF1和CsRopGEF3基因全长序列,并利用生物信息学手段对2个基因编码的氨基酸序列的结构和功能进行分析;在此基础上,利用qRT-PCR分析了CsRopGEF1和CsRopGEF3基因在茶树不同组织中的表达模式及外源ABA处理对CsRopGEF1和CsRopGEF3基因表达的影响,以期为进一步研究CsRopGEFs基因在茶树生长发育中的作用及其对逆境的响应机制提供基础研究数据。
1 材料和方法
1.1 材料
供试材料为2年生茶树品种‘龙井长叶’的扦插幼苗,由江苏省南京市高淳区南京雅润茶业有限公司提供,于昼/夜温度28 ℃/22 ℃、光照时间12 h·d-1、光照强度240 μmol·m-2·s-1、空气相对湿度75%~80%的人工气候箱预培养1周。
于2017年7月2日选取长势基本一致、生长良好的幼苗,用手持喷雾器向叶面喷施100 μmol·L-1ABA[23],充分喷湿整株植株;共设置3组生物学重复,每重复35株幼苗。分别在处理0(CK)、1、2、4、8、12和24 h采集顶端第1枚芽叶,用精度1 mg的电子天平分别称取约0.1 g;另采集未经ABA处理的幼苗根(吸收根)、茎(顶端的新生茎)和叶片(顶端第1枚芽叶)各约0.1 g。所有样品均使用液氮速冻,于-80 ℃冰箱中保存,用于qRT-PCR分析。
1.2 方法
1.2.1 基因克隆 用EASYspin Plus Complex Plant RNA Kit试剂盒〔艾德莱生物科技(北京)有限公司〕提取叶片总RNA;用Thermo NanoDrop 2000c微量紫外分光光度计(美国Thermo Scientific公司)和质量体积分数1%琼脂糖凝胶电泳检测RNA的浓度和完整性。用PrimeScriptTMRT reagent Kit with gDNA Eraser (Perfect Real Time)反转录试剂盒〔宝生物工程(大连)有限公司〕合成cDNA,操作详见试剂盒说明书。
由茶树基因组数据库中获得茶树CsRopGEF1和CsRopGEF3基因序列,用IDT(http:∥sg.idtdna.com/calc/analyzer) 软件设计各自的上游引物和下游引物。CsRopGEF1-F: 5′-CATTGACGACGAGATGTGC ATC-3′; CsRopGEF1-R: 5′-CCTGATTAATGTCACT TATGGTGG-3′。CsRopGEF3-F: 5′-ATGGACAACT TGTCTAATTCGGATG-3′; CsRopGEF3-R:5′-GCAA GAGAGGAAGCTCATTATATGGAG-3′。以叶片cDNA为模板进行PCR扩增,扩增体系包含2×Hieff CanaceTMPCR Master Mix 25.0 μL、cDNA 5.0 μL、100 μmol·L-1上游特异性引物和下游特异性引物各2.5 μL,用双蒸水补足至50.0 μL。扩增程序为:94 ℃预变性5 min;94 ℃变性30 s、60 ℃退火30 s、72 ℃延伸2 min,35个循环;最后于72 ℃延伸10 min。PCR产物经质量体积分数1.2%琼脂糖凝胶电泳检测,并用Gel Extraction Kit试剂盒(美国OMEGA Bio-tek公司)进行回收;回收产物连接至pMD19-T Vector〔宝生物工程(大连)有限公司〕并转入DH5α感受态细胞,由南京金斯瑞生物科技有限公司测序。
1.2.2 生物信息学分析 用BioXM 2.6软件分析获得的2个基因序列的开放阅读框(ORF),并在NCBI(https:∥www.ncbi.nlm.nih.gov/)数据库中查找同源序列,用BLASTp软件分析保守结构域。
用ExPASy ProtParam tool(http:∥web.expasy.org/protparam)预测2个基因编码的氨基酸序列的理论相对分子质量、理论等电点、亲水性平均系数和不稳定系数。用DNAMAN 6.0软件对茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列与其他植物RopGEF1和RopGEF3基因编码的氨基酸序列进行多重比对;并用MEGA 7.0软件,采用邻接法(neighbor-joining)构建茶树CsRopGEF1和CsRopGEF3基因与其他植物RopGEF基因编码的氨基酸序列的系统进化树,bootstrap设置为1 000。用ProtScale (http:∥web.expasy.org/cgi-bin/protscale/protscale.pl)软件分析各氨基酸序列的亲水性和疏水性,并以0为分界值判断亲水性和疏水性,得分大于0的为疏水性区域,得分小于0的为亲水性区域;用NetPhos 3.1 Server(http:∥www.cbs.dtu.dk/services/NetPhos/)软件预测各氨基酸序列的磷酸化位点,并用MEME (http:∥meme-suite.org/)软件分析其保守基序。
1.2.3 基因表达分析 根据茶树CsRopGEF1和CsRopGEF3基因的cDNA序列和定量PCR引物设计原则,使用IDT软件设计各自的正向定量引物和反向定量引物。CsRopGEF1-qF: 5′-GTGTTTCGTCTGAA GATGGCT-3′; CsRopGEF1-qR: 5′-GACATCGAAC TCGAAGCTCCAT-3′。CsRopGEF3-qF: 5′-GGATG AGAATTATGATCTGGGT-3′;CsRopGEF3-qR:5′-AGCCTGACATCTCAGAGTTTG-3′。以茶树β-action基因为内参设计正向引物和反向引物(β-action-F:5′-GCCATCTTTGATTGGAATGG-3′;β-action-R:5′-GGTGCCACAACCTTGATCTT-3′)。引物合成由南京金斯瑞生物科技有限公司完成。
用LightCycler® 480 Ⅱ荧光定量PCR仪(瑞士Roche公司),并按照SYBR®PremixExTaqTMⅡ试剂盒〔宝生物工程(大连)有限公司〕使用说明书进行qRT-PCR反应,反应体系包括SYBR®PremixExTaqTMⅡ 10.0 μL、cDNA 2.0 μL、100.0 μmol·L-1上游引物和下游引物各0.8 μL,用DEPC水补足至20.0 μL。扩增程序为:95 ℃预变性3 min;95 ℃变性10 s、60 ℃退火30 s、72 ℃延伸 30 s, 40个循环;最后于72 ℃延伸7 min。采用2-ΔΔCT法[24]计算茶树CsRopGEF1和CsRopGEF3基因的相对表达量。
1.3 数据统计和分析
采用EXCEL 2003软件对数据进行统计,用SPSS 22.0软件对qRT-PCR数据进行统计学分析,并采用Duncan’s新复极差法进行多重比较。
2 结果和分析
2.1 茶树CsRopGEF1和CsRopGEF3基因的全长序列分析
以茶树品种‘龙井长叶’的叶片cDNA为模板,通过PCR扩增,获得的条带见图1。测序结果显示:茶树CsRopGEF1基因全长1 844 bp,包含长度1 731 bp的开放阅读框,编码576个氨基酸,GenBank登录号为MH160832;茶树CsRopGEF3基因全长1 509 bp,包含长度1 434 bp的开放阅读框,编码477个氨基酸,GenBank登录号为MH160833。
2.2 茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列信息分析
2.2.1 结构域预测和保守基序分析 分析结果表明:在茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列中均存在保守的PRONE结构域(图2)。且与茶树、拟南芥、水稻、可可(TheobromacacaoLinn.)和大豆〔Glycinemax(Linn.) Merr.〕的RopGEF1和RopGEF3氨基酸序列的PRONE结构域(图3)比,均含有保守基序Motif 1至Motif 7,且除拟南芥外,其他植物的RopGEF1氨基酸序列的C端还具有保守基序Motif 8,而所有植物的RopGEF3氨基酸序列均不含保守基序Motif 8。
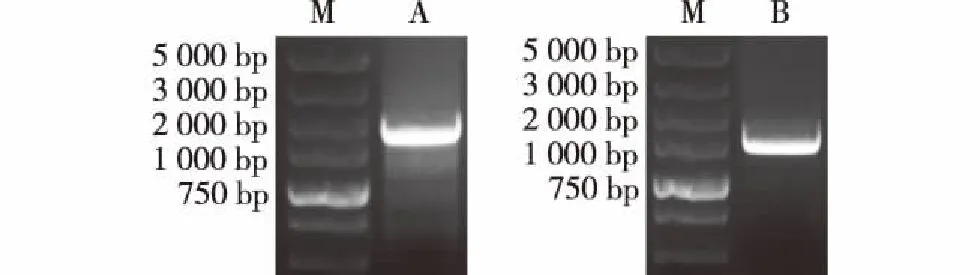
M: DNA marker; A: CsRopGEF1基因 CsRopGEF1 gene; B: CsRopGEF3基因 CsRopGEF3 gene.图1 茶树CsRopGEF1和CsRopGEF3基因的PCR扩增结果Fig. 1 PCR amplication result of CsRopGEF1 and CsRopGEF3 genes from Camellia sinensis (Linn.) O. Ktze.

图2 茶树CsRopGEF1(A)和CsRopGEF3(B)基因编码的氨基酸序列中保守结构域预测Fig. 2 Prediction of conserved domain in amino acid sequences encoded by CsRopGEF1 (A) and CsRopGEF3 (B) genes from Camellia sinensis (Linn.) O. Ktze.

Cs: 茶树Camellia sinensis (Linn.) O. Ktze.; At: 拟南芥Arabidopsis thaliana (Linn.) Heynh.; Os: 水稻Oryza sativa Linn.; Tc: 可可Theobroma cacao Linn.; Gm: 大豆Glycine max (Linn.) Merr. 1-8: 表示Motif 1至Motif 8 Representing Motif 1 to Motif 8.图3 茶树CsRopGEF1和CsRopGEF3基因与其他植物RopGEF1和RopGEF3基因编码的氨基酸序列中保守基序分析Fig. 3 Analysis on conserved motifs in amino acid sequences encoded by CsRopGEF1 and CsRopGEF3 genes from Camellia sinensis (Linn.) O. Ktze. and RopGEF1 and RopGEF3 genes from other plants
2.2.2 多重比对分析 多重比对分析结果(图4)显示:茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列分别与拟南芥AtRopGEF1(NP_195556.1)和AtRopGEF3(NP_001118903.1)、水稻OsRopGEF1(XP_015651165.1)和OsRopGEF3(XP_015636129.1)、可可TcRopGEF1(XP_007041712.2)和TcRopGEF3 (XP_007051804.2)以及大豆GmRopGEF1(XP_003537755.3)和GmRopGEF3(XP_003521141.1)的氨基酸序列具有较高的同源性,相似度均在75%以上。

Cs: 茶树Camellia sinensis (Linn.) O. Ktze.; At: 拟南芥Arabidopsis thaliana (Linn.) Heynh.; Os: 水稻Oryza sativa Linn.; Tc: 可可Theobroma cacao Linn.; Gm: 大豆Glycine max (Linn.) Merr.图4 茶树CsRopGEF1和CsRopGEF3基因与其他植物RopGEF1和RopGEF3基因编码的氨基酸序列的多重比对分析Fig. 4 Multiple alignment analysis on amino acid sequences encoded by CsRopGEF1 and CsRopGEF3 genes from Camellia sinensis (Linn.) O. Ktze. with RopGEF1 and RopGEF3 genes from other plants
2.2.3 系统进化分析 为更加明确茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列与其他植物RopGEF氨基酸序列的进化关系,选用拟南芥AtRopGEF1、AtRopGEF2(NP_171676.1)、AtRopGEF3、AtRopGEF4(NP_182113.2)、AtRopGEF5(NP_196213.2)、AtRopGEF6(NP_191125.1)、AtRopGEF7(NP_195821.1)、AtRopGEF8(NP_189105.1)、AtRopGEF9(NP_193060.1)、AtRopGEF10(NP_197457.1)、AtRopGEF11(NP_175634.6)、AtRopGEF12(NP_178104.1)、AtRopGEF13(NP_001319564.1)和AtRopGEF14(NP_174446.2)的氨基酸序列,水稻OsRopGEF1、OsRopGEF3、OsRopGEF7(XP_015622383.1)、OsRopGEF9(XP_015640605.1)、OsRopGEF10(XP_015622938.1)和OsRopGEF11(XP_015628594.1)的氨基酸序列,玉米(ZeamaysLinn.)ZmRopGEF1(NP_001132788.1)、ZmRopGEF3(XP_008653079.1)、ZmRopGEF9(XP_008675023.1)、ZmRopGEF10(XP_008645515.1)和ZmRopGEF14(NP_001130409.1)的氨基酸序列,可可TcRopGEF1、TcRopGEF3、TcRopGEF5(XP_017984042.1)、TcRopGEF7(XP_017977890.1)、TcRopGEF8(XP_007013139.2)、TcRopGEF12(XP_017978795.1)、TcRopGEF14-1(XP_007040892.2)和TcRopGEF14-2(XP_017973488.1)的氨基酸序列,烟草(NicotianatabacumLinn.)NtRopGEF1(XP_016445632.1)、NtRopGEF3(XP_016491474.1)、NtRopGEF5(XP_016439450.1)、NtRopGEF7(XP_016465518.1)、NtRopGEF8(XP_016484735.1)、NtRopGEF12(XP_016460052.1)和NtRopGEF14(XP_016504442.1)的氨基酸序列,高粱〔Sorghumbicolor(Linn.) Moench〕SbRopGEF1(XP_002462770.1)、SbRopGEF3(XP_002464972.1)、SbRopGEF7(XP_002439905.1)、SbRopGEF9(XP_002441507.1)、SbRopGEF12-1(XP_021312196.1)、SbRopGEF12-2(XP_002456125.2)和SbRopGEF14(XP_002452689.1)的氨基酸序列,与上述2个茶树基因编码的氨基酸序列共同构建系统进化树,结果见图5。结果显示:茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列分别与双子叶植物拟南芥、可可和烟草的RopGEF1和RopGEF3的氨基酸序列同源性较高,而与单子叶植物水稻、玉米和高粱的RopGEF1和RopGEF3的氨基酸序列同源性较低。
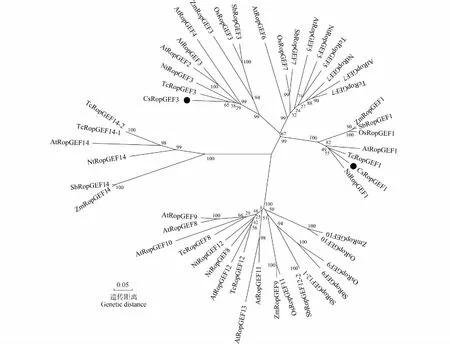
Cs: 茶树Camellia sinensis (Linn.) O. Ktze.; At: 拟南芥Arabidopsis thaliana (Linn.) Heynh.; Os: 水稻Oryza sativa Linn.; Zm: 玉米Zea mays Linn.; Tc: 可可Theobroma cacao Linn.; Nt: 烟草Nicotiana tabacum Linn.; Sb: 高粱Sorghum bicolor (Linn.) Moench. 分支上的数值表示bootstrap百分数 Data above the branches indicate percentages of bootstrap.图5 茶树CsRopGEF1和CsRopGEF3基因与其他植物RopGEF基因编码的氨基酸序列的系统进化树Fig. 5 Phylogenetic tree of amino acid sequences encoded by CsRopGEF1 and CsRopGEF3 genes from Camellia sinensis (Linn.) O. Ktze. with RopGEF genes from other plants
2.2.4 理化性质预测 ExPASy ProtParam tool预测结果显示:茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列的分子式分别为C2797H4402N762O856S32和C2356H3696N648O752S29,理论相对分子质量分别为63 420和54 060,理论等电点分别为pI 6.69和pI 5.10;分别有69和70个带负电荷的氨基酸残基(天冬氨酸和谷氨酸)及65和33个带正电荷的氨基酸残基(精氨酸和赖氨酸);蛋白质三维结构不稳定系数分别为57.57和49.88,不稳定系数均大于40,呈不稳定状态。推测茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列均属于不稳定的酸性蛋白质。
亲水性和疏水性预测结果(图6)显示:在茶树CsRopGEF1基因编码的氨基酸序列中,疏水性最强的位点是第211位的亮氨酸,亲水性最强的位点是第369位的天冬酰胺;在茶树CsRopGEF3基因编码的氨基酸序列中,疏水性最强的位点是第175位的半胱氨酸,亲水性最强的位点是第259位的赖氨酸。总体上看,茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列的亲水性区域均大于疏水性区域,且亲水性平均系数分别为-0.368和-0.552,表明二者都属于亲水性蛋白。
磷酸化位点预测结果(图7)显示:在茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列中存在多个磷酸化位点,其中丝氨酸磷酸化位点最多,表明这2个基因编码的氨基酸序列在发挥生物学功能时可能受到磷酸化的调控。
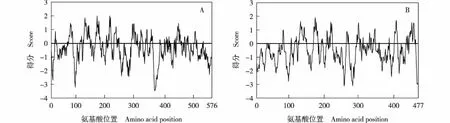
图6 茶树CsRopGEF1(A)和CsRopGEF3(B)基因编码的氨基酸序列的亲水性和疏水性分析Fig. 6 Analysis on hydrophilicity and hydrophobicity of amino acid sequences encoded by CsRopGEF1 (A) and CsRopGEF3 (B) genes from Camellia sinensis (Linn.) O. Ktze.

: 丝氨酸Serine; : 苏氨酸Threomine; : 酪氨酸Tyrosine; : 阈值Threshold.图7 茶树CsRopGEF1(A)和CsRopGEF3(B)基因编码的氨基酸序列中磷酸化位点预测Fig. 7 Prediction on phosphorylation site in amino acid sequences encoded by CsRopGEF1 (A) and CsRopGEF3 (B) genes from Camellia sinensis (Linn.) O. Ktze.
2.3 茶树CsRopGEF1和CsRopGEF3基因的表达分析
2.3.1 组织特异性分析 茶树CsRopGEF1和CsRopGEF3基因在不同组织中相对表达量的差异见图8。结果显示:茶树CsRopGEF1和CsRopGEF3基因表达存在组织特异性。其中,CsRopGEF1基因的相对表达量在不同组织间差异显著(P<0.05),以茎中最高、叶片中最低;CsRopGEF3基因的相对表达量则在根中最高,在叶片中最低,但根与茎中CsRopGEF3基因的相对表达量无显著(P>0.05)差异,而叶片中CsRopGEF3基因的相对表达量与根和茎差异显著。整体上看,CsRopGEF1基因的相对表达量高于CsRopGEF3基因。
2.3.2 对外源ABA处理的响应 经100 μmol·L-1外源ABA处理后茶树CsRopGEF1和CsRopGEF3基因表现出不同的表达模式(图9)。其中,CsRopGEF1基因的相对表达量在24 h内整体显著下调;而CsRopGEF3基因的相对表达量则在处理前期显著上调,在处理4 h时达到峰值,随后逐渐下降,但仍显著高于对照(0 μmol·L-1ABA),整体仍为上调。

: CsRopGEF1基因 CsRopGEF1 gene; □: CsRopGEF3基因 CsRopGEF3 gene.不同的小写字母表示同一个基因的相对表达量在不同组织间差异显著(P<0.05) Different lowercases indicate the significant (P<0.05) difference in relative expression level of the same gene among different tissues.图8 茶树不同组织中CsRopGEF1和CsRopGEF3基因的相对表达水平比较Fig. 8 Comparison on relative expression level of CsRopGEF1 and CsRopGEF3 genes in different tissues of Camellia sinensis (Linn.) O. Ktze.
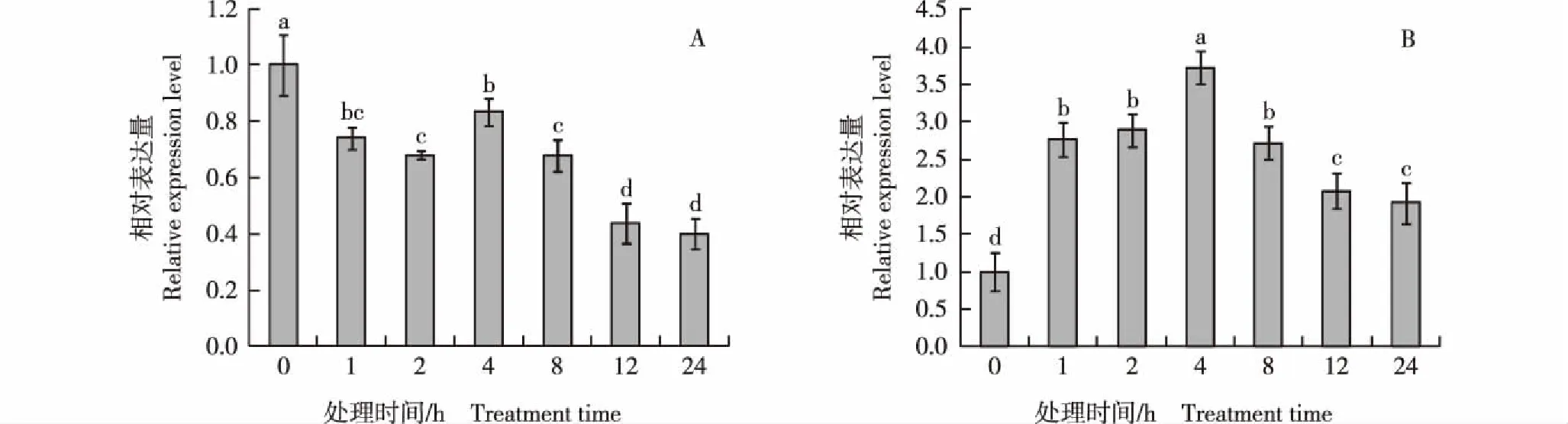
不同的小写字母表示差异显著(P<0.05)Different lowercases indicate the significant (P<0.05) difference.图9 经100 μmol·L-1外源ABA处理后茶树CsRopGEF1(A)和CsRopGEF3(B)基因相对表达量的比较Fig. 9 Comparison on relative expression level of CsRopGEF1 (A) and CsRopGEF3 (B) genes from Camellia sinensis (Linn.) O. Ktze. after 100 μmol·L-1 exogenous ABA treatment
3 讨论和结论
在植物的生长发育以及信号传导过程中RopGEF蛋白起了至关重要的作用,其生物学功能及分子调控机制越来越受到关注[25-27]。RopGEF蛋白的PRONE结构域的C端和N端对中间的结构域有调节功能,具有自主抑制作用;当RopGEF蛋白的C端丝氨酸(Ser)残基被受体激酶(receptor-like kinases)磷酸化后,PRONE结构域的自主抑制作用被释放,结合并激活Rop蛋白活性,从而调节花粉管极性与根系的生长发育[13,28]。而在本研究中,茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列均含有PRONE结构域,并包含数个Ser磷酸化位点,推测茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列也具有类似功能。本研究结果还表明:RopGEF氨基酸序列的PRONE结构域均含有保守基序Motif 1至Motif 7,其中,除拟南芥外,茶树、水稻、可可和大豆的RopGEF1氨基酸序列的C端都含有保守基序Motif 8,而茶树、拟南芥、水稻、可可和大豆的RopGEF3氨基酸序列均不含有保守基序Motif 8,说明不同植物种类的RopGEF1蛋白总体上较RopGEF3蛋白具有更高的保守性。从系统进化树看,茶树CsRopGEF1和CsRopGEF3基因编码的氨基酸序列分别与双子叶植物拟南芥、可可和烟草的RopGEF1和RopGEF3氨基酸序列划分为一支,而单子叶植物水稻、玉米和高粱的RopGEF1和RopGEF3氨基酸序列分别单独为一支,推测在进化过程中RopGEF1和RopGEF3基因的分化在双子叶植物和单子叶植物之间具有不同的进化起源。
相关的研究结果表明:拟南芥AtRopGEF基因具有显著的组织特异性,AtRopGEF7、AtRopGEF8、AtRopGEF9和AtRopGEF13基因在花中具有较高的表达量[18],而AtRopGEF4和AtRopGEF10基因则在根毛中具有较高的表达量[29];AtRopGEF1、AtRopGEF2、AtRopGEF13和AtRopGEF14基因在根中的表达量低于叶片,而AtRopGEF3、AtRopGEF4、AtRopGEF8和AtRopGEF10基因在根中的表达量则显著高于叶片[30]。此外,拟南芥AtRopGEF1、AtRopGEF4和AtRopGEF10蛋白还被证实参与拟南芥根部的生长发育和形态建成[21,31]。本研究中,茶树CsRopGEF1基因主要在茎中表达,CsRopGEF3基因则在根和茎中具有较高的表达量,推测茶树CsRopGEF1和CsRopGEF3基因在根和茎的生长发育过程中可能具有重要作用。此外,经100 μmol·L-1外源ABA处理后,茶树CsRopGEF1基因的相对表达量整体显著下调,而CsRopGEF3基因则整体上调;拟南芥RopGEFs基因对外源ABA处理的响应也存在类似的规律[16],提示茶树CsRopGEF1和CsRopGEF3基因在ABA信号转导途径中受不同的调控作用。另外,大量研究已经证实,逆境胁迫(如干旱、盐和低温)能够促进植物体内ABA的合成[32-33]。Li等[34]的研究结果表明:在存在ABA的条件下,拟南芥的蛋白磷酸酶2C(PP2C)失去活性,AtRopGEF1蛋白逐渐水解,使得下游SNF1相关蛋白激酶2(SnRK2)去磷酸化,进一步触发ABA信号转导;而在缺乏ABA的条件下,AtRopGEF1蛋白激活Rop蛋白,使PP2C保持活性,且具有活性的PP2C能够维持AtRopGEF1蛋白活性,并继续下游SnRK2的磷酸化,终止ABA信号传导。然而,在逆境条件下茶树CsRopGEF1蛋白是否也具有类似的转导机制,则有待更深入的研究。
本研究中,茶树CsRopGEF1和CsRopGEF3基因分别受到外源ABA的负调控和正调控,推测外源ABA对茶树CsRopGEF1和CsRopGEF3基因具有不同的调控方式,从而参与茶树的生长发育和对胁迫应答的信号转导,但具体的调控机制有待后期进一步深入研究。

