Dickeya sp.DCE-01关键脱胶酶基因克隆及表达
曾洁,成莉凤,段盛文,冯湘沅,郑科,刘志远,周映君,杨琦,刘正初
(中国农业科学院麻类研究所,长沙410205)
麻类等韧皮纤维在加工利用过程中必须去除麻纤维伴生物,俗称胶质,才能获得可供利用的纤维[1]。脱胶是为了脱去韧皮纤维中的伴生物,其主要成分为果胶、半纤维素和木质素。果胶是一种存在于高等植物细胞壁中的杂多糖,与植物组织中的半纤维素、木质素、纤维素和蛋白质等相互交联[2]。半纤维素是连接木质素和纤维素的一类杂聚多糖,广泛存在于各种植物纤维的细胞壁中。木聚糖和甘露聚糖都属于半纤维素[3-4]。因此,果胶酶、木聚糖酶、甘露聚糖酶是生物脱胶的关键酶。与传统的沤麻和化学脱胶方法相比,苎麻生物脱胶方法具有“高效、节能、减排”等优势,是一种环境友好型加工方式[5-7]。
Dickeya sp.DCE-01是本团队长期从事麻类等草本纤维生物精制科学与工程研究选育到的一株“广谱性”、“高效性”脱胶菌,该菌株胞外分泌果胶酶、甘露聚糖酶和木聚糖酶,对苎麻、红麻、黄麻、亚麻、罗布麻、龙须草、麦草等草本纤维原料均能起到很好的脱胶效果,且6 h能独立完成苎麻脱胶,不需要其他化学方法后处理补充。其脱胶功能的广谱性和高效性在国内外都处于领先地位[8-9]。
本研究拟通过对DCE-01菌株关键脱胶酶基因(果胶酶基因、甘露聚糖酶基因和木聚糖酶基因)的克隆及表达分析,深入挖掘DCE-01菌株中脱胶酶基因,旨在为研发高效麻类脱胶酶制剂提供科学依据。
1 材料
1.1 菌株与质粒
菌株DCE-01是由中国农业科学院麻类研究所生物加工课题组分离筛选与保存。E.coli BL21,E.coli Top 10菌株和质粒pET-28a购自TaKaRa生物公司。
1.2 主要试剂
DNA胶回收试剂盒、细菌基因组抽提试剂盒和小量质粒提取试剂盒均购自OMEGA公司;rTaq PCR酶、TA连接试剂盒(质粒pMD 19-T)、KOD-Plus-高保真PCR试剂盒和T4 ligase试剂盒均购自TOYOBO生物公司;DNA Marker DL 15000和FastDigest内切酶均购自Thermo scientific生物公司;卡那霉素、X-gal、IPTG、氨苄青霉素、酵母提取物、琼脂粉和胰蛋白胨购自 Sigma公司;其余化学试剂均为分析纯级产品。引物合成和核酸测序由上海生工生物工程技术服务有限公司完成。
1.3 主要培养基
LB培养基:胰蛋白胨1%,NaCl 1%,酵母提取物0.5%。固体培养基需加1.5%的琼脂粉[10]。
SOC培养基:胰蛋白胨 2%,NaCl 0.05%,酵母提取物 0.5%,2.5 mmol/L KCl,0.01 mol/L MgCl2和 0.02 mol/L葡糖糖[10]。
活化培养基:蛋白胨0.5%,NaCl 0.5%,葡萄糖1%,牛肉膏0.5%[10]。
2 方法
2.1 菌株DCE-01的活化
挑取一环课题组斜面管保存的DCE-01菌种菌苔接种于5 mL活化培养基中,振荡悬浮,35℃培养5 h。稀释涂平板,35℃培养16~18 h。挑取单菌落接种至5 mL LB培养基,35℃,180 r/min震荡培养 12~16 h,用于基因组 DNA提取[10]。
2.2 DCE-01菌株基因组DNA提取
采用细菌基因组DNA提取试剂盒抽提DCE-01基因组 DNA,具体操作参照《细菌基因组DNA抽提试剂盒说明书》。用0.7%的琼脂糖凝胶电泳检测基因组DNA提取质量。于-20℃保存备用。
2.3 引物设计
根据菌株DCE-01的基因组DNA序列信息,利用生物信息学软件DNAMAN设计特异引物。

表1 生物脱胶关键酶基因的扩增引物Tab.1 Primers of key genes of bio-degumming enzymes
2.4 PCR扩增DCE-01关键脱胶酶基因
以DCE-01菌株基因组DNA为模板,采用表1的引物和高保真聚合酶 KOD Plus-Neo-进行 PCR扩增。PCR反应体系为:10×PCR Buffer 5μL,dNTPs 5μL,MgSO43μL,Fa1 1μL,Ra1 1μL,KOD Plus-DNA聚合酶1μL,基因组DNA 1μL,用无菌ddH2O补充到总反应体积为50μL,快速离心混和,参照TOYOBO《KOD-Plus-高保真PCR试剂盒说明书》进行PCR扩增。
2.5 目的片段回收
采用1%琼脂糖凝胶电泳检测PCR产物,切取目的基因片段进行回收,具体步骤参照OMEGA公司的《DNA凝胶回收试剂盒说明书》。
2.6 平末端DNA加A尾
加 A尾反应体系为:10×buffer 2μL,dNTPs 3μL,MgCl21μL,rTaq酶0.5μL,PCR产 物13.5μL。72℃ 保温30 min,电泳,切胶回收加尾产物。
2.7 目的片段的TA连接
参照试剂盒说明进行TA连接,具体步骤如下:
(1)将Solution I溶液与pMD18-T载体等试剂置于冰上融化;
(2)于200μL灭菌反应管中依次加入下列反应体系:目的DNA片段4.5μL,Solution I 5μL,pMD18-T载体1μL;
(3)轻缓混匀,低速短暂离心使反应体系集中于管底,将连接体系置于16℃孵育过夜。
2.8 重组质粒构建与转化
(1)将E.coli Top10感受态细胞置于冰上融化;
(2)将酶连产物按1∶100的比例加入Top10感受态细胞中,轻缓混匀,置于冰水中孵育30 min;
(3)取孵育后的混合液,42℃热激准确反应90 s,迅速置于冰上静置5~10 min;
(4)加入 800μL SOC液体培养基,37℃、180 r/min振荡培养 90 min;
(5)取 100μL菌液涂布于 LB固体平板上(已加 20μL 20 mg/mL IPTG、40μL 20 mg/mL X-gal、30μL 100 mg/mL Amp),于 37℃过夜培养。
2.9 重组子筛选与鉴定
(1)菌落PCR验证
从筛选平板上挑取阳性克隆单菌,用10μL无菌双蒸水溶解悬浮后,取1μL混合菌液用各自目的基因的引物进行PCR扩增,鉴定重组子是否正确[10]。
(2)重组子的酶切鉴定
参照OMEGA公司的《小量质粒提取试剂盒说明书》提取质粒,用限制性内切酶Bam H I和Hand III对重组质粒进行双酶切,验证目的基因片段是否正确。
2.10 高效表达重组菌的构建与验证
用限制性内切酶Bam H I和Hand III分别对重组质粒pMD 19-T-P、pMD 19-T-X和pMD 19-T-M及载体pET-28a双酶切。经电泳、切胶、纯化,将目的片段与线性的pET-28a载体进行连接,并转化到E.coli BL21中,通过含100μg/mL卡那霉素的LB平板对重组菌进行筛选。挑取典型单菌落于LB培养液,过夜培养后提取质粒,用限制性内切酶Bam H I和Hand III对重组质粒pET-28a-P、pET-28a-X和pET-28a-M双酶切,电泳检测,验证目的片段是否正确插入。鉴定后的重组质粒由上海生工生物公司进行DNA序列测定。
2.11 重组菌的诱导表达与产酶分析
挑取正确插入目的基因片段的重组菌接种于5 mL LB培养液(含100μg/mL卡那霉素)中,37℃、180 r/min过夜培养。取1 mL菌液接种至100 mL LB培养液(含100μg/mL卡那霉素),37℃、180 r/min培养至OD600达到0.4~0.6时,添加 IPTG到终浓度1 mmol/L,放入水浴摇床中,120 r/min,30℃诱导产酶。以未插入目的片段的空载体菌株pET-28a/BL21为对照,与重组菌进行相同处理。
取1 mL菌液10000 r/min离心5 min,收集菌体,用50μL无菌双蒸水与等体积2×蛋白上样缓冲液悬浮菌体,沸水浴5 min,离心取上清,用于SDS-PAGE电泳点样[11-13]。
分别取诱导6、9、12、15、18、21、24 h的发酵菌液进行超声破碎,超声参数设置为:强度30%,超声5 s,间隔5 s,时间30 min[10]。将裂解后的菌体溶液10000 r/min离心10 min,上清液用于酶活力测定。果胶酶、甘露聚糖酶及木聚糖酶活力测定方法分别参照文献[14-17]。
3 结果与分析
3.1 DCE-01菌株基因组DNA提取
由图1可以看出,检测到清晰的单一条带。OD260/OD280值为1.6~1.8,说明该核酸纯度较高,可供后续试验备用。
3.2 关键酶基因的扩增
以提取的DCE-01基因组DNA为模板,进行果胶酶基因(pel E)、木聚糖酶基因(xyn)和甘露聚糖酶基因(man)的PCR扩增。采用1%琼脂糖凝胶电泳,由图2可知,该PCR产物条带清晰、无杂带,基因片段大小为1000~1300 bp,与预测结果一致。经测序分析,果胶酶基因片段为1179 bp、木聚糖酶基因片段为1251 bp、甘露聚糖酶基因片段为1137 bp。初步说明关键酶基因扩增成功。

图1 DCE-01基因组DNAFig.1 Genomic DNA of DEC-01 strain
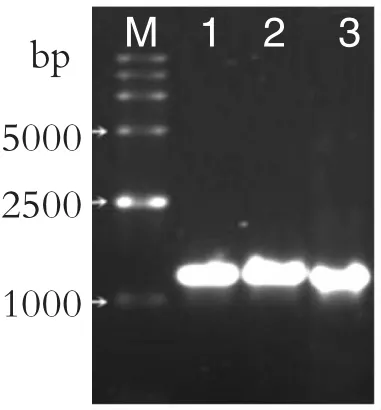
图2 关键酶基因PCR扩增产物Fig.2 PCR products of key enzyme gene
3.3 TA克隆重组子筛选与鉴定
将PCR扩增得到的目的基因片段加 A尾,再与pMD 19-T进行TA连接,导入E.coli Top10受体,构建获得重组质粒pMD 19-T-P、pMD 19-T-X和pMD 19-T-M。对阳性克隆进行菌落PCR,图3结果表明:1200 bp附近出现单一清晰的条带,条带大小与关键酶基因扩增结果和预计基因片段大小基本一致。说明3种关键脱胶酶基因片段成功连接到了pMD 19-T载体上。
对PCR验证后的重组菌提质粒,双酶切验证,电泳检测结果见图4。通过双酶切获得两条清晰单一条带,分别为线性载体pMD 19-T与目的基因片段。线性载体pMD 19-T片段约2600 bp,目的基因片段在1000~1300 bp附近,与预计DNA片段大小一致,说明重组子构建成功。

图3 菌落PCR检测重组子Fig.3 Detection of recombinants by PCR

图4 酶切检测重组子Fig.4 Detection of recombinants
3.4 高效表达重组菌的鉴定与产酶分析
经连接转化,成功构建重组菌 pET-28a-P/BL21、pET-28a-X/BL21和 pET-28a-M/BL21。提取质粒,用限制性内切酶Bam H I和Hand III对重组子进行酶切验证,酶切鉴定电泳检测结果见图5。通过双酶切获得两条清晰单一条带,分别为 pET-28a线性空载体与目的基因片段。线性载体pET-28a约5000 bp,目的片段条带在1000~3000 bp,与预计片段大小一致。
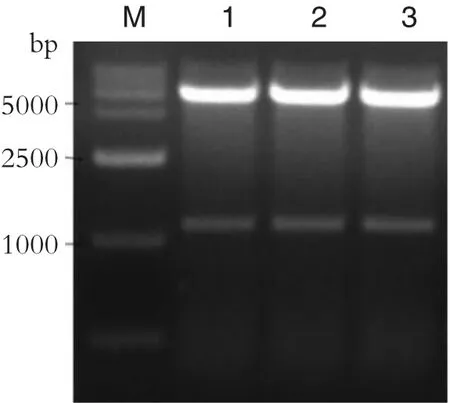
图5 酶切检测重组子Fig.5 Detection of recombinants
将构建的重组菌株诱导表达后进行SDS-PAGE电泳,检测蛋白表达量,以未插入目的片段的空载体菌株pET-28a/BL21为对照,与重组菌进行相同处理,结果(见图6)可看到明显表达条带,其表观分子量与预测的带有部分载体的融合蛋白分子量接近,即果胶酶(pel E)、木聚糖酶(xyn)、甘露聚糖酶(man)蛋白的表观分子量分别为43.8、45.7、41.8 kDa。由此可见,从DCE-01菌株中克隆出来的3个关键酶基因转化至菌株pET-28a/BL21中均达到了特异性表达的效果。

图6 SDS-PAGE检测亲本酶、多基因重组菌株表达Fig.6 Expression of parent enzyme and multiple gene recombinant strains by SDS-PAGE
以未插入目的片段的空载体菌株pET-28a/BL21为对照,与重组菌进行相同处理。分别对重组菌pET-28a-P/BL21、pET-28a-X/BL21和 pET-28a-M/BL21进行诱导表达,测定不同诱导时间的酶活,结果(见图7~9)发现3种酶酶活均较高,尤其以果胶酶和甘露聚糖酶最为明显,果胶酶酶活最高达471.97 U/mL,甘露聚糖酶则达到16882.86 U/mL。
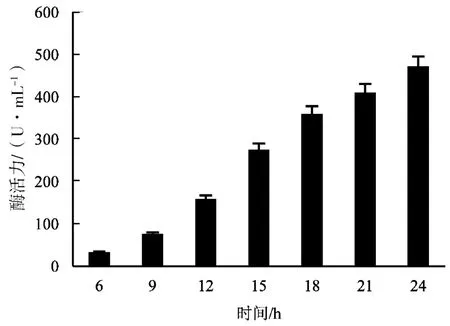
图7 不同诱导时间的果胶酶活力Fig.7 Activity of pectinase in different induction time

图8 不同诱导时间的木聚糖酶活力Fig.8 Activity of xylanase in different induction time
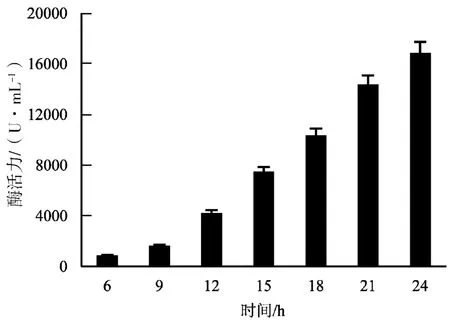
图9 不同诱导时间的甘露聚糖酶活力Fig.9 Activity ofβ-mannanase in different induction time
4 结论与讨论
通过对广谱性高效脱胶菌株DCE0-1的关键性脱胶酶基因的研究,成功获得了高效表达果胶酶、甘露聚糖酶和木聚糖酶的重组菌pET-28a-P/BL21、pET-28a-M/BL21和pET-28a-X/BL21。经酶活测定,果胶酶、甘露聚糖酶、木聚糖酶活力分别高达471.97、16882.86、64.32 U/mL。
与其他报道相比,本研究构建的甘露聚糖酶重组菌pET-28a-M/BL21具有明显优势。杨家兴[18]在β-甘露聚糖酶在黑曲霉中的表达研究中报道的粗酶液的最高酶活为521 U/mL;张立霞[19]在硫色曲霉来源的β-甘露聚糖酶的异源表达及酶学性质研究中报道的甘露聚糖酶的产量为587 U/mL;刘项羽等[20]关于β-甘露聚糖酶基因在枯草芽孢杆菌中的克隆及表达中报道的酶活力最高为2748.82 U/mL。本研究中构建的甘露聚糖酶表达菌株比上述报道的菌株甘露聚糖酶活力高14~31倍,为脱胶用甘露聚糖酶制剂的开发利用奠定了良好的基础。
本团队后续将会对基因工程菌种产酶的诱导条件(诱导剂浓度、时间、温度等)进一步优化,也将对基因工程菌种所产的果胶酶、甘露聚糖酶和木聚糖酶进行纯化及末端氨基酸肽段测序(或western blot分析),验证SDS-PAGE图谱中的目标蛋白即为对应的脱胶关键酶蛋白。
[1]Deng L,Geng M J,Zhu DW,etal.Effect of chemical and biological degumming on the adsorption of heavymetal by cellulose xanthogenates prepared from Eichhornia crassipes[J].Bioresource Technology,2012,10(7):41-45.
[2]谢明勇,李精,聂少平.果胶研究与应用进展[J].中国食品学报,2013,13(8):1-14.
[3]黎征帆.苎麻半纤维素组分研究[D].上海:东华大学,2015.
[4]余紫苹,彭红,林妲,等.植物半纤维素结构研究进展[J].高分子通报,2011(6):48-54.
[5]刘正初,彭源德,冯湘沅,等.苎麻生物脱胶新技术工业化生产应用研究[J].纺织学报,2001(2):27-29.
[6]Du B,Zheng L J.Bio-degumming process on jute fiber for textile[J].Journal of Biotechnology,2008,136(4):S474-S474.
[7]Liu Z C,Duan SW,Sun Q X,et al.A rapid process of ramie bio-degumming by Pectobacterium sp.CXJZU-120[J].Textile Research Journal,2012,82(15):1553-1559.
[8]Duan SW,Feng X Y,Cheng L F,et al.Bio-degumming technology of jute bast by Pectobacterium sp.DCE-01[J].Amb Express,2016,6(1):86.
[9]Cheng Y,Liu ZC,Zeng J,etal.Construction and co-expression of polycistronic plasmids encoding bio-degumming-related enzymes to improve the degumming process of ramie fibres[J].Biotechnology Letters,2016,38(12):28-34.
[10]成莉凤.DCE-01菌株果胶酶基因克隆与表达及其多样性研究[D].北京:中国农业科学院,2013.
[11]李琦,成莉凤,曾洁,等.麻类脱胶高效菌株 DCE-01的 Pel4I8基因克隆与表达[J].中国麻业科学,2015,37(4):206-210.
[12]李琦,成莉凤,曾洁,等.麻类脱胶高效菌株 DCE-01的 Pel325基因克隆与表达[J].中国麻业科学,2015,37(3):157-161.
[13]Huang G L,Wong K L.Analysis of interactions between carbohydrates and proteins using fluorescent labeling and SDS-PAGE[J].Zeitschrift für Naturforschung C A Journal of Biosciences,2008,63(3-4):311-312.
[14]徐君飞.枯草芽孢杆菌BE-91木聚糖酶基因多样性及其克隆与表达研究[D].长沙:湖南农业大学,2010.
[15]顾佳佳,刘正初,张运雄,等.CXJZ95-198菌株果胶酶活力检测方法研究[J].中国麻业科学,2006,28(6):309-312.
[16]张飞,岳田利,费坚,等.果胶酶活力的测定方法研究[J].西北农业学报,2004,13(4):134-137.
[17]曾莹,钟晓凌,夏服宝.木聚糖酶活力测定条件研究[J].生物技术,2003,13(5):21-22.
[18]杨家兴.β-甘露聚糖酶在黑曲霉中的表达及性质研究[D].天津:天津科技大学,2016.
[19]张立霞.硫色曲霉来源的β-甘露聚糖酶的异源表达及酶学性质研究[D].天津:天津科技大学,2017.
[20]刘项羽,徐美娟,杨套伟,等.β-甘露聚糖酶基因在枯草芽孢杆菌中的克隆及表达[J].应用与环境生物学报,2012,18(4):672-677.

