番茄多胺氧化酶(PAO)基因家族鉴定及表达分析
李 会,王 超,黄思杰,陈 健,杨立飞*
(1 南京农业大学 园艺学院,南京 210095;2 环境保护部南京环境科学研究所,南京 210042;3 江苏省农业科学院 农产品质量安全与营养研究所,南京 210014)
天然多胺 (polyamine,PA) 包括腐胺(putrescine,Put)、亚精胺(spermidine,Spd) 和精胺(spermine,Spm),是真核生物与原核生物细胞代谢过程中必需的一类小分子量多聚有机阳离子,其普遍分布于高等植物中[1]。目前,人们认为PA参与植物一系列生理过程,包括生长、发育和应激反应[2-3]。内源性PA含量取决于合成和分解代谢之间的动态平衡,负责PA分解代谢的2类胺氧化酶分别为铜胺氧化酶 (copper amine oxidase,CuAO) 和多胺氧化酶 (polyamine oxidase,PAO)[4-5]。PAO在维护细胞多胺平衡方面扮演了重要的角色,植物PAO负责含次级氨基PAs的分解代谢,氧化Spd和Spm形成相应的氨基醛和二氨基丙烷,并释放过氧化氢 (H2O2)[6-7]。
越来越多的研究表明,PAO催化产生的H2O2在调控一系列生理生化过程中发挥重要作用,并直接或间接地参与植物生长发育的调控,例如,PAO催化Spd产生的H2O2可以激活Ca2+渗透通道来调控花粉管生长[8],PAO还参与植物的氮代谢[9],PAOs介导的分解代谢与程序性细胞死亡有关[10]。与此同时,PAO催化PAs的反应,对于植物抵抗生物或非生物胁迫也有重要的影响,例如,通过PA氧化产生的H2O2参与超敏反应,调控植物对病原菌的抗性[11]。此外,PAO还可以通过基因表达的差异性影响氧化还原相关蛋白的表达,从而使番茄增强对盐胁迫的抵抗能力[12]。
植物PAO在不同的亚细胞位置具有特异性表达,例如,Slocum等[13]研究显示豆科植物的PAO主要存在于胚轴组织的细胞壁上,而覃广泉等[14]首次报道大豆PAO在细胞膜和液泡膜上也有分布。具有不同的生化功能的PAO,其亚细胞定位也具有特异性,负责PAs终极氧化反应的ZmPAO1(玉米)、OsPAO7(水稻)位于质外体[15-16],而参与Spm逆向转化的AtPAO1~5和OsPAO1、OsPAO3~5定位于细胞质及过氧化物酶体中[17-18],这表明了PAO在不同亚细胞中的表达对于其发挥不同的生理作用有着重要的意义。
植物PAOs由一个或几个成员组成的小基因家族编码,例如,拟南芥基因组中含有5个PAO基因 (AtPAO1~AtPAO5);水稻基因组中包含7个PAO基因 (OsPAO1~OsPAO7)[19-20];Cervelli等[21]也从玉米中分离到3个PAO基因。目前已从拟南芥[17]、水稻[18]、玉米[21]、大麦[22]、烟草[23]、苹果[24]和杨树[25]等植物中鉴定分离出了一系列编码PAO的基因,但是其中只有拟南芥的PAO基因家族得到广泛的关注和研究,在双子叶植物中,对PAO基因家族成员的研究和分析还比较少。
番茄作为主要的蔬菜作物之一,它不但是重要的农艺作物,也是研究双子叶植物的模式植物之一。目前,对于番茄PAO的研究多集中于总酶活测定,而对番茄PAO基因家族信息的全面分析鲜有报道。本研究以番茄基因组数据为基础,对番茄PAO基因家族进行了鉴定和分析,系统地分析了番茄PAO基因家族的基本信息以及组织表达情况,旨在为进一步对番茄PAO基因的克隆以及功能鉴定提供基础。
1 材料和方法
1.1 材料与仪器
试验材料番茄 (Solanumlycopersicum)品种为‘合作906’,购自上海长种番茄种业有限公司;RNA提取试剂盒购自TIANGEN公司;反转录试剂盒和荧光定量PCR分析试剂盒 (SYBR Premix Ex Taq) 均购自大连TaKaRa公司。
所用实验仪器有光照培养箱 (宁波江南仪器有限公司)、电子天平 (AR5120,美国AHOMS公司)、实时荧光定量PCR仪(美国Applied Biosystems 7500 Fast Real-Time PCR System)、凝胶成像系统 (Gel Doc 2000,上海天能科技有限公司)、普通PCR仪 (美国BIORAD/伯乐,C1000)等。
1.2 方 法
1.2.1材料培养选取饱满的番茄种子用1% NaClO溶液消毒10 min,用蒸馏水冲洗3遍,然后用去离子水浸种6~8 h,将其播种于培养皿中于黑暗处催芽。待根长1 cm时挑选生长状况一致的幼苗,转入Hoagland营养液中培养,光照培养箱的培养条件是:温度26 ℃/22 ℃、光周期16/8 h、相对湿度75%、光照强度200 μmol·m-2·s-1。培养10 d,分别取真叶、子叶、茎、根提取总RNA。
1.2.2番茄SlPAOs的筛选及进化分析从拟南芥基因组数据库 TAIR (http://www.arabidopsis.org/) 中下载已经报道的AtPAOs序列,作为检索依据在SGN (https://solgenomics.net/) 的番茄基因组数据库中进行Blast检索,获得番茄同源SlPAOs基因序列及其氨基酸序列信息。已报道的水稻OsPAOs序列从水稻基因组数据库RGAP (http://rice.plantbiology.msu.edu/) 获得[18]。针对筛选到的番茄SlPAOs氨基酸序列以及拟南芥AtPAOs、水稻OsPAOs序列,利用Clustal X 2.1进行多重比对,再通过MEGA 6.0构建系统发育进化树。
表1 引物序列信息
1.2.3番茄SlPAOs基因结构分析将SGN中获得的番茄SlPAOs基因组序列和CDS序列,分别输入GSDS2.0 (http://gsds.cbi.pku.edu.cn/),进行内含子和外显子结构分析[26]。
1.2.4番茄SlPAOs蛋白二级结构预测与分析利用在线软件ExPaSy中提供的Compute pI工具 (http://web.expasy.org/compute_pi/) 计算番茄SlPAOs的分子量、理论等电点;ProtScale工具 (http://web.expasy.org/protscale/) 分析其亲/疏水性;ProtParam工具 (http://web.ex-pasy.org/protparam/) 分析蛋白的氨基酸组成及理化性质;SOPMA工具 (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl? page=npsa_sopma.html) 预测分析其α-螺旋、β-折叠和无卷曲情况。蛋白结构域利用在线网站(http://www. ebi.ac.uk/ interpro/search/sequence-search)进行分析;亚细胞定位由WoLF PSORT Prediction程序 (http://wolfpsort.org.html) 进行预测。
1.2.5番茄SlPAOs组织表达分析取干燥的种子及新鲜的真叶、子叶、茎、根提取组织总RNA,按照TaKaRa PrimeScriptTMRT reagent试剂盒说明书合成cDNA,4 ℃保存,作为后续PCR扩增的模板。SlPAOs引物利用在线网站http://sg.idtdna.com/primerquest/Home/Index设计,引物序列信息见表1。根据荧光定量PCR分析试剂盒 (SYBR Premix Ex Taq) 说明,采用Applied Biosystems 7500 Fast Real-Time PCR System对目的基因相对表达量进行qPCR分析。以看家基因Actin作为内标对表达结果进行校准。
1.2.6统计分析采用SPSS2.0对实验数据进行单因素方差分析 (ANOVA),然后采用最小显著极差法 (LSD) 进行多重比较,分析任意两个处理之间在0.05水平的差异显著性。
2 结果与分析
2.1 番茄SlPAOs序列的筛选与鉴定
以拟南芥AtPAOs基因家族为检索靶标,从番茄基因组中检索到了11个PAO基因,分别命名为SlPAO1-11 (表2)。这11个SlPAOs基因分布在7条不同的染色体上。SlPAO1位于1号染色体;SlPAO2、SlPAO7、SlPAO9、SlPAO10位于7号染色体;SlPAO3和SlPAO8位于12号染色体;SlPAO4、SlPAO5、SlPAO6、SlPAO11分别位于2、3、5、4号染色体。番茄11个SlPAOs可以编码含478 (SlPAO5) 至2078 (SlPAO11) 个氨基酸的蛋白。SlPAOs蛋白的分子量范围是52.86~227.87 kD,SlPAO9~11的分子量较之SlPAO1~8偏大。理论等电点分析结果显示SlPAO9编码碱性蛋白,其余呈酸性。
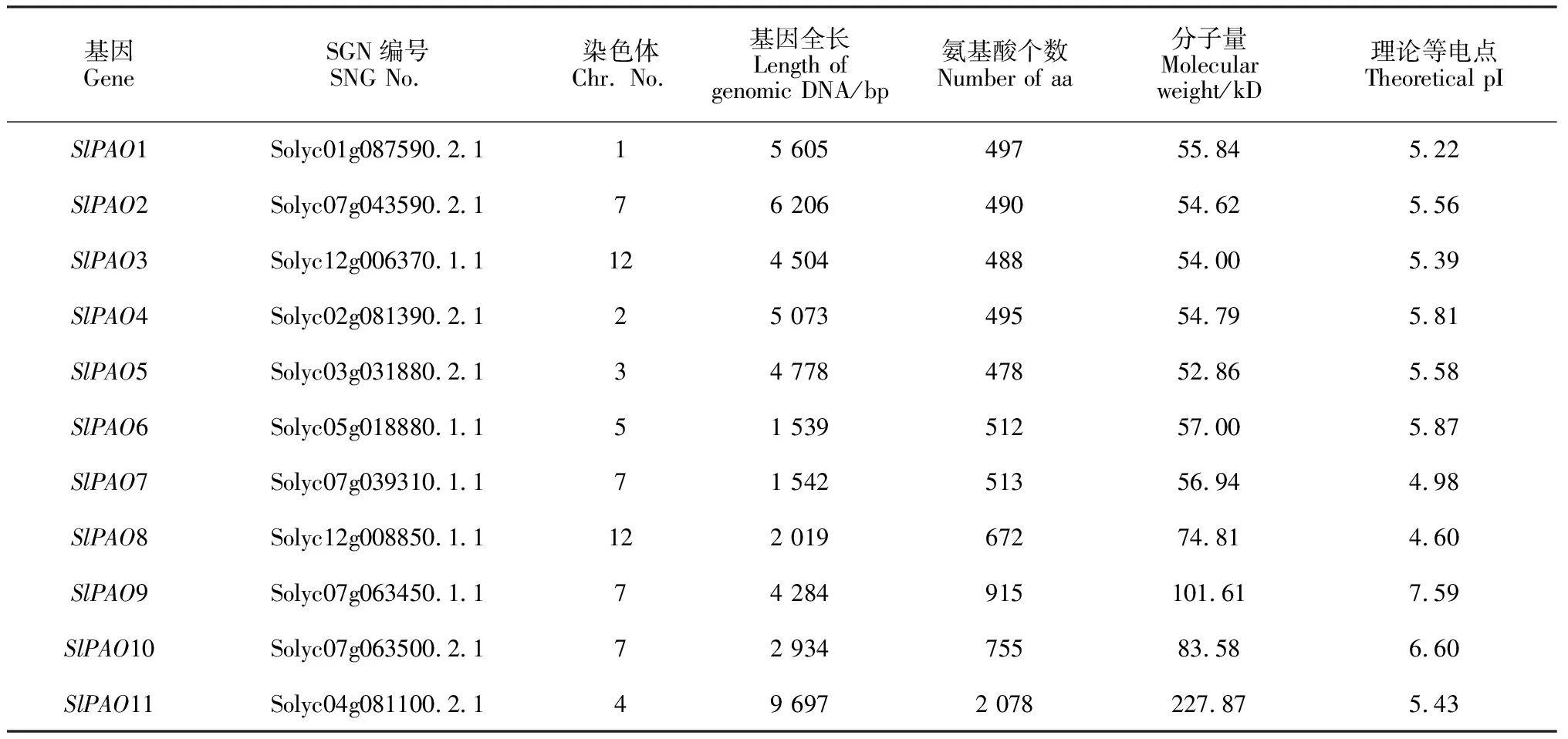
表2 番茄SlPAOs基因家族基本信息
2.2 番茄SlPAOs基因结构分析
基因结构 (内含子、外显子、5′/3′非翻译区) 分析结果表明:番茄SlPAOs基因结构特征亦呈现明显的亚家族分布规律 (图1)。亚家族Ⅰ和Ⅱb中的SlPAO1-5具备完全一致的基因结构特征,即:10个外显子,9个内含子,且都具有5′/3′非翻译区;亚家族Ⅱa中的SlPAO6~8只有1个外显子,无内含子和5′/3′非翻译区;亚家族Ⅲ中的SlPAOs基因结构无明显的规律特征,SlPAO9含有7个外显子,无非翻译区存在;SlPAO10和SlPAO11具有3′非翻译区,外显子分别为2个和8个。
2.3 番茄SlPAOs的进化分类及蛋白结构域分析
为了分析SlPAOs的进化关系,利用11个SlPAOs、9个拟南芥AtPAOs[22]、11个水稻OsPAOs[26]的氨基酸序列构建了系统发育树 (图2,A)。番茄SlPAO家族分为Ⅰ、Ⅱ、Ⅲ 三大亚家族。其中SlPAO1属于亚家族Ⅰ;SlPAO2~8归于亚家族Ⅱ;SlPAO9~11归于亚家族Ⅲ。根据Chen等[20]对水稻OsPAOs家族的功能分类,Ⅰ中的SlPAOs作用于PAs的最终分解代谢,Ⅱ中的SlPAOs作用于催化Spm向Spd的转化。Ⅲ 中的SlPAOs虽然也具备典型的氨基氧化酶保守结构域 (Amino_oxidase),但其上游还具备独特的SWIRM结构域 (图2,B),其主要功能是催化H3K4组蛋白赖氨酸的脱甲基化,属于组蛋白赖氨酸特异性脱甲基化酶类,而非真正的PAO。因此,Ⅰ和Ⅱ中的SlPAO1~8是典型的PAO,而Ⅲ 中SlPAO9~11是非典型PAO。对于亚家族Ⅱ中的SlPAOs,又可进一步分为2种类型 (Ⅱa、Ⅱb),Ⅱa包括SlPAO6~8,Ⅱb包括SlPAO2~5,这是基于其亚细胞定位有所不同。
2.4 番茄SlPAOs的亚细胞预测分析
利用WoLF PSORT Prediction预测结果显示SlPAOs的亚细胞定位呈现明显的亚家族特征(表3)。亚家族Ⅰ中的SlPAO1位于液泡;亚家族Ⅱb中的SlPAO2-5位于过氧化物酶体;亚家族Ⅱa中的SlPAO6和SlPAO8位于细胞质内,SlPAO7位于细胞核;亚家族Ⅲ分布较为广泛,SlPAO9位于细胞质,SlPAO10位于细胞核内,SlPAO11位于质膜。
2.5 番茄SlPAOs蛋白理化性质与二级结构预测分析
SlPAOs亲水性预测结果显示 (表4),SlPAO1-SlPAO11预测值均小于0,结合脂肪系数来看,表明SlPAOs蛋白均为亲水性蛋白。不稳定指数的结果显示:SlPAO1~SlPAO6以及SlPAO8 (不稳定指数<40) 属于较稳定蛋白,而SlPAO7及SlPAO9~SlPAO11 (不稳定指数>40) 稳定性相对较差。总体来看,亚家族Ⅰ和Ⅱ中的SlPAOs相对稳定。蛋白质二级结构预测结果显示 (表4):11个SlPAOs二级结构主要组成部分是α-螺旋和无规则卷曲,延伸片段次之,β-折叠占很小的一部分(10%左右),均无β-转角结构存在。
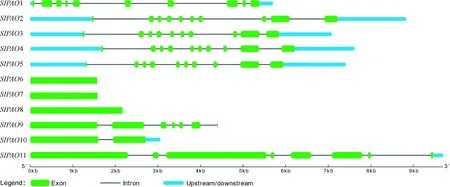
图1 番茄SlPAOs的基因结构Fig.1 Gene structure of tomato SlPAOs
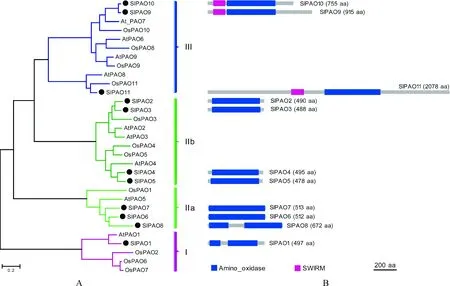
A. 番茄(SlPAOs)、水稻(OsPAOs)和拟南芥(AtPAOs)基因家族系统进化树;B. SlPAOs蛋白结构域分布图2 PAO基因家族系统进化树及番茄SlPAOs蛋白结构域分布A. Phylogenetic tree of PAO protein family in tomato (SlPAOs), rice (OsPAOs) and Arabidopsis (AtPAOs);B. Protein domain structure of tomato SlPAOsFig.2 Phylogenetic tree of PAO gene family and protein domain structure of tomato SlPAOs
2.6 番茄SlPAOs的组织表达分析
由于进化和结构分析中发现番茄中的SlPAO1~8编码典型的PAO,我们进一步分析了这8个基因在番茄植株不同组织 (种子、真叶、子叶、茎、根) 的相对表达量。结果如图3所示,这些基因在不同组织中均有表达,但是相对表达量却有很大差异。SlPAO1在茎中显著高表达,而SlPAO2在干燥种子中显著高表达;SlPAO3相对在叶片和茎中表达较高;SlPAO4的表达多集中于种子、茎、根;SlPAO5显著富集于子叶和茎中,且极大高于其他组织;SlPAO6主要在真叶和根中表达;SlPAO7在真叶中表达显著;SlPAO8主要表达于种子中。对于不同组织而言,干燥种子中相对表达量较高的有SlPAO2、SlPAO4、SlPAO8;真叶中相对表达量较高的有SlPAO3、SlPAO6、SlPAO7;子叶中相对表达量较高的有SlPAO3和SlPAO5;茎中相对表达量较高的有SlPAO1、SlPAO3、SlPAO5;根中相对表达量较高的有SlPAO4、SlPAO5、SlPAO6、SlPAO7。
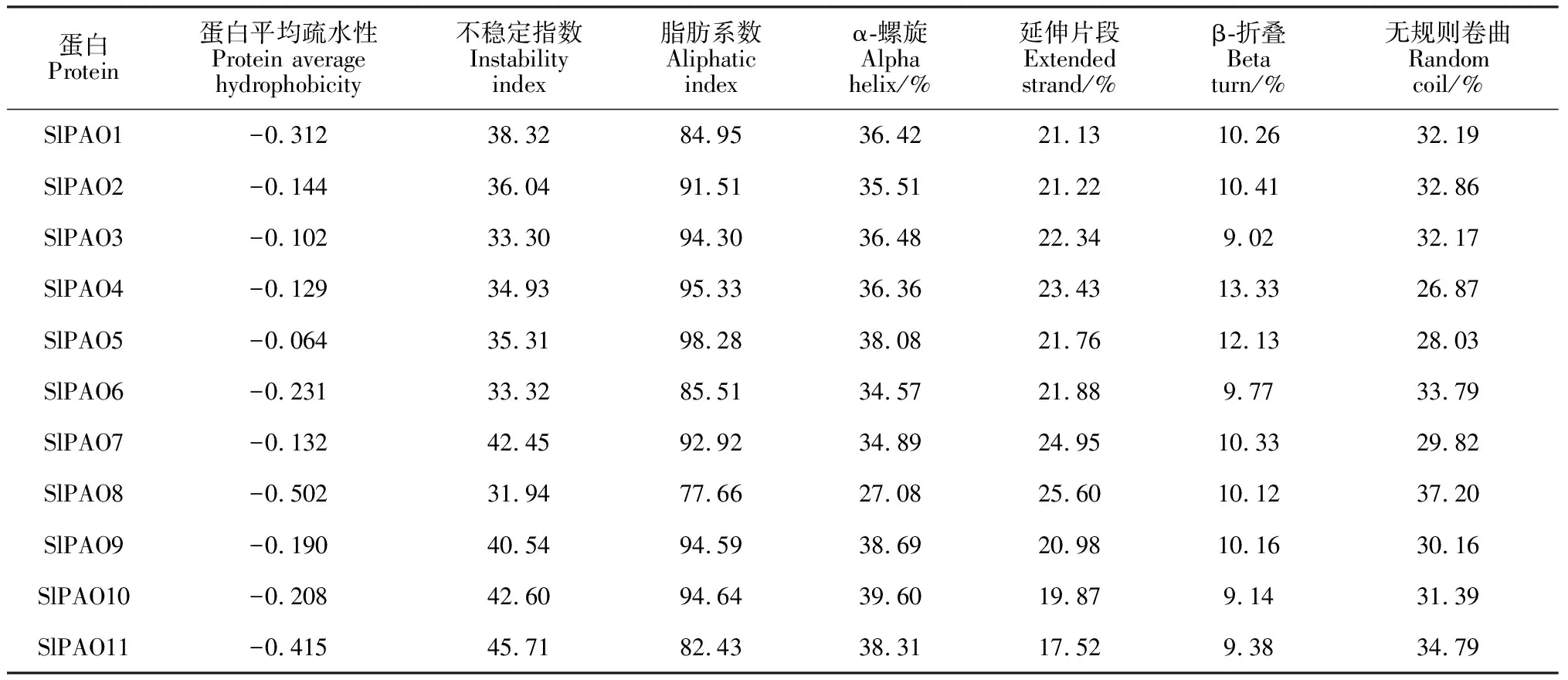
表4 番茄SlPAOs蛋白理化性质及二级结构预测
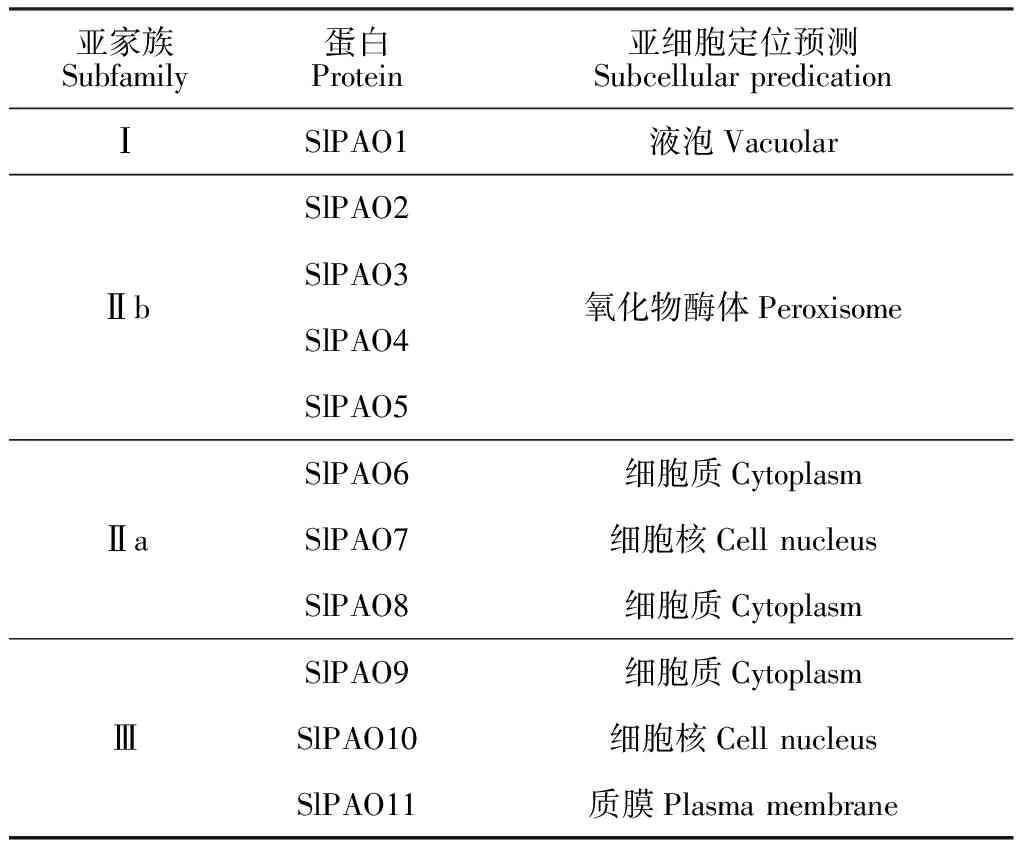
表3 番茄SlPAOs亚细胞定位预测分析
3 讨 论
本研究从番茄基因组中鉴定出11个PAO基因 (SlPAO1~11),进化树和结构与分析显示亚家族Ⅰ和Ⅱ (包括Ⅱa和Ⅱb) 的SlPAO1~8编码真正的PAO,而亚家族Ⅲ 的SlPAO9~11编码蛋白为脱甲基化酶类 (属于非典型性PAO)。基因结构分析、蛋白质二级结构和亚细胞定位预测分析亦显示出:亚家族Ⅰ和Ⅱ具有明显的规律特征。Angelini等[27]认为植物中的PAO根据生化功能分为两大类:一类负责PA的终极氧化反应,一类负责PA的逆向转化反应。本研究分析所得番茄SlPAOs家族分类同样符合这一特征。另外,进一步发现的多重亚家族分类表明了番茄SlPAOs可能的功能多样性。AtPAO1和OsPAO7已被证明催化PA的最终代谢[16, 19],由此可推测与它们同属亚家族Ⅰ的番茄SlPAO1亦可能具备此生化功能。生化和遗传学证据表明:OsPAO1和AtPAO5都是定位于细胞质的直系同源功能蛋白,催化PA的逆向转化[28]。进化分析亦证实OsPAO1和AtPAO5属于同一个亚家族 (Ⅱa),其中包括番茄SlPAO6~8。基因结构分析显示SlPAO6~8具备区别于其他SlPAOs的独特特征,即:只有1个外显子 (无内含子)。通过对OsPAO1和AtPAO5的基因组序列分析发现两者也是只具有1个外显子,这表明亚家族Ⅱa中不同物种的PAOs基因在进化上相对保守一致,而基因功能的重要性可能是导致这种自然保守进化的原因[29],无内含子意味着不需要转录过程的拼接修饰过程,从而可能快速完成转录翻译过程并发挥生理功能。有报道证明水稻中的OsPAO3/4/5定位于过氧化物酶体,催化PA的逆向转化[18],进化分析显示亚家族Ⅱb中包含了OsPAO3/4/5和番茄SlPAO2~5,从而可以推测出SlPAO2~5可能具备与OsPAO3/4/5相似的生化功能。另外,亚家族I和IIb中的所有的SlPAOs(SlPAO1~5) 基因结构特征非常一致,但较为复杂 (10外显子/9内含子)。番茄转座子对于基因结构的进化和形成发挥重要作用[30],进一步解析基因组中转座子分布规律对这些基因中内含子形成的影响,将有助于揭示SlPAO1~5进化规律与生理功能之间的相关性。
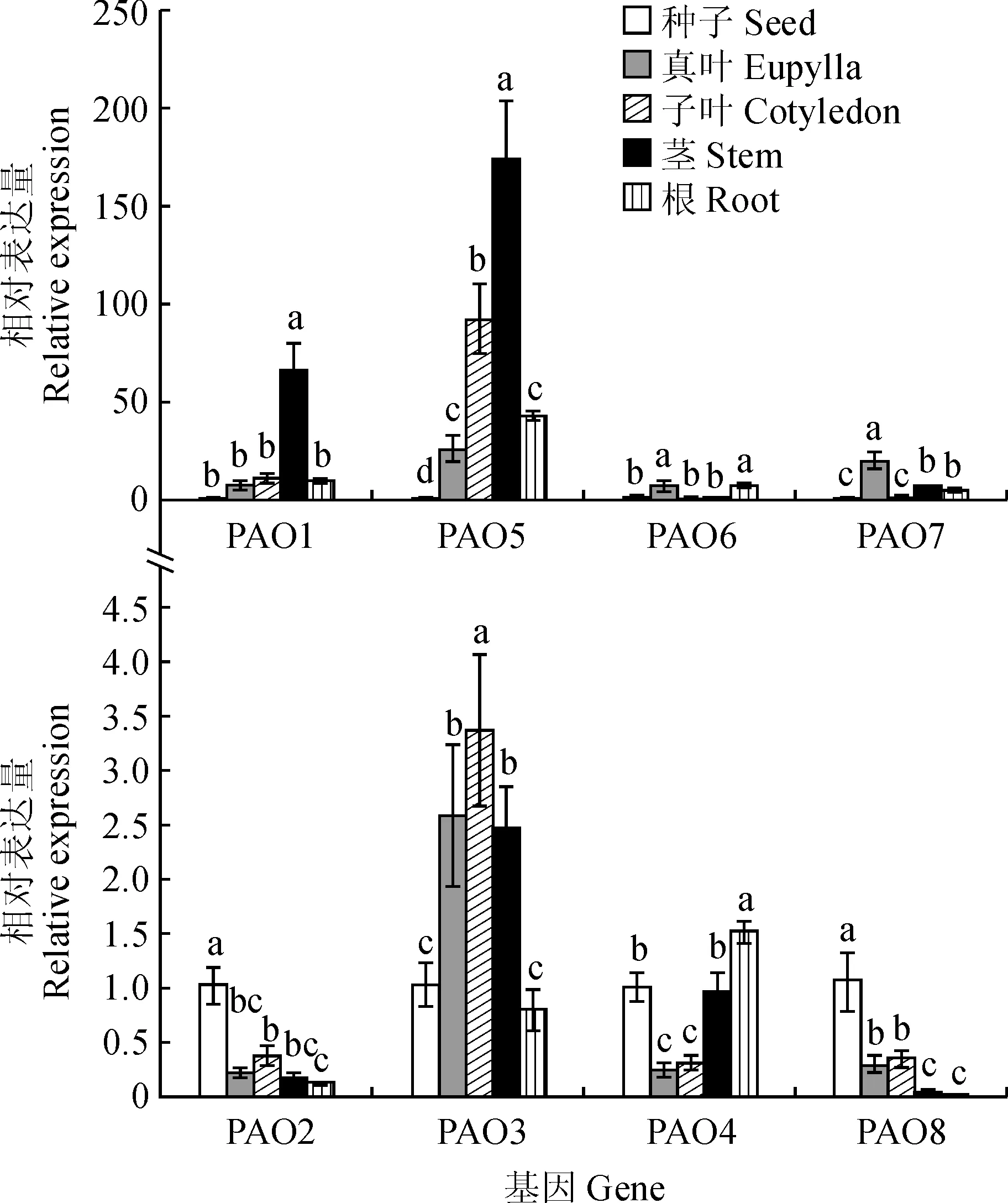
不同小写字母表示同一基因在不同组织中的表达差异显著(P <0.05)图3 番茄SlPAOs家族8个基因在不同组织(种子、真叶、子叶、茎、根)中的相对表达分析Different normal letters indicate significant differences among treatments at 0.05 levelFig.3 Analysis of relative expression of SlPAOs in different tissues(seed, eupylla, cotyledon, stem, root)
基因表达结果显示SlPAO1~8在不同组织中的表达呈现丰富的多样性。有报道证明水稻OsPAO5及其产生的H2O2参与调控水稻种子的萌发过程[20]。我们研究发现与OsPAO5亲缘关系较近的SlPAO4在种子和幼苗根中的表达量相对较高,说明SlPAO4和OsPAO5可能在分别调控双子叶和单子叶植物生理过程中发挥相似作用。本研究发现SlPAO7在叶片中具有相对较高的表达量,而最近在拟南芥中的研究可能解释此生理现象,SlPAO7与拟南芥AtPAO5亲缘关系较近,遗传学证据显示AtPAO5通过调控叶绿素和脱落酸信号应对盐胁迫[31],同时脱落酸是调控叶片气孔关闭的关键信号分子。另外,我们还发现SlPAO5在各组织中的表达量比种子高达27~174倍。进化分析显示番茄SlPAO5与AtPAO4亲缘关系较近,Sequeramutiozabal等[32]通过AtPAO4功能缺失突变体的代谢组学分析,发现AtPAO4能够通过影响植物整个代谢组循环来调控植物衰老过程,由此可推测高表达量的SlPAO5可能在调控植物生理过程方面发挥多重作用。
综上所述,本研究从番茄基因组中分析出了SlPAOs家族基因,分类分析和组织表达分析显示出其可能的生化和生理功能多样性,为后续这些基因的功能鉴定提供了重要参考。
参考文献:
[1] KAUR SAWHNEY R, TIBURCIO A F, ALTABELLA T,etal. Polyamines in plants: an overview[J].JournalofCell&MolecularBiology, 2003,2(1): 1-12.
[2] KUSANO T, BERBERICH T, TATEDA C,etal. Polyamines: essential factors for growth and survival[J].Planta, 2008,228(3): 367-381.
[3] ALCAZAR R, ALTABELLA T, MARCO F,etal. Polyamines: molecules with regulatory functions in plant abiotic stress tolerance[J].Planta, 2010,231(6): 1 237-1 249.
[4] SMITH T A, BARKER J H A. The di- and polyamine oxidases of plants[J].AdvancesinExperimentalMedical&Biology, 1985,13(2): 319-322.
[5] MOSCHOU P N, WU J, CONA A,etal. The polyamines and their catabolic products are significant players in the turnover of nitrogenous molecules in plants[J].JournalofExperimentalBotany, 2012,63(14): 5 003-5 015.
[6] ANGELINI R. Functions of amine oxidases in plant development and defence[J].TrendsinPlantScience, 2006,11(2): 80-88.
[7] AHOU A, MARTIGNAGO D, ALABDALLAH O,etal. A plant spermine oxidase/dehydrogenase regulated by the proteasome and polyamines[J].JournalofExperimentalBotany, 2014,65(6): 1 585-1 603.
[8] WU J Y, SHANG Z L, WU J,etal. Spermidine oxidase-derived H2O2regulates pollen plasma membrane hyperpolarization-activated Ca2+-permeable channels and pollen tube growth[J].PlantJournalforCell&MolecularBiology, 2010,63(6): 1 042-1 053.
[9] 樊怀福. 外源NO缓解黄瓜幼苗盐胁迫伤害的生理基础研究[D]. 南京:南京农业大学, 2007.
[10] MOSCHOU P N, PASCHALIDIS K A, DELIS I D,etal. Spermidine exodus and oxidation in the apoplast induced by abiotic stress is responsible for H2O2signatures that direct tolerance responses in tobacco[J].PlantCell, 2008,20(6): 1 708-1 724.
[11] FU X Z, CHEN C W, WANG Y,etal. Ectopic expression of MdSPDS1 in sweet orange (CitrussinensisOsbeck) reduces canker susceptibility: involvement of H2O2production and transcriptional alteration[J].BmcPlantBiology, 2011,11(1): 55-70.
[12] 王 强, 王 娟, 张慧君,等. 番茄幼苗盐胁迫响应蛋白质组分析[J]. 西北植物学报, 2016,36(11): 2 226-2 232.
WANG Q, WANG J, ZHANG H J,etal. Analysis of salt stress response proteome in tomato seedlings[J].ActaBotanicaBoreali-OccidentaliaSinica, 2016,36(11): 2 226-2 232.
[13] SLOCUM R D, III M J F. Electron-microscopic cytochemical localization of diamine and polyamine oxidases in pea and maize tissues[J].Planta, 1991,183(3): 443-450.
[14] 覃广泉, 何生根, 王明祖,等. 大豆初生幼苗多胺氧化酶活性的细胞化学定位[J]. 热带亚热带植物学报, 2006,14(4): 327-332.
TAN G Q, HE S G, WANG M Z,etal. Cytochemical localization of polyamine oxidase activity in soybean newborn seedlings[J].JournalofTropicalandSubtropicalBotany, 2006,14(4): 327-332.
[15] TAVLADORAKI P, SCHININA M E, CECCONI F,etal. Maize polyamine oxidase: primary structure from protein and cDNA sequencing[J].FebsLetters, 1998,426(1): 62-66.
[16] LIU T, DONG W K, NIITSU M,etal. Polyamine oxidase 7 is a terminal catabolism-type enzyme inOryzasativaand is specifically expressed in anthers[J].Plant&CellPhysiology, 2014,55(6): 1 110-1 122.
[17] FINCATO P, MOSCHOU P N, SPEDALETTI V,etal. Functional diversity inside theArabidopsispolyamine oxidase gene family[J].JournalofExperimentalBotany, 2011,62(3): 1 155-1 168.
[18] ONO Y, DONG W K, WATANABE K,etal. constitutively and highly expressedOryzasativapolyamine oxidases localize in peroxisomes and catalyze polyamine back conversion[J].AminoAcids, 2012,42(2): 867-876.
[19] TAKAHASHI Y, CONG R, SAGOR G H,etal. Characterization of five polyamine oxidase isoforms inArabidopsisthaliana[J].PlantCellReports, 2010,29(9): 955-965.
[20] CHEN B X, LI W Y, GAO Y T,etal. Involvement of polyamine oxidase-produced hydrogen peroxide during coleorhiza-limited germination of rice seeds[J].FrontiersinPlantScience, 2016,7(401): 1 219-1 232.
[21] CERVELLI M, TAVLADORAKI P, AGOSTINO S D,etal. Isolation and characterization of three polyamine oxidase genes fromZeamays[J].PlantPhysiology&Biochemistry, 2000,38(9): 667-677.
[22] CERVELLI M, CDNA A, ANGELINI R,etal. A barley polyamine oxidase isoform with distinct structural features and subcellular localization[J].FEBsJournal, 2001,268(13): 3 816-3 830.
[23] YODA H, HIROR Y, SANO H. Polyamine oxidase is one of the key elements for oxidative burst to induce programmed cell death in tobacco cultured cells[J].PlantPhysiology, 2006,142(1): 193-206.
[24] KITASHIBA H, HONDA C, MPRIGUCHI T. Identification of polyamine oxidase genes from apple and expression analysis during fruit development and cell growth[J].PlantBiotechnology, 2006,23(4): 425-429.
[25] TUSKAN G A,etal. The genome of black cottonwood,Populustrichocarpa(Torr. & Gray)[J].Science, 2006,313(5 793): 1 596-1 604.
[26] HU B, JIN J, GUO A Y,etal. GSDS 2.0: an upgraded gene feature visualization server[J].Bioinformatics, 2015,31(8): 1 296-1 297.
[27] ANGELINI R, CONA A, FEDERICO R,etal. Plant amine oxidases “on the move”: an update[J].PlantPhysiology&Biochemistry, 2010,48(7): 560-564.
[28] LIU T, DONG W K, NIITSU M,etal. POLYAMINE OXIDASE 1 from rice (Oryzasativa) is a functional ortholog ofArabidopsisPOLYAMINE OXIDASE 5[J].PlantSignaling&Behavior, 2014,9(9): e29773.
[29] YAN J, MA Z, XU X,etal. Evolution, functional divergence and conserved exon-intron structure of bHLH/PAS gene family[J].MolGenetGenomics, 2014,289: 25-36.
[30] XU Y, DU J. Young but not relatively old retrotransposons are preferentially located in gene-rich euchromatic regions in tomato plants[J].ThePlantJournal. 2014,80(4): 582-591.
[31] ZARZA X, ATANASOV K E, MARCO F,etal. Polyamine oxidase 5 loss-of-function mutations inArabidopsisthalianatrigger metabolic and transcriptional reprogramming and promote salt stress tolerance[J].PlantCell&Environment, 2017,40(4): 1 703-1 713.
[32] SEQUERAMUTIOZABAL M I, ERBAN A, KOPKA J,etal. Global metabolic profiling ofArabidopsispolyamine oxidase 4 (AtPAO4) loss-of-function mutants exhibiting delayed dark-induced senescence[J].FrontiersinPlantScience, 2016,7(173): 173-186.

