全基因组预测里氏木霉QM6a的碳水化合物活性酶类蛋白
韩长志,祝友朋,许 僖
(西南林业大学生物多样性保护与利用学院/云南省森林灾害预警与控制重点实验室,云南 昆明 650224)
【研究意义】为了更好地明确里氏木霉QM6a中碳水化合物活性酶功能和作用,本研究对其进行预测,并对其数量和特征进行分析。【前人研究进展】CAZymes是碳水化合物活性酶类(Carbohydrate-Active Enzymes)的简称,作为植物丝状真菌中重要的一类蛋白,在其生长发育过程中发挥着重要的作用。碳水化合物结构(功能)活性酶类数据库(Carbohydrate-Active enZYmes Database),作为一个专业描述结构相关的酶催化或者是碳水化合物绑定或者是功能域模块的数据库,上述基因功能包括修饰、转移以及水解、断裂糖苷键等[1],从而有助于进一步解析植物病原菌胞外酶细胞壁降解酶活性基因的注释与聚类分析[2]。近些年关于CAZymes的研究呈现较快的增长[3]。根据最新研究成果,以涵盖木质纤维素降解所需要的相关酶类进行分类,主要涉及以下6大类,糖苷水解酶(Glycoside Hydrolases, GHs)[4]、糖基转移酶(Glycosyl Transferases, GTs)[5]、多糖裂解酶(Polysaccharide Lyases, PLs)[6]、碳水化合物酯酶(Carbohydrate Esterases, CEs)[6]、辅助酶类家族(Auxiliary Activities, AAs)[7]以及碳水化合物绑定结构(Carbohydrate-Binding Modules, CBMs)[8]。截止2016年5月18日,CAZy数据库中涉及上述类别酶的家族分别有135、98、24、16、13以及74个。【本研究切入点】里氏木霉(TrichodermareeseiQM6a)作为重要的产纤维素酶工业用菌种之一[9],与其他木霉属真菌所具有的抑菌功能不同,为了更好地解析该工业菌种与其他木霉内在的差异,前期基于真菌分泌蛋白所具有的主要特征,利用SignalP、ProtComp、TMHMM、big-PI Fungal predictor、TargetP等在线分析程序对T.reeseiQM6a中分泌蛋白进行找寻,明确该菌中包括356个分泌蛋白。【拟解决的关键问题】本研究基于上述356个分泌蛋白序列,利用CAT在线CAZymes预测工具,从而明确T.reeseiQM6a中CAZymes相关酶的不同亚家族的分布情况,以期为进一步开展该菌CAZymes功能研究打下坚实的理论基础。
1 材料与方法
1.1 里氏木霉QM6a中分泌蛋白序列来源
前期对T.reeseiQM6a中9143条蛋白序列(http://genome.jgi.doe.gov/TriviGv29_8_2/TriviGv29_8_2.home.html)(BioProject ID为PRJNA183001)进行SignalP、ProtComp、TMHMM、big-PI Fungal predictor、TargetP等在线分析,明确其含有356个分泌蛋白[10]。
1.2 CAZymes预测
利用在线工具CAZymes Analysis Toolkit[2]来开展预测工作,并对预测的蛋白进行整理、汇总。
2 结果与分析
2.1 里氏木霉QM6a中含有201个CAZymes
通过CAT分析,明确里氏木霉QM6a中有201个CAZymes,其中,以GH的数量最多,有93个,占46.27 %,其次是CBM,有52个,占25.87 %,再次为AA、CE、PL、GT(图1)。同时,除上述含有单种酶特性外,还有部分蛋白具有两种酶类型,即复合型,有GH/CBM、CE/CBM、AA/CBM等。由于不同的CAZymes具有不同的功能,因此,具有2种酶类型的蛋白在行使CAZymes功能方面具有多样性特征。
2.2 里氏木霉QM6a中所含93个GH的特征
93个GH蛋白分别属于41个大类,以GH18数量最多,为8个,其次为GH55、GH27、GH92,数量分别为7、6、5个(表1)。有22个GH类别仅含有1个蛋白,如GH6、GH7、GH10、GH12、GH13、GH20等,上述类别所占比例为53.66 %,其所含蛋白数量仅有22个,所占比例仅为23.66 %。上述结果表明,里氏木霉QM6a中所含糖苷水解酶的类型较多,在行使糖苷水解方面发挥着重要的作用。
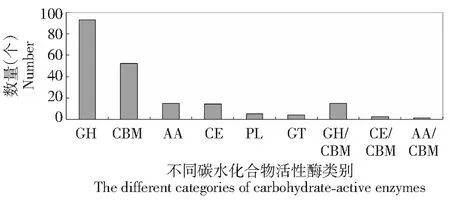
图1 里氏木霉QM6a中CAZymes的分布Fig.1 Distribution of CAZymes from T. reesei QM6a
续表1 Continued table 1

不同类别TypeID数量NumberGH18jgi|Trire2|119859、jgi|Trire2|2735、jgi|Trire2|43873、jgi|Trire2|59791、jgi|Trire2|80833、jgi|Trire2|110317、jgi|Trire2|124043、jgi|Trire2|683478GH20jgi|Trire2|233461GH24jgi|Trire2|108585、jgi|Trire2|1092782GH25jgi|Trire2|1034581GH27jgi|Trire2|55999、jgi|Trire2|65986、jgi|Trire2|72632、jgi|Trire2|59391、jgi|Trire2|72704、jgi|Trire2|750156GH28jgi|Trire2|103049、jgi|Trire2|112140、jgi|Trire2|122780、jgi|Trire2|701864GH30jgi|Trire2|110894、jgi|Trire2|3094、jgi|Trire2|69276、jgi|Trire2|697364GH31jgi|Trire2|822351GH35jgi|Trire2|802401GH39jgi|Trire2|731021GH43jgi|Trire2|120784、jgi|Trire2|121136、jgi|Trire2|68064、3GH45jgi|Trire2|499761GH47jgi|Trire2|45717、jgi|Trire2|51365、jgi|Trire2|810703GH54jgi|Trire2|553191GH55jgi|Trire2|108776、jgi|Trire2|121746、jgi|Trire2|44366、jgi|Trire2|54242、jgi|Trire2|56418、jgi|Trire2|70845、jgi|Trire2|732487GH62jgi|Trire2|762101GH64jgi|Trire2|1236391GH67jgi|Trire2|725261GH74jgi|Trire2|490811GH75jgi|Trire2|42152、jgi|Trire2|66789、jgi|Trire2|703413GH79jgi|Trire2|106575、jgi|Trire2|730052GH81jgi|Trire2|732561GH89jgi|Trire2|697001GH92jgi|Trire2|111733、jgi|Trire2|55733、jgi|Trire2|60635、jgi|Trire2|69493、jgi|Trire2|741985GH95jgi|Trire2|111138、jgi|Trire2|588022GH103jgi|Trire2|1207841GH117jgi|Trire2|1093921GH127jgi|Trire2|1214751GH128jgi|Trire2|596281合计Total—93
2.3 里氏木霉QM6a中所含52个CBM的特征
52个CBM蛋白分属于13个大类,以CBM1的数量最多,为12个,其次是CBM50、CBM32、CBM13,数量分别为10、7、6个(表2)。除此之外,里氏木霉QM6a还具有CBM32/CBM13类型,其ID为jgi|Trire2|58880,该蛋白不仅涉及CBM32类别,也涉及CBM13类别,上述研究结果表明CBM中具体类别不同,其具有的功能也不尽相同。
2.4 里氏木霉QM6a中所含15个AA的特征
通过对里氏木霉QM6a中15个辅助酶家族的分析,发现以AA2的数量最多,为4个,其次为AA1、AA7、AA9,均为3个,上述蛋白所占比例高达86.67 %(表3)。同时,还涉及AA3、AA5等类型的蛋白。
2.5 里氏木霉QM6a中所含14个CE的特征
通过对里氏木霉QM6a中14个CE蛋白的分析,发现以CE10数量最多(8个),其次是CE16(2个),上述蛋白所占比例高达71.43 %(表4)。同时,还涉及CE1、CE3、CE5、CE15等蛋白,其数量均为1个。
2.6 里氏木霉QM6a中所含5个PL的特征
通过对里氏木霉QM6a中5个多糖裂解酶蛋白的分析,其类别分别为PL1、PL7、PL20,数量分别为1、2、2个(表5)。

表2 里氏木霉QM6a碳水化合物绑定结构分布情况

表3 里氏木霉QM6a中AAs的分布情况
2.7 里氏木霉QM6a中所含4个GT的特征
通过对里氏木霉QM6a中4个GT蛋白的分析,其具体类别包括GT2、GT4、GT8以及GT20。在上述每个类别中,均仅含有1个蛋白,其ID分别为jgi|Trire2|69061、jgi|Trire2|104461、jgi|Trire2|108655、jgi|Trire2|72379。

表4 里氏木霉QM6a中CE蛋白分布情况

表5 里氏木霉QM6a中PL的分布情况

表6 里氏木霉QM6a中复合类型蛋白的分布情况
2.8 里氏木霉QM6a中含有17个复合类型的蛋白
里氏木霉QM6a中除含有典型的GH、CBM、AA、CE、GT以外,还含有一些复合类别的蛋白,其包括以下几种类型:AA/CBM、GH/CBM以及CE/CBM等,其中以GH/CBM居多,为14个,所占比例为82.35 %(表6)。
3 讨 论
本研究所获得里氏木霉QM6a中CAZmes,是基于前期通过全基因组序列预测所获分泌蛋白,同时,依据现有网站所获的CAZymes,上述蛋白是否囊括了里氏木霉QM6a中全部的CAZymes,有待于今后进一步通过生物学试验进行验证。同时,前人对于该菌中CAZymes所报道的数量均高于本研究所得到的结果[9],就每一类中的数量报道也不尽相同,究其原因,前人所开展CAZymes的具体类别有5个(GH、PL、CE、CBM、GT),而根据最新的分类类别有6个(GH、PL、CE、CBM、GT、AA),造成具体不同类别分项数量方面的差异。前人对于GH、GT数量的报道分别为200、103个[9],远高于本研究所获得的93、4个,原因如何,有待进一步明确。前期对禾谷炭疽菌、希金斯炭疽菌中CAZymes开展的蛋白预测研究,也存在着数量方面的差异[11-12]。
随着大量植物病原菌基因组测序的完成[13-14],通过比较基因组学分析病原菌的寄生性与基因组变异的关系已经成为研究热点[15]。一般而言,植物病原丝状真菌中的CAZymes蛋白可以作为其侵染过程中突破寄主细胞壁的非常关键的因素之一[16]。前人选择一些营养类型不同的植物病原真菌全基因组数据,通过生物信息学分析[17],明确非活体营养型病原菌基因组较活体营养型病原菌具有更多的CAZymes[16]。前人对于木霉的研究涉及在土壤的定殖规律、拮抗能力等内容[18],然而,里氏木霉QM6a显然不是植物病原菌,所含有的CAZymes所发挥的功能与植物病原菌不同,即同一类别蛋白在不同菌中所具有的功能不尽相同。
4 结 论
本研究明确里氏木霉QM6a中含有201个CAZymes蛋白,可以分为主要类别、复合类别,其中,主要类别涉及93个GHs、52个CBMs以及15个AAs、14个CEs、5个PLs、4个GTs;复合类别则涉及15个GH/CBMs、1个AA/CBMs以及2个CE/CBMs。
参考文献:
[1]Cantarel B L, Coutinho P M, Rancurel C, et al. The Carbohydrate-Active EnZymes database (CAZy): an expert resource for Glycogenomics[J]. Nucleic Acids Res, 2009, 37(Database issue): D233-238.
[2]Park B H, Karpinets T V, Syed M H, et al. CAZymes Analysis Toolkit (CAT): web service for searching and analyzing carbohydrate-active enzymes in a newly sequenced organism using CAZy database[J]. Glycobiology, 2010, 20(12): 1574-1584.
[3]Lombard V, Golaconda Ramulu H, Drula E, et al. The carbohydrate-active enzymes database (CAZy) in 2013[J]. Nucleic Acids Res, 2014, 42(Database issue): D490-495.
[4]Bourne Y, Henrissat B. Glycoside hydrolases and glycosyltransferases: families and functional modules[J]. Current Opinion in Structural Biology, 2001, 11(5): 593-600.
[5]Campbell J A, Davies G J, Bulone V, et al. A classification of nucleotide-diphospho-sugar glycosyltransferases based on amino acid sequence similarities[J]. Biochem J, 1997, 326 ( Pt 3):929-939.
[6]Lombard V, Bernard T, Rancurel C, et al. A hierarchical classification of polysaccharide lyases for glycogenomics[J]. Biochem J, 2010, 432(3): 437-444.
[7]Levasseur A, Drula E, Lombard V, et al. Expansion of the enzymatic repertoire of the CAZy database to integrate auxiliary redox enzymes[J]. Biotechnol Biofuels, 2013, 6(1): 41.
[8]Boraston A B, Bolam D N, Gilbert H J, et al. Carbohydrate-binding modules: fine-tuning polysaccharide recognition[J]. Biochem J, 2004, 382(Pt 3): 769-781.
[9]Martinez D, Berka RM, Henrissat B, et al. Genome sequencing and analysis of the biomass-degrading fungusTrichodermareesei(syn.Hypocreajecorina)[J]. Nat Biotechnol, 2008, 26(5): 553-560.
[10]韩长志,王 娟.基于全基因组序列预测里氏木霉QM6a的分泌蛋白[J].华中农业大学学报, 2017, 36(2): 28-32.
[11]韩长志.基于全基因组测序的禾谷炭疽菌中碳水化合物酶类蛋白预测[J].河南师范大学学报(自然科学版), 2016, 44(4): 118-124.
[12]韩长志.全基因组预测希金斯炭疽菌中碳水化合物酶类蛋白[J].江苏农业科学, 2017, 45(2): 24-28.
[13]聂燕芳,周 淦,黄嘉瑶,等.尖孢镰刀菌甜瓜专化型基因组规模分泌蛋白的预测与分析[J].华中农业大学学报, 2016, 35(3): 24-29.
[14]李云锋,聂燕芳,王振中.植物病原真菌分泌蛋白质组学研究进展[J].微生物学通报, 2015, 42(6): 1101-1107.
[15]Schmidt S M, Panstruga R. Pathogenomics of fungal plant parasites: what have we learnt about pathogenesis?[J]. Curr Opin Plant Biol, 2011, 14(4): 392-399.
[16]韩长志,许 僖.植物病原丝状真菌分泌蛋白及CAZymes的研究进展[J].南京林业大学学报(自然科学版), 2017, 41(5): 152-160.
[17]陈相永,陈捷胤,肖红利,等.植物病原真菌寄生性与分泌蛋白组CAZymes的比较分析[J].植物病理学报, 2014, 44(2): 163-172.
[18]杜婵娟,付 岗,潘连富,等.哈茨木霉gz-2菌株在土壤中的定殖规律研究[J].西南农业学报, 2016, 29(1): 138-142.

