SCNT牛胚胎中PWS/AS基因簇母源印记控制区甲基化模式研究
王 巍,方东辉,付茂忠,甘 佳,唐 慧,石 溢,王 淮,易 军
(四川省畜牧科学研究院,动物遗传育种四川省重点实验室,成都 610066)
基因组印记(Genomic imprinting),又称为遗传印记(Genetic imprinting),是指在配子期或合子期,来自亲本(父源或母源)的等位基因或染色体发生特异性的修饰,导致不同亲本来源等位基因具有不同表达活性的一种不遵从孟德尔定律的单亲性特殊遗传现象[1-2]。绝大多数的印记基因都是成簇存在的;在印记簇中的印记基因的表达或沉默是由印记控制区(Imprinting control regions,ICRs)通过顺式作用来调控的[3]。ICRs实质为一段差异性甲基化区域(Differentially methylated regions,DMRs),这种差异性甲基化一般是在生殖细胞中建立的,以调控整个印记基因簇的基因沉默情况[4]。因此,印记基因簇可以分为两种,即在卵母细胞发生过程中进行修饰而产生的母源印记以及在精子发生过程中进行修饰而产生的父源印记[5-6]。
普-威综合征(Prader-Willi syndrome,PWS)和安吉尔曼综合征(Angelman syndrome,AS)是两种先天性的神经行为发育异常综合症。这两种疾病的临床症状和遗传形式虽不相同,但病因都与人类染色体15q11~13区域的母源性印记基因簇有关。PWS/AS基因簇包含
PWS和AS两个亚区,其中PWS亚区又包含有5个父源表达的蛋白编码基因,即MKRN3、MAGEL2、NDN、SNURF 和 SNRPN[7]。PWS/AS 基因簇的 ICRs区由 PWSIC和AC-IC两部分组成;PWS-IC被定位于包括SNRPN的启动子和外显子1的4.3 kb区域内[8],而AC-IC区则被定位于SNRPN基因转录起始点上游35 kb前的880 bp区域内[9]。敲除父源性的PWS-IC区可致PWS亚区所有父源基因表达的沉默[10]。PWS/AS基因簇等母源印记区是良好的分子标签。应用甲基化测序方法检测克隆动物早期胚胎中这些印记区的甲基化水平,并与体外受精胚胎进行比较,可直观地了解克隆胚中母源染色体的甲基化模式,也可为提高克隆胚胎的成功率提供理论指导。
1 材料与方法
1.1 实验样本
处于配子期(受精后8 h或激活后5 h)、S期、2-cell期、8-cell期和桑葚期的西门塔尔牛体外受精(IVF)胚胎和体细胞核移植(SCNT)胚胎每个时期各40枚,由四川农业大学动物遗传育种与繁殖实验室提供。
1.2 总DNA的亚硫酸盐变性及DNA提取
按照全细胞亚硫酸盐DNA甲基化修饰试剂盒(Epigentek)的操作说明,取20 μL的样品进行DNA的亚硫酸盐变性及提取,-20℃冻存备用。
1.3 引物设计
以PWS/AS基因簇的ICR区为主要的分析对象,根据NCBI上公布的牛SNRPN基因序列(GenBank ID:NM_001079797)查找其启动子序列,使用methyl primer express software v1.0引物设计软件分别在两个基因的启动子内选取一个CPG岛设计引物,用于BSP测序。引物设计情况见图1。

图1 PCR扩增片段在SNRPN基因启动子区的相对位置示意图及引物设计
1.4 目标片段的扩增
参照天根生化科技(北京)有限公司MasterMix PCR试剂盒的使用说明,在2×25 μL及应体系中进行PCR反应,条件如下:94℃预变性5 min;94℃变性45 s,53.6℃退火 45 s,72℃延伸 45 s,38 个循环;72℃末端扩增10 min。
1.5 目标片段的克隆
用干净的手术刀割下含有目的DNA片段的琼脂糖凝胶,放入1.5 mL离心管中。采用UNIQ-10柱式DNA胶回收试剂盒(上海生物工程技术服务有限公司),按照说明书操作进行胶回收。回收的DNA通过pMD19-T载体(大连宝生物公司)进行连接,连接产物均匀加入到30 μL的大肠杆菌DH5α感受态细胞中,冰浴30 min;42℃水浴热激45 s,然后立即冰上放置冰浴1 min;每管中加入900 μL的LB肉汤培养基,37℃震荡培养2 h,使细菌复苏,并表达对抗生素的抗性;然后涂布于含Amp的LB琼脂平板中,37℃倒置培养过夜。
1.6 菌落PCR鉴定
挑选平板上的单个菌落,接种于含Amp的LB肉汤中培养过夜,用菌液作PCR模板进行菌落PCR鉴定。从SCNT和IVF胚胎5个发育阶段菌落样本中各挑选12~15个阳性克隆,送上海英俊公司进行序列测定。
1.7 数据分析
使用BIQ甲基化分析软件对重亚硫酸盐测序结果分析,并对CpG位点的甲基化状态进行统计。实验数据以x±S表示,采用SPSS 17.0统计软件处理,相同阶段不同类型胚胎、相同胚胎不同阶段的甲基化水平和甲基化位点比较采用两因素方差分析(One-way ANOVA)。
2 结果与分析
2.1 目标基因扩增
PCR产物经1%琼脂糖凝胶电泳检测后,在250 bp至500 bp之间接近250 bp处出现目的条带,与目的片段的长度相符,见图2。

图2 PCR产物凝胶电泳检测结果
2.2 SNRPN基因启动子总体甲基化值测定
各个发育阶段的IVF胚胎和SCNT胚胎中,SNRPN基因启动子的甲基化测序结果见表1。
由表1可知,SCNT胚胎发育时期PWS/AS亚区ICR甲基化水平的变化模式为:S期甲基化水平显著低于其他各时期(P<0.05),且其他4个时期的甲基化水平差异均不显著(P>0.05);IVF胚胎发育时期PWS/AS亚区ICR甲基化水平的变化模式为:2-cell期甲基化水平显著低于其他各时期(P<0.05),且其他4个时期的甲基化水平差异也均不显著(P>0.05)。SCNT胚胎发育至2-cell期时,PWS/AS亚区ICR甲基化水平显著高于IVF胚胎(P<0.05)。
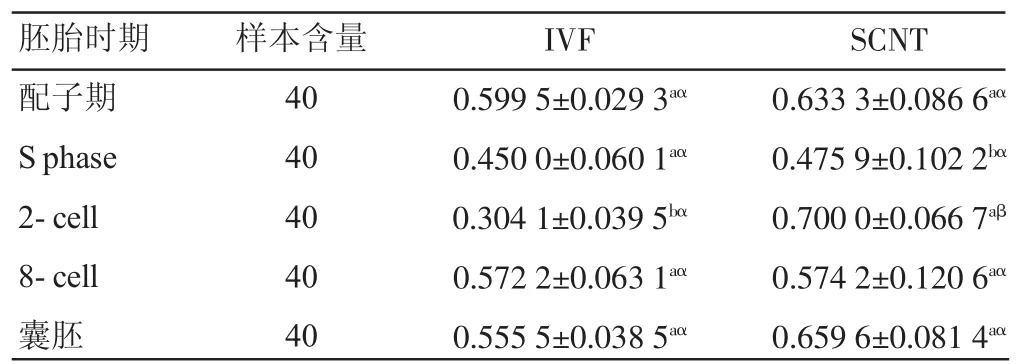
表1 PWS-IC亚区ICR区在早期胚胎发育期中的甲基化值
2.3 SNRPN启动子区5个位点甲基化情况分析
如图3所示,SNRPN转录起始点1 693 bp开始的258 bp序列中共检测到5个甲基化位点。

图3 SNRPN基因启动子的5个甲基化位点分布

图4 SCNT和IVF胚胎各发育时期5个位点甲基化水平
由图4-1可见,配子期SCNT胚胎PWS/AS亚区ICR中位点1的甲基化水平显著低于IVF(P<0.05),两种胚胎其他4个位点的甲基化水平差异均不显著(P>0.05);由图4-2可见,S期SCNT胚胎和IVF胚胎的PWS/AS亚区ICR中5个位点甲基化水平差异均不显著(P >0.05);由图 4-3可见,2-cell期 SCNT胚胎 PWS/AS亚区ICR中位点1和位点5的甲基化水平均显著高于IVF(P<0.05),两种胚胎其他3个位点的甲基化水平差异均不显著(P>0.05);由图4-4可见,8-cell期SCNT胚胎PWS/AS亚区ICR中位点1的甲基化水平显著高于IVF(P<0.05),两种胚胎其他4个位点的甲基化水平差异均不显著(P>0.05);由图4-5可见,囊胚期SCNT胚胎PWS/AS亚区ICR中位点1的甲基化水平显著高于IVF(P<0.05),两种胚胎其他4个位点的甲基化水平差异也均不显著((P>0.05)。因此,位点1和位点5为去甲基化抑制机制的易感位点。
3 讨论
PWS/AS基因簇的ICR区表现为母源印记状态,因此,其ICR区在早期胚胎发育过程中的甲基化变化模式可以反映母源染色体的甲基化模式[11]。该ICR印记区在IVF胚胎中的甲基化模式为2-cell期之前进行去甲基化,并于8-cell期开始DNA甲基化的重建。该结果与前面的免疫荧光结果相吻合,与Fatima等[3]的试验结果吻合[3]。IVF 胚胎中 PWS/AS基因簇的 ICR 区(PWS-IC)在S期前去甲基化并不显著(P>0.05),发育到S期,DNA大量复制后才进行了显著的去甲基化(P<0.05)。该检测结果表明,母源基因的印记区段在S期之前先进行微弱的主动去甲基化,在S期DNA大量复制时进行大规模的被动去甲基化。该结论可以支持Chan、Goll和Morgan等的观点[12],但还有待更多的试验验证。
SCNT胚胎PWS/AS基因簇的ICR区(PWS-IC)甲基化模式较IVF胚胎变化比较剧烈,在卵裂之前的SCNT胚胎中PWS/AS基因簇的ICR区的甲基化水平较IVF胚胎差异不显著(P>0.05),见表1。卵裂后SCNT的甲基化水平发生了极显著的升高,表明母源DNA在S期至2-cell期的发育阶段进行了异常的甲基化重建,该结果证明了Dean等[13]的观点,克隆胚胎甲基化过高的原因为过早地进行了甲基化的重建。同时也提示在去核的卵母细胞中存在着大量过表达或功能得到增强的甲基化重建相关酶,影响基因组的正常去甲基化。超甲基化水平必定会影响ICR区对于整个基因簇的调控情况,从而对细胞分化、增殖、胚胎发育等产生重要关系[14]。McAllister等[15]研究已经证实,SNRPN 基因印记的紊乱还会导致神经系统的发育异常。
4 结论
PWS-IC区域甲基化模式改变,导致了SCNT胚胎母源DNA的高甲基化;同时筛选出了2个去甲基化抑制机制的易感位点,即SNRPN启动子的1 693 bp开始的258 bp序列中的1号和5号位点。推测是母源DNA过早地进行了甲基化的重建,造成了克隆胚胎的甲基化过高,建议在核移植过程中优化卵母细胞的筛选机制和卵母细胞成熟机制。
[1]Thorvaldsen J L,Duran K L,Bartolomei MS.Deletion of the H19 differentially methylated domain results in loss of imprinted expression of H19 and Igf2[J].Genes Dev,1998,12:3693-3702.
[2]Drewell R A,Arney K L,Arima T,et al.Novel conserved elements upstream of the H19 gene are transcribed and act as mesodermal enhancers[J].Development,2002,129:1205-1213.
[3]Fatima Santos,Valeri Zakhartchenko,Miodrag Stojkovic,et al.Epigenetic marking correlates with developmental potential in cloned bovine preimplantation embryos[J].Current Biology,2003,13:1116-1121.
[4]Dean W,Santos F,Stojkovic M,et al.Conservation of methylation reprogramming in mammalian development:Aberrant reprogramming in cloned embryos[J].Proc Natl Acad Sci USA,2001,98:13734-13738.
[5]Wolf D P.Assisted reproductive technologies in rhesus macaques[J].Reprod Biol Endocrinol,2004,2:37.
[6]李裕强,安志兴,张涌.牛卵母细胞质内注射体细胞核移植胚的早期发育[J].畜牧兽医学报,2005,36(1):33-37.
[7]Barlow D P.Gametic imprinting in mammals[J].Science,1995,270(5242):1610-1613.
[8]Juan V,Crain C,Wilson C.Evidence for evolutionarily conserved secondary structure in the H19 tumor suppressor RNA[J].Nuclear Acids Res,2000,28(5):1221-1227.
[9]Lustig Yariv O,Schulze E,Komitowski D,et al.The expression of the imprinted genes H19 and IGF-2 in choriocarcinoma cell lines.Is H19 a tumor suppressor gene[J].Oncogene,1997,15(2):169-177.
[10]Ariel I,Sughayer M,Fellig Y,et al.The imprinted H19 gene is a marker of early recurrence in human bladder carcinoma [J].Mol-Pathol,2000,53(6):320-323.
[11]Ohta T,Gray T A,Rogan P K,et al.Imprinting-mutation mechanisms in Prader-Willi syndrome[J].Am J Hum Genet,1999,64:397-413.
[12]Chan S W,Henderson I R,Jacobsen S E.Gardening the genome:DNA methylation in Arabidopsis thaliana[J].Nat Rev Genet,2005,6(5):351-360.
[13]Dean W,Santos F,Stojkovic M,etal.Conservation of methylation reprogramming in mammalian development:aberrant reprogramming in cloned embryos[J].Proc Natl Acad Sci USA,2001,98(24):13734-13738.
[14]Couldrey C,Lee R S F.DNA methylation patterns in tissues from mid-gestation bovine foetuses produced by somatic cell nuclear transfer show subtle abnormalities in nuclear reprogramming[J].BMC Developmental Biology,2010,10:27.
[15]McAllister G,Amara S G,Lerner M R.Tissue-specific expression and cDNA cloning of small nuclear ribonucleoprotein-associated polypeptide N[J].Proceeding of the National Academy of Sciences of the United States of America,1988,85(14):5296-5300.

