TAR克隆技术及其在微生物次级代谢产物研究中的应用
童瑞年,吴旭日,陈依军
(中国药科大学生命科学与技术学院化学生物学研究室,南京210009)
微生物次级代谢产物结构多样、种类丰富,是新药研发的重要来源之一[1]。微生物的次级代谢产物通常由相关生物合成基因簇(biosynthetic gene clusters BGCs)编码的生物合成途径逐步生物合成产生。微生物次级代谢产物的生物合成基因簇大小一般为几十到几百kb不等,包含编码化合物合成前体、骨架装配、修饰的相关基因,有时也会存在抗性基因和/或调控基因[2]。据推测,实验室可培养的微生物数量不及微生物总数的1%,而且很多微生物在人类设定的培养条件下会出现基因簇沉默或低表达,严重阻碍了微生物来源药物的发现和开发,亟待革新研究策略[3]。完整生物合成基因簇的异源表达策略为从不可培养微生物和基因簇沉默微生物中获取有价值的次级代谢产物提供了有效手段[4],其优点主要包括:(1)可直接关联不同生物合成基因簇与其产生的次级代谢产物;(2)采用通用性强的异源表达宿主和遗传操作系统,实现对任何来源的生物合成基因簇进行简便的遗传操作;(3)异源表达宿主多为遗传背景清晰且稳定的菌株,有利于异源基因簇的稳定表达和次级代谢产物的累积;(4)可通过理性改造生物合成基因簇,实现次级代谢产物结构的多样性。
作为一种大片段DNA克隆技术,TAR(transformation associated recombination)克隆技术近年来被广泛应用于微生物次级代谢产物生物合成基因簇的异源表达和次级代谢产物异源生物合成[5]。与常规的生物合成基因簇异源表达策略相比,基于TAR的基因簇异源表达策略的优势体现为:(1)可从复杂基因组中选择性分离目标DNA;(2)所选择分离的 DNA片段较大,最长可达到 300 kb;(3)操作简便,实验周期短;(4)能够与CRSPR/Cas9技术偶联,进一步提高效率的同时便于基因簇的理性改造,能够满足绝大多数微生物次级代谢产物生物合成基因簇完整异源表达的需求,对微生物来源新药的研发具有重要价值。本文从TAR克隆技术的基本原理出发,简要介绍TAR克隆技术在微生物次级代谢产物生物合成基因簇异源表达方面的研究进展,为微生物来源新药研发提供新的研究策略。
1 TAR克隆技术及其基本原理
TAR克隆技术是指利用酵母的重组系统从复杂基因组中选择性分离目的DNA片段的策略,也是目前唯一能够选择性分离长达300 kb片段的方法[6-7]。20世纪 80年代,Botstein等[8-9]首次证明了在酵母细胞内两个含同源序列的DNA分子可以同源重组,并将其开发用于更普遍的DNA体内连接或拼接,后被命名为TAR克隆技术。但是,当初的TAR克隆技术对于选择性分离大片段的效率很低,只有1% ~5%[5,10]。经过长期和持续的策略改变和操作方法优化,改良后的TAR克隆技术在分离长度为250 kb的目的DNA片段时阳性克隆效率被提高至32%,分离长度较小的片段时效率则更高,而传统的基因组BAC文库筛选的阳性克隆率仅为 0.003%[11-12]。相比之下,TAR克隆技术极大地提高了大片段DNA的获取效率,降低了筛选的工作量,缩短了实验周期。由于上述优势,TAR克隆技术近期被不断尝试应用于合成生物学领域的研究,为克隆和改造生物合成基因簇以及通过异源表达获得活性次级代谢产物注入了新的活力。
TAR克隆技术是基于含同源序列的DNA片段的自由末端在酵母细胞内能够高效同源重组的原理而建立[13]。TAR克隆载体两端各含一个特异性靶向目的基因的序列即目的DNA片段“捕获臂”,长度一般达到60 bp以上就能实现目的片段的选择性捕获。同时,TAR载体还包含酵母着丝粒序列(CEN)和酵母选择性标记(His3),便于阳性载体在酵母细胞中的复制和筛选。线性化的TAR载体与含有目的DNA片段的基因组共转入基因改造的酿酒酵母细胞Saccharomyces cerevisiae VL6-48。借助酵母的重组系统,TAR载体两端的捕获臂与目的DNA片段两端的同源序列发生同源重组,重组后的质粒在酵母中自我复制和游离,从而实现目的DNA片段的选择性获取。在Larionov等[14]对TAR载体改造前的初级版中,被克隆的目的DNA片段中需要含有至少一个自主复制序列(autonomously replicating sequence,ARS),作为在酵母细胞中的复制起点。由于真核生物基因组中每20~40 kb就出现一段ARS样序列,理论上即使TAR载体不含任何ARS序列也能够实现目的DNA片段的选择性获取和复制。但是,原核生物基因组中缺少ARS样序列,导致TAR克隆技术无法应用于细菌来源次级代谢产物生物合成基因簇的选择性获取。2003年,Larionov等[14]通过在TAR克隆载体中插入来源于酵母的ARS元件(ARSH4),使得TAR克隆技术快速高效获取的生物合成基因簇或DNA片段具备了真核和原核微生物的通用性,拓宽了该策略的应用范围。
2 TAR克隆技术的应用
2.1 基于TAR克隆技术的基因簇组装
虽然大量的微生物在现有实验室条件下无法培养,但是可以从环境DNA(environmental DNA,eDNA)中选择性克隆这些微生物的生物合成基因簇,并在模式宿主中异源表达,为从无法培养微生物中筛选获得结构新颖、活性强的次级代谢产物提供了可能。
以前,基于cosmid/fosmid文库的DNA克隆技术是从eDNA筛选微生物次级代谢产物生物合成基因簇的经典手段。美国洛克菲勒大学Brady教授课题组在该方面开展了一系列研究,筛选获得了多种结构新颖且具有抗菌活性的次级代谢产物[15-18]。然而,基因组功能分析显示,不少微生物次级代谢产物的生物合成基因簇如PKS-NRPS杂合基因簇长达上百kb,但cosmid/fosmid文库可插入的DNA片段大小一般限制在45 kb以内,难以将一个大型生物合成基因簇完整克隆,需要将克隆的多个片段通过重叠延伸PCR技术拼接组装成完整的基因簇[19-20]。随后,再通过在模式宿主菌中异源表达所拼接的基因簇,生物合成特定的微生物次级代谢产物。显而易见,基于PCR的片段装配存在操作过程繁琐,阳性克隆率低和实验周期长等缺陷。针对这些问题,Kim等[21]于2010年构建了TAR克隆载体pTARa(图1-A),包含酵母、大肠埃希菌和链霉菌3部分元件。pTARa载体可以在酵母细胞中进行基因簇片段的快速拼接和装配,也可以转化至大肠埃希菌进行目的基因簇的分析和扩增,还可以借助接合转移将目的生物合成基因簇整合到链霉菌染色体而实现异源表达,合成目标次级代谢产物。在此基础上,Kim等[21]将构建的eDNA cosmid克隆和pTARa载体共转染至酿酒酵母,借助其高效同源重组系统快速组装出完整的次级代谢生物合成基因簇,包括PKS、NRPS和FRI 3个基因簇,长度分别为39、89和90 kb。与传统PCR拼接技术相比,基于TAR的基因簇组装技术基本克服了操作繁琐和实验周期长的问题,提高了从eDNA中获得完整生物合成基因簇的效率。
2.2 基于TAR克隆技术的微生物次级代谢产物的异源生物合成
TAR克隆技术不仅可用于从eDNA任意组装不可培养微生物的基因簇,也可用于从已知基因组信息的微生物中直接特异性分离特定的生物合成基因簇,避免了构建和筛选文库,有助于加快次级代谢产物异源生物合成的研究进程。
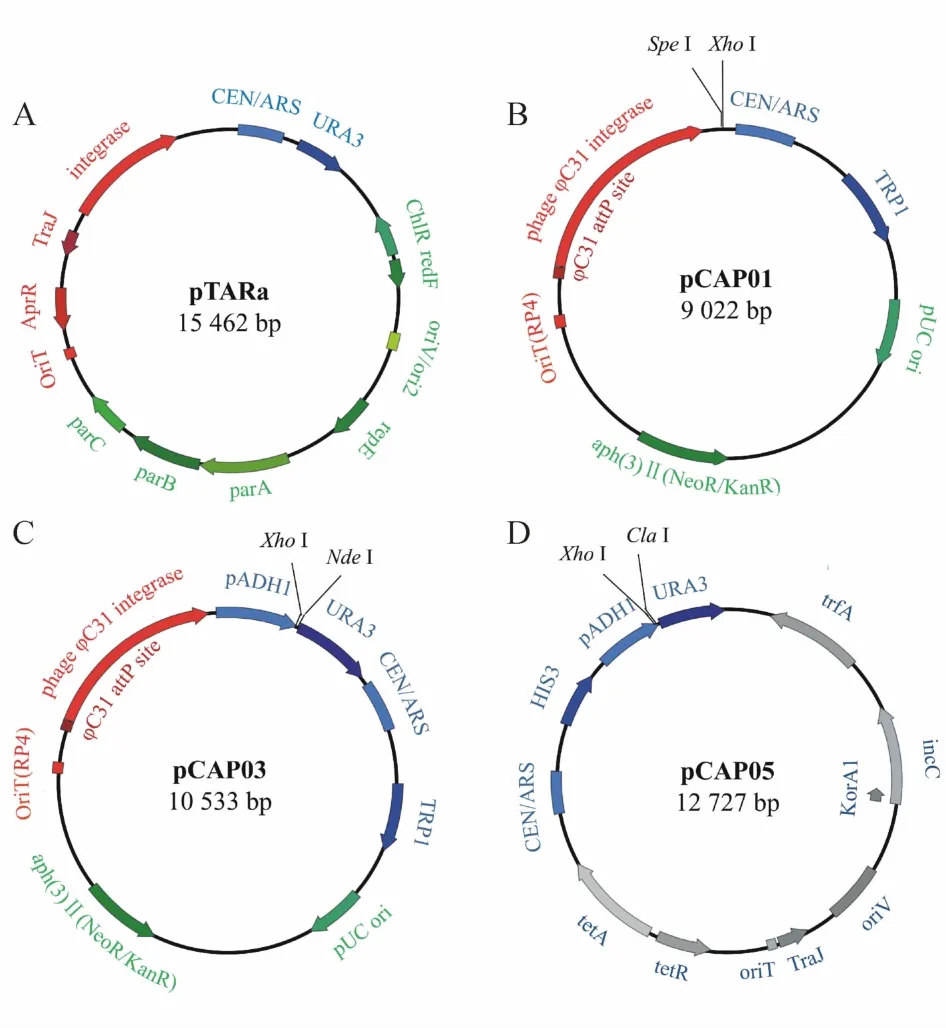
Figure 1 Physicalmap of the TAR vectorCapture arms are inserted in pCAP01,pCAP03 and pCAP05 to construct specific capture vector by restriction sites shown in the vector maps.Capture arms are incorporated into pTARa using recombination system in S.cerevisiae yeast element(blue);E.coli element(green);actinobacter element(red)and broad-host-range element(gray)
2.2.1 G+菌基因簇的异源表达 放线菌是典型的G+菌,其产生的次级代谢产物种类繁多,是抗生素等活性化合物的重要来源。据不完全统计,迄今发现的抗生素近70%为放线菌产生的次级代谢产物。因此,TAR克隆技术在G+菌生物合成基因簇的异源表达应用较多[22-25]。为更加有效地异源表达 G+菌的生物合成基因簇,美国加州大学Scripps海洋研究所Moore教授课题组在pTARa基础上构建了酵母-大肠埃希菌穿梭链霉菌整合型捕获质粒TAR载体pCAP01,并已商业化销售(图1-B)。pCAP01含有pUC的复制起始位点ori,便于在大肠埃希菌中进行多拷贝复制,并能够稳定携带大于50 kb的外源DNA片段。同时,pCAP01载体还含有卡那霉素/新霉素抗性基因aph(3)Ⅱ,用于在大肠埃希菌和链霉菌中进行阳性克隆的筛选。此外,pCAP01载体的骨架上还存在φ13整合元件,能通过接合转移将目的基因簇从大肠埃希菌转移至异源表达的链霉菌宿主并定点整合在染色体。由于φ13整合元件适用范围广,很多链霉菌可以作为基因簇异源表达合成次级代谢产物的宿主菌。此外,pCAP01在酵母细胞中为单拷贝,有利于维持大型质粒的稳定性,避免了在TAR克隆过程中产生随机同源重组,有效降低了假阳性率。如图2所示,基于TAR克隆载体pCAP01的次级代谢产物异源生物合成流程大体分为:(1)构建含捕获臂的pCAP01捕获质粒;(2)将pCAP01捕获质粒与含目的基因簇的基因组DNA同时转至酿酒酵母原生质体;(3)通过 PCR筛选捕获目的基因簇的pCAP01质粒,并测序验证;(4)大肠埃希菌扩增制备该重组质粒;(5)利用属间接合转移将重组质粒转至链霉菌宿主,经筛选获得阳性克隆菌株;(6)发酵培养阳性菌株,实现目的基因簇的异源表达和次级代谢产物的异源生物合成。
Yamanaka等[26]利用基于pCAP01质粒的TAR克隆技术捕获海洋放线菌Saccharomonospora sp.CNQ-490中一个长为67 kb的NRPS沉默生物合成基因簇,并对其调控基因进行了理性改造,将改造过的基因簇异源表达于模式链霉菌Streptomyces coelicolor,发酵产生了该基因簇编码的二氯化脂肽类抗生素taromycinA。Larson等[27]利用全合成同源臂和简单的酶切、连接反应替代了所有的载体装配步骤,简化了pCAP01捕获载体的构建,使其更适用于高通量筛选。利用改进后的TAR克隆技术,选择性分离了Streptomyces sp.CNT-302中长度为54 kb的Ⅱ型PKS途径的生物合成基因簇,并异源表达于S.coelicolor M512,成功获得了具有抗肿瘤活性的芳香聚酮化合物宇宙霉素cosmomycin及其类似物。
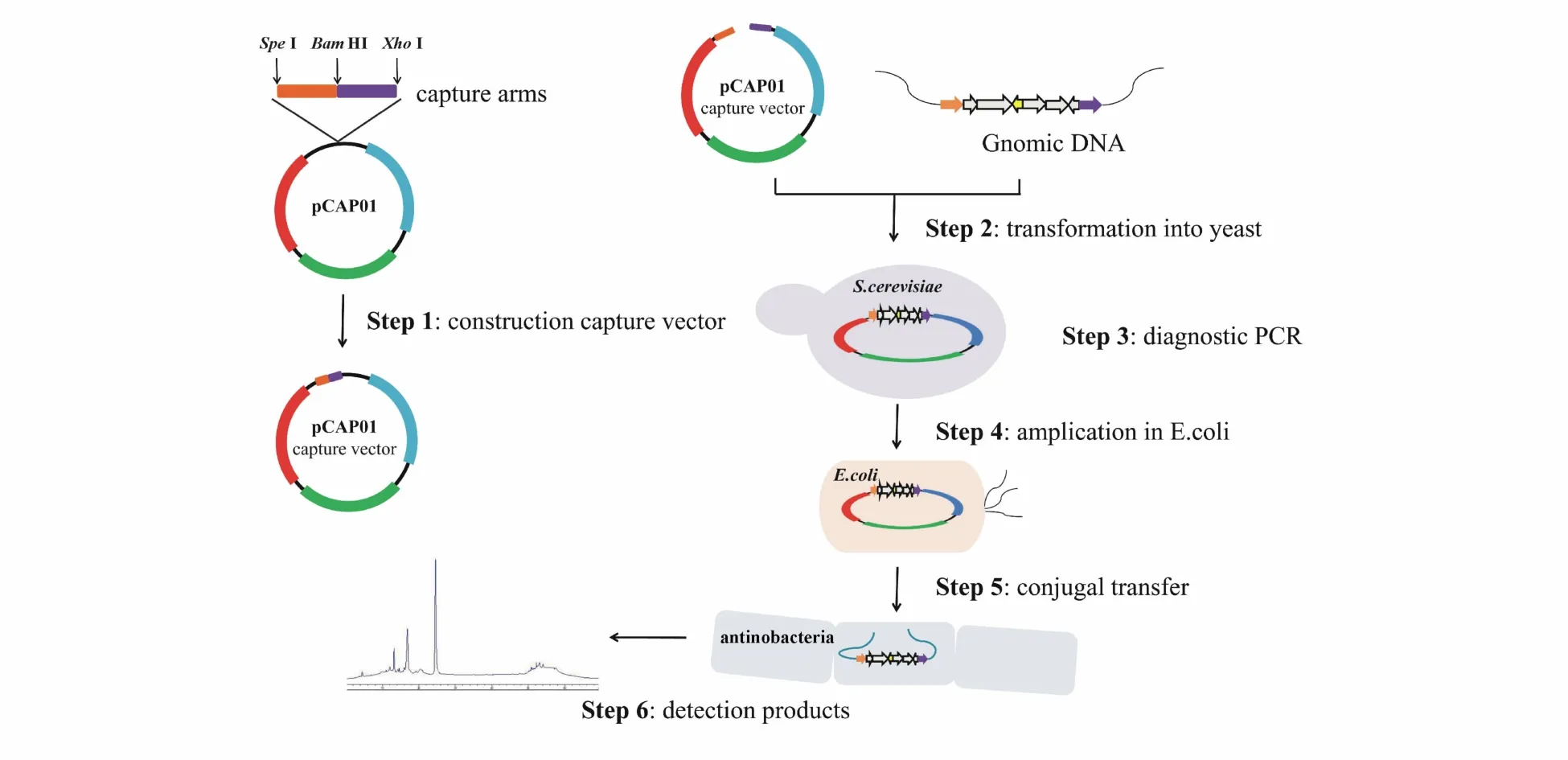
Figure 2 Strategy of TAR-based heterologous expression
2.2.2 G-菌基因簇的异源表达 TAR克隆技术不仅在G+菌基因簇的异源表达合成次级代谢产物中的应用较为广泛,在发掘G-菌产生的结构新颖次级代谢产物中的应用逐渐增多[28-30]。通常,G-菌基因簇的异源表达宿主应与原宿主的遗传背景相似或相近,目前已开发的一些G-菌宿主如Myxococcus xanthus、Pseudomonas putida和 E.coli具有生长迅速、操作简便等优势。为建立基于TAR克隆的G-菌基因簇异源表达系统,Moore教授课题组[31]将酵母元件与G-菌广宿主元件RK2家族相组合,构建了可用于变型杆菌宿主的多宿主TAR克隆载体pCAP05(图1-D)。pCAP05包含ori V复制起点和trfA基因,能在多种G-菌中低拷贝数复制。同时,它具有与pCAP03(G+菌TAR克隆载体,图1-C)相同的酵母细胞元件,能利用反相选择标记以减少背景影响,提高了阳性克隆的筛选效率。该课题组利用该 G-菌 TAR克隆载体从Pseudoalteromonas luteoviolacea 2ta16基因组中捕获了紫色杆菌素的生物合成基因簇,并在两种变形杆菌宿主Pseudomonas putida KT2440和Agrobacterium tumefaciens LBA4404实现了紫色杆菌素的异源生物合成[29]。与此同时,他们发现异源宿主与原宿主相近与否并不是决定成功异源表达的唯一因素,特殊的宿主调控机制和生物合成元件均会影响次级代谢产物的异源生物合成,这对进一步开发G-菌异源表达宿主具有重要的指导意义。
3 基于TAR克隆技术基因簇异源表达策略的改进
虽然TAR克隆技术能够从基因组中捕获目的基因簇并异源表达和合成微生物次级代谢产物,但线性载体通过非同源末端连接自连导致该技术的阳性率很低,只有0.5%~2%,由此带来的筛选工作量巨大、实验周期仍然较长。针对这一局限,研究者从多个角度对基于TAR克隆技术的微生物次级代谢产物异源生物合成策略进行了改进,大致可归纳为以下两类。
3.1 CRISPR/Cas9系统偶联TAR克隆技术
在理论上,目的基因簇捕获臂两端各引入一个双链断裂可以提高TAR克隆的效率[32]。但是,由于待克隆的目的基因簇较大且处于复杂的基因组中,一般很难在临近基因簇两端选择合适的核酸内切酶剪切位点而不破坏目的基因簇的完整性[14]。与核酸内切酶相比,CRISPR/Cas9系统不依赖于特定的酶切位点,只需识别长度为20 nt的特异性序列,几乎可以在任意位点引入双链断裂。因此,CRISPR/Cas9系统与TAR克隆技术的偶联在微生物次级代谢产物异源生物合成方面的优势显著。Nicholas等[33]通过 CRISPR/Cas9偶联 TAR克隆技术在目的DNA片段两端引入双链断裂使TAR克隆的阳性克隆率提高至32%,减少了筛选工作量,缩短了实验周期。CRISPR/Cas9系统偶联TAR克隆技术的操作流程如图3所示:(1)设计并转录合成靶向目的基因簇两端的sgRNA,体外装配Cas9-sgRNA复合物;(2)利用Cas9-sgRNA复合物处理含目的基因簇的基因组,并在目的基因簇两端切割基因组;(3)将Cas9-sgRNA处理的基因组和线性化的捕获质粒共转至酵母原生质体,以通过同源重组捕获目的基因簇;(4)PCR筛选捕获目的基因簇的TAR质粒。含目的基因簇的TAR质粒则可被转化至大肠埃希菌扩增,并通过接合转移转移至模式宿主菌实现特定微生物次级代谢产物的异源生物合成。Meng等[34]利用 CRISPR/Cas9偶联 TAR克隆技术从Streptomyces pristinaespiralis中选择性克隆pristinamycin I基因簇,敲除负调控基因后整合至基因改造宿主 S.pristinaespiralisΔPIIΔpapR3,实现了pristinamycin I的高效生物合成。
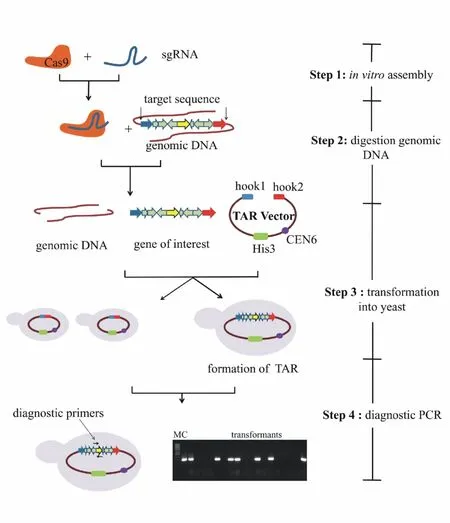
Figure 3 Selective cloning of biosynthetic gene cluster of interest by CRISPR/Cas9-meditated TAR
3.2 阳性克隆的反向筛选
在TAR克隆过程中,降低TAR克隆载体自环化所致的高背景同样可以提高阳性克隆率。Moore教授课题组在TAR克隆载体pCAP01的基础上,引入强启动子pADH1基因和反向选择性标记URA3基因,构建了可减少高背景影响的新TAR克隆载体 pCAP03(图2-C)。结果显示,加入 URA3反向标记后,含有自环化载体的酵母克隆不能在含5-FOA的平板上生长,酵母克隆数量大幅减少,阳性克隆率达到80%,极大地缩短了筛选阳性克隆的工作量和时间。借助pCAP03载体,Moore教授课题组[35]成功地从86株海洋放线菌基因组中分离出PKS-NRPS杂交途径的生物合成基因簇tlm及其相关的ttm基因簇,并在模式链霉菌S.coelicolor M1152中异源表达,合成了一系列硫代季酮酸类次级代谢产物,其中包括脂肪酸合成酶抑制剂硫乳霉素。
4 展 望
本文概述了TAR克隆技术的基本原理及其在微生物次级代谢产物异源生物合成的中的应用,充分展现了该技术作为发现微生物来源药物的重要工具的科学意义和应用价值。虽然基于TAR克隆技术的基因簇异源表达策略正在不断发挥其功能和价值,但目前尚存在一些明显的缺点,有待于进一步改进:(1)TAR克隆技术对高GC含量基因簇的捕获能力较弱;(2)TAR克隆技术无法用于研究非成簇存在的基因所编码的次级代谢产物;(3)有些基因簇编码的酶或次级代谢产物对酵母有毒性,这些基因簇无法通过TAR克隆技术克隆和异源表达;(4)理论上TAR克隆技术对捕获片段无长度限制,但目前能捕捉的基因簇最大约为300 kb。然而,随着对微生物次级代谢研究的不断深入和不同学科或技术间的交叉融合,基于TAR克隆技术的微生物次级代谢产物异源生物合成策略将会日趋完善,为微生物生物合成基因功能的研究和微生物来源新药发现提供强有力的工具和手段。
[1] Newman DJ,Cragg GM.Natural products as sources of new drugs over the 30 years from 1981 to 2010[J].J Nat Prod,2012,75(3):311-335.
[2] Castro JF,Razmilic V,Gomez-Escribano JP,et al.Identification and heterologous expression of the chaxamycin biosynthesis gene cluster from Streptomyces leeuwenhoekii[J].Appl Environ Microbiol,2015,81(17):5820-5831.
[3] Galm U,Shen B.Expression of biosynthetic gene clusters in heterologous hosts for natural product production and combinatorial biosynthesis[J].Expert Opin Drug Discov,2006,1(5):409-437.
[4] Cobb R E,Luo Y,Freestone T,etal.Chapter10-Drug Discovery and Development via Synthetic Biology[M]//Synthetic Biology.Elsevier Inc.2013:183-206.
[5] Kouprina N,Larionov V.Transformation-associated recombination(TAR)cloning for genomics studies and synthetic biology[J].Chromosoma,2016,125(4):621-632.
[6] Kouprina N,Annab L,Graves J,et al.Functional copies of a human gene can be directly isolated by transformation-associated recombination cloning with a small 3′end target sequence[J].Proc Natl Acad Sci U SA,1998,95(8):4469-4474.
[7] Larionov V,Kouprina N,Solomon G,et al.Direct isolation of human BRCA2 gene by transformation-associated recombination in yeast[J].Proc Natl Acad Sci U S A,1997,94(14):7384-7387.
[8] Kunes S,Botstein D,Fox MS.Transformation of yeastwith linearized plasmid DNA.Formation of inverted dimers and recombinant plasmid products[J].JMol Biol,1985,184(3):375-387.
[9] Ma H,Kunes S,Schatz PJ,et al.Plasmid construction by homologous recombination in yeast[J].Gene,1987,58(2/3):201-216.
[10]Kouprina N,Larionov V.TAR cloning:insights into gene function,long-range haplotypes and genome structure and evolution[J].Nat Rev Genet,2006,7(10):805-812.
[11]Kouprina N,Larionov V.Exploiting the yeast Saccharomyces cerevisiae for the study of the organization and evolution of complex genomes[J].FEMSMicrobiol Rev,2003,27(5):629-649.
[12]Leem SH,Noskov VN,Park JE,et al.Optimum conditions for selective isolation of genes from complex genomes by transformation-associated recombination cloning[J].Nucleic Acids Res,2003,31(6):e29.
[13]Orr-Weaver TL,Szostak JW,Rothstein RJ.Yeast transformation:a model system for the study of recombination[J].Proc Natl Acad Sci U SA,1981,78(10):6354-6358.
[14]Noskov VN,Kouprina N,Leem SH,et al.A general cloning system to selectively isolate any eukaryotic or prokaryotic genomic region in yeast[J].BMCGenomics,2003,4(1):16.
[15]Katz M,Hover BM,Brady SF.Culture-independent discovery of natural products from soilmetagenomes[J].J Ind Microbiol Biotechnol,2016,43(2/3):129-141.
[16]Zhou Z,Xu Q,Bu Q,et al.Genomemining-directed activation of a silent angucycline biosynthetic gene cluster in Streptomyces chattanoogensis[J].ChemBioChem,2015,16(3):496-502.
[17]Kang HS,Brady SF.Mining soil metagenomes to better understand the evolution of natural product structural diversity:pentangular polyphenols as a case study[J].JAm Chem Soc,2014,136(52):18111-18119.
[18]Kang HS,Brady SF.Arixanthomycins A-C:phylogeny-guided discovery of biologically active eDNA-derived pentangular polyphenols[J].ACSChem Biol,2014,9(6):1267-1272.
[19]Brady SF.Construction of soil environmental DNA cosmid libraries and screening for clones that produce biologically active small molecules[J].Nat Protoc,2007,2(5):1297-1305.
[20]Schmidt EW,Nelson JT,Rasko DA,et al.Patellamide A and C biosynthesis by a microcin-like pathway in Prochloron didemni,the cyanobacterial symbiont of Lissoclinum patella[J].Proc Natl Acad Sci,2005,102(20):7315-7320.
[21]Kim JH,Feng Z,Bauer JD,et al.Cloning large natural product gene clusters from the environment:piecing environmental DNA gene clusters back together with TAR[J].Biopolymers,2010,93(9):833-844.
[22]Jordan PA,Moore BS.Biosynthetic pathway connects cryptic ribosomally synthesized posttranslationally modified peptide genes with pyrroloquinoline alkaloids[J].Cell Chem Biol,2016,23(12):1504-1514.
[23]Tan GY,Deng K,Liu X,et al.Heterologous biosynthesis of spinosad:an omics-guided large polyketide synthase gene cluster reconstitution in Streptomyces[J].ACS Synth Biol,2017,6(6):995-1005.
[24]Losada AA,Cano-Prieto C,García-Salcedo R,et al.Caboxamycin biosynthesis pathway and identification of novel benzoxazoles produced by cross-talk in Streptomyces sp.NTK 937[J].Microb Biotechnol,2017,10(4):873-885.
[25]Wu N,Huang H,Min T,et al.TAR cloning and integrated overexpression of6-demethylchlortetracycline biosynthetic gene cluster in Streptomyces aureofaciens[J].Acta Biochim Biophys Sin(Shanghai),2017,49(12):1129-1134.
[26]Yamanaka K,Reynolds KA,Kersten RD,etal.Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J].Proc Natl Acad Sci,2014,111(5):1957-1962.
[27]Larson CB,Crüsemann M,Moore BS.PCR-Independentmethod of transformation-associated recombination reveals the cosmomycin biosynthetic gene cluster in an ocean Streptomycete[J].JNat Prod,2017,80(4):1200-1204.
[28]Kong FD,Zhou LM,Ma QY,et al.Metabolites with Gram-negative bacteria quorum sensing inhibitory activity from the marine animal endogenic fungus Penicillium sp.SCS-KFD08[J].Arch Pharm Res,2017,40(1):25-31.
[29]Pham TH,Boon N,Aelterman P,et al.Metabolites produced by Pseudomonas sp.enable a Gram-positive bacterium to achieve extracellular electron transfer[J].Appl Microbiol Biotechnol,2008,77(5):1119-1129.
[30]Masschelein J,Jenner M,Challis GL.Antibiotics from Gram-negative bacteria:a comprehensive overview and selected biosynthetic highlights[J].Nat Prod Rep,2017,34(7):712-783.
[31]Zhang JJ,Tang X,Zhang M,et al.Broad-host-range expression reveals native and host regulatory elements that influence heterologous antibiotic production in Gram-negative bacteria[J].MBio,2017,8(5):e01291-1317.
[32]Fu J,Bian X,Hu S,etal.Full-length RecE enhances linear-linear homologous recombination and facilitates direct cloning for bioprospecting[J].Nat Biotechnol,2012,30(5):440-446.
[33] Lee NCO,Larionov V,Kouprina N.Highly efficient CRISPR/Cas9-mediated TAR cloning of genes and chromosomal loci from complex genomes in yeast[J].Nucleic Acids Res,2015,43(8):e55.
[34]Meng J,Feng R,Zheng G,et al.Improvement of pristinamycin I(PI)production in Streptomycespristinaespiralis bymetabolic engineering approaches[J].Synth Syst Biotechnol,2017,2(2):130-136.
[35]Tang X,Li J,Millán-Aguiñaga N,et al.Identification of thiotetronic acid antibiotic biosynthetic pathways by target-directed genome mining[J].ACS Chem Biol,2015,10(12):2841-2849.

