牦牛SCD基因的克隆和生物信息学分析
付东海,吴晓云,王宏博,褚 敏,郭 宪,裴 杰,包鹏甲,丁学智,阎 萍,梁春年
(1.中国农业科学院兰州畜牧与兽药研究所,兰州 730050;2.甘肃省牦牛繁育重点实验室,兰州 730050)
硬脂酰辅酶A去饱和酶(Stearoyl-CoA desaturase,SCD)是调控乳中不饱和脂肪酸合成的限速酶,也是反刍动物肉奶中共轭亚油酸内源合成的关键酶[1-2]。在参与脂肪代谢的基因中,SCD能够催化C14到C19的脂肪酸乙酰-CoA底物上Δ9位加一个双键,在反刍动物的乳腺和肝脏脂类代谢过程中发挥重要作用[3-5]。SCD负责SFA向单不饱和脂肪酸 (Monounsaturated fatty acids,MUFA)的转变[3,6]。哺乳动物 SCD cDNA 首次在老鼠肝脏中发现[7]。随后,SCD 基因在大鼠[8]、猪[9]、绵羊[10]、山羊[11]、牛[12]、人[13]等主要哺乳动物体内相继被分离。牛SCD基因位于26号染色体(26q21),整个基因长17 088 bp,转录所得的cDNA全长5 287 bp,CDS序列共1 080 bp,编码 359个氨基酸[14]。研究发现,SCD可能影响牛奶中SFAs/MUFAs比率、肌间脂肪沉积和成分,是改善牛奶和牛肉质量的主要候选基因[15]。无角牦牛新品种是中国农业科学院兰州畜牧与兽药研究所“牦牛资源与育种”创新团队与青海大通种牛场经过20余年密切合作培育的牦牛新品种,因产于海拔4 380 m的阿什旦雪山脚下而得名。由于SCD在不饱和脂肪酸合成过程中起关键作用,因此,深入了解牦牛SCD基因的结构和功能,对无角牦牛新品种的培育和乳肉品质的改良都有重要意义。
1 材料与方法
1.1 材料
选取屠宰后健康母牦牛,迅速取乳腺组织,液氮保存,带回实验室,-70℃备用。
1.2 主要试剂
RNA提取试剂盒、DNA胶回收试剂盒购自大连宝生物有限公司;TaKaRa反转录试剂盒、200 bp ladder DNA marker、pMD19-T克隆载体试剂盒购自大连宝生物工程有限公司;Trans-5α感受态细胞购自北京全式金生物技术有限公司;2×Power Taq PCR MasterMix购自大连宝生物有限公司。
1.3 方法
1.3.1 无角牦牛乳腺组织总RNA提取及RT-PCR反应用RNA试剂盒对牦牛乳腺组织总RNA进行提取,去除基因组中残留的DNA,用紫外分光光度计与1%琼脂糖凝胶电泳检测提取物质量。按照反转录试剂盒说明书操作步骤反转录为cDNA,-70℃保存备用。1.3.2 引物设计 根据GenBank中普通牛SCD基因mRNA序列(登录号:NM_173959.4),利用NCBI网站的pick Primer在线程序设计特异性引物,预计扩增大小为1 000 bp。扩增引物由兰州励合生物有限公司合成。引物序列为上游 F:5′-TCTACACTCAGTTTGGACTGCC-3′;下游 R:3′-GCTTTTGGAAAAGGAACCCAAG-5′。
1.3.3 PCR扩增反应 总反应体系20 μL:上下游引物各 1 μL,RNase-Free ddH2O 7 μL,2×Power Taq PCR ,MasterMix 10 μL,cDNA 模板 1 μL。PCR 反应条件:99℃预变性 5 min;99℃变性 30 s,57℃退火 30 s,65℃ 延伸1 min,30个循环;65℃终延伸 10 min,4℃保存。PCR 产物用0.8%的琼脂糖凝胶电泳检测。
1.3.4 目的片段TA克隆与鉴定 使用0.8%琼脂糖凝胶电泳检测扩增产物,用琼脂糖凝胶回收试剂盒回收目的片段,与pMD19-T克隆载体在 4℃条件下连接30 mins;连接产物转化至50 μL Trans-5α感受态细胞,转化后加900 μL LB培养液摇匀复苏10 mins,将复苏后的菌液涂布于含0.1 mg/mL Amp的LB平板培养皿表面,37℃避光培养10 h;挑取白色阳性单克隆菌落,用LB(Amp+)培养液37℃扩大培养,之后进行菌液 PCR鉴定。PCR 反应体系为 20 μL:菌液 1 μL,上下游引物各 1 μL,2×Power Taq PCR MasterMix 10 μL,水 7 μL。PCR 反应条件:99℃预变性 5 min;99℃变性 30 s,57℃退火 30 s,65℃延伸 1 min,30个循环;65℃延伸 10 min,4℃保存,产物用0.8%的琼脂糖凝胶电泳检测,筛选阳性菌液送北京擎合生物有限公司测序。
1.3.5 生物信息学分析 试验所用的分析网站和软件如下。
基因开放阅读框(ORF):DNASTAR 和 Lasergene;理化性质分析:http://web.expasy.org/protparam;二级结构预测:http://www.predictprotein.org;亚细胞定位:https://wolfpsort.hgc.jp/; 同 源 性 比 较 :https://www.ebi.ac.uk/Tools/emboss;系统发生树:MEGA5.1软件中的Phylogeny程序;信号肽预测:http://cbs.dtu.dk/service/SignalIP;功能结构域预测:http://cbs.dtu.dk/service/TMHMM Signal IP。
2 结果与分析
2.1 牦牛SCD基因总RNA检测
提取无角牦牛新品种乳房组织总RNA,用1.5%琼脂糖凝胶电泳检测到带型完整的3条带,分别为28 S、18 S和5 S rRNA;测定的A260/A280比值在1.8~2.0之间,说明RNA完整性较好,无蛋白质和DNA污染,适合下一步RT-PCR反应。
2.2 牦牛SCD基因目的片段扩增与TA克隆
RT-PCR扩增产物经0.8%琼脂糖凝胶电泳检测,并做两个平行。如图1所示。出现单一DNA条带,片段长度介于1 000~1 300 bp,与预扩增片段长短一致,可用于克隆测序。将克隆后的阳性菌液送公司测序,获得无角牦牛SCD基因CDS区序列。
2.3 牦牛SCD基因CDS区核苷酸序列及氨基酸序列分析
应用Lasargene的edit squence程序分析无角牦牛SCD基因序列,获得长1 271 bp的ORF,起始密码子ATG位于122 bp处,终止密码子TAG位于1 202 bp处,由此得出,无角牦牛SCD基因CDS区全长1 080 bp。软件分析其碱基组成分别为A=24.7%、G=23.6%、T=24.2%、C=27.5%,共编码359个氨基酸残基(图2)。G+C(51.1%),高于A+T(48.9%),说明SCD基因编码区的DNA双链比较稳定,变异较少。

图1 牦牛SCD基因RT-PCR扩增产物琼脂糖凝胶电泳

图2 牦牛SCD基因CDS区核苷酸及编码氨基酸序列
2.4 牦牛与普通牛SCD基因序列及氨基酸序列比对
使 用 Pairwise Sequence Alignment(https://www.ebi.ac.uk/Tools/emboss/)在线网站分析程序,对无角牦牛新品种测定序列的CDS区与普通牛的SCD核酸序列(登录号:NM_173959)进行比对,结果表明,两者相似性99.35%。相比普通牛,无角牦牛新品种SCD基因的扩增片段共检测出7个碱基差异,分别为:ATC→CTC、CGG→AGG、CCA →CCG、CTG →TTG、TCC→TCA、GCG →GTG、TAC→AAC。对氨基酸序列进行比对发现:第1、第6、第7处的差异使得对应的氨基酸发生变化,而其他4处的核苷酸差异未引起编码的氨基酸改变。用SPBDV软件对普通牛的SCD蛋白结构分析发现:与无角牦牛存在的3个氨基酸差异均未发生在SCD蛋白的核心区域,而是出现在SCD蛋白质的中层至外层结构之间。这表明两者的SCD生物学功能可能相同,不存在差异。
2.5 牦牛SCD的理化性质分析
蛋白质理化性质包括相对氨基酸组成、分子质量、等电点及消光系数等数据[10]。用ProtParam与Protean软件预测牦牛SCD的理化性质。结果显示:无角牦牛新品种SCD由359个氨基酸残基组成,分子式为C1922H2912N506O514S11,分子量 41.7 kD,理论等电点(pI)9.37,说明该蛋白为碱性蛋白质。其中氨基酸残基中Leu=11.6%、Thr=7.0%的频率较高,极性氨基酸占56.5%,疏水性氨基酸占43.5%,带电荷氨基酸占20.9%,其包括11.7%的碱性氨基酸及9.2%的酸性氨基酸。其水溶液在280 nm处的消光系数83 895,不稳定系数44.41,属不稳定类蛋白质。而且预计在哺乳动物网织红细胞内的半衰期为30 h。疏水指数86.96,平均亲水性-0.235,属不可溶性蛋白。
2.6 牦牛SCD二级结构预测
蛋白质二级结构主要指多肽链主链骨架中局部的构象,对其进行预测分析有助于认识蛋白的空间结构[11]。使用Predict Protein软件预测牦牛SCD的二级结构,见图3。结果表明:螺旋所占例最大,为40.8%,其次为无规则卷曲比例,为35.3%,延伸链和折叠所占比例较低,分别为14%和10.2%。

图3 SCD蛋白二级结构预测
2.7 牦牛SCD亚细胞定位预测分析
利用WoLF PSORT在线程序对SCD的亚细胞定位进行预测分析。结果显示,SCD在内质网、液泡、细胞膜、分线粒体中均有分布,在内质网中分布的可能性最大。
2.8 牦牛SCD信号肽和跨膜结构域预测
利用SignalP和TMHMM sever v2.0对其信号肽和跨膜结构域分析,见图4和图5。信号肽预测结果表明:该蛋白的C值最大,为0.130,最大Y值(0.018)以及最大S值(0.163)均小于阈值0.5,说明无角牦牛新品种SCD无信号肽。跨膜结构域预测结果表明:该蛋白的期望跨膜螺旋数值为4,均为跨膜蛋白。使用SMART对无角牦牛SCD功能结构域预测,见图6,结果表明,无角牦牛SCD蛋白包含一个低复杂区域和4个跨膜结构。
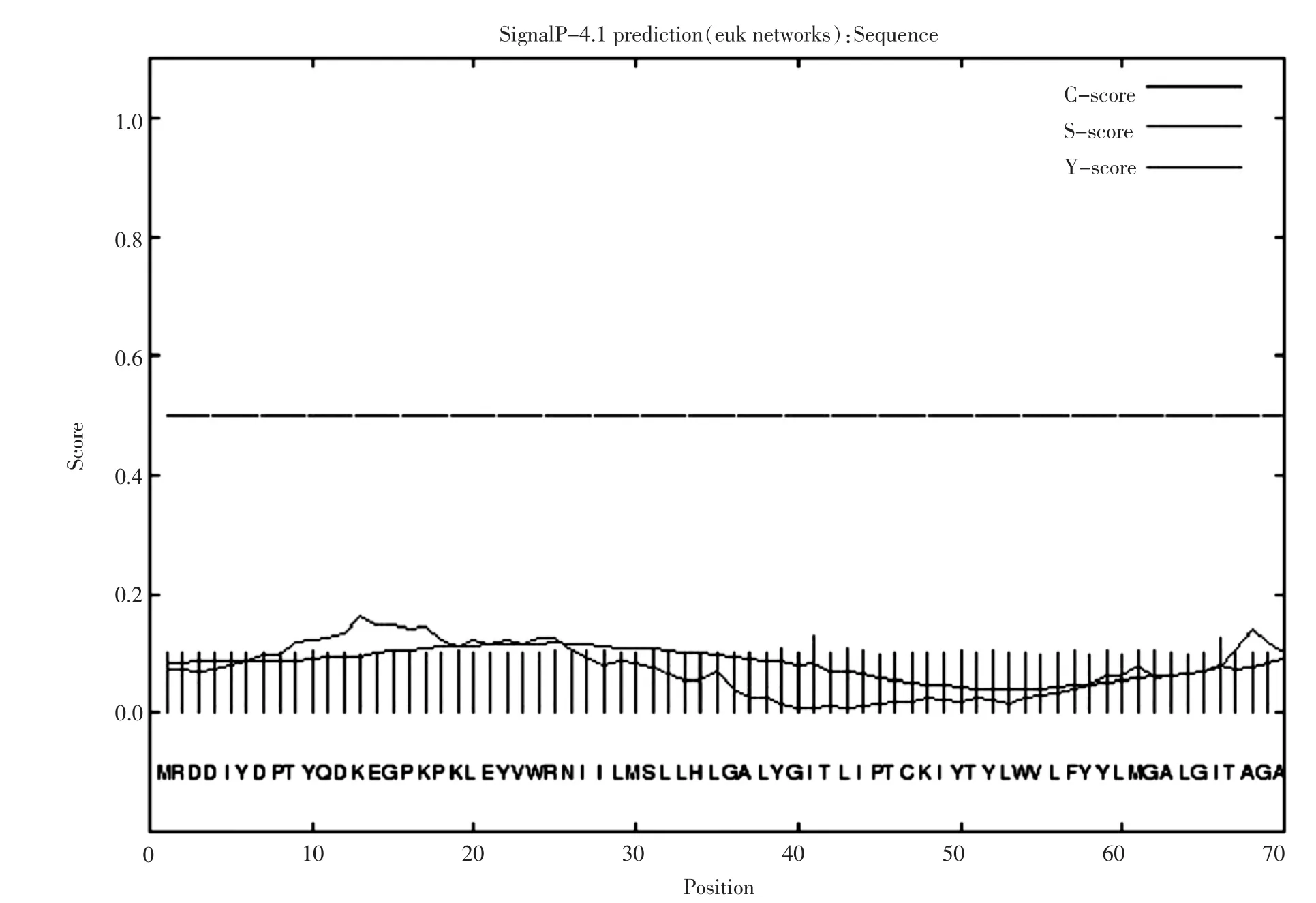
图4 信号肽预测
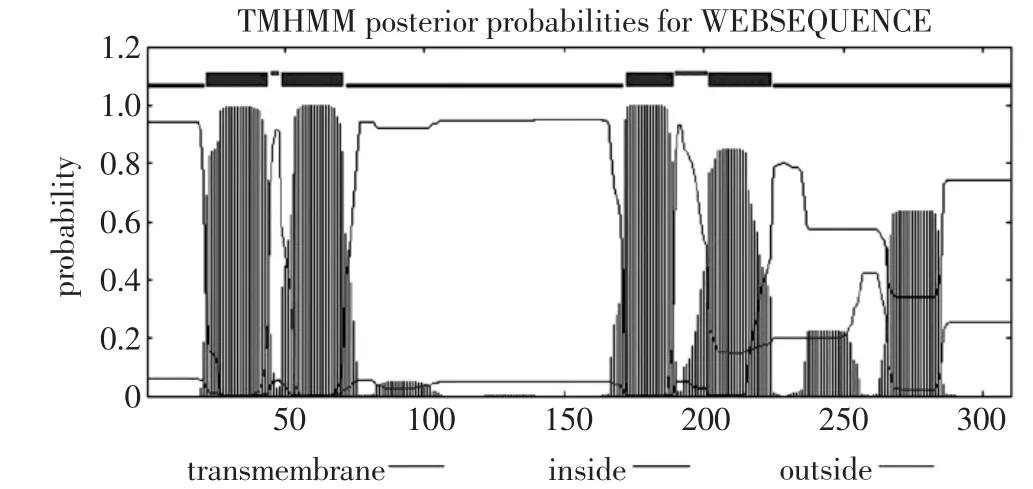
图5 跨膜结构域预测
2.9 牦牛SCD同源性及系统发育分析
利用Mega软件5.1版本中Align和Pylogeny程序对无角牦牛新品种、黑猩猩、普通牛、大鼠、小鼠、原鸡、野猪、野马、人等物种的SCD氨基酸序列进行同源性比较并构建系统发育树(表2、图7)。结果表明:无角牦牛新品种SCD氨基酸序列与普通牛亲缘关系最近,同源性达99.4%;其他依次为人、黑猩猩、野马、野猪、大鼠、小鼠,同源性达89.4%~94.2%,与原鸡亲缘关系较远。并且SCD发育树与生物进化的物种树基本一致,符合物种进化规律,说明SCD基因编码区在哺乳动物种间比较保守。

表2 牦牛与8个物种SCD氨基酸序列同源性比较%
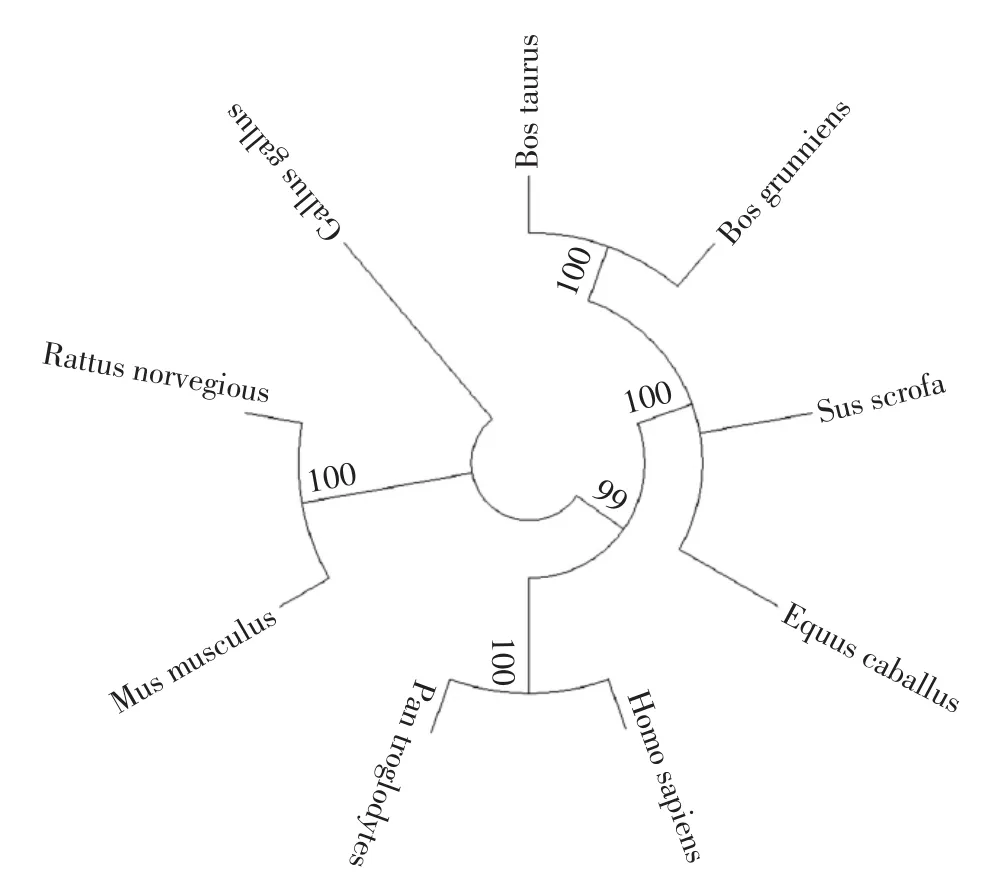
图7 牦牛与其他8个物种的SCD进化发生树
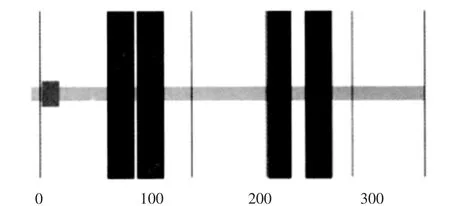
图6 SCD结构域预测
3 讨论
本次试验对无角牦牛新品种SCD基因进行克隆和编码蛋白分析,发现其编码长度为359个氨基酸残基,且与普通牛、人等物种的SCD蛋白质的氨基酸数量相差很少,与个别物种如原鸡和小鼠差异较大。这些差异与物种间的亲缘关系差异以及物种演化关系差异是保持高度一致的。无角牦牛的SCD核苷酸序列与普通牛的相比,两者存在7个碱基差异,导致编码的氨基酸发生3处改变。这3个氨基酸差异位点未出现在核心编码位点,而是发生在蛋白结构的中层至外层。表明无角牦牛与普通牛的SCD之间可能并不存在生物学功能上的差异。
蛋白质半衰期与其稳定性之间存在密切联系,一般来说,半衰期长则蛋白质稳定性高。本试验分析结果显示,SCD具有较长的半衰期,但却属于不稳定蛋白,这需要进一步的验证。蛋白同源性在一定程度上反映物种间亲缘关系的远近。牦牛、普通牛、黑猩猩、大鼠、小鼠、野猪、野马、人7个物种的氨基酸亲缘关系较近,同源性均在89%~100%,而与原鸡相差较远,差异达20.8%。而且牦牛SCD氨基酸序列的系统发生树与生物进化的物种树以及动物分类学观点也近乎保持一致。说明SCD基因编码区在长期生物进化过程中具有较强的保守性。
本研究采用RT-PCR技术与TA克隆技术,从无角牦牛新品种乳腺组织获得了SCD基因的cDNA序列,全长1 080 bp,编码359个氨基酸残基,这与罗毅浩等[16]对玉树牦牛的研究结果相吻合。对无角牦牛新品种SCD基因的生物信息学分析结果表明,牦牛SCD基因编码的为可溶碱性蛋白质,且大部分定位于内质网,在能量代谢和饱和脂肪酸脱氢途径中起重要作用。SCD氨基酸序列与普通牛、黑猩猩、人等物种间同源性较高,与其亲缘关系的远近一致,说明SCD基因编码区在长期生物进化过程中具有较强的保守性[17]。该基因的成功克隆及相关信息分析为揭示牦牛脂蛋白代谢和细胞信号的转导提供了一定的理论依据。
[1]Miyazaki M,Kim Y C,Ntambi J M.A lipogenic diet in mice with a disuption of the stearoyl-CoA desaturase 1 gene reveals a stringent requirement of endogenous monounsaturated fatty acids for triglyceride synthesis[J].Journal of Lipid Research,2001,42(7):1018-1024.
[2]陶璇,张健,魏学良.牛SCD基因研究进展[J].中国草食动物,2009,29(4):57-59.
[3]邱思源,罗军,晏原,等.奶山羊SCD的多克隆抗体制备与鉴定[J].农业生物技术学报,2017,25(7):1111-1118.
[4]曾硕士,江黎明,元冬娟.脂肪酸去饱和酶的研究进展[J].生命科学,2008(5):816-820.
[5]Jessica C Ralston,Adam H Metherel,Ken D Stark,et al.SCD1 mediates the influence of exogenous saturated and monounsaturated fatty acids in adipocytes:Effects on cellular stress,inflammatory markers and fatty acid elongation[J].The Journal of Nutritional Biochemistry,2015,27:241-248.
[6]韩立强,曹菁菁,付彤,等.奶牛硬脂酰辅酶A去饱和酶基因(SCD)启动子的克隆及活性分析[J].农业生物技术学报,2013,21(4):435-440.
[7]Thiede MA,Ozols J,Strittmatter P.Construction and sequence of cDNA for rat liver stearyl coenzyme A desaturase[J]. The Journal of Biological Chemistry,1986,261(28):13230-13235.
[8]Mihara K.Structure and regulation of rat liver microsomal stearoyl-CoA desaturase gene[J].Journal of Biochemistry,1990,108(6):1022-1029.
[9]Ren J,Knorr C,Huang L,et al.Isolation and molecular characterization of the porcine stearoyl-CoA desaturase gene[J].Gene,2004,340(1):19-30.
[10]Ward R J,Travers M T,Richards S E,et al.Stearoyl-CoA desaturase mRNA is transcribed from a single gene in the ovine genome[J].Biochimica et Biophysica Acta,1998,1391(2):145-156.
[11]Yahyaoui M H,Sánchez A,Folch J M.Rapid communication:partial nucleotide sequence of the goat stearoyl coenzyme A desaturase cDNA and gene structure [J].Journal of Animal Science,2002,80(3):866-867.
[12]Campbell E MQ,Gallagher D S,Davis S K,et al.Rapid communication:mapping of the bovine stearoyl-coenzyme A desaturase(SCD)gene to BTA26[J].J Anim Sci,2001,79:1954-1955.
[13]Zhang L,Ge L,Parimoo S,et al.Human stearoyl-CoA desaturase:alternative transcripts generated from a single gene by usage of tandem polyadenylation sites[J].The Biochemical Journal,1999,340(1):255-264.
[14]Mele M,Conte G ,Castiglioni B,et al.Stearoyl-coenzyme A desaturase gene polymorphism and milk fatty acid composition in Italian Holsteins[J].Journal of Dairy Science,2007,90(9):4458-4465.
[15]Jiang Z,Michal J J,Tobey D J,et al.Significant associations of stearoyl-CoA desaturase(SCD1)gene with fat deposition and composition in skeletal muscle[J].International Journal of Biological Sciences,2008 ,4(6):345-351.
[16]罗毅皓,孙万成.牦牛去饱和脂肪酶1(SCD1)基因的克隆及分析[J].黑龙江畜牧兽医,2017(17):237-240.
[17]Smith S B,Gill C A,Lunt D K,et al.Regulation of fat and fatty acid composition in beef cattle[J].Asian-Aust J Anim Sci,2009,22(9):1225-1233.

