GSTM1基因型与炎症性肠病关系的Meta分析
陈影影,甄玲玲,费素娟
1.徐州医科大学研究生院,江苏 徐州 221000;2.徐州医科大学附属医院消化内科
炎症性肠病(inflammatory bowel disease, IBD)包括克罗恩病(Crohn’s disease, CD)和溃疡性结肠炎(ulcerative colitis, UC),是一种慢性、复发性、非特异性肠道炎性疾病,目前发病机制尚不清楚,可能与肠道菌群失调、黏膜免疫障碍、环境因素和易感基因等有关。谷胱甘肽S-转移酶(GST)在人体内广泛存在,除了有促进毒物代谢的作用,还可以保护DNA免受损伤、促进DNA修复[1]。研究[2]表明,谷胱甘肽S-转移酶M1(GSTM1)基因型患者对化学致癌物质的解毒能力下降,增加了个体对肿瘤及其他疾病的易感性。本研究实时评估GSTM1基因型与IBD易感性的关系。
1 资料与方法
1.1检索策略检索PubMed、SinoMed、万方、维普、知网电子数据库。检索关键词:谷胱甘肽S-转移酶M1(GSTM1);基因多态性(polymorphism);炎症性肠病(inflammatory bowel disease)。检索时间:建库至2017年7月。
1.2纳入与排除标准纳入标准:(1)公开发表全文;(2)文章研究GSTM1基因型与IBD相关性;(3)病例对照研究,队列研究,横断面研究。排除标准:(1)重复发表的文献;(2)研究未设立对照组。
1.3数据提取及质量评估阅读文献,提取文献基本特征,包括:作者,发表年份,国家,人种,疾病类型,病例组和对照组样本量及GSTM1基因型分布。通常,使用Hardy-Weinberg平衡(HWE)分析3种及以上单核苷酸基因多态性的分布,但我们的研究涉及两种基因型,因此,Hardy-Weinberg平衡不被用于本研究质量的评估。使用Newcastle-Ottawa量表(NOS)[3]评价文献质量,7~9分为高质量文献,4~6分为中等质量,<4分为低质量文献。
1.4统计学分析采用Review Manager 5.3对纳入文献进行Meta分析。对文献进行异质性检验,P≥0.1,I2≤50%,表明异质性较低,使用固定效应模型。如果P<0.1,I2>50%,表明异质性较高,使用随机效应模型。为寻找潜在的异质性来源,我们对研究对象进行亚组分析及敏感性分析。计数资料采用OR作为分析统计量,并计算其95%CI,使用Begg漏斗图评估发表偏倚,P<0.05为差异有统计学意义。
2 结果
2.1文献基本特征共13篇文献纳入研究,包括20个研究,其中8项研究了GSTM1基因型与CD的相关性,12项研究了GSTM1基因型与UC的关系。IBD患者共4 179例,对照组8 198例。此外,13项研究涉及高加索人种,5项涉及亚洲人种,非洲人种2项。纳入研究均使用NOS评估文献质量(见表1)。
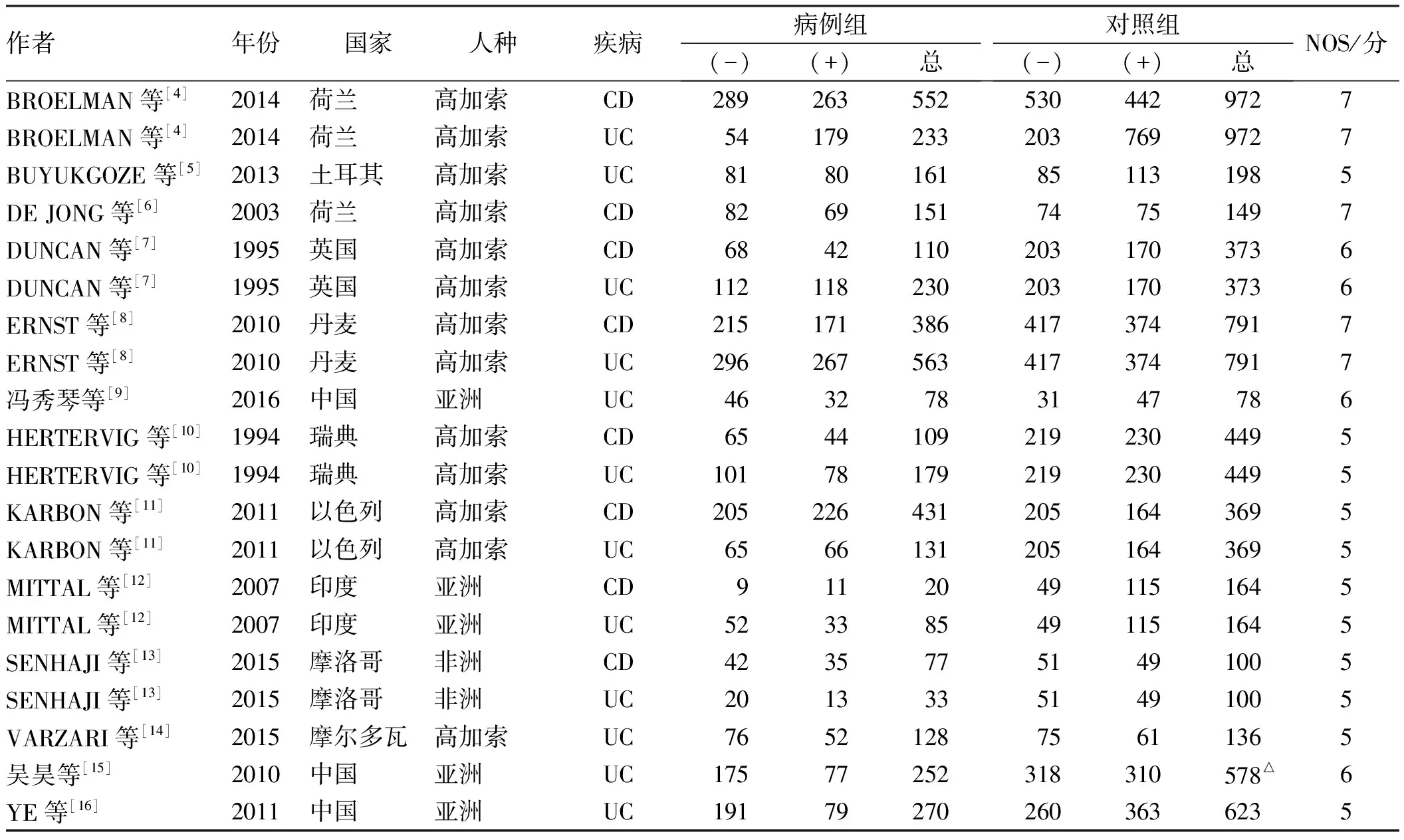
表1 纳入文献基本特征及GSTM1基因型分布Tab 1 Basic characteristics of inclusive literature and distribution of GSTM1 genotypes
注:Δ:该文献未给出后部分患者未纳入原因。
2.2Meta分析结果
2.2.1 GSTM1基因型与IBD的相关性分析:对20项研究进行Meta分析,异质性检验显示研究之间存在异质性(I2=82%,P<0.00001),采用随机效应模型。Meta分析表明,GSTM1(-)基因型与IBD有相关性,GSTM1(-)基因型发生IBD的风险是GSTM1(+)基因型的1.32倍(OR=1.32, 95%CI: 1.09~1.61,P<0.05,见图1)。
2.2.2 人种亚组分析结果:为分析异质性来源,我们对纳入文献进行了人种亚组分析。异质性检验显示,高加索人(I2=46%)和亚洲人(I2=58%)的异质性较前均降低,高加索人使用固定效应模型进行分析,亚洲人使用随机效应模型分析。结果表明,在亚洲人中,GSTM1(-)基因型患IBD的风险是GSTM1(+)基因型的2.57倍(OR=2.57, 95%CI: 1.85~3.57,P<0.05)。但高加索人GSTM1(-)基因型与IBD无明显相关性(OR=1.03, 95%CI: 0.94~1.12,P>0.05)(见表2、图2~3)。
2.2.3 疾病亚组分析结果:将纳入研究分为UC组和CD组,对两组进行亚组分析。异质性检验显示,UC组(I2=86%)与CD组(I2=54%)均存在异质性,两组均采用随机效应模型进行分析。结果显示,携带GSTM1(-)基因型发生UC的几率是GSTM1(+)基因型的1.46倍(OR=1.46, 95%CI: 1.09~1.96,P<0.05)。但CD与GSTM1(-)基因型无显示明显相关性(OR=1.09, 95%CI: 0.90~1.33,P>0.05)(见表2、图4)。
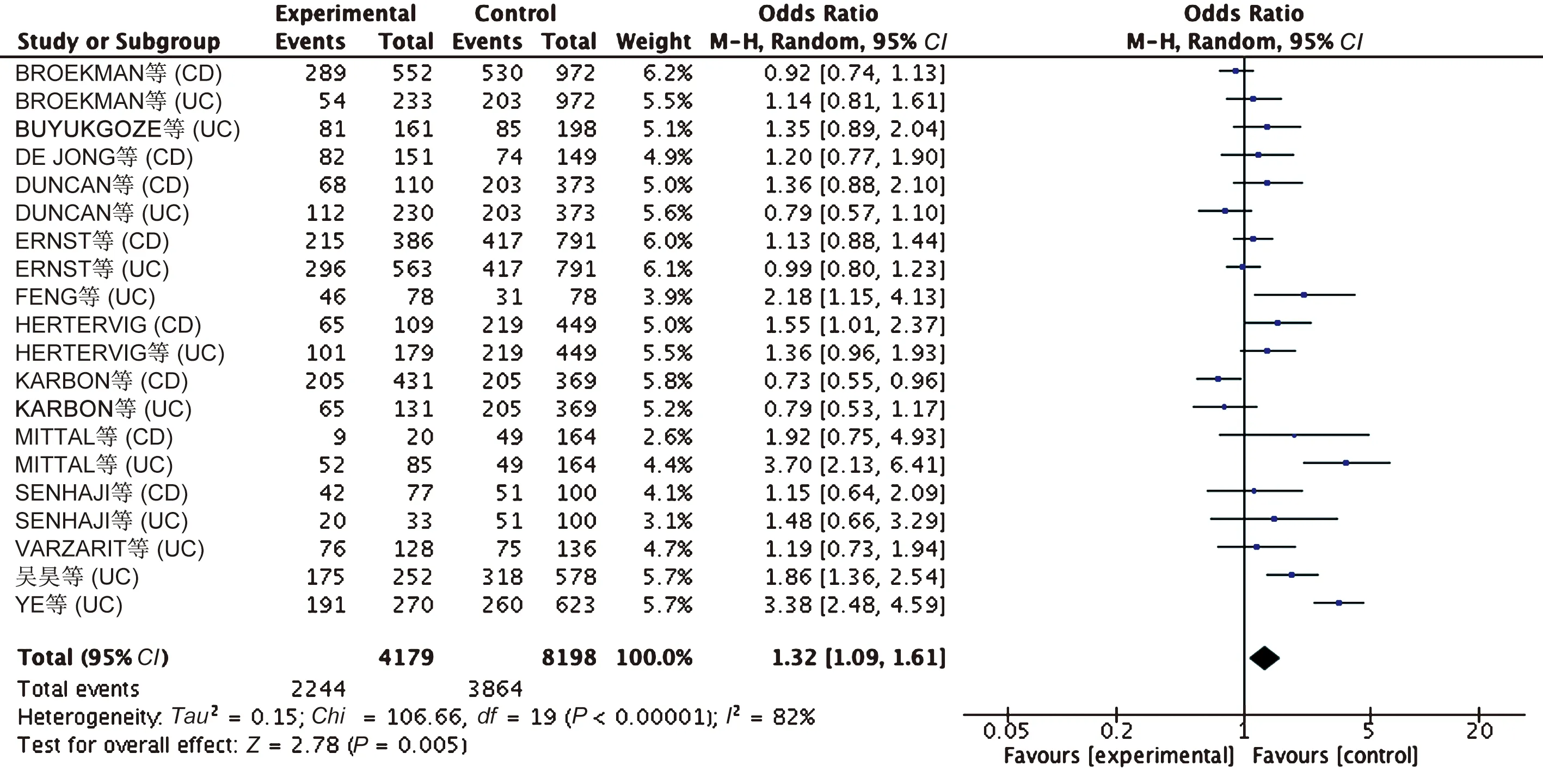
图1 GSTM1基因型与IBD相关性Meta分析森林图Fig 1 Analysis of forest map of GSTM1 genotypes and IBD
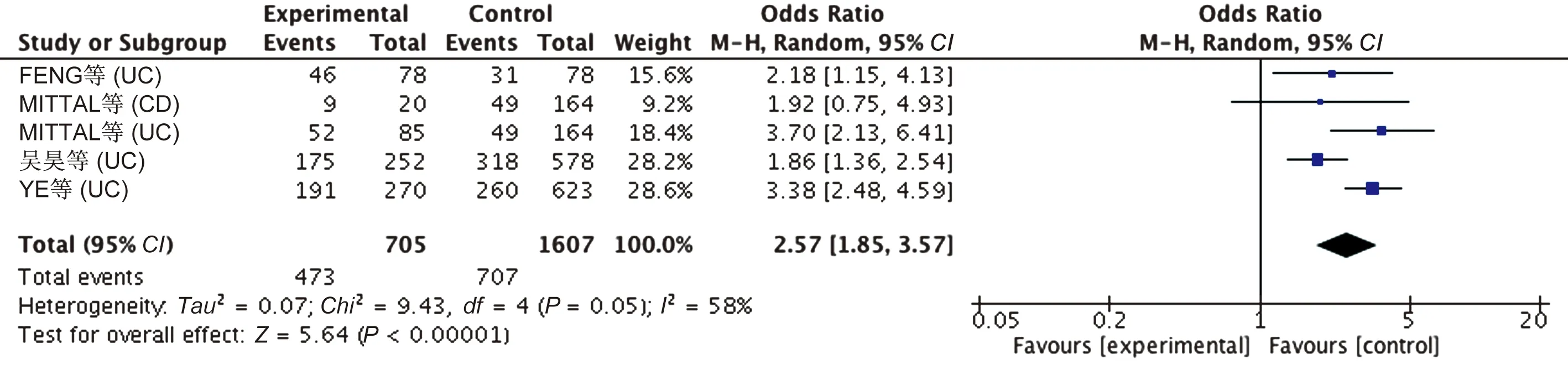
图2 GSTM1基因型与亚洲人种分析的森林图Fig 2 Analysis of forest map of GSTM1 genotype and Asian ethnic
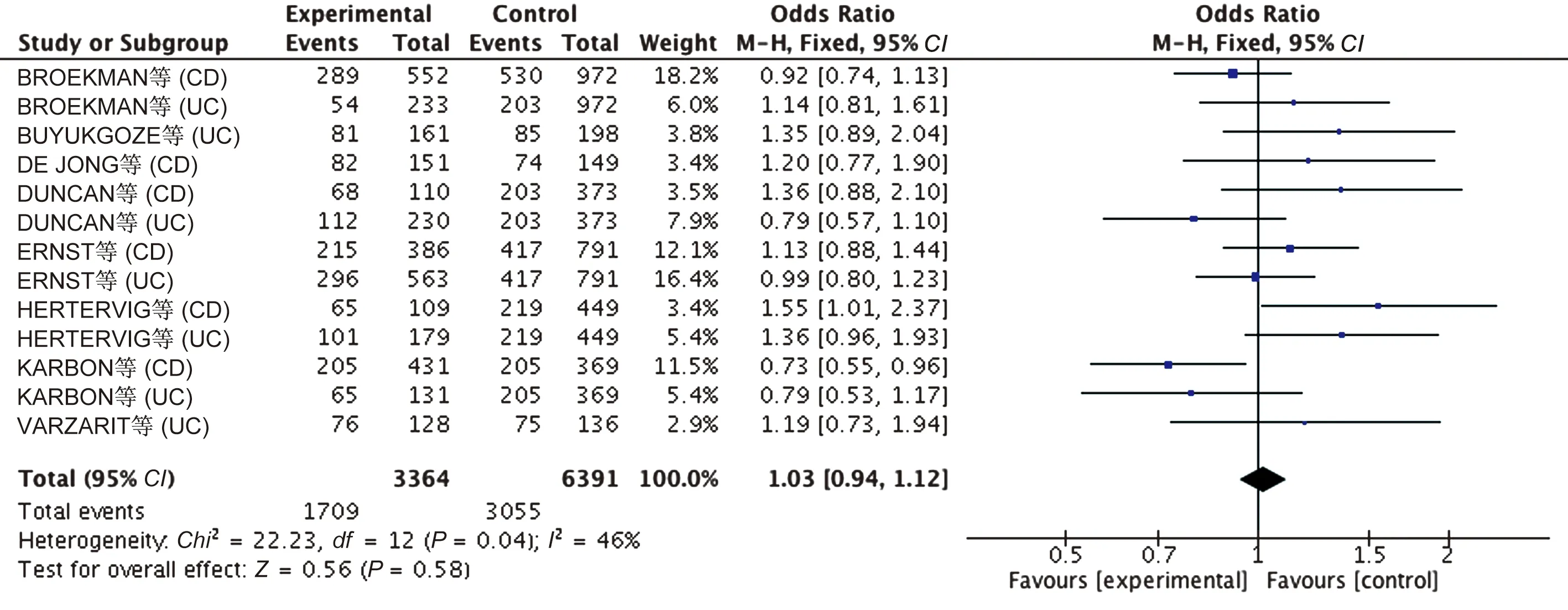
图3 GSTM1基因型与高加索人种分析的森林图Fig 3 Analysis of forest map of GSTM1 genotype and Caucasian race
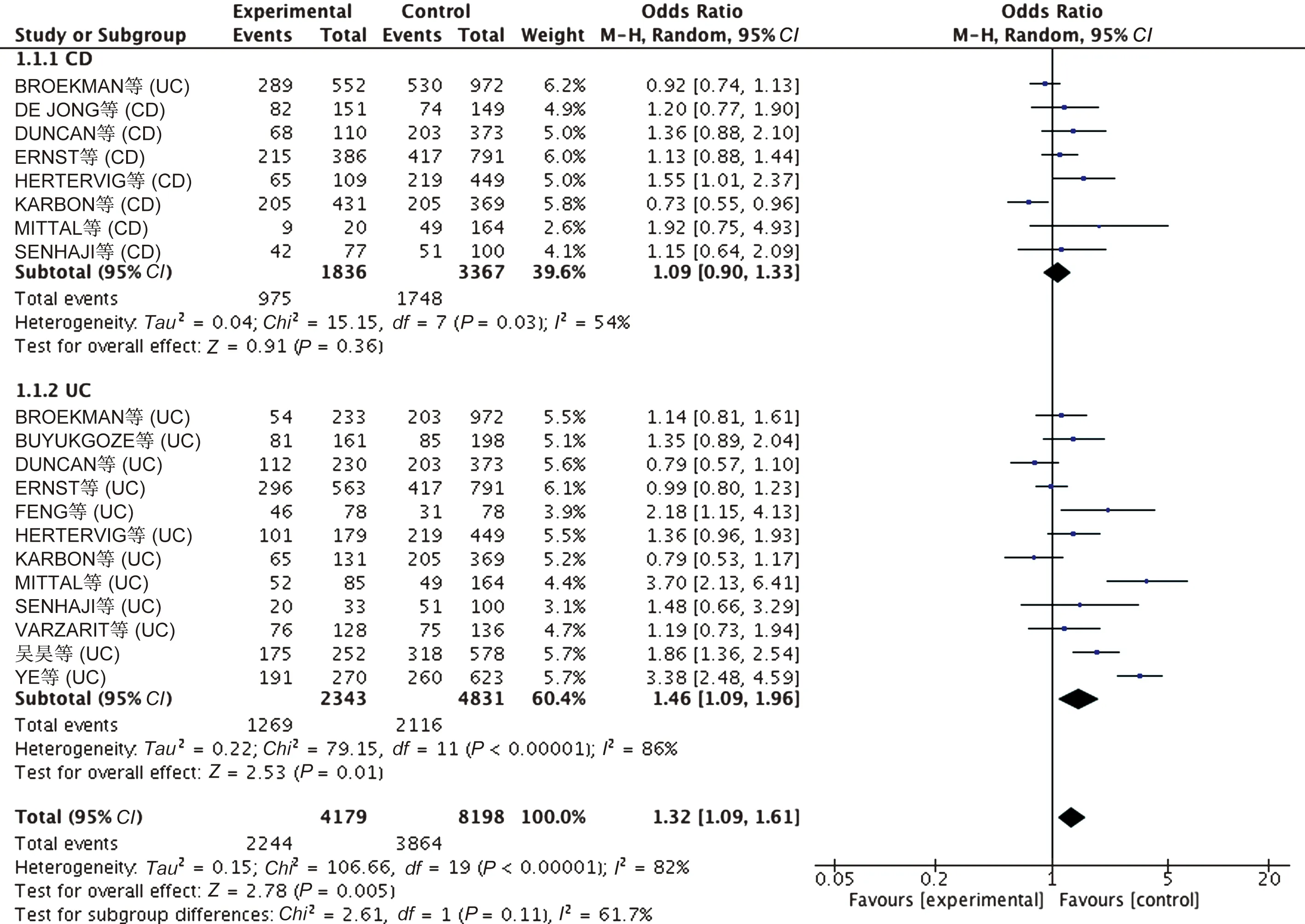
图4 GSTM1基因型与疾病亚组分析的森林图Fig 4 Analysis of forest map of GSTM1 genotype and disease subgroup
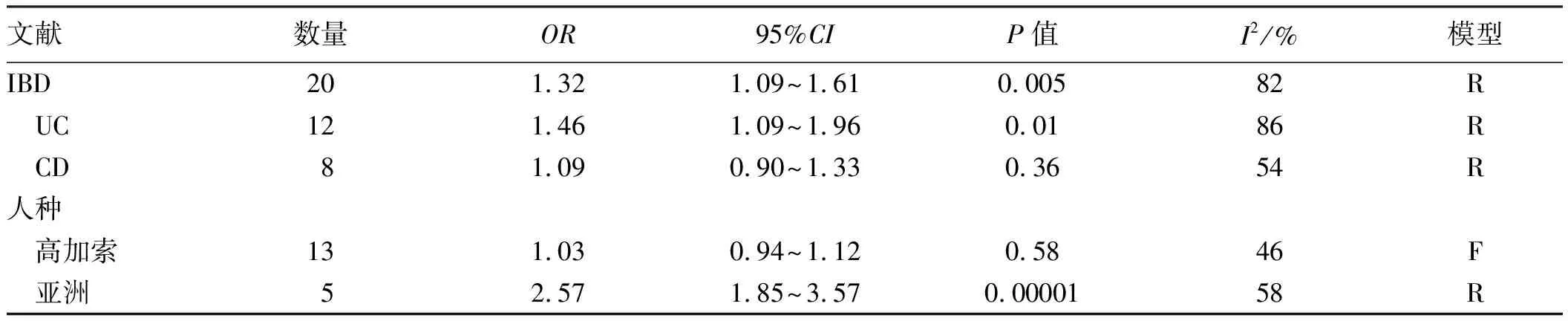
表2 Meta分析结果Tab 2 The results of Meta analysis
注:F:固定效应模型;R:随机效应模型。
2.3敏感性分析采用RevMan 5.3进行敏感性分析,每排除一个病例-对照研究,就重新进行Meta分析以确保总体结果不受任何一项研究的较大影响。结果显示,在人种亚组分析中,去除吴昊等[15]的研究后,亚洲人群与对照组之间的异质性明显降低(I2=0)(见图5)。
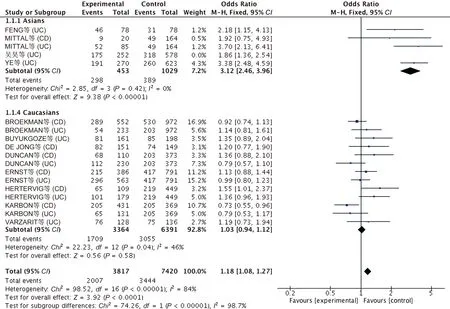
图5 敏感性分析的森林图Fig 5 Analysis of forest map of sensitivity
2.4发表偏倚采用RevMan 5.3绘制Begg图,评估纳入研究是否存在潜在的发表偏倚。结果显示,纳入研究的Begg图呈基本对称的倒漏斗形,未见明显的不对称,提示研究的发表偏倚较小(见图6)。
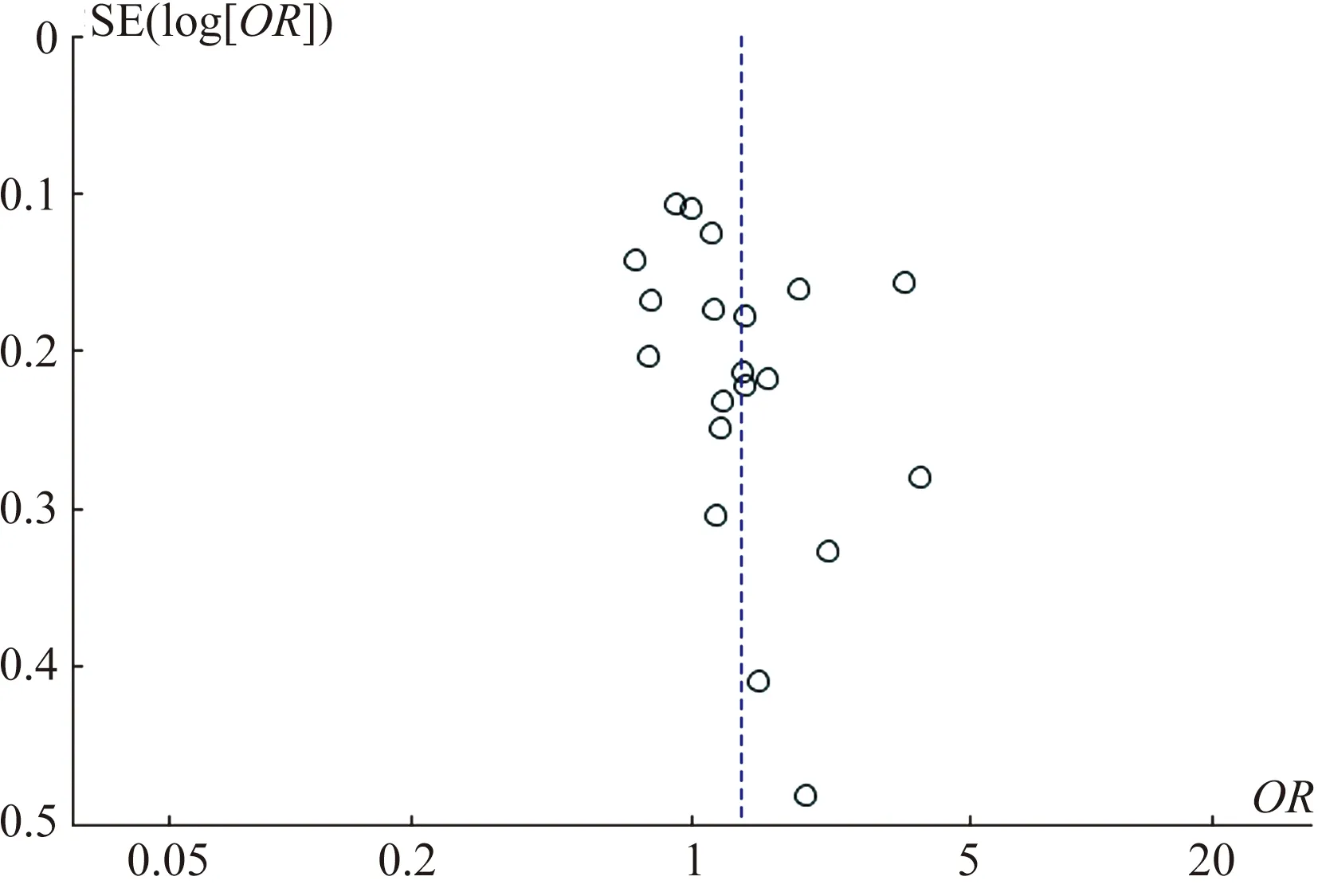
图6 Meta分析的Begg图Fig 6 Begg diagram of Meta-analysis
3 讨论
谷胱甘肽是人体中具有去除潜在毒物能力的保护性化合物,GST催化谷胱甘肽与各种活性氧结合并进行生物转化,维持氧化-抗氧化系统平衡[17-18],GST是细胞抗损伤和抗癌的重要解毒系统[19]。GST根据其基因序列分为A、M、T、P亚家族,且酶系中许多成员的编码基因具有多态性,GST基因多态性可导致代谢酶活性变化从而导致疾病。研究表明,GSTM1基因多态性与肺癌[20]、乳腺癌[21]、胃癌[22]、骨髓增生异常综合征[23]等疾病有关。GSTM1定位于染色体1p13.3,GSTM1基因型在不同人种中的分布存在波动[24],非洲人为33%~48%,亚洲人为33%~63%,欧洲人为39%~62%,美国人为23%~62%。
IBD是一种多病因、异常免疫介导的肠道疾病,具有终身复发倾向,与环境、感染、遗传、免疫等因素有关。寻找与IBD发生、发展相关的基因标记十分重要,将有助于我们筛选具有遗传易感性的群体,并采取有针对性的措施来预防IBD的发生。本研究分析了不同种族和IBD不同疾病类型的GSTM1基因型的分布。结果表明,GSTM1(-)基因型增加了IBD的易感性。此外,在亚洲人群中,GSTM1(-)基因型与IBD的发生显著相关,但在高加索人群中,GSTM1(-)基因型与IBD患者无明显相关性。同时,本研究表明,GSTM1(-)基因型与UC的发生密切相关,但与CD的发生无明显相关性。
当然,我们的研究有很多局限性。首先GSTM1、GSTA1、GSTP1、GSTT1基因多态性并不是IBD的独立危险因素。单独GST基因型无法正确预测GST的确切表达;其次,IBD的发生和发展涉及基因-基因和基因-环境的相互作用,因此我们需要更多的高质量研究来验证此次Meta分析的结果。
[1] DEAKIN M, ELDER J, HENDRICKSE C, et al. Glutathione S-transferase GSTT1 genotypes and susceptibility to cancer: studies of interactions with, GSTM1 in lung, oral, gastric and colorectal cancers [J]. Carcinogenesis, 1996, 17(4): 881.
[2] PINARBASI H, SILIG Y, GURELIK M. Genetic polymorphisms of GSTs and their association with primary brain tumor incidence [J]. Cancer Genet Cytogenet, 2005, 156(2): 144-149. DOI: 10.1016/j.cancergencyto.2004.04.020.
[3] STANG A. Critical evaluation of the Newcastle-Ottawa scale for the assessment of the quality of nonrandomized studies in meta-analyses [J]. Eur J Epidemiol, 2010, 25(9): 603-605. DOI: 10.1007/s10654-010-9491-z.
[4] BROEKMAN M M, BOS C, MORSCHE R H, et al. GST Theta null genotype is associated with an increased risk for ulcerative colitis: a case-control study and meta-analysis of GST Mu and GST Theta polymorphisms in inflammatory bowel disease [J]. J Hum Genet, 2014, 59(10): 575-580. DOI: 10.1038/jhg.2014.77.
[5] BUYUKGOZE O, OSMANOGLU N, ARSLAN S, et al. Association of the CYP1A1*2A, GSTT1 null, GSTM1 null, mEPHX*3, and XRCC1-399 genetic polymorphisms with ulcerative colitis [J]. Int J Colorectal Dis, 2013, 28(4): 593. DOI: 10.1007/s00384-012-1507-6.
[6] DE JONG D J, VAN DER LOGT E M, VAN SCHAIK A, et al. Genetic polymorphisms in biotransformation enzymes in Crohn’s disease: association with microsomal epoxide hydrolase [J]. Gut, 2003, 52(4): 547-551.
[7] DUNCAN H, SWAN C, GREEN J, et al. Susceptibility to ulcerative colitis and Crohn’s disease: interactions between glutathione S-transferase GSTM1 and GSTT1 genotypes [J]. Clin Chim Acta, 1995, 240(1): 53-61.
[8] ERNST A, ANDERSEN V, ØSTERGAARD M, et al. Genetic variants of glutathione S-transferases μ, θ, and π display no susceptibility to inflammatory bowel disease in the Danish population [J]. Scand J Gastroenterol, 2010, 45(9): 1068-1075. DOI: 10.3109/00365521.2010.490594.
[9] 冯秀琴, 刘婉薇. 谷胱甘肽转硫酶基因多态性与溃疡性结肠炎的关系[J]. 宁夏医学杂志, 2016, 38(11): 988-990. DOI: 10.13621/j.1001-5949.2016.11.0988.
FENG X Q, LIU W W. The relationship of glutathione S-transferase gene polymorphism and ulcerative colitis [J]. Ningxia Med J, 2016, 38(11): 988-990. DOI: 10.13621/j.1001-5949.2016.11.0988.
[10] HERTERVIG E, NILSSON A, SEIDEGÅED J. The expression of glutathione transferase mu in patients with inflammatory bowel disease [J]. Scand J Gastroenterol, 1994, 29(8): 729-735.
[11] KARBAN A, KRIVOY N, ELKIN H, et al. Non-Jewish Israeli IBD patients have significantly higher glutathione S-transferase GSTT1-null frequency [J]. Dig Dis Sci, 2011, 56(7): 2081-2087. DOI: 10.1007/s10620-010-1543-4.
[12] MITTAL R D, MANCHANDA P K, BID H K, et al. Analysis of polymorphisms of tumor necrosis factor-alpha and polymorphic xenobiotic metabolizing enzymes in inflammatory bowel disease: study from northern India [J]. J Gastroenterol Hepatol, 2007, 22(6): 920-924. DOI: 10.1111/j.1440-1746.2006.04538.x.
[13] SENHAJI N, KASSOGUE Y, FAHIMI M, et al. Genetic polymorphisms of multidrug resistance gene-1 (MDR1/ABCB1) and glutathione S-Transferase gene and the risk of inflammatory bowel disease among moroccan patients [J]. Mediators Inflamm, 2015: 248060. DOI: 10.1155/2015/248060.
[14] VARZARI A, DEYNEKO I V, TUDOR E, et al. Polymorphisms of glutathione S-transferase and methylenetetrahydrofolate reductase genes in Moldavian patients with ulcerative colitis: Genotype-phenotype correlation [J]. Meta Gene, 2015, 7: 76-82. DOI: 10.1016/j.mgene.2015.12.002.
[15] 吴昊, 郑波, 王建嶂, 等. 谷胱甘肽转硫酶基因多态性与浙江汉族溃疡性结肠炎易感性的相关性[J]. 世界华人消化杂志, 2010, 18(26): 2780-2784.
WU H, ZHENG B, WANG J Z, et al. Relationship between genetic polymorphisms of glutathione S-transferase genes and susceptibility to ulcerative colitis in Zhejiang Han population [J]. World Chinese Journal of Digestion, 2010, 18(26): 2780- 2784.
[16] YE X, JIANG Y, WANG H, et al. Genetic polymorphisms of glutathione S-transferases are associated with ulcerative colitis in central China [J]. Cell Biochem Biophys, 2011, 60(3): 323-328. DOI: 10.1007/s12013-011-9154-z.
[17] AGRAWAL D, SHAJIL E M, MARFATIA Y S, et al. Study on the antioxidant status of vitiligo patients of different age groups in Baroda [J]. Pigment Cell Res, 2010, 17(3): 289-294. DOI: 10.1111/j.1600-0749.2004.00149.x.
[18] HAZNECI E, KARABULUT A B, ÖZTÜRK M D, et al. A comparative study of superoxide dismutase, catalase, and glutathione peroxidase activities and nitrate levels in vitiligo patients [J]. Int J Dermatol, 2005, 44(8): 636-640. DOI: 10.1111/j.1365-4632.2004.02027.x.
[19] FEDETS O M. Structure and functions of glutathione transferases [J]. Ukr Biochem J, 2014, 86(3): 23-32.
[20] NAZAR-STEWART V, VAUGHAN T L, STAPLETON P, et al. A population-based study of glutathione S-transferase M1, T1 and P1 genotypes and risk for lung cancer [J]. Lung Cancer, 2003, 40(3): 247-258.
[21] AMBROSONE C B, SWEENEY C, COLES B F, et al. Polymorphisms in glutathione S-transferases (GSTM1 and GSTT1) and survival after treatment for breast cancer [J]. Cancer Res, 2001, 61(19): 7130-7135.
[22] TAMER L, ATEN A, ATEC, et al. Glutathione S-transferase M1, T1 and P1 genetic polymorphisms, cigarette smoking and gastric cancer risk [J]. Cell Biochem Funct, 2005, 23(4): 267-272. DOI: 10.1002/cbf.1148.
[23] TSABOURI S E, GEORGIOU I, ALAMANOS I, et al. Increased prevalence of GSTM (1) null genotype in patients with myelodysplastic syndrome: a case-control study [J]. Acta Haematologica, 2000, 104(4): 169-173. DOI: 10.1159/000046510.
[24] COTTON S C, SHARP L, LITTLE J, et al. Glutathione S-transferase polymorphisms and colorectal cancer: a HuGE review [J]. Am J Epidemiol, 2000, 151(1): 7-32.

