蛇床子RPS13蛋白的体外表达及生物信息学分析
王晨凯 ,廖 怡, 刘苗苗, 储 俊, 吴家文
(1. 安徽中医药大学 新安医学教育部重点实验室 科研实验中心, 合肥 230038; 2.安徽中医药大学 药学院, 合肥230012; 3. 安徽道地中药材品质提升协同创新中心,合肥 230038; 4. 安徽省中医药科学院,合肥 230038)
蛇床子[Cnidiummonnieri(L.) Cuss.]是一年生的蛇床属植物蛇床的成熟果实,主要生长于中国安徽、江苏、河北等地[1]。通常人们熟知蛇床子具有抗病毒、抑菌、治疗支气管哮喘等作用。近几年来,随着国内外学者对中药蛇床子化学成分和主要药理作用方面的研究发现香豆素类物质蛇床子素具有预防骨质疏松、治疗虫咬皮炎、抗心律失常等作用,蛇床子挥发油具有杀虫、止痒作用[2]。随着对中草药研究的深入,人们对蛇床子抗诱变和抗肿瘤新作用的认识越来越深[3]。
核糖体(ribosomal) 是由小亚基和大亚基两个重要部分结合而成,是完成蛋白质多肽链生物合成的重要生物细胞器,其中核糖体蛋白(RP)是核糖体的主要成分,核糖体蛋白S13位于核糖体小亚基的头部,一方面与大亚基连接,另一方面其伸展的C末端与P位点tRNA反密码子区域结合,同时维持核糖体的小亚基与tRNA之间以及大亚基与小亚基之间的动态相互作用,直接参与细胞内蛋白质生物合成的调节[4]。
核糖体蛋白除了在基因转录、细胞增殖、发育和凋亡等过程中发挥重要作用,王辉等研究还发现在肝癌、胃癌、食管癌等肿瘤组织中核糖体蛋白的一部分基因表达有变化[5];Wang等[6]报道在多个胃癌细胞株(AGS、MKN45、SGC7901和KATO Ⅲ)中RPL15的表达量较高;在直肠癌中S8、S12、S18、S24、L13a、L18、L28、L32和L35a与其他正常细胞的核糖体蛋白表达量相比表达量降低,而S11和L7的表达量明显上调[7]。姜润德等[8]研究发现红细胞色素氧化酶亚单位1(COX1)、核糖体蛋白S27和L27在人鼻咽癌耐药细胞中表达被抑制;也有研究报道胃癌某些细胞中的RPS13蛋白表达量明显比亲本细胞高[9],这些研究都表明了核糖体蛋白存在的差异性表达与人类的某些特别的疾病相关。
目前有些物种的rps13已经被研究,如非洲爪蟾蜍rps13被发现存在于染色体11P区域,有5个内含子和6个外显子[10];在植物研究中发现RPS13的突变体导入植物体内,由于细胞分裂减少可延缓根的生长、导致叶的生长紊乱、开花期的推后[11]。
本研究首次对蛇床子核糖体蛋白S13进行基因克隆、体外原核细胞中表达以及系统的生物信息学分析,为进一步纯化其蛋白、研究其结构功能,进而探索通过基因调控的方法干扰肿瘤细胞的发生、发展奠定理论基础。
1 材料与方法
1.1 材料及试剂
野生蛇床子[Cnidiummonnieri(L.) Cuss.]植株采自安徽省桐城市,由安徽中医药大学王德群教授鉴定。把新鲜的蛇床子根、茎、叶分开并用灭菌的蒸馏水清洗干净,分别快速置于液氮中冷冻后放入-80℃冰箱保存备用。E.Z.N.A Plant RNA Kit (50)购于OMEGA Bio-Tek (USA)公司;RevertAid First Stand cDNA Synthesis Kit购于Thermo公司;AxyPrepTMDNA Gel Extraction Kit (50 prep)和AxyPrepTMPlasmid miniprep kit (50 prep)购于Axygen公司;两种限制酶(NdeI /XhoI)购于TOKOYO;DNA连接酶购于TaKaRa公司;蛋白鉴定中的一抗(AM1010a)购于ABGENT公司;二抗(W402B)购于Promega公司。
1.2 菌株和载体质粒
实验用的大肠杆菌克隆菌株E.coliDH5α、表达菌株E.coliBL21、pET22b(+)质粒、pMD19-T载体质粒均购于TaKaRa生物公司。
1.3 方法
1.3.1总RNA的提取
取2 g左右的蛇床子根、茎、叶组织,液氮中充分研磨,采用E.Z.N.A Plant RNA Kit (50)提取总RNA,在260 nm和280 nm处测紫外吸收值并计算OD260/OD280的比值检测RNA的浓度,用琼脂糖凝胶电泳检测RNA的完整性,之后将其用于cDNA的合成。
1.3.2 引物设计及RT-PCR
基于蛇床子转录组测序的Unigene数据,我们根据目的基因rps13 ORF的cDNA序列,利用Primer 5.0 软件设计一对特异性扩增引物:
rps13-S:5′-CATATGATGGGGCGTATGCACAGTCATG-3′(28 bp);rps13-A:5′-CTCGAGGGCAACCAGGGTGCTGGC-3′(24 bp)下划线部分分别为NdeI和XhoI的酶切位点。以纯度高、完整性好的总RNA为模板,用RevertAid First Stand cDNA Synthesis Kit进行cDNA的合成。以合成的cDNA为模板,采用25 μL反应体系进行PCR扩增:2.5 μL的10×PCR buffer缓冲液、13.2 μL ddH2O、1 μL 10 mmol/L dNTP混合物、上下游引物各1.5 μL、2 μL cDNA、0.8 μLTaqDNA聚合酶。反应条件:94℃预变性3 min;94℃变性30 s,45℃退火30 s,72℃延伸1 min,共10个循环;随后94℃变性30 s,55℃退火45 s,72℃延伸1 min,共25个循环;最后72℃再延伸10 min。用1%的琼脂糖凝胶电泳检测PCR产物,用AxyPrepTMDNA Gel Extraction Kit (50 prep)回收目的基因。
1.3.3构建克隆重组质粒pMD19-rps13
按照以下条件将纯化得到的目的基因与克隆载体pMD19-T连接:pMD19-T载体1.5 μL,回收的rps13基因ORF片段11 μL,10×T4DNA连接酶缓冲液1.5 μL,T4DNA 连接酶1 μL,16℃连接8 h。利用热激法将其转化至E.coliDH5α感受态细胞,37℃培养10~12 h,挑单克隆扩大培养。用AxyPrepTMPlasmid miniprep kit (50 prep)提取纯化质粒,采用双酶切(NdeI/XhoI )和菌液PCR进行鉴定。挑选鉴定正确的重组质粒pMD19-rps13送至生工基因有限公司进一步测序鉴定。
1.3.4构建表达重组质粒pET22b(+)-rps13
表达载体pET22b(+)和重组质粒pMD19-rps13用NdeI和XhoI双酶切后,将酶切后的pET22b(+)载体和胶回收纯化的rps13目的基因于16℃连接8 h后转化至E.coliDH5α感受态细胞中,挑单个克隆扩大培养。进行菌落PCR初筛后用AxyPrepTMPlasmid miniprep kit (50 prep)提取纯化质粒,进行NdeI /XhoI双酶切进一步鉴定,再将鉴定正确的重组质粒pET22b(+)-rps13送至生工基因进行测序。
1.3.5重组质粒pET22b(+)-rps13诱导表达
将pET22b(+)-rps13重组质粒转化到E.coliBL21感受态细胞,于37℃的恒温箱中培养,1 h后倒置平板,再培养7~8 h,挑单克隆到15 mL LB培养基中扩大培养,以220 r/min转速,37℃培养至OD600为0.6~0.8时,加IPTG诱导表达蛋白(IPTG浓度分别为0、0.1、0.5和1 mmol/L),在16℃、25℃和37℃,150 r/min的条件下分别诱导24、8及6 h,再离心并收集菌体。因为目的蛋白的等电点为10.31,我们选择pH 6.5的PBS缓冲液(含500 mmol/L NaCl) 2 mL重悬菌体,用超声将菌体打碎使其裂解,高速离心 (13 000 r/min 10 min) 后将沉淀和上清进行分离。
1.3.6Western-blotting鉴定
将高速离心后分离的上清蛋白进行聚丙烯酰胺凝胶电泳(浓缩胶:75 V,30 min;分离胶:115 V,90 min)转硝酸纤维素膜(400 mA以上恒定电流,电压15 V,20~30 min),脱脂奶粉(2 g/40 mL PBST)封闭1.5 h,PBST洗3次(10 min /次),加一抗(His-tag单克隆小鼠抗体)4℃孵育过夜,10 h后用PBST洗3次(10 min/次),再加二抗(兔抗)25℃避光孵育2 h,PBST洗3次(10 min/次),暗室曝光。
1.3.7生物信息学分析
利用在线翻译序列软件(http://www.bio-soft.net/sms/index.html)将rps13碱基序列翻译成氨基酸序列;采用Protparam、Motif Scan软件分析RPS13蛋白理化特性、基元及其结构域;通过DNAMAN 6.0.3.99软件对同源性最高的5种不同植物RPS13蛋白氨基酸序列进行多序列比对;利用MEGA 5.0[12]、Clustalx 1.83[13]、Swiss Model、PyMOL[14]软件进行系统发育进化树关系分析、二级结构预测以及三级结构模拟。
2 结果与分析
2.1 rps13 ORF克隆和鉴定
2.1.1总RNA的提取及鉴定
E.Z.N.A Plant RNA Kit (50)提取后的蛇床子总RNA用紫外分光光度计测得样品的OD260/OD280比值为1.90,表明所提RNA纯度较高。琼脂糖凝胶电泳和凝胶成像仪检测结果显示凝胶中有3条明亮可见的条带从上至下分别为28 S、18 S及5 S (图未显示),表明所提RNA具有较好的完整性。
2.1.2rps13 ORF的克隆
以富集的mRNA为模板进行逆转录反应,再以逆转录产物cDNA为模板利用目的基因rps13的一对引物进行PCR扩增,经琼脂糖凝胶电泳和凝胶成像仪检测显示接近500 bp处有一条清晰的目的条带(图未显示),与目的基因rps13长度一致。
2.1.3RPS13表达载体的构建及鉴定
回收纯化目的基因条带,然后将其与pMD19-T 载体连接,转化至E.coliDH5α感受态细胞,扩大培养后抽提质粒,利用菌液PCR和双酶切重组质粒鉴定正确后再用NdeI /XhoI两种酶对重组质粒pMD19-rps13和表达载体pET22b(+)进行双酶切,将纯化的rps13 ORF目的基因片段和酶切后的pET22b(+)载体连接,转化至E.coliDH5α感受态细胞,涂板培养,挑取平板上的单个克隆进行扩大培养,再进行菌液PCR和双酶切鉴定,目的基因条带位置正确(图未显示)。进一步将阳性重组质粒送至生工进行序列测定,测序结果与蛇床子转录组测序得到的序列完全一致,表明此表达重组载体构建成功。 蛇床子RPS13的ORF序列已经提交至GenBank数据库中,其登录号为KY608745。
2.1.4rps13 ORF表达及Western-blotting分析
将测序正确的重组质粒pET22b-rps13转化至E.coliBL21感受态细胞中,加入不同浓度的IPTG (0、0.1、0.5和1 mmol/L)分别在16℃、25℃和37℃进行诱导24、8 和6 h,超声破碎收集上清进行Western-blotting分析,暗室曝光后膜上显示在分子质量15~25 ku之间有一条明显的目的蛋白条带,结果(如图1-A)与RPS13蛋白理论分子质量17 ku相符,表明重组质粒在E.coliBL21中成功表达,利用AIC.ALphaView.exe和Graph Pad Prism.6软件对蛋白表达量进行统计学分析(如图1-B),可以看出,37℃条件下0.1 mmol/L IPTG诱导蛋白表达量最高。蛇床子RPS13蛋白体外成功表达为进一步研究这一类蛋白的结构和功能奠定了基础。

图1 RPS13蛋白表达分析
A为RPS13蛋白表达的结果,1、2、3、4分别为在16℃和0、0.1、0.5和1 mmol/L诱导表达的目的蛋白上清;5、6、7、8分别为在25℃和0、0.1、0.5和1 mmol/L诱导表达的上清目的蛋白;9、10、11、12分别为在37℃和0、0.1、0.5和1 mmol/L诱导表达的上清目的蛋白。B为A中RPS13蛋白表达量统计学分析, *代表在相同条件RPS13蛋白表达量最高
2.2 生物信息学分析结果
2.2.1rps13序列分析
rps13的cDNA序列经翻译软件(http://www.bio-soft.net/sms/index.html)翻译成氨基酸序列,结果(如图2)所示该目的基因的读码框(ORF)长度为456 bp,编码151个氨基酸。
电子标签数据由128个比特组成。标签被激活后,对数据进行编码,加同步帧头,形成数据帧信号,采用反向散射原理(ASK调制信号)将数据发送给读写器。标签的数据编码形式如图2a和b所示。同步帧头形式如图2c所示。标签返回数据速率为10 kbps;返回整个数据帧,包括帧头,需13 ms。
2.2.2RPS13蛋白理化特性分析
采用Protparam(http://web.expasy.org/cgi-bin/protparam/protparam)软件分析表明RPS13蛋白分子结构式C772H1272N226O207S4,分子质量约为17 ku,理论等电点为10.31。其中赖氨酸(k)有20个,含量最高,占13.2%;有31个正电荷氨基酸(其中Arg 11个,Lys 20个),12个负电荷氨基酸(其中Asp 6个,Glu 6个),这与该蛋白偏碱性的等电点一致;该蛋白属于不稳定型蛋白,溶液不稳定指数为51.09,高于阈值40。此蛋白在280 nm处的摩尔消光系数为18 450 M-1·cm-1,0.1%浓度(1 g/L)的Abs为1.075。

图2 rps13氨基酸序列和ORF序列
2.2.3RPS13蛋白基元、结构域和定位分析
通过Motif Scan在线软件(http://hits.isb-sib.ch/cgi-bin/motif_scan/)分析RPS13编码蛋白,显示其含有不同基元(motif),包含一个酪蛋白激酶II磷酸化位点位于P29-V32,2个N-蛋白质豆蔻酰化位点分别位于G10-S15和G44-Q49,一个蛋白激酶C磷酸化位点在T132-K134,在K93-K107 和K93-D110位置含有2个双向核定位信号 (表1)。采用PSORT在线软件(http://psort.hgc.jp/form.html)预测rps13定位于线粒体基质的概率为10%,位于核中的概率为91.9%。

表1 RPS13蛋白基元及结构域分析
2.2.4RPS13的同源蛋白比对及进化树分析
2.2.5RPS13蛋白二级结构分析
通过MEGA 5.0和Clustalx 1.83软件将5个不同物种的核糖体蛋白S13进行了比对(图4),最上面显示的是蛇床子RPS13基于Swiss Model预测的二级结构,预测结果表明RPS13有6个α 螺旋结构(29S~43K、46T~58H、70K~79G、85P~105N、108D~132T、142E~151A)和1个转角(TT)结构(137P~138V)。
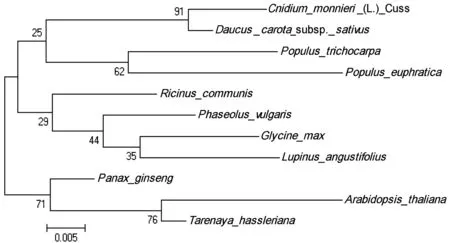
图3 蛇床子RPS13与其他物种的RPS13同源进化树分析
该进化树是采用MEGA5.0软件中的邻接法(NJ)并用 Bootstrap 统计分析进行了100次重抽样构建;0.005 代表遗传距离

图4 蛇床子RPS13的二级结构示意图及与其他物种RPS13的比对
红底白字示意的是相同的氨基酸;白底红字示意的是性质相似的氨基酸;黑字显示的是不同的氨基酸
2.2.6RPS13蛋白三级结构预测及分析
利用Swiss Model软件基于蛋白质数据库(PDB)中5k0y蛋白三级结构得到了蛇床子RPS13的三级结构模型[15-17](图5)。蛇床子RPS13蛋白的N端和C端各有3个α 螺旋自成一簇,这两簇α 螺旋通过长的无规卷曲连接,C末端的α 螺旋也通过长的无规卷曲与邻近的α 螺旋相连,这与RPS13动态连接大小亚基,以及其伸展的C末端动态连接P位点tRNA反密码子区域的功能是一致的。
3 结论与展望
以前人们对核糖体蛋白功能的认识仅局限于蛋白质的生物合成过程,近些年来发现核糖体蛋白可能与人类一些特殊疾病有关。1990年,Fisher等[18]报道位于Y染色体和X染色体上的rps4缺失是造成Turner综合征的重要原因;Denis等[19]研究发现rps13不但在结肠肿瘤细胞中高表达,还在其他增殖较快的细胞中高表达。

图5 蛇床子RPS13蛋白三级结构模型
本文主要通过基因重组技术对蛇床子rps13 ORF的cDNA片段进行了克隆,体外表达了RPS13蛋白,并对其蛋白理化性质和结构进行了分析。将蛇床子RPS13氨基酸序列与胡萝卜、人参、毛茛、大豆、拟南芥、羽扇豆、山杨、醉蝶花、蓖麻和菜豆10个不同物种的RPS13氨基酸序列进行了同源性比较,发现它们的同源性都超过90%,表明蛇床子RPS13蛋白在不同的物种间具有较高的保守性;另外,蛇床子RPS13与胡萝卜的RPS13同源性最高,这与植物的种属关系是一致的,蛇床子别称野胡萝卜子,与胡萝卜同属伞形科植物。RPS13位于核糖体小亚基的头部,它动态地连接核糖体大小亚基,并通过它的碳末端与核糖体大亚基P位点tRNA反密码子区域结合。三级结构预测整个RPS13分子由一条长的无规则卷曲连接两个α 螺旋簇,C末端的α 螺旋通过更长的无规卷曲与邻近的α 螺旋相连,这与RPS13维持核糖体大亚基与小亚基之间的动态连接以及调节翻译周期中的tRNA运动的功能是高度一致的[20]。
本研究为进一步研究RPS13蛋白的生物学功能、探索rps13调控肿瘤细胞的发生发展提供了理论和实验依据。
[1]向仁德, 韩 英. 中药蛇床子的研究概况[J]. 天然产物研究与开发, 1993,5(4): 57-61.
[2]董 欣. 中药蛇床子的研究概况[J]. 吉林中医药, 2006, 26(3):58-59.
[3]周则卫, 刘培勋. 蛇床子化学成分及抗肿瘤活性的研究进展[J]. 中国中药杂志, 2005, 30(17): 1309-1313.
[4]CUKRAS A R, GREEN R. Multiple effects of S13 in modulating the strength of intersubunit interactions in the ribosome during translation[J]. Journal of Molecular Biology, 2005, 349(1): 47-59.
[5]王 辉, 刘伟利. 核糖体蛋白基因表达与肿瘤的关系[J]. 生理科学进展, 2007, 38(4): 376-379.
[6]WANG H, ZHAO L N, LI K Z, et al. Overexpression of ribosomal protein L15 is associated with cell proliferation in gastric cancer[J]. BMC Cancer, 2006, 6: 91-98.
[7]KASAI H, NADANO D, HIDAKA E, et al. Differential expression of ribosomal proteins in human normal and neoplastic colorectum[J]. Journal of Histochemistry & Cytochemistry, 2003, 51(5): 567-574.
[8]姜润德, 张立新, 岳 文, 等. 人鼻咽癌顺铂耐药细胞系(CNE2/DDP)的建立及耐药相关基因的筛选[J]. 癌症, 2003, 22(4):337-345.
[9]郭雪艳, 时永全, 翟惠虹, 等. 人核糖体蛋白S13的原核表达、纯化及多克隆抗体的制备[J]. 细胞与分子免疫学杂志, 2007, 23(4): 363-366.
[10]CHEN M S, LASZLO A. Unique features of Chinese hamster S13 gene relative to its human and Xenopus analogs[J]. DNA and Cell Biology, 1999, 18(6):463-470.
[11]ITO T, KIM G T, SHINOZAKI K. Disruption of an Arabidopsis cytoplasmic ribosomal protein S13-homologous gene by transposon-mediated mutagenesis causes aberrant growth and development[J]. The Plant Journal: for Cell and Molecular Biology, 2000, 22(3): 257-264.
[12]TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739.
[13]JEANMOUGIN F, THOMPSON J D, GOUY M, et al. Multiple sequence alignment with Clustal X[J]. Trends in Biochemical Sciences, 1998, 23(10): 403-405.
[15]ARNOLD K, BORDOLI L, KOPP J, et al. The SWISS-MODEL workspace: a web-based environment for protein structure homology modelling[J]. Bioinformatics, 2006, 22(2): 195-201.
[16]BIASINI M, BIENERT S, WATERHOUSE A, et al. SWISS-MODEL: modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research, 2014, 42(W1): 252-258.
[17]KIEFER F, ARNOLD K, KUNZLI M, et al. The SWISS-MODEL Repository and associated resources[J]. Nucleic Acids Research, 2009, 37(S1): D387-D392.
[18]FISHER E M, BEER-ROMERO P, BROWN L G, et al. Homologous ribosomal protein genes on the human X and Y chromosomes: escape from X inactivation and possible implications for Turner syndrome[J]. Cell, 1990, 63(6): 1205-1218.
[19]DENIS M G, CHADENEAU C, LECABELLEC M T, et al. Over-expression of the S13 ribosomal protein in actively growing cells[J]. International Journal of Cancer, 1993, 55(2): 275-280.
[20]DEL C M, OFENGAND J, MALHOTRA A. Crystal structure of the catalytic domain of RluD, the only rRNA pseudouridine synthase required for normal growth of Escherichia coli[J]. RNA, 2004, 10(2): 231-239.

