人HNRNPA1基因及蛋白质的生物信息学分析
马素珍,刘丹丹,张方方,潘晓丽,刘胜利,朱艳琴
(河南中医药大学 基础医学院实验教学中心,河南 郑州 450008)
采用生物信息学的方法分析人HNRNPA1基因的启动子情况及蛋白质的理化性质、信号肽及NLS、亲疏水性、跨膜区域、蛋白质结构、相互作用的蛋白质及GO注释.选择合适软件对HNRNPA1相关信息进行分析.结果显示,预测的HNRNPA1基因存在1个启动子;HNRNPA1蛋白质是由372个氨基酸组成的具有NLS、无跨膜结构的亲水不稳定蛋白质,其等电点为9.17;哺乳动物中氨基酸序列高度保守;二级结构以无规卷曲为主,预测的三级结构经拉曼图分析可信度高;HNRNPA1多定位于细胞核,与RNA的选择性剪接及mRNA运输有关.此外,启动子的甲基化对HNRNPA1表达影响明显,其蛋白质具有NLS、无跨膜结构且不稳定,属于亲水蛋白质,分布于细胞核,对mRNA的成熟具有重要作用.
HNRNPA1;RNA;选择性剪接;生物信息学
HNRNPA1(heterogeneous nuclear ribonucleoprotein A1)是核不均一核糖核蛋白家族中重要的一员,是一种重要的RNA(ribonucleic acid)结合蛋白质,通过调控转录及转录后RNA的剪接修饰调控了mRNA(messenger ribonucleic acid)的成熟及运输过程[1-2].HNRNPA1可以影响多种生物学过程,如细胞的自我更新[3]、蛋白质正常成熟[4]、免疫反应[5]等.HNRNPA1与多种疾病的发生发展具有重要的关系,如HNRNPA1的突变会引起人遗传性包涵体肌病[6],抑制HNRNPA1的表达会通过影响细胞周期而抑制肺腺癌的增殖[7],通过调控雄激素受体的表达影响前列腺癌的发生[8].另外,它还与乳腺癌[9]、胃癌[10]、胰腺癌[11]等肿瘤的发生发展相关.分析HNRNPA1的生物信息学数据,期望对深入研究该蛋白质在机体生长发育、病情发生发展中的作用提供有价值的理论数据支持.
1 材料与方法
1.1 材 料
从蛋白质Uniprot数据库中下载人(P09651)及黑猩猩(A5A6H4)、鼠(P49312)、牛(P09867)、鼠鸟(A0A091KA33)、斑马鱼(F1QS20)的HNRNPA1蛋白质序列;其相应的HNRNPA1基因及人上游的核酸序列由NBCI(National Center for Biotechnology Information)获得.
1.2 方 法
通过NCBI(http://www.ncbi.nlm.nih.gov/)获得基因相关信息;利用Promoter Scan(http://www-bimas.cit.nih.gov/molbio/proscan/)预测启动子及相关转录因子;ProtParam(http://web.expasy.org/protparam)分析HNRNPA1蛋白质的理化性质、蛋白质分子组成、等电点等相关信息;ClustalX2.1和njplot对多物种间HNRNPA1基因及氨基酸进行同源性和系统进化树进行分析;SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP)、核定位序列(nuclear localization sequence,简称NLS) Mapper(http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)和TMHMM(http://www.cbs.dtu.dk/services/TMHMM)分析蛋白质的信号肽、核定位序列以及蛋白质的跨膜结构;ProtScale(http://expasy.org/tools/protscale.html)分析了蛋白质亲/疏水性;SOMPA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_sopma.html)、Swiss-Model(http://swissmodel.expasy.org)和The Structure Analysis and Verification Server(http://services.mbi.ucla.edu/SAVES)对蛋白质的二三级结构及拉曼图进行预测分析;String(http://string-db.org)和Gene Ontology Consortium(http://amigo1.geneontology.org/cgi-bin/amigo/go.cgi)分析与HNRNPA1相互作用蛋白质及其参与的生物学功能.
2 结果与分析
2.1 HNRNPA1基因的启动子及转录因子分析
通过NCBI检索发现HNRNPA1基因位于人12号染色体的长臂1区3带(12q13)上,具有13个外显子.Promoter Scan是基于与真核Pol II启动子序列的同源性来预测目标基因的启动子区域的,对HNRNPA1基因上游2 000 bp和基因前1 000 bp的核酸序列,通过Promoter Scan对该基因的启动子进行预测,得到如表1所示的启动子.
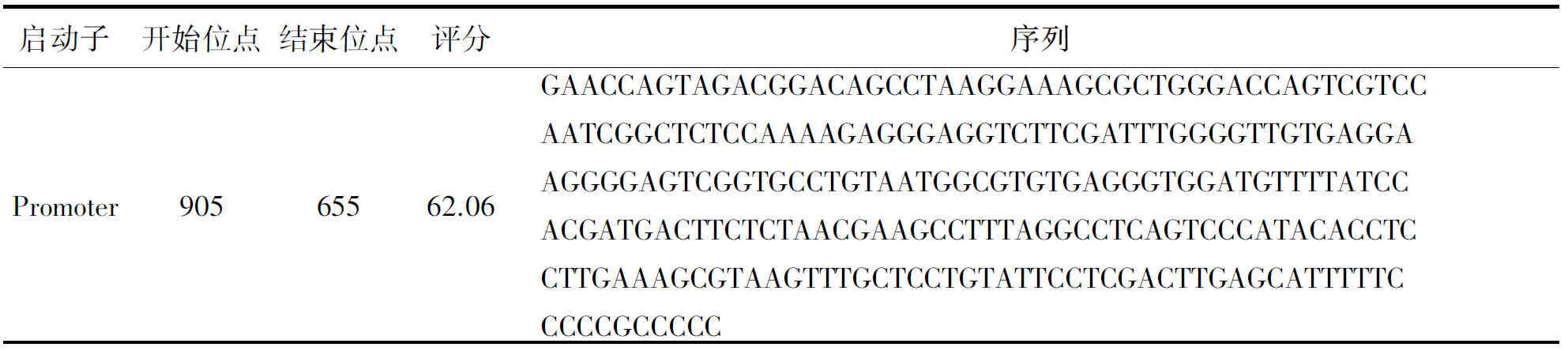
表1 人HNRNPA1基因启动子预测
由表1可知,该启动子位于负链区,即905—655 bp之间.同时得到与该启动子相结合的转录因子如表2所示.

表2 Promoter结合的转录因子
由表2发现,存在大量与甲基化相关的SP-1、AP-2与该启动子结合,这说明DNA的甲基化对该基因的表达调控具有重要的意义.
2.2 人HNRNPA1基因及蛋白质的同源性预测和分析
ClustalX2.1是一种常用的序列比对软件,不仅可以对核酸进行比对,也可以对蛋白质的序列进行比对.它首先对不同来源的序列进行两两比对,构建合理的系统进化树,然后根据已构建的系统进化树从最相近的两条序列开始,逐步引入邻近的序列,直至所有序列都完成比对.通过ClustalX2.1不仅可以完成序列比对,也可以构建相应的系统进化树,进一步对研究目标进行分析.
使用序列比对软件对不同物种来源的HNRNPA1基因进行多重序列比对,通过序列比对发现这些不同物种来源的基因在外显子区域序列相似度较高,而在内含子等非编码区相似度低,如图1所示(显示部分序列).进一步分析由HNRNPA1基因构建的系统进化树发现人与黑猩猩的进化关系最近,与鼠较远,与斑马鱼之间进化距离最远.说明该基因也随着物种的进化不断发生变化.
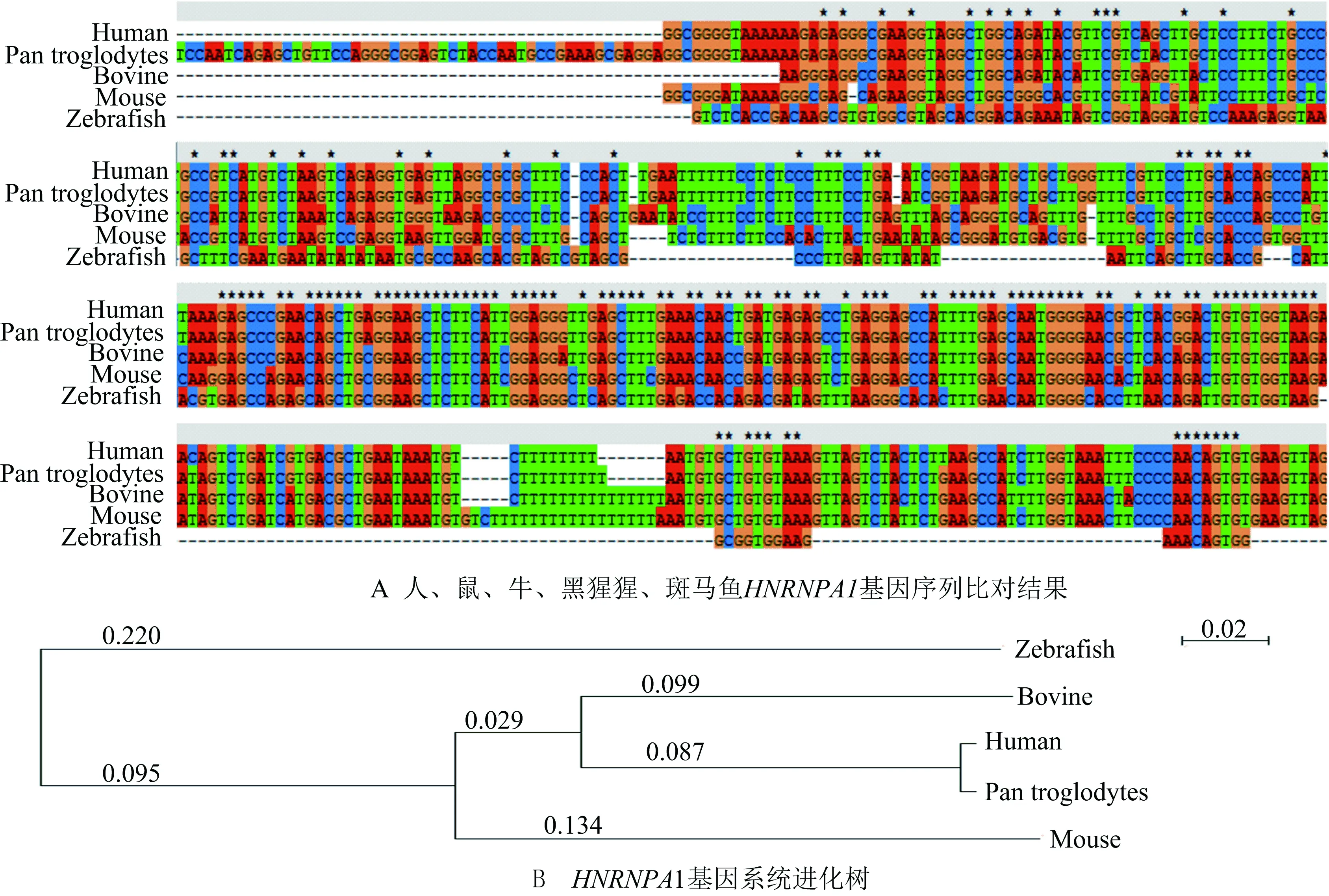
图1 不同物种间HNRNPA1基因的同源性分析
序列比对软件对Uniprot蛋白质数据库中下载的多种物种的蛋白质序列进行多重序列比对,使用可视化分析软件njplot对构建的系统进化树进行分析,得到如图2所示结果.
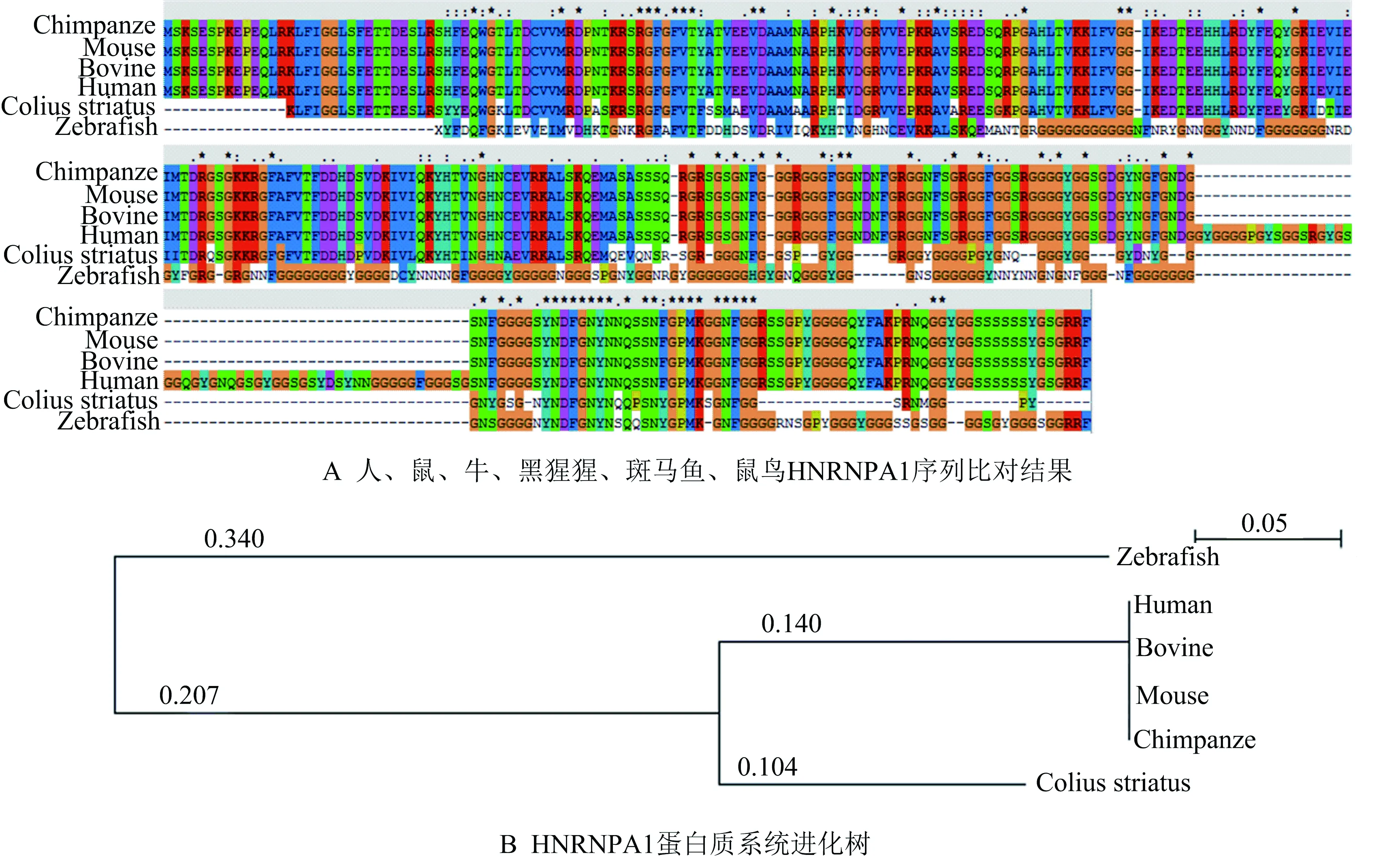
图2 不同物种间HNRNPA1蛋白质的同源性分析
图2的序列比对发现,在人的第305—335位氨基酸突变率极低,保守性强,可能该区域是该蛋白的重要功能区域,这些区域可能与其与DNA和RNA的结合有关.哺乳动物之间该蛋白质的序列相似度极高,但人比其他哺乳动物多了52个氨基酸.分析系统进化树发现,人与黑猩猩及其他哺乳动物之间的进化距离极小,但鸟类和斑马鱼之间的进化距离较大,分别是0.244和0.787,该蛋白质在哺乳动物之间具有极高的保守性.
综合分析HNRNPA1基因和蛋白质序列比对结果和构建的系统进化树可以发现,两者所得出的亲缘关系基本相同,与达尔文进化论基本吻合,但通过基因分析所得出的进化树更加精确,可以计算出人和黑猩猩与牛、小鼠之间的进化距离,而由蛋白质序列构建的进化树则无法将其区分.这是由于基因不仅包括编码区还包括了内含子等非编码区,而这些区域不会改变蛋白质的氨基酸,因此这些区域的突变更容易积累.此外由于每3个碱基构成1个密码子,每个密码子对应1种氨基酸,而密码子又具有简并性,第3个碱基的改变多数情况下不会影响到蛋白质,这种突变在进化过程中也被保存了下来.因此通过核酸序列构建的系统进化树更能反映进化的亲缘关系,也能反映出HNRNPA1蛋白质在人和黑猩猩之间的表达调控及功能上极为相似.
2.3 人HNRNPA1蛋白质的理化性质分析
ProtParam是由瑞士生物信息学中心维护并提供的蛋白质理化分析工具,以检索目的蛋白质的理化性质,并基于这些理化性质分析未知蛋白质的类别,为后续实验提供数据支持.分析人HNRNPA1蛋白质的理化性质发现:HNRNPA1共有372个氨基酸残基;理论等电点为9.17;蛋白质总分子式为C1661H2491N515O551S8,其原子总数为5 226;分子质量为38 746.6;带负电荷氨基酸残基Asp(aspartic acid)和Glu(glutamic acid)占9.68%(36/372),带正电荷氨基酸残基Arg(arginine)和Lys(lysine)占11.56%(43/372);HNRNPA1的不稳定系数为42.00,属于不稳定蛋白质.
2.4 人HNRNPA1蛋白质的亲水性/疏水性预测与分析
ProtScale程序是一种简便的蛋白质亲水性与疏水性分析的工具,该工具提供多种算法,常选择Hphob. / Kyte & Doolittle算法.该算法将不同氨基酸进行赋值,如Ala(alanine)为1.800,Arg为-4.500,Ile(isoleucine)为4.500等,通过分析蛋白质中所有氨基酸疏水值的分布情况,判定蛋白质的亲疏水性.通过分析HNRNPA1蛋白质的亲疏水性发现蛋白质的氨基酸处于正值区的均小于1.5,其中最大值是第61位苏氨酸的1.411.有25个氨基酸的分值小于-2,其中最小值是51位苏氨酸的-3.056,预测分析结果如图3所示.
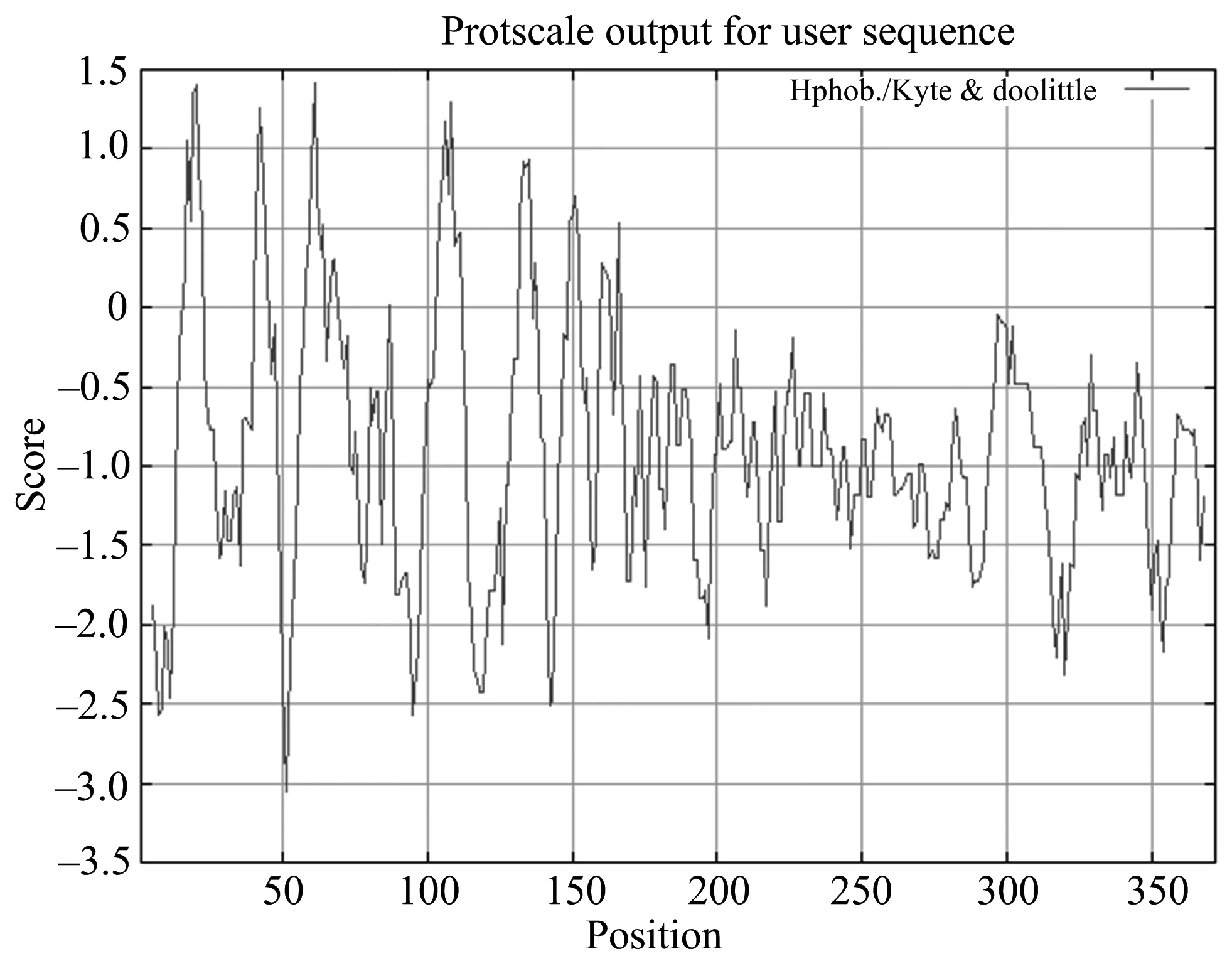
图3 人HNRNPA1亲水性/疏水性分析
由图3可知,ProtScale分析的364个氨基酸(5-368)有87.64%(319个)分布在低分值区,总得分-350.68;2.36%(45个)分布在Score>0区,总得分为28.166.HNRNPA1具有大量的亲水氨基酸,可形成亲水域,属亲水蛋白质.多种亲水结构的存在,有利于该蛋白质在细胞质中进行游离扩散,多种亲水结构的存在可以使其在细胞质或细胞核中以游离状态存在,当行使功能时,迅速与DNA或RNA结合,外部的亲水结构保护了内部的疏水区域.
2.5 人HNRNPA1蛋白质的信号肽预测与分析
SignalP 4.1 Server是根据信号肽位于新合成肽链的N(nitrogen)端,且在完成引导功能后切除的特性而开发的信号肽预测软件,通过对目的肽链前70个氨基酸间潜在酶切位点的预测而推断是否存在信号肽.将HNRNPA1蛋白质氨基酸序列提交到信号肽预测服务器SignalP 4.1 Server,设置Cut-off值为0.450,得到如图4的预测结果.
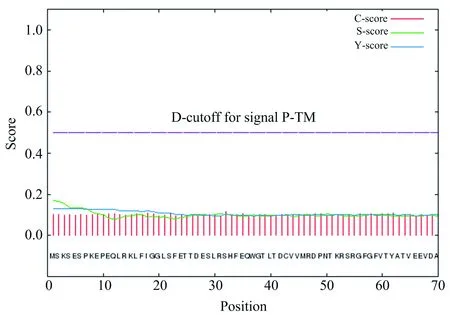
图4 人HNRNPA1信号肽分析
图4中C、Y、S的最大值分别为0.115、0.127、0.171,S-mean、D值分别为0.134、0.131,分析该数据可以得知人HNRNPA1蛋白质无信号肽.HNRNPA1主要在细胞核内参与RNA的形成,不需要进入其他膜性细胞器,但需进入核内,因此进一步通过cNLS Mapper分析其核定位序列.不同氨基酸在cNLS中会有有利或不利的作用,cNLS Mapper给予相应氨基酸或正或负的分数,通过计算每个氨基酸残基对整个NLS活性的贡献而计分,根据不同段的总分与设定阈值进行比较而确定NLS存在与否,该结果可以用来指导设计特异于importin αβ的核输入途径的有效抑制剂.通过分析发现在HNRNPA1蛋白质中存在一段序列为RGSGKKRGFAFVTFDDHDSVDKIVIQKYHTV(140-170)的NLS,其得分值为5.9分,高于设定的阈值(5分)
2.6 人HNRNPA1蛋白质的跨膜区预测与分析
TMHMM是目前蛋白质跨膜区域分析结果可信度最高的软件[12],因此选用TMHMM 2.0,各选项按其默认选项,分析人HNRNPA1蛋白质序列,进行分析后得到如图5的预测结果.
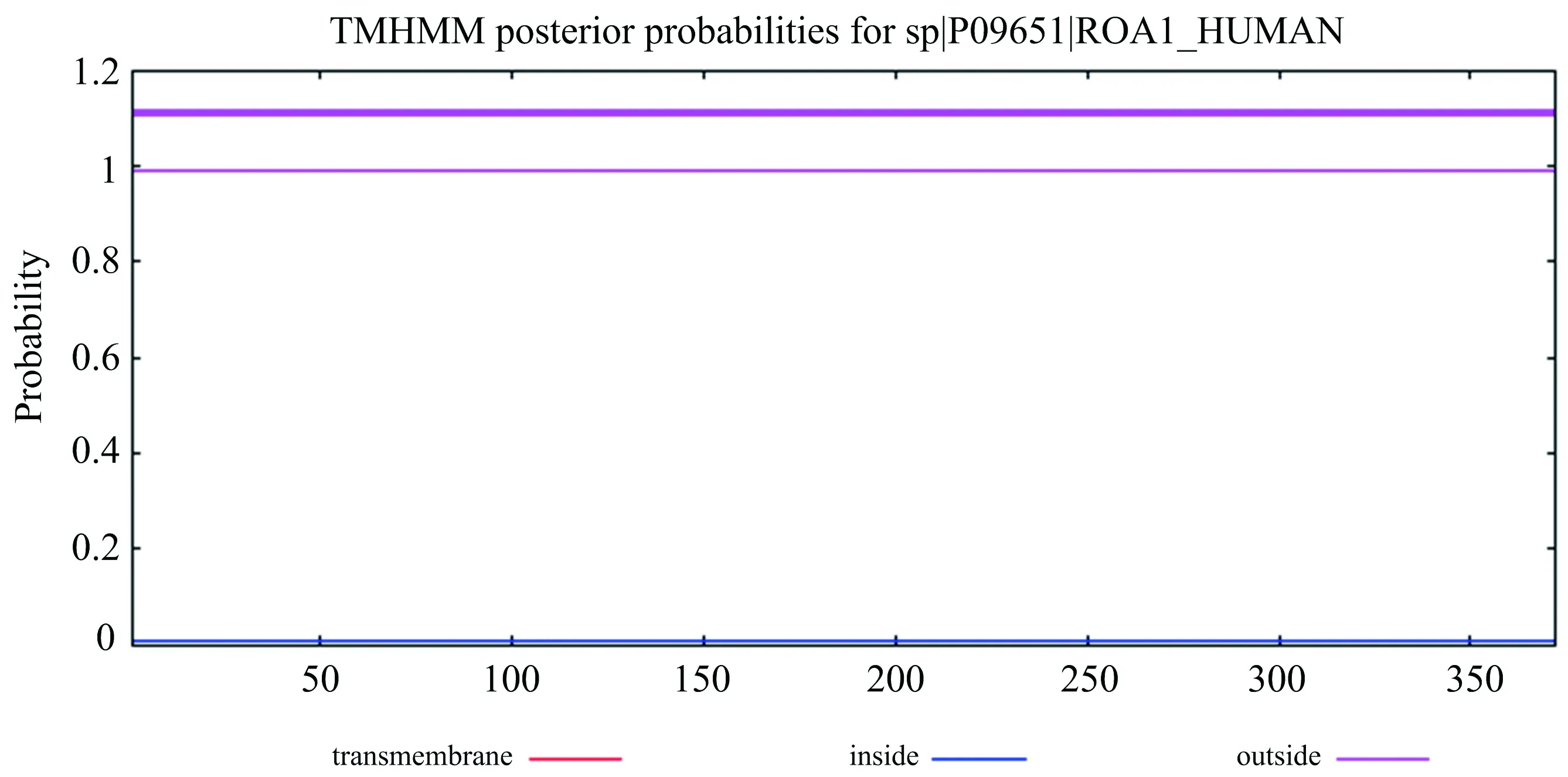
图5 人HNRNPA1跨膜区预测
由图5发现,HNRNPA1不存在跨膜区域,这说明HNRNPA1蛋白质不是跨膜蛋白质.HNRNPA1主要进入细胞核中参与RNA的形成过程,主要作用区域分布在细胞核,因此,HNRNPA1不需要跨膜转运的过程,该蛋白质可能是在细胞质中游离核糖体上合成,不经过内质网和高尔基体修饰运输,而是经由NLS引导,通过核孔复合体直接进入细胞核,参与RNA的形成,因此,该蛋白质无跨膜结构.
2.7 人HNRNPA1蛋白质二级结构的预测与分析
SMOPA是通过已知二级结构的氨基酸数据库,通过自优化的预测方法对目标蛋白质的二级结构进行预测分析的方法.采用SOMPA方法分析HNRNPA1蛋白质所形成的二级结构,构象状态数选择3(Helix,Sheet,Coil),相似性阈值选择8,分析结果如图6所示.

h:α螺旋;e:β折叠;c:无规卷曲.图6 人HNRNPA1二级结构预测
图6的分析发现HNRNPA1具有6个α螺旋(h所示区域),6个β折叠片层(e所示区域),其余大多数处于无规卷曲状态,α螺旋占15.05%(56/372),β折叠占11.29%(42/372),剩余274个氨基酸(73.66%)处于无规卷曲的状态,这与该蛋白质高甘氨酸含量有关.甘氨酸侧链只有一个氢,二面角取值范围较大,不易形成稳定构象.
2.8 人HNRNPA1蛋白质三级结构预测与分析
蛋白质的高级结构决定了其生物学功能,分析蛋白质的高级结构对探索其生物学功能具有重要的指导意义.蛋白质高级结构的预测有串线法、同源建模法和从头预测的方法,常用的预测方法是同源建模法.Swiss-Model是一个全自动化的蛋白质结构同源建模服务器,提交蛋白质序列至Swiss-Model,选择默认选项,通过与数据库中已有的蛋白进行序列比对,选择相似度、覆盖度最高的模板进行建模,得到相应的高级结构的预测结果及相似性波形图,如图7所示.
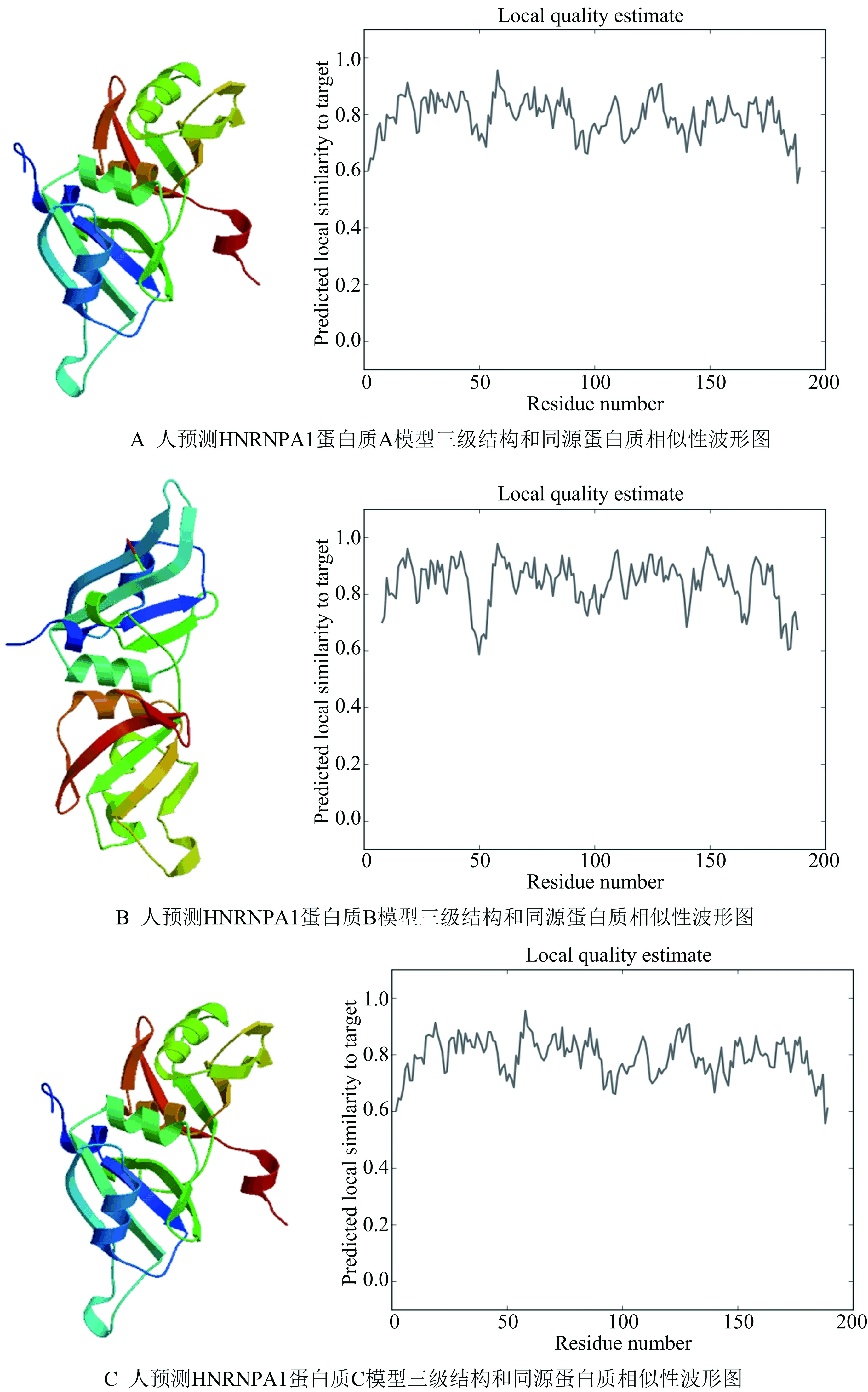
图7 人HNRNPA1三级结构预测结果
图7中,A、C两个结果采用了相同的同源模板,预测结果相似度极高;B结果采用另一模板,但波形图数值波动较大.综合分析序列相似度、覆盖度及与同源蛋白质的相似性波形图可以发现,预测结构A相似性波形图预测值高,较稳定,因此,预测结构A可信度较高.
蛋白质的高级结构由于氨基酸侧链基团大小的区别,对形成的二面角有不同的要求.为了进一步验证结构A的可信度,通过蛋白质拉曼图分析网站The Structure Analysis and Verification Server对预测模型各氨基酸的二面角进行分析,从而判断预测模型的可靠性.通过拉曼图分析得到如图8所示结果.
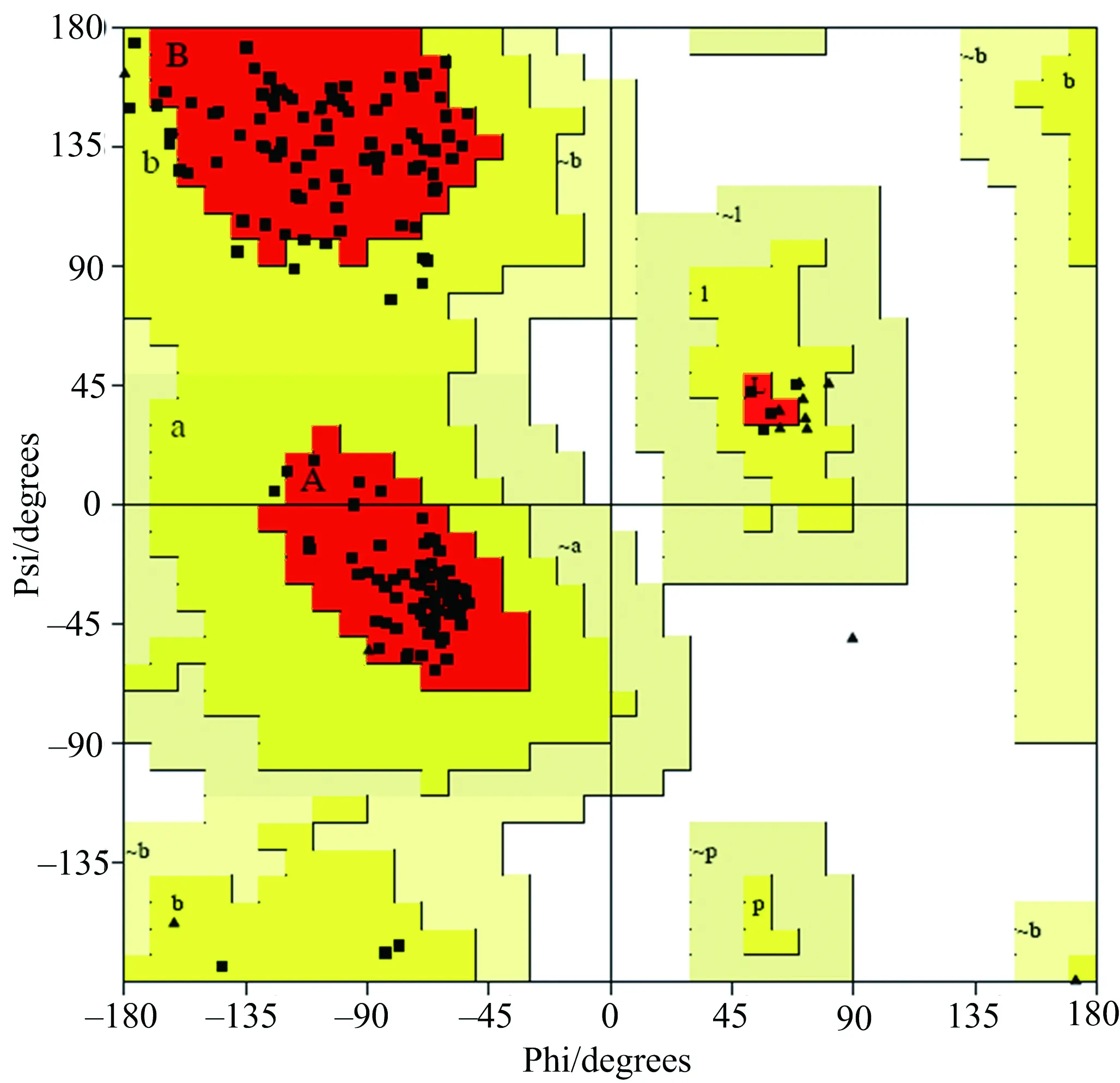
▲:甘氨酸;■:其他氨基酸.图8 人HNRNPA1预测三级结构模型的拉曼图分析
图8所示的预测结构中所涉及的188个氨基酸中147个处于最佳区域,即图中红色区域;19个处于允许区域,即黄色区域,另外还有14个甘氨酸对二面角的要求较低,因此该预测结构形成的二面角稳定可靠.
2.9 人HNRNPA1相互作用蛋白质预测及分析
蛋白质在机体内不能单独完成生物过程,需要与其他蛋白质相互作用才可以正常行使生命过程.String是一个有效的相关功能蛋白质相互作用网络预测服务器,通过该服务器对与HNRNPA1相互作用的蛋白质进行了预测分析,输入蛋白质名称为hnrnpa1,选择生物类型为人类,通过检索分析后对得分高的前10个蛋白质进行了统计介绍,如表3和图9所示.

表3 与人HNRNPA1相互作用可能性较大的10种蛋白质
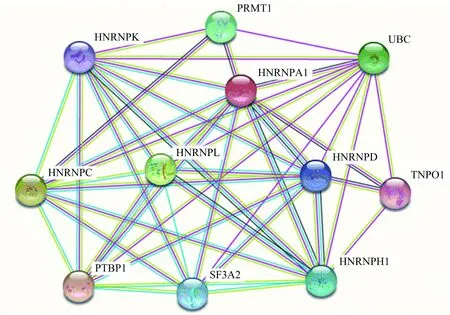
图9 人HNRNPA1蛋白质相互作用预测
与HNRNPA1相互作用较强的蛋白质主要是HNRNP家族的蛋白,另外还有一些mRNA前体形成中参与的剪切因子,这说明HNRNPA1需要与家族中其他成员共同参与mRNA前体的剪接成熟.同泛素C的结合说明HNRNPA1的降解会通过泛素化途径.综合相互作用蛋白质的结果,可进一步说明HNRNPA1进入细胞核后与家族其他成员及mRNA前体形成相关蛋白质结合后共同调控mRNA前体的形成.
2.10 人HNRNPA1的GO注释分析
基因本体(gene ontology,简称GO)可对基因及其产物的细胞组分(cellular component)、生物过程(biological process)和分子功能(molecular function)进行统一的归纳、解释和分析.Gene Ontology Consortium服务器可以对基因本体进行详尽的分析,以“hnrnpa1”为关键词,选择“genes or proteins”条目进行检索,检索后选择人类相对应的GO条目进行分析.表4所示为人HNRNPA1进行基因本体论分析后得到的结果.
从表4所示的细胞组成上看,HNRNPA1分布在整个细胞中,但细胞核中相对较多,在剪接体及剪接体复合物中较多;从分子功能上来看,HNRNPA1主要是与蛋白质和核酸结合,通过与核酸的结合调控mRNA的形成、端粒活性,与蛋白质的结合影响其出入核.
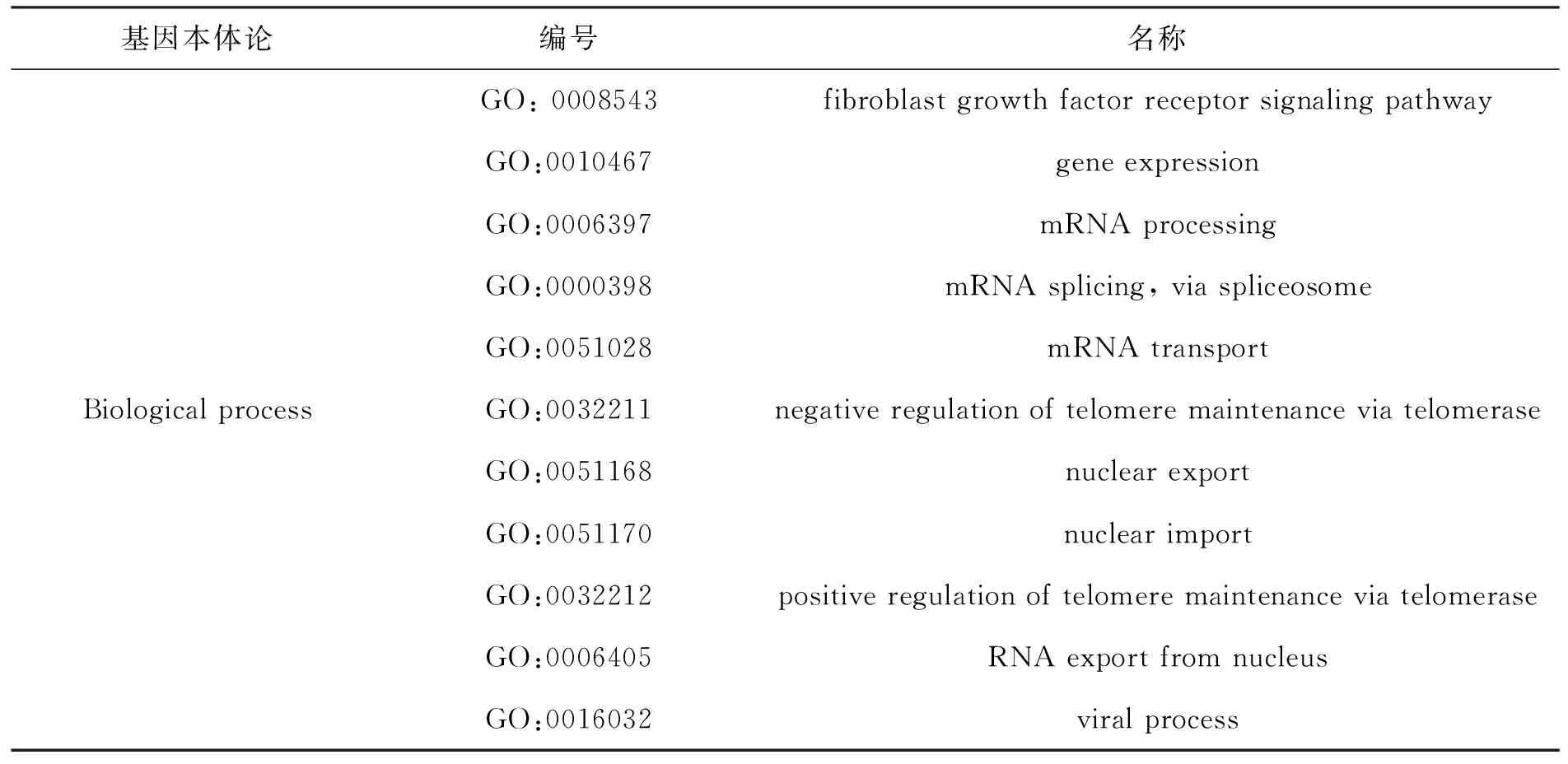
表4 人HNRNPA1 基因注释分析结果
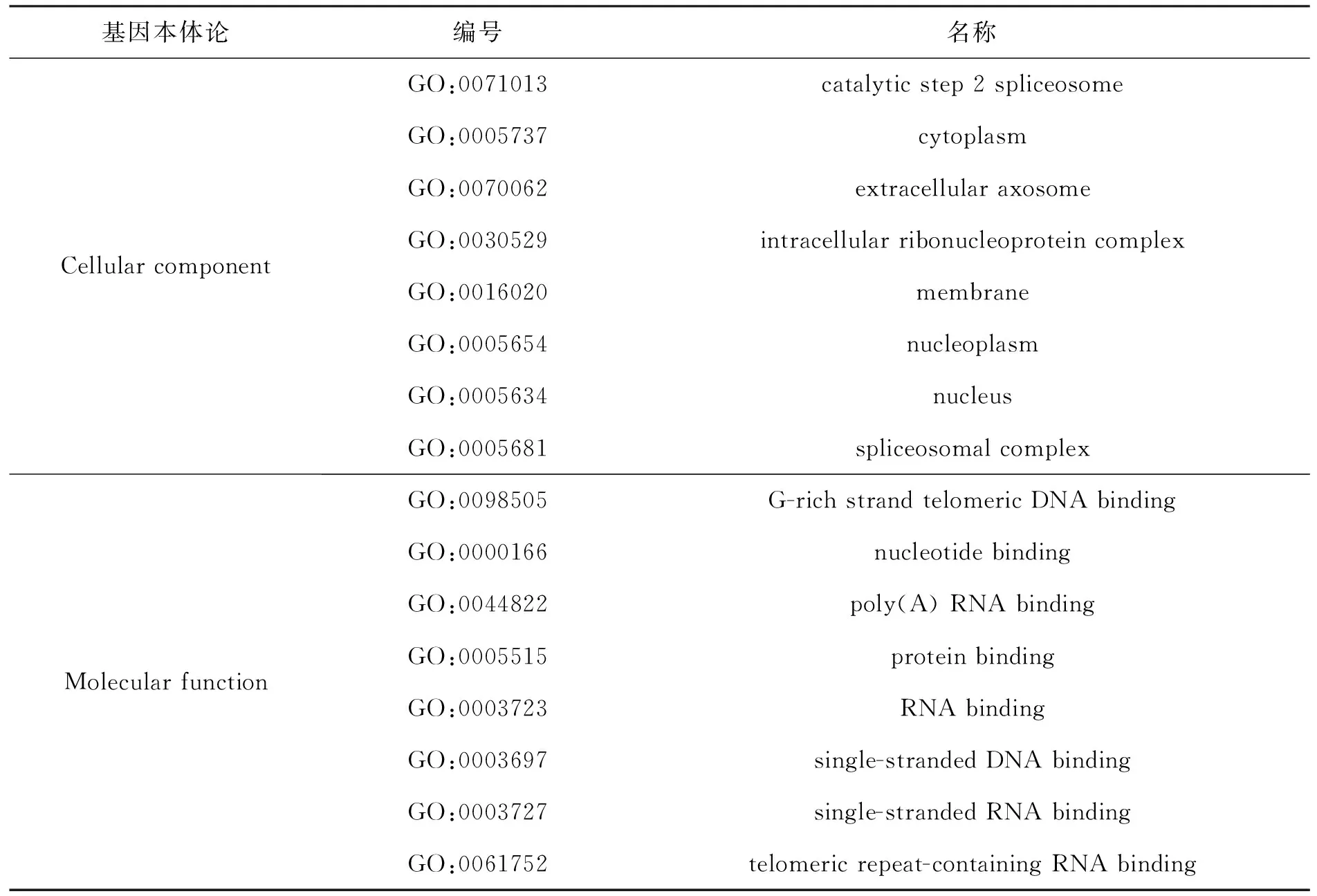
续表4
3 结束语
HNRNPA1作为核不均一核糖核蛋白家族中重要的一员,是RNA结合蛋白中重要的成分[13],与RNA转录及产生hnRNP(heterogeneous nuclear ribonucleoprotein)颗粒有关,参与信使RNA的代谢调控过程,但在RNA的选择剪接过程中与SR蛋白的作用相拮抗[14],HNRNPA1通过与多种蛋白质及DNA、RNA相互作用,对RNA的剪接成熟具有重要的调控作用.近来,越来越多的研究发现HNRNPA1与多种疾病的发生具有重要的关系,除了与乳腺癌、前列腺癌等肿瘤外,与神经退行性疾病的发生也具有重要的关系[15-17],如与额颞叶的变性具有重要的关系[4],在阿尔兹海默症中表达量也会下降[4].因此,对HNRNPA1的深入研究可以为疾病的研究及诊断治疗带来新的思路.
笔者应用生物信息学的方法,对HNRNPA1进行深入的分析,发现其基因存在1个启动子,其转录受甲基化的影响较大.HNRNPA1蛋白质是由372个氨基酸组成的不具有核定位序列、无跨膜结构的亲水不稳定蛋白质,其等电点为9.17.通过对多物种的序列比对发现其305—335氨基酸序列保守性强,是重要的功能区域,该蛋白质在哺乳动物中具有极高的同源性.蛋白质相互作用及GO分析表明,HNRNPA1多分布于细胞核中,参与mRNA的选择性剪接.
[1] DREYFUSS G, KIM V N, KATAOKA N. Messenger-RNA-binding proteins and the messages they carry[J]. Nat Rev Mol Cell Biol, 2002, 3 (3): 195-205.
[2] GLISOVIC T, SODERBERG M, CHRISTIAN K, et al. Interplay between transcriptional and post-transcriptional regulation of Cyp2a5 expression[J]. Biochem Pharmacol, 2003, 65 (10): 1653-1661.
[3] JI Y, TULIN A V. Poly (ADP-ribose) controls DE-cadherin-dependent stem cell maintenance and oocyte localization[J]. Nat Commun, 2012, 3: 760.
[4] KIM H J, KIM N C, WANG Y D, et al. Mutations in prion-like domains in hnRNPA2B1 and hnRNPA1 cause multisystem proteinopathy and ALS[J]. Nature, 2013, 495 (7442): 467-473.
[5] YIN S Y, EFFERTH T, JIAN F Y, et al. Immunogenicity of mammary tumor cells can be induced by shikonin via direct binding-interference with hnRNPA1[J]. Oncotarget, 2016, 7 (28): 43629-43653.
[6] IZUMI R, WARITA H, NIIHORI T, et al. Isolated inclusion body myopathy caused by a multisystem proteinopathy-linked hnRNPA1 mutation[J]. Neurol Genet, 2015, 1 (3): e23.
[7] LIU X, ZHOU Y, LOU Y, et al. Knockdown of HNRNPA1 inhibits lung adenocarcinoma cell proliferation through cell cycle arrest at G0/G1 phase[J]. Gene, 2016, 576 (2): 791-797.
[8] NADIMINTY N, TUMMALA R, LIU C, et al. NF-kappaB2/p52: c-Myc: hnRNPA1 pathway regulates expression of androgen receptor splice variants and enzalutamide sensitivity in prostate cancer[J]. Mol Cancer Ther, 2015, 14 (8): 1884-1895.
[9] VAD-NIELSEN J, JAKOBSEN K R, DAUGAARD T F, et al. Regulatory dissection of the CBX5 and hnRNPA1 bi-directional promoter in human breast cancer cells reveals novel transcript variants differentially associated with HP1alpha down-regulation in metastatic cells[J]. BMC Cancer, 2016, 16: 32.
[10] ZHOU B, WANG Y, JIANG J, et al. The long noncoding RNA colon cancer-associated transcript-1/miR-490 axis regulates gastric cancer cell migration by targeting hnRNPA1[J]. IUBMB Life, 2016, 68 (3): 201-210.
[11] CHU P C, YANG M C, KULP S K, et al. Regulation of oncogenic KRAS signaling via a novel KRAS-integrin-linked kinase-hnRNPA1 regulatory loop in human pancreatic cancer cells[J]. Oncogene, 2015, 35 (30): 3897-3908.
[12] MOLLER S, CRONING M D, APWEILER R. Evaluation of methods for the prediction of membrane spanning regions[J]. Bioinformatics, 2001, 17 (7): 646-653.
[13] HE Y, SMITH R. Nuclear functions of heterogeneous nuclear ribonucleoproteins A/B[J]. Cell Mol Life Sci, 2009, 66 (7): 1239-1256.
[14] BILODEAU P S, DOMSIC J K, MAYEDA A, et al. RNA splicing at human immunodeficiency virus type 1 3' splice site A2 is regulated by binding of hnRNP A/B proteins to an exonic splicing silencer element[J]. J Virol, 2001, 75 (18): 8487-8497.
[15] GILPIN K M, CHANG L, MONTEIRO M J. ALS-linked mutations in ubiquitin-2 or hnRNPA1 reduce interaction between ubiquitin-2 and hnRNPA1[J]. Hum Mol Genet, 2015, 24 (9): 2565-2577.
[16] HONDA H, HAMASAKI H, WAKAMIYA T, et al. Loss of hnRNPA1 in ALS spinal cord motor neurons with TDP-43-positive inclusions[J]. Neuropathology, 2015, 35 (1): 37-43.
[17] LIU X Y, LI H L, SU J B, et al. Regulation of RAGE splicing by hnRNP A1 and Tra2β-1 and its potential role in AD pathogenesis[J]. J Neurochem, 2015, 133 (2): 187-198.

