棉花细胞质雄性不育恢复基因Rf1候选区域SSCP标记开发与候选基因表达分析
刘利彩,郭立平,戚廷香,王海林,唐会妮,张学贤,乔秀琴,吴建勇,邢朝柱
(棉花生物学国家重点实验室/中国农业科学院棉花研究所,河南安阳455000)
杂种优势利用是提高棉花产量的重要手段,传统的人工制种方式费时费工,且容易造成种子混杂。细胞质雄性不育系 (Cytoplasmic male sterility,CMS)的应用则可以节省大量劳动力,且种子纯度有保障,已成为杂交棉制种的最佳途径之一。细胞质雄性不育“三系”(不育系、保持系和恢复系)材料中恢复系的恢复性能是决定能否选育到强优势组合的关键因素之一,而恢复系育性的恢复由细胞核恢复基因决定。因此,近年来围绕恢复基因开展了大量研究。Liu等[1]利用F2群体,鉴定到2个随机扩增多态性DNA(Random amplified polymorphic DNA,RAPD)标记和3个简单重复序列 (Simple sequence repeats,SSR)标记与 Rf1基因紧密连锁。Feng等[2]利用 1个Ark8518回交群体,有4个RAPD标记被鉴定到,其中1个标记UBC607500与恢复基因Rf1的遗传距离为2.7 cM。Yin等[3]鉴定到1个与恢复基因Rf1遗传距离仅为0.2 cM的标记NAU4047,通过BAC文库筛选,恢复基因被定位在100 kb区间内。杨路明等[4]利用BC1群体,鉴定到6个表达序列标签 (Expressed sequence tag,EST)-SSR 标记,距Rf1遗传距离均为0.327 cM。本课题利用近等基因系群体和F2群体,筛选到13个与Rf1基因紧密连锁的分子标记,距Rf1最近距离为0.042 cM[5]。
多种作物中的研究表明,由PPR(Pentatricope ptiderepeat)基序串联组成的PPR蛋白与恢复基因存在一定联系[6-13]。Wang等[14]从棉花中获得了1个与恢复基因Rf2连锁的PPR扩增片段长度多态性 (Amplified fragment length polymorphism,AFLP)标记,随后,Wang等[15]又鉴定到1个与Rf1紧密连锁的PPR目标区域扩增多态性(Target region amplified polymorphism,TRAP)标记。杨路明等[16]从目标细菌人工染色体(Bacterial artificial chromosome,BAC)中获得了恢复基因Rf1的可能候选PPR基因orf3,但仍需通过转基因进行功能互补验证。本课题结合恢复基因Rf1初定位和棉花D基因组中PPR序列的分析,在包含与恢复基因Rf1紧密连锁的标记BNL3535的160kb区间内鉴定到成簇存在的9个PPR基因,利用距离标记最近的Cotton_D_gene_10013437基因获得了1个与Rf1基因共分离的PPR-CAPS标记[5]。上述研究表明棉花中PPR基因可能与恢复基因存在重要联系,但是相关研究仍有待深入。
在前期研究基础上,本研究期望通过对目标区间内相关基因的分析进一步增加恢复基因目标区间内标记密度,同时通过荧光定量分析相关基因在花蕾不同发育阶段的表达模式,从而为恢复基因的进一步研究奠定基础。
1 材料与方法
1.1 试验材料
本研究以哈克尼西棉细胞质雄性不育系(ZBA)、保持系(ZB)和恢复系(中恢 -46)为亲本,构建恢复基因近等基因系和F2群体[5]。
1.2 试验方法
1.2.1 材料采集。2013、2014年4月下旬,试验材料分别种植于中国农业科学院棉花研究所东场试验田(河南省安阳县),田间常规管理。在苗期采集群体单株叶片;在7―8月盛花期,分别剥取不育系、保持系和恢复系材料长度为1 mm、2 mm、3 mm、4 mm的花蕾,保存于-70℃超低温冰箱中备用。
1.2.2 性状调查。在7月上旬到8月中下旬的开花期间,调查F2群体中2 500个单株育性,为了准确,共调查5次,每株查看3次,每株查看至少3个花朵。根据手指捻破花药是否出现花粉为标准,判断棉花植株的育性。
1.2.3 植物DNA、RNA提取。叶片DNA的提取采用改良的CTAB法[17]。花蕾RNA提取采用天根生化科技(北京)有限公司的多糖多酚植物总RNA提取试剂盒,RNAase-free处理的枪头、离心管购自生工生物工程(上海)股份有限公司。
1.2.4 构建基因池。利用前期构建的近等基因系群体[5],采用群体分离分析法(Bulked segregate analysis,BSA)分别随机选出10株可育和10株不育的单株分别提取DNA,然后等量混合,构建可育DNA池和不育DNA池。
1.2.5 目标区域基因分析。利用初定位的SSR标记通过D基因组本地Blast分析候选区域基因结构分析,并在NCBI网站Blast寻找分析目标区域内的预测基因的同源基因。
1.2.6 单链构象多态性(Single-strand conformation polymorphism,SSCP)引物筛选与验证。利用Primer Premier 5软件分段设计出覆盖18个基因的155对引物,经琼脂糖凝胶检测能扩增出目标条带的引物分别在可育池和不育池中进行SSCP筛选。扩增体系为 20 μL,包括 10×Buffer 2 μl,5 mmol·L-1dNTP 0.5 μL,5 U·μL-1Taq 聚合酶 0.2 μL,10 mmol·L-1的上下游引物各 1 μL, 模板DNA 1.5 μL,灭菌 ddH2O 13.8 μL;反应程序如下:94℃3 min,(94℃ 30 s,58℃ 30 s,72℃ 1 m in)30个循环。银染参照张军等[18]。
在可育基因池和不育基因池中,使用SSCP引物分别扩增,将扩增产物分别与T载体(Promega,USA)连接,然后转化和涂板培养,每个产物随机挑取8个克隆送至生物公司测序,使用DNASTAR软件进行序列拼装,多序列比对采用Genedoc软件进行分析。
1.2.7 F2群体图谱构建。在本课题前人研究基础上[5],3 对 SSR 标 记 (BNL3535、TMB1295 和NAU3652),以及本试验筛选得到的15对多态性SSCP标记包含2 500个单株F2群体的DNA中进行扩增。采用JoinMap 4.0作图软件,构建遗传连锁图谱。
1.2.8 荧光定量。总RNA反转录程序参照试剂盒说明书进行,Oligo(dT)用于反转录引物。将反转录产物稀释10倍后用于荧光定量分析。荧光定量根据北京康为世纪生物科技有限公司试剂盒说明书进行操作,反应体系包含稀释后反转录产物 2 μL,2×UltraSYBR M ixture 10 μL,10 μmol·L-1的上下游引物各 0.4 μL, 用 RNase-free H2O定量到20 μL总体系。PCR反应程序如下:95℃10 m in,(95 ℃ 5 s,58 ℃ 20 s,72 ℃ 30 s)40 个循环。每个材料、每个基因做3次重复,actin为内参基因。
1.2.9 引物设计。本实验9个基因的引物是利用Primer Prem ier 5软件设计,引物序列参见表1。
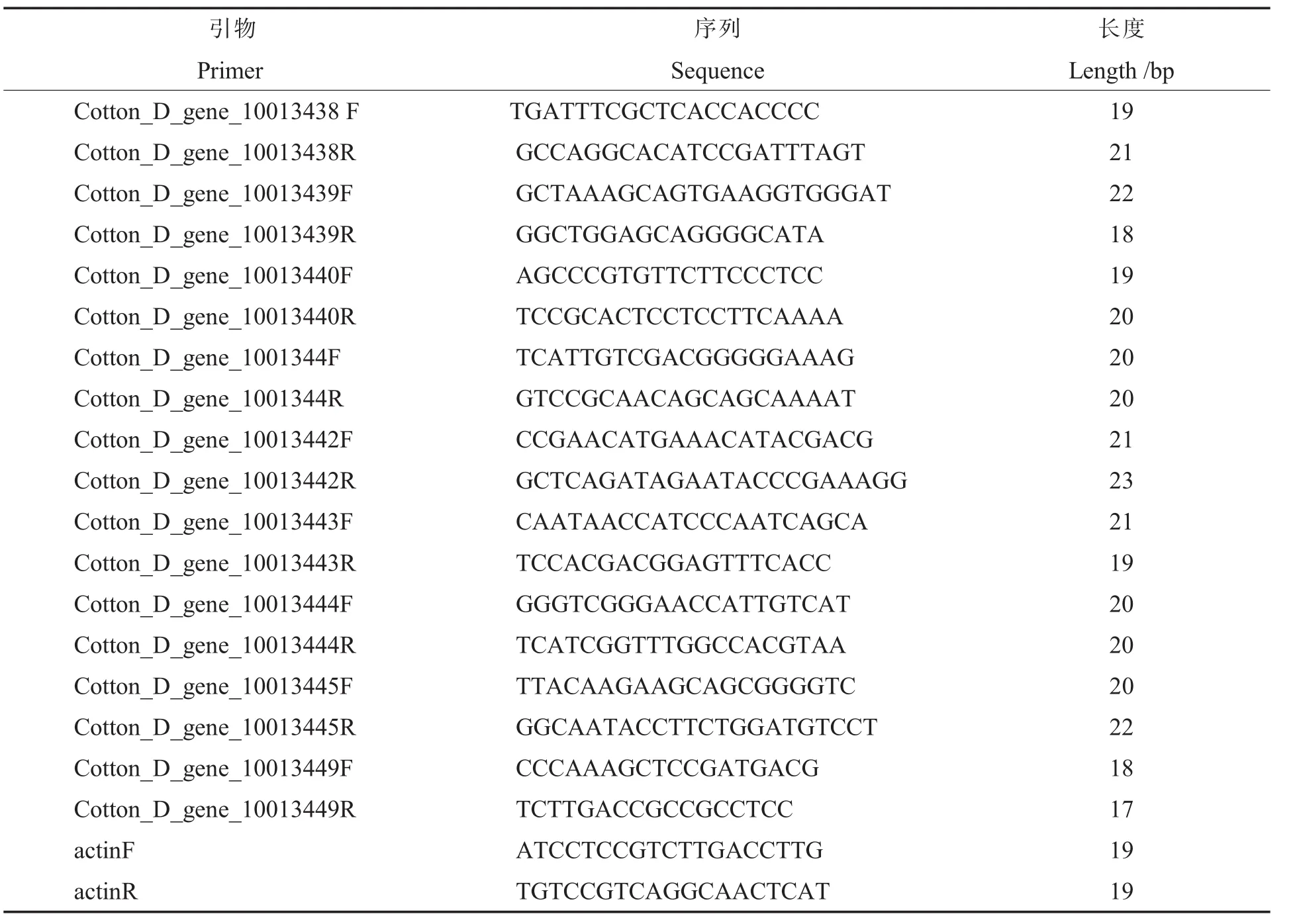
表1 RT-PCR荧光定量引物序列Table 1 Primers for quantitative RT-PCR
2 结果与分析
2.1 F2群体单株育性调查
根据F2群体单株育性统计,2 500个单株群体中,可育株 1 870株,不育630株,经 χ2检验(P=0.8174),可育株与不育株分离比例符合3∶1,表明哈克尼西棉不育恢复基因是受1对显性基因控制。
2.2 目标区域基因分析
前期研究中[5],恢复基因Rf1所在160 kb区间内共鉴定到18个基因,除成簇存在的9个PPR基因外,还存在9个其他类型的基因(表2)。通过Blast分析发现,其中有部分基因,如AP2/EREBP转录因子参与调控花器官的发育,并且在环境胁迫反应中发挥重要作用[19]。
2.3 多态性引物筛选及分析
在恢复基因所在的160 kb区间内分布18个PPR基因,用155对引物进行筛选,其中有115对引物能扩增得到目标条带,随后通过对115对引物的SSCP筛选,其中有15对SSCP在不育池和可育池间存在多态性,如图1所示。这15对引物主要分布在6个PPR基因(Cotton_D_gene_10013435,Cotton_D_gene_10013437,cotton_D_gene_10013446 ,Cotton_D_gene_10013447 ,Cotton_D_gene_10013448,Cotton_D_gene_10013450)和Cotton_D_gene_10013441,Cotton_D_gene_10013442,Cotton_D_gene_10013444。

表2 目标区域内18个基因的功能注释Table 2 Functional annotation of the 18 genes in the target region
2.4 多态性SSCP标记测序分析
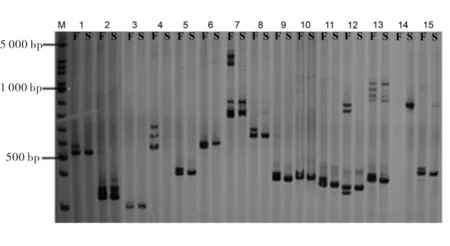
图1 可育池和不育池SSCP多态性Fig.1 SSCP polymorphism s within fertile and sterile pool
为验证SSCP标记是否真实,随机挑选7个SSCP标记的扩增产物,对其克隆测序。序列比对发现:有6个引物的扩增在2个材料间存在SNP或者Indel(图2),说明筛选到的SSCP标记真实可靠。

图2 2个多态性SSCP引物扩增序列比对结果Fig.2 Multiple sequences alignment of the am plifications of two polymorphic SSCR primers
2.5 多态性标记群体分析
利用F2群体结合表型分析,采用JionMap 4.0软件,用3个SSR标记和15个SSCP标记构建出Rf1遗传连锁图谱(图3)。从遗传连锁图看出:与Rf1基因最远的标记是343503-1和344204-4,相距4.8 cM,距Rf1基因最近的标记是BNL3535,其遗传距离为 2.9 cM;SSCP标记343501-1和344805-2距Rf1基因最近,其遗传距离为3.2 cM。
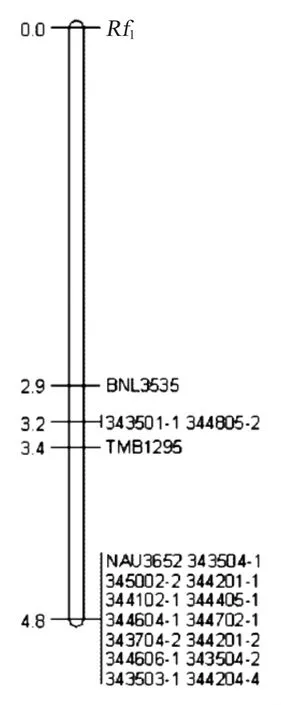
图3 棉花恢复基因Rf1的遗传连锁图Fig.3 Genetic linkage map of restorer gene Rf1
2.6 目标区域内基因的表达分析
在前期的研究中,目标区域内的PPR基因作为恢复基因候选基因,已经对其在“三系”间的表达模式进行了研究(未发表)。在本研究中,为了进一步了解目标区域内其他9个其他类型基因的表达模式,对其中8个基因在“三系”花蕾发育4个时期(花蕾长度为 1mm、2mm、3mm、4mm)的表达进行分析(图4)。结果表明8个基因在不同材料和不同发育时期的表达水平各不相同,其中Cotton_D_gene_10013443和Cotton_D_gene_10013444这2个基因在不同材料中有相似的表达模式,其表达量均随花蕾发育逐渐升高;此外,4个基因(Cotton_D_gene_10013439,Cotton_D_gene_10013441,Cotton_D_gene_10013443和Cotton_D_gene_10013444基因)在不育系材料中随花蕾发育表达量逐渐升高,Cotton_D_gene_10013438和Cotton_D_gene_10013440这2个基因在不育系中的表达量在花蕾2 mm长时降低,随后随花蕾发育升高,而Cotton_D_gene_10013442基因在不育系和恢复系中表达量随花蕾发育分别呈现下降和升高的趋势。
3 讨论
SSCP标记体现的是基因序列内部的多态性,可以检测生物中广泛存在的SNP和InDel位点等,已经成为1种高效的功能标记,并在动物育种[20]、微生物研究[21]、人类疾病研究[22]、以及植物遗传作图等方面都获得了很好的应用[23]。在棉花中,SSCP技术也已经成功应用在油分相关基因和纤维发育相关基因的研究中[24-25]。本研究主要参照谢晓兵等[24-25]在棉花中建立的成熟的技术体系和分析方法,获得了重复性较好的15对多态性引物,并且通过对相关多态性标记在可育池和不育池中的克隆测序分析,从克隆的7个序列中明确了6个被检测序列在可育和不育材料间确实存在着SNP和InDel位点。这充分证明了SSCP技术的高效性和可靠性。
农艺性状优良的高配合力恢复系选育是决定三系杂交种选育成败关键,所以对恢复基因进行精细定位,以致克隆恢复基因,创造新的恢复系,意义重大。近年来,在棉花中,围绕恢复基因定位的相关研究[26-33]开展较多,但是仍未见有关恢复基因克隆和功能验证方面的报道。这表明对恢复基因进行精细定位为下一步对其克隆仍有必要。近年来,相继完成了3个棉种(AA、DD、AADD)测序[34-36],为精细定位恢复基因提供了理想的参考序列。本研究在前期研究的基础上,参考了棉花基因组测序数据,根据目标区域内PPR的相关基因序列,作进一步定位,不仅丰富了目标区域相关标记数目,同时也对相关基因的表达模式进行了分析。本研究利用相关基因序列获得了一些与恢复基因紧密连锁的SSCP标记,但是,所得到的这些标记距离目标基因仍较远。因此,有必要开发更多的标记,进一步对目标基因进行精细定位,如利用棉花基因组序列开发出涵盖全基因组的SSR、SNP和InDel标记等。另一方面,所获得的标记容易成簇存在。因此,为了提高恢复基因定位的准确性和分辨率,有必要进一步扩大分离群体,并通过多个群体同时进行定位,以期相互验证。而目标区域内的基因随着花药的发育表现出了不同的表达模式,猜测这些基因由于受到不育胞质或者恢复基因的影响,其表达模式受到一定程度的影响。至于这些基因与不育胞质或恢复基因的关系,则有待于进一步的研究。
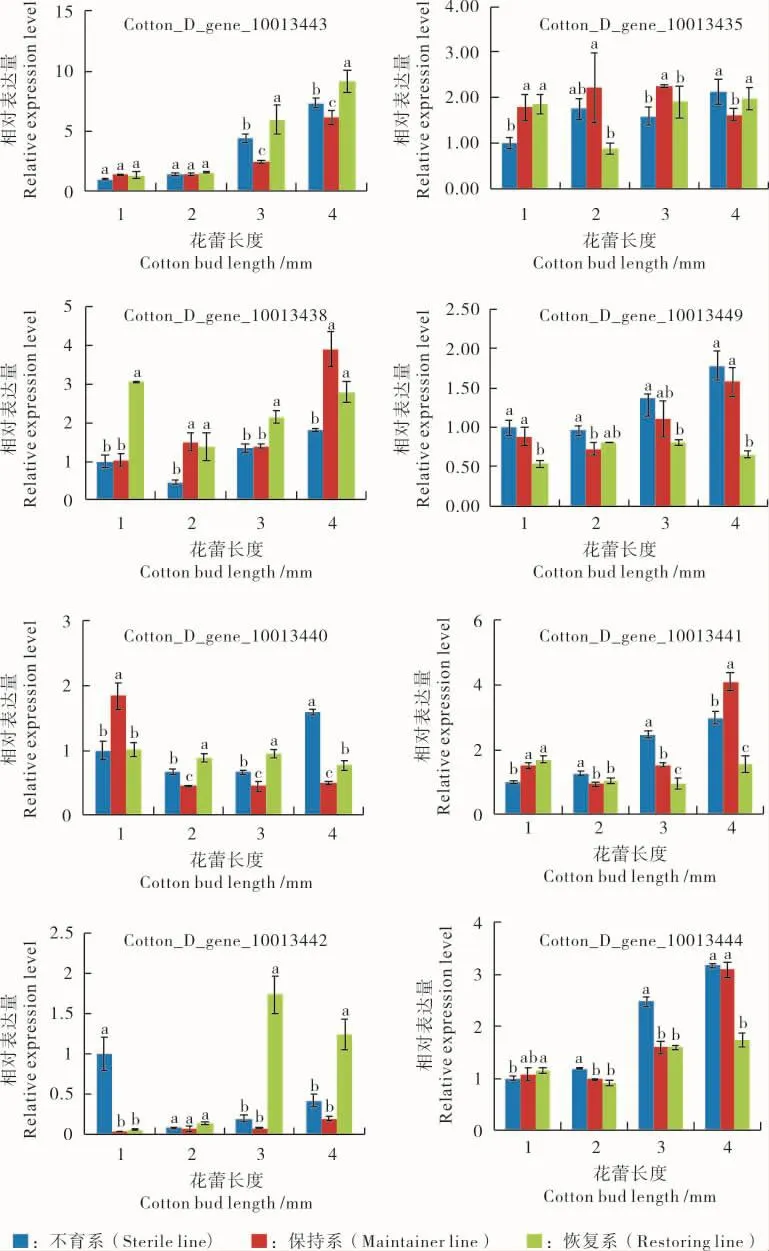
图4 8个基因在不育系、保持系、恢复系材料花蕾发育不同阶段表达模式Fig.4 Expression pattern of eight genes on different buds developing stages am ong restoring lines
[1]Liu Liwang,Guo Wangzhen,Zhang Tianzhen.Inheritance and fine mapping of fertility restoration for Cytoplasmic male sterility in Gossypium hirsutum L.[J].Theoretical and Applied Genetics,2003,106(3):461-469.
[2]Feng Cunda,Stewart J M,Zhang Jinfa,et al.STS markers linked to the Rf1 fertility restorer gene of cotton[J].Theoretical and Applied Genetics,2005,110(2):237-243.
[3]Yin Jianmei,Guo Wangzhen,Yang Luming,et al.Physical mappingof the Rf1 fertility-restoringgene to a 100 kb region in cotton[J].Theoreticaland Applied Genetics,2006,112(7):1318-1325.
[4]Yang Lum ing,Zhu Huayu,Guo Wangzhen,et al.Molecular cloning and characterization of five genes encoding pentatricopeptide repeat proteins from upland cotton(Gossypium hirsutum L.)[J].Molecular Biology Reports,2010,37(2):801-808.
[5]Wu Jianyong,Cao Xiuxia,Guo Liping,et al.Development of a candidate gene marker for Rf1 based on a PPR gene in Cytoplasmic male sterile CMS-D2 upland cotton[J].Molecular Breeding,2014,34(1):231-240.
[6]Cui X,Wise R P,Schnable P S.The rf2nuclear restorer gene of male-sterile T-cytoplasm maize[J].Science,1996,272(5266):1334-1336.
[7]Itabashi E,Iwata N,Fujii S,et al.The fertility restorer gene,Rf2,for Lead Rice-type Cytoplasmic male sterility of rice encodes a mitochondrial glycine-rich protein[J].The Plant Journal,2011,65(3):359-367.
[8]Bentolila S,Alfonso A A,Hanson M R.A pentatricopeptide repeat containing gene restores fertility to Cytoplasmic malesterile plants[J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(16):10887-10892.
[9]Brown G G,Formanova N,Jin H,et al.The radish Rf o restorer gene of Ogura Cytoplasmic male sterility encodes a protein with multiple pentatricopeptide repeats[J].The Plant Journal,2003,35(2):262-272.
[10]Li Y,Liu Z,Cai Q,et al.Identification of a m icrosatellite maker linked to the fertility-restoring gene for a polima Cytoplasmic male-sterile line in Brassica napus L.[J].A frican Journal of Biotechnology,2011,10(47):9563-9569.
[11]K lein R R,Klein P E,Mullet J E,et al.Fertility restorer locus Rf1of soghum(Soghum bicolor L.)encodes a pentatricopeptide repeat protein not present in the colinear region of rice chromosome 12[J].Theoretical and Applied Genetics,2005,111(6):994-1012.
[12]Komori T,Ohta S,Murai N,et al.Map-based cloning of a fertility restorer gene,Rf-1,in rice(Oryza sativa L.)[J].The Plant Journal,2004,37(3):315-325.
[13]A karg H,Nakamura A,Yokozeki-M isono Y,et al.Positional cloning of the rice Rf-1 gene,a restorer of Bt-type Cytoplasmic male sterility that encodes a m itochondria-targeting PPR protein[J].Theoreticaland Applied Genetics,2004,108(8):1449-1457.
[14]倪西源,王学德,程超华,等.棉花分子标记图谱的构建和一些重要性状的定位[J].棉花学报,2007,19(1):71-73.Ni Xiyuan,Wang Xuede,Cheng Chaohua,et al.Cotton molecular markers chromatogram and some important properties of positioning[J].Cotton Science,2007,19(1):71-73.
[15]张小全,王学德,蒋培东,等.细胞质雄性不育海岛棉恢复系恢复基因的遗传分析[J].中国农业科学,2009,42(6):1896-1900.Zhang Xiaoquan,Wang Xuede,Jiang Peidong,et al.Restoring genes of cytoplasmic male sterility(CMS)sea island cotton restorer of genetic analysis[J].Scientia Agricultura Sinica,2009,42(6):1896-1900.
[16]杨路明.棉花细胞质雄性不育恢复基因的图位克隆及PPR基因家族的分析[D].南京:南京农业大学,2009.Yang Lum ing.Map-based cloning of fertility restoring gene of CMS and analysis of PPR gene family in cotton[D].Nanjing:Nanjing Agricultural University,2009.
[17]宋国立,崔荣霞,王坤波,等.改良CTAB法快速提取棉花DNA[J].棉花学报,1998,10(5):273-275.Song Guoli,Cui Rongxia,Wang Kunbo,et al.The modified CTAB method to extract the cotton DNA[J].Cotton Science,1998,10(5):273-275.
[18]张军,武耀廷,郭旺珍,等.棉花微卫星标记的PAGE/银染快速检测[J].棉花学报,2000,12(5):267-269.Zhang Jun,Wu Yaoting,Guo Wangzhen,et al.Fast screening of m icrosatellite markers in cotton with PAGE/silver staining[J].Cotton Science,2000,12(5):267-269.
[19]Dietz K J,Vogel M O,Viehhauser A.AP2/EREBP transcription factorsarepartofgeneregulatorynetworksand integratemetabolic,hormonal and environmental signals in stress acclimation and retrogradesignaling[J].Protoplasma,2010,245(1-4):3-14.
[20]李绍华,熊远著,郑嵘,等.猪MSTN基因多态性及其SNPs的研究[J].遗传学报,2002,29(4):326-331.Li Shaohua,Xiong Yuanzhu,Zheng Rong,et al.Polymorphism of porcine myostatin gene[J].Acat Genetica Sinica,2002,29(4):326-331.
[21]Callon C,Delbès C,Duthoit F,et al.Application of SSCP-PCR fingerprinting to profile the yeast community in raw m ilk salers cheeses[J].Systematic and Applied M icrobiology,2006,29(2):172-180.
[22]Shintani S,Nakahara Y,Waghmare V N,et al.Inactivation of the p14ARF,p15INK4B and p16INK4A genes is a frequent event in human oral squamous cell carcinomas[J].Oral Oncology,2001,37(6):498-504.
[23]李媛媛.SSCP技术及在植物中的应用[J].北方园艺,2009(5):122-124.Li Yuanyuan.Single-strand conformation polymorphism and its application in plant research[J].Northern Horticulture,2009,(5):122-124.
[24]谢晓兵,于霁雯,吴嫚,等.棉花油分合成相关基因的SSCP标记开发[J].分子植物育种,2011,9(3):336-342.Xie Xiaobing,Yu Jiwen,Wu Man,et al.Development of SSCP markers for genes related to oil synthesis in cotton[J].Molecular Plant Breeding,2011,9(3):336-342.
[25]谢晓兵,于霁雯,吴嫚,等.利用SSCP技术分析棉花纤维差异表达的基因[J].棉花学报,2011,23(4):306-310.Xie Xiaobing,Yu Jiwen,Wu Man,et al.AnaIysis of SSCP for cotton differentially expressed genes related to fiber development[J].Cotton Science,2011,23(4):306-310.
[26]Zhang J F,Stewart J M.CMS-D8 restoration in cotton is conditioned by one dom inant gene[J].Crop Science,2001,41(2):283-288.
[27]Zhang F,Stewart J M.Inheritance and genetic relationships of the D8 and D2-2 restorer genes for cotton cytoplasmic male sterility[J].Crop Science,2001,41(2):289-294.
[28]李朋波,曹美莲,刘慧民,等.棉花晋A细胞质雄性不育恢复基因定位[J].西北植物学报,2007,27(10):1937-1942.Li Pengbo,Cao Meilian,Liu Huim in,et al.Assignment of genes of cotton in A cytoplasmic male sterile restorer gene[J].Acta Botanica Boreali-Occidentalia Sinica,2007,27(10):1937-1942.
[29]郭旺珍,张天真,潘家驹,等.棉花细胞质雄性不育育性恢复基因的RAPD-PCR标记筛选[J].科学通报,1997,42(24):2645-2647.Guo Wangzhen,Zhang Tianzhen,Pan Jiaju,et al.Cytoplasmic male sterility (CMS)in cotton fertility restoring genes of RAPD-PCR marker screening[J].Science Bulletin,1997,42(24):2645-2647.
[30]Guo W Z,Zhang T Z,Pan J J,et al.Identification of RAPD marker linked with fertility-restoring gene of cytoplasmic male sterile lines in upland cotton[J].Chinese Agricultural Science Bulletin,1998,(43):52-54.
[31]Lan T,Cook C G,Paterson A H.Identification of a RAPD marker linked to a male fertility restoration gene in cotton(Gossypium hirsutum L.)[J].Journal of Agricultural Genomics,1999,1(4):299.
[32]Zhang J F,Stewart J M.Identification of molecular markers linked to the fertility restorer genes for CMS-D8 in cotton[J].Crop Science,2004,44(4):1209-1217.
[33]Wang F,Stewart J M,Zhang J.Molecular markers linked to the Rf2 fertility restorer gene in cotton[J].Genome,2007,50(9):818-824.
[34]Wang Kunbo,Wang Zhiwen,Li Fuguang.et al.The draft genome of a diploid cotton Gossypium raimondii[J].Nature Genetics,2012,44(10):1098-1103.
[35]Paterson A H,Wendel J F,Gundlach H,et al.Repeated polyploidization of Gossypium genomes and the evolution of spinnable cotton fibres[J].Nature,2012,492(7429):423-427.
[36]Li Fuguang,Fan Guangyi,Wang Kunbo,et al.Genome sequence of the cultivated cotton Gossypium arboretum[J].Nature Genetics,2014,46(6):567-572.

