棉花FAR1/FHY3基因家族的全基因组分析
袁娜,王彤,刘廷利,杨郁文,郭月,刘静,张保龙,杜建厂
(江苏省农业科学院种质资源与生物技术研究所/江苏省农业生物学重点实验室,南京210014)
光是影响植物生长和发育最为重要的环境因素之一[1]。高等植物在进化过程中形成了多个光受体网络,如光敏色素(Phytochromes)、隐花色素(Cryptochromes)、向光素(Phototropins)和中波紫外线(UV-B)受体等用以感受不同波长的光[2-3]。光敏色素是1类主要感受红光(600~700 nm)与远红光(700~800 nm)的色素蛋白[4]。光敏色素通过响应红光和远红光直接或间接调控下游多个转录因子,从而控制下游基因表达,影响植物种子萌发、幼苗去黄化、茎秆伸长、叶绿体运动、植物避荫反应和开花等发育过程[5]。充分理解植物对红光及远红光的响应将有助于促进农作物的关键发育过程的进行,从而对保障农作物的产量起到积极作用[6]。
在光敏色素家族中,phyA是植物最主要的远红光受体。在黑暗环境中,phyA以非活性的红光吸收型(Pr)形式位于细胞质中。当受到短暂的红光、远红光、蓝光或持续的远红光照射时,phyA便转化成活性的远红光吸收型(Pfr)状态,从细胞质转移到细胞核内[7-8],直接与一系列转录因子相互作用,进而调控一系列远红光介导的光形态反应相关基因的表达[9-10]。在过去的20年间,phyA信号通路中的一些重要元件相继被鉴定,例如FAR-RED ELONGATED HYPOCOTYL 1(FHY1) 及 其 同 源 的 FHY1-like (FHL) 和FAR-RED ELONGATED HYPOCOTYL 3(FHY3)及其同源的FAR-RED IMPAIRED RESPONSE 1 (FAR1)[11-12]。 FAR1/FHY3 与 玉 米Mutator编码的MURA转座酶及移动因子Jittery的转座酶具有较高的序列相似性,但没有明显的末端反向重复序列 (Term inal inverted repeats,TIR)和其他的转座子类似结构[13],因此这是1类起源于转座子酶,经“分子驯化”而形成的转录因子家族。目前关于FAR1/FHY3蛋白家族的研究主要集中于拟南芥。拟南芥中共有14个FAR1/FHY3家族成员被鉴定,编码531~851个氨基酸。除FAR1-RELATED SEQUENCE 10(FRS10)外的所有成员在叶、茎、花和果中均有表达[14]。FAR1/FHY3蛋白包含3个功能结构域,包括具有DNA结合活性的C2H2型锌指结构域(C2H2 zinc finger domain)、核心转座酶结构域(Putative core transposase domain)以及具有转录激活活性的SW IM锌指结构域(SW IM zinc finger domain)[15]。研究发现,拟南芥中FHY3和FAR1分别与FHY1和FHL的启动子上的FBS motif结合,从而激活FHY1和FHL的转录。FHY1和FHL直接与Pfr相互作用,协助phyA进入细胞核[14]。因此,FAR1/FHY3在启动phyA信号通路中具有非常关键的作用,直接决定在光诱导后phyA是否可以进入细胞核行使生物学功能。
研究发现通过转化光敏色素相关基因可以降低作物株高、增加作物产量等[16]。在马铃薯中过表达拟南芥的phyB基因可以使植株株型变得紧凑,从而增加高密度下块茎的数量。在水稻中过表达拟南芥的phyA基因可以使转基因水稻在远红光和红光下胚芽鞘变短、节间缩短、成株高度显著降低,并且单株籽粒产量显著增加[17-18]。这些研究表明光敏色素相关基因也可以影响作物的重要农艺性状,因此对这些基因的挖掘及分析具有重要的应用价值。
棉花是世界上最重要的经济作物之一。随着雷蒙德氏棉 (Gossypium raimondii Ulbrich)[19]、亚洲 棉 (Gossypium arboreum L.)[20]和 陆 地 棉(Gossypium hirsutum L.)[21-22]基因组测序的完成,许多功能基因已被发掘和鉴定,也为全基因组水平分析棉花中重要基因家族的演化提供可能。光照时间、光照强度以及不同光质如红光和蓝光等对棉花的产量和质量都有很大的影响。然而有关棉花光敏色素信号通路相关基因家族的研究还未见报道。本研究利用生物信息学方法,对棉花FAR1/FHY3基因家族成员进行鉴定,分析其蛋白的理化特性、序列特征及系统进化关系,以期为后续棉花FAR1/FHY3的功能研究和应用提供理论参考和依据。
1 材料与方法
1.1 棉花中光信号转录因子FAR1/FHY3家族成员的鉴定
在 Pfam 数据库(http://pfam.xfam.org/)中 标号为PF03101、PF10551和 PF04434的结构域是植物光信号转录因子FAR1/FHY3的结构域。首先从棉花基因组数据库(https://www.cottongen.org/)中下载亚洲棉、雷蒙德氏棉、陆地棉的蛋白序列数据,利用HMMER 3.0软件(http://www.ebi.ac.uk/Tools/hmmer/search/hmmscan)和本地blastp方法联合进行检索,HMMER 3.0的限定E值为 0.01,blastp的 E 值设定为<1×e-10,筛选含有FAR1/FHY3结构域的序列。将获得的序列在InterPro(http://www.ebi.ac.uk/interpro/)和 SMART(http://smart.embl-heidelberg.de/)上进一步确认序列包含FAR1/FHY3保守域。同时获取这些FAR1/FHY3的 DNA、CDS以及染色体位置信息。根据FAR1/FHY3基因在拟南芥(Arabidopsis thaliana L.)的相关文献报道[14],从 TAIR 10(https://www.arabidopsis.org)下载拟南芥的FAR1/FHY3序列,同时下载可可全基因组序列(http://www.cacaogenomedb.org),利用上述方法获得可可FAR1/FHY3蛋白序列。
利用ExPASy Proteom ics Server软件(http://www.expasy.org)对棉花FAR1/FHY3基因家族成员的氨基酸序列进行理化性质分析,并利用PSORT(http://psort.hgc.jp/form.htm l)对棉花 FAR1/FHY3家族成员进行亚细胞定位预测。
1.2 棉花FAR1/FHY3基因结构和蛋白序列保守基序分析
利用在线工具 GSDS[23](http://gsds.cbi.pku.edu.cn/)分析和绘制棉花FAR1/FHY3基因家族成员的基因结构图。利用在线软件MEME(http://meme-suite.org/)对FAR1/FHY3蛋白保守域结构进行分析。基序最大发现数量设置为6,其他参数为默认值。
1.3 多序列比对与系统进化树的构建
利用MEGA 7.0[24]软件对获得的FAR1/FHY3蛋白序列进行多重序列比对。以多序列比对结果为基础,采用邻接(Neighbor-Joining)算法构建系统进化树,Bootstrap重复测试(Replications)设置为1 000。
1.4 染色体定位和基因同源性分析
根据棉花FAR1/FHY3的基因位置信息和棉花染色体的长度,使用Mapinspect(http://www.dpw.wau.nl/pv/PUB/MapComp/)和 Adobe illustrator CS3[25]软件绘制其染色体分布图。根据蛋白序列比对结果识别同源基因对,经过比对后短序列如果能覆盖超过长序列70%的区域,认为这对同源基因为重复基因(Duplication genes)。利用In-Paranoid[26]软件对3个棉种的FAR1/FHY3基因的直系同源性关系进行分析,利用可视化工具Circos(http://circos.ca/)将直系同源关系进行可视化展示。利用KaKs_Calculator 2.0[27]软件计算同源基因对的Ka(非同义突变率)、Ks(同义突变率)值,确定其在进化过程中受何种选择。
1.5 基因组织表达分析
在NCBI SRA数据库(http://www.ncbi.nlm.nih.gov/sra/)下载亚洲棉的叶片,雷蒙德氏棉的叶片、花瓣和陆地棉的茎、叶片、花瓣、花托转录组测序数据,下载序列号分别为SRR530430,SRR-389183,SRR943769,SRR1695174,SRR1695175,SRR1695177,SRR1695176。 基于以上数据,利用Tophat[28]和Cufflink[29]软件包分析转录组测序数据并进行表达量计算,提取FAR1/FHY3基因家族成员的基因表达数据,将表达数据标准化后,使用 Hem I (Heatmap Illustrator,version 1.0)[30]软件绘制热图,将表达数据可视化。
2 结果与分析
2.1 棉花FAR1/FHY3基因家族成员的鉴定
鉴定获得88个FAR1/FHY3基因,其中亚洲棉有27个、雷蒙德氏棉有35个、陆地棉有26个,分别命名为GaFAR1~GaFAR27、GrFAR1~GrFAR 35和 GhFAR1~GhFAR26(表 1)。 这 88个FAR1/FHY3基因编码的蛋白长度在158~1 134个氨基酸,最大和最小分子量分别为124 996.6 Da和18 598.26 Da。GhFAR14的等电点最小,为4.9;GaFAR1的等电点最大,为10.73。这些FAR1/FHY3蛋白质的等电点差异较大,表明其酸碱性不同。大多数FAR1/FHY3定位在细胞核,少数基因定位在细胞质和细胞膜,仅Gh-FAR4定位在线粒体。
2.2 棉花FAR1/FHY3蛋白的系统进化分析
利用MEGA 7.0软件通过邻接法对88个棉花FAR1/FHY3蛋白,18个拟南芥和 36个可可FAR1/FHY3蛋白序列构建了系统进化树,按照其亲缘关系可将这些成员分成3个亚组 (图1)。大部分的棉花FAR1/FHY3蛋白(44个)被归类到亚组Ⅰ中(黄色);亚组Ⅱ中(紫色)包含了36个棉花、11个可可和4个拟南芥FAR1/FHY3家族成员;亚组Ⅲ (绿色)则仅包含了8个棉花(Gr-FAR20,GhFAR15,GrFAR12,GhFAR4,GaFAR25,GhFAR8,GhFAR21,GrFAR26) 和 3 个 可 可FAR1/FHY3蛋白。单独进行棉花FAR1/FHY3蛋白聚类结果与上述进化树较为一致,也分为3个亚组(图 2A)。

表1 棉花FAR1/FHY3基因家族的部分成员信息Table 1 Information of the partial FAR1/FHY3 genes identified in cotton
2.3 棉花FAR1/FHY3基因结构及保守序列分析

图1 3个物种FAR1/FHY3蛋白序列的系统进化树Fig.1 Phylogenetic tree of the FAR1/FHY3 protein sequences in three species

图2 棉花FAR1/FHY3蛋白序列的系统进化树(A),保守基序(B)和基因结构(C)分析Fig.2 Phylogenetic tree(A),conserved motif(B)and gene structure(C)of cotton FAR1/FHY3 protein sequences in three Gossypium species
利用GSDS构建了棉花FAR1/FHY3基因结构图,结果显示不同亚组成员的内含子数量存在差异。亚组Ⅰ中有4个基因无内含子,其余基因的内含子数量均不超过9个。亚组Ⅱ的成员的含有1~4个内含子。亚组Ⅲ的8个成员的内含子数量较多,最少的有7个内含子,最多的有21个内含子。同一亚组内较为相似的外显子、内含子的组织结构进一步证实了系统发育树的可靠性(图 2A、图 2C)。
利用MEME软件对FAR1/FHY3蛋白保守基序的分析发现有6个比较保守的基序Motif 1-Motif 6(图2B)。在亚组Ⅰ中,24条蛋白序列各包含6种基序,这些序列的基序组织模式均为Motif 1_5_4_6_2_3,只有GrFAR14的保守基序组织模式为Motif 1_4_5_6_2_3。15条蛋白序列各包含5个基序,除了GrFAR13缺少Motif3外,其余序列均缺少Motif5。剩余的GhFAR14,Gh-FAR17,GhFAR26,GaFAR10,GaFAR15 各 包 含2~4个基序。在亚组Ⅱ中,29条蛋白序列各包含3 种基序(Motif 1、Motif 5、Motif 4),剩下的 7 条序列仅各包含2个基序 (3个Motif 5_4和4个Motif 1_4)。在亚组Ⅲ中,除去GrFAR20由于序列或软件原因无法检测外,剩余的7条序列仅各包含2个基序(Motif 1和Motif 4),基序组织模式均为M 4_1。比较保守基序和功能结构域发现,FAR1/FHY3蛋白的FAR1结构域(PF03101)与保守 基 序 1、4、5 相 互 重 叠 ,MULE (PF10551)和SW IM(PF04434)则分别与保守基序2、6和3部分重叠。
2.4 棉花FAR1/FHY3基因家族染色体定位和同源性关系分析
根据位置信息将79个FAR1/FHY3基因定位到棉花染色体上,另外9个FAR1/FHY3基因未被定位到染色体上。亚洲棉的27个FAR1/FHY3基因分布在11条染色体上,4号染色体上有5个,1号和9号染色体各分布4个,2号和7号染色体上均没有发现(图3A)。雷蒙德氏棉的33个基因分布在13条染色体上,其中11号染色体上分布较多,有7个基因;其次5号和6号染色体分别包含6个和5个FAR1/FHY3基因(图3B)。陆地棉的26条染色体中只有7条染色体包含FAR1/FHY3基因,其中A亚组10号染色体、D亚组2号和10号染色体上最多,各有4个FAR1/FHY3基因(图3C)。此外,FAR1/FHY3基因在3种棉花染色体上的分布都存在串联重复现象。亚洲棉中发现3个串联重复的基因簇(GaFAR1 和 GaFAR2,GaFA11 和 GaFAR12,Ga-FAR18、GaFAR19和 GaFAR20)。 在雷蒙德氏棉中仅发现 1个串联重复基因簇(GrFAR27、Gr FAR28和GrFAR29)并检测到1次基因片段复制事件;陆地棉中共发现3个串联重复的基因簇,包含8个基因,同时还检测到2次基因片段复制事件。
在3个棉种中利用InParanoid软件共检测到42对直系同源基因对。排除3对无法定位到染色体上的同源基因对后(GhFAR26,GrFAR34,Gr-FAR35,图 4),我们利用 KaKs_Calculator对剩余的36对同源基因对进行Ka/Ks分析,结果显示61%的直系同源基因的Ka/Ks值小于0.5,这表明这些基因经历了较强的负选择作用过程,暗示这些复制基因在进化中较为保守,结构比较稳定,功能具有一致性。另外,6对直系同源基因的Ka/Ks值在 0.5~1 之 间(GhFAR1/GaFAR12,Gh FAR5/Ga FAR3,GhFAR23/GaFAR19,GrFAR2/GaFAR3,GrFAR7/GaFAR12,GrFAR14/GaFAR13),仅有2对直系同源基因的Ka/Ks值大于 1(Gh-FAR8/GaFAR25,GrFAR29/GaFAR1), 表明其受到了正选择作用(表2)。
2.5 棉花FAR1/FHY3基因的组织表达分析
根据已有的亚洲棉叶片,雷蒙德氏棉叶片、花瓣和陆地棉的茎、叶片、花瓣、花托转录组数据库的分析,获得FAR1/FHY3基因表达量的FPKM(Fragments per kilobase of transcript per million fragments mapped)值,利用Hem I 1.0软件绘制出基因表达模式图(图5)。结果显示,在叶片中,亚组Ⅰ和亚组Ⅱ的部分成员具有较高的表达量,亚组Ⅲ的成员表达量均较低。在亚洲棉的叶片中,GaFAR17和GaFAR6具有较高的表达量;在雷蒙德氏棉和陆地棉叶片中,GrFAR34和Gh FAR14分别是表达量最高的基因。在花瓣中,GrFAR30和GhFAR14分别在雷蒙德氏棉和陆地棉中优势表达,剩余基因的表达量均较低。在陆地棉中,GhFAR14在茎、叶、花瓣和花托中的表达量都是最高,表明该基因可能在陆地棉远红光调控过程中起到重要的作用。经同源比对发现,GhFAR14与拟南芥FHY3基因具有较高的同源性,表明该基因可能在启动phyA信号通路中起到关键作用。
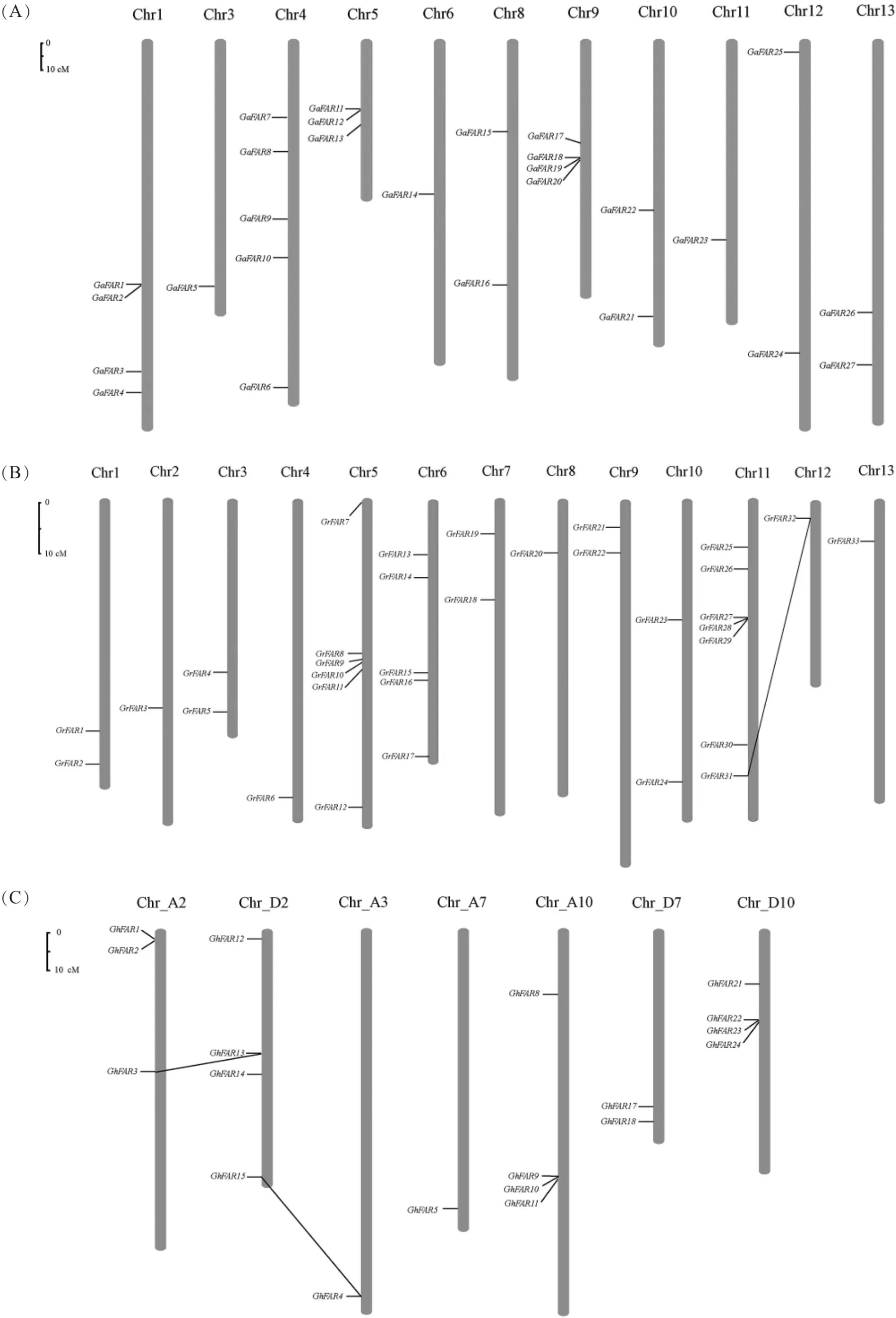
图3 亚洲棉(A)、雷蒙德氏棉(B)和陆地棉(C)FAR1/FHY3基因在染色体上的位置Fig.3 Chromosomal distribution of FAR1/FHY3 genes in Gossypium arboretum L.(A),G.raimondii Ulbrich(B)and G.hirsutum L.(C)

图4 棉花FAR1/FHY3基因家族成员的同源关系分析Fig.4 Homologous relationships of FAR1/FHY3 gene family members in cotton
3 讨论与结论
红光和远红光在植物生长发育过程中具有重要的调控作用。光敏色素是植物接收红光/远红光的主要受体,大量的实验研究已在分子水平、生化水平上证实其在光信号传递的过程中具有重要的作用[31-32]。在phyA信号通路的众多调控因子中,FAR1/FHY3是1类重要的正向调控因子。它们可以直接与FHY1和FHL的启动子结合,被激活表达的FHY1和FHL则协助phyA进入细胞核。在模式植物拟南芥中还发现1 000多个可能的FHY3结合基因,包括ERF4、ARC2、HEMB1和COP1等,表明FAR1/FHY3可能广泛地参与植物生长发育各个方面的调控[33]。我们通过生物信息学手段在亚洲棉和雷蒙德氏棉中分别鉴定出27和35个FAR1/FHY3基因。在陆地棉中仅获得了26个基因。根据染色体定位分析发现,这26个基因中有14个是由串联重复或者片段复制事件形成,这表明陆地棉在A、D亚组杂交加倍形成后,丢失了大量的FAR1/FHY3基因,然后又通过串联重复和片段复制进行基因扩增。
系统发育分析将棉花和拟南芥、可可的
FAR1/FHY3转录因子成员分为3个亚组,每个物种的FAR1/FHY3转录因子在亚组Ⅰ和亚组Ⅱ中均有分布,表明亚组Ⅰ和亚组ⅡFAR1/FHY3转录因子家族成员的分化可能早于拟南芥和棉花的物种分化。亚组Ⅲ仅包含可可和棉花基因,表明该支分化形成在与拟南芥物种分化之后。单独对棉花88个基因的聚类分析显示,亚组Ⅰ主要由亚洲棉和雷蒙德氏棉的FAR1/FHY3基因成员组成,陆地棉的FAR1/FHY3基因成员主要聚类在亚组Ⅱ和亚组Ⅲ中。同一亚组内的FAR1/FHY3转录因子的基因结构以及蛋白序列的保守基序组织模式都较为相似,进一步反应了聚类结果的可靠性。

表2 直系同源基因对的K a/K sTable 2 K a/K s ratio of orthologous gene pairs
Ka/Ks值常用来检验基因是否经历了选择作用[34]。受正选择的直系同源基因通常与生物合成代谢、酶和生物胁迫相关[35]。本研究鉴定的棉花FAR1/FHY3直系同源基因对中,仅有GhFAR8和 GaFAR25、GrFAR29 和 GaFAR18 的 Ka/Ks值大于1,这2对基因经同源比对发现与甲基丙二酸半醛脱氢酶 (Methylmalonate-sem ialdehyde de hydrogenase)和FHY3同源性较高。因此,我们推测这些基因在物种长期进化过程中经历了正选择作用。
组织表达分析表明,FAR1/FHY3基因主要在棉花叶片中表达。亚洲棉中的GaFAR17,雷蒙德氏棉中的GrFAR34和陆地棉中的GhFAR14分别是3种棉花叶片中表达量最高的基因。由于亚洲棉和雷蒙德氏棉其他组织转录组数据的不完善,本研究在仅有雷蒙德氏棉和陆地棉的花瓣,以及陆地棉的茎、花托转录组数据中发现FAR1/FHY3基因在这些组织中的表达较少。这些基因主要与FRS11,FRS5和FHY3具有较高的同源性。然而在FAR1/FHY3基因家族中,仅有FHY3基因的功能及作用机理被研究,其他基因还未曾被关注,这些基因是否在phyA信号通路中,或者在棉花的生长发育过程中具有重要影响作用,还需要进一步的验证研究。
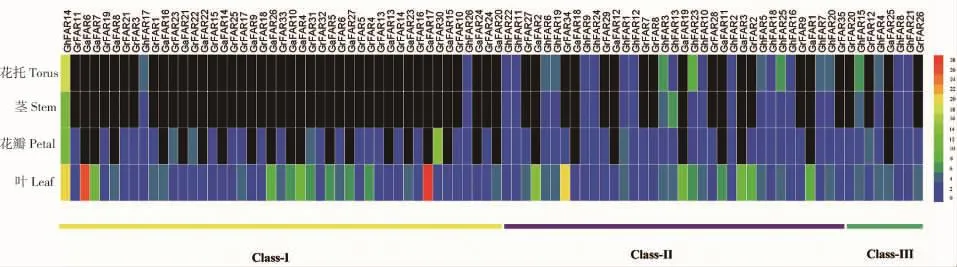
图5 FAR1/FHY3家族基因在棉花组织中的表达模式Fig.5 Expression patterns of FAR1/FHY3 gene family numbers in different cotton tissues
[1]Deng X W,Quail P H.Signalling in light-controlled development[J].Seminars in Cell&Developmental Biology,1999,10(2):121-129.
[2]Casal J J.Phytochromes,cryptochromes,phototropin:photoreceptor interactions in plants[J].Photochemistry&Photobiology,2000,71(1):1-11.
[3]Heijde M,Ulm R.UV-B photoreceptor-mediated signalling in plants[J].Trends in Plant Science,2012,17(4):230-237.
[4]刘圈炜,何云,齐胜利,等.光敏色素研究进展[J].中国农学通报,2005,21(5):237-241.Liu Quanwei,He Yun,Qi Shengli,et al.Advances in researches on phytochrome[J].Chinese Agricultural Science Bulletin,2005,21(5):237-241.
[5]Furuya M.Phytochromes:their molecular species,gene families,and functions[J].Annual Review of Plant Physiology and Plant Molecular Biology,1993,44(1):617-645.
[6]陈芳,邓兴旺.远红光受体伴侣蛋白FHY1在介导基因表达和植物生长发育过程中的独特作用[J].遗传,2014,36(9):958.Chen Fang,Deng Xingwang.Far red light receptor chaperone FHY1 plays a unique role in mediating gene expression and plant grow th and development[J].Hereditas(Beijing),2014,36(9):958.
[7]Lamparter T.Evolution of cyanobacterial and plant phytochromes[J].FEBS Letters,2004,573(1-3):1-5.
[8]Kircher S,Kozma-Bognar L,Kim L,et al.Light quality-dependent nuclear import of the plant photoreceptors phytochrome A and B[J].Plant Cell,1999,11(8):1445-1456.
[9]Tepperman J M,Zhu T,Chang H S,et al.Multiple transcription-factor genes are early targets of phytochrome A signaling[J].Proceedings of the National Academy of Sciences of the United States of America,2001,98(16):9437-9442.
[10]Lin R C,Ding L,Casola C,et al.Transposase-derived transcription factors regulate light signaling in Arabidopsis[J].Science,2007,318(5854):1302-1305.
[11]Hiltbrunner A.FHY1 and FHL act together to mediate nuclear accumulation of the phytochrome A photoreceptor[J].Plant&Cell Physiology,2006,47(8):1023-1034.
[12]Hudson M E,Lisch D R,Quail P H.The FHY3 and FAR1 genes encode transposase-related proteins involved in regulation of gene expression by the phytochrome A-signaling pathway[J].The Plant Journal,2003,34(4):453-471.
[13]Xu Z N,Dooner H K.Jittery,a mutator distant relative with a paradoxical mobile behavior:Excision without reinsertion[J].Plant Cell,2004,16(5):1105-1114.
[14]Lin R C,Wang H Y.Arabidopsis FHY3/FAR1 gene family and distinct roles of its members in light control of Arabidopsis development[J].Plant Physiology,2004,136(4):4010-4022.
[15]Lin R C,Teng Y B,Park H J,et al.Discrete and essential roles of the multiple domains of Arabidopsis FHY3 in mediating phytochrome A signal transduction[J].Plant Physiology,2008,148(2):981-992.
[16]马燕斌,吴霞,张林水,等.利用光敏色素基因改良棉花农艺经济性状的探讨 [C]//中国棉花学会2011年年会论文汇编.安阳:中国棉花杂志社,2011:71-73.Ma Yanbin,Wu Xia,Zhang Linshui,et al.Improvement of agronomic and economic characteristics of cotton by phytochrome genes[C]//The 2011 Annual Conference of China Society of Cotton Sci-Tech.Anyang:China Cotton Magazine House,2011:71-73.
[17]Thiele A,Herold M,Lenk I,et al.Heterologous expression of Arabidopsis phytochrome B in transgenic potato influences photosynthetic performance and tuber development[J].Plant Physiology,1999,120(1):73-82.
[18]Garg A K,Sawers R J,Wang H,et al.Light-regulated overex-pression of an Arabidopsis phytochrome A gene in rice alters plant architecture and increases grain yield[J].Planta,2006,223(4):627-636.
[19]Wang K B,Wang Z W,Li F G,et al.The draft genome of a diploid cotton Gossypium raimondii[J].Nature Genetics,2012,44(10):1098-1103.
[20]Li F G,Fan G Y,Wang K B,et al.Genome sequence of the cultivated cotton Gossypium arboreum[J].Nature Genetics,2014,46(6):567-572.
[21]Li F G,Fan G Y,Lu C R,et al.Genome sequence of cultivated upland cotton(Gossypium hirsutum TM-1)provides insights into genome evolution[J].Nature Biotechnology,2015,33(5):524-530.
[22]Zhang T Z,Hu Y,Jiang W K,et al.Sequencing of allotetraploid cotton(Gossypium hirsutum L.acc.TM-1)provides a resource for fiber improvement[J].Nature Biotechnology,2015,33(5):531-537.
[23]郭安源,朱其慧,陈新,等.GSDS:基因结构显示系统[J].遗传,2007,29(8):1023-1026.Guo Anyuan,Zhu Qihui,Chen Xin et al.GSDS:a gene structure display server[J].Hereditas(Beijing),2007,29(8):1023-1026.
[24]Kumar S,Stecher G,Tamura K.MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J].Molecular Biology&Evolution,2016,33(7):1870-1874.
[25]Adobe C.Adobe illustrator CS3[M].San Francisco:Adobe Press,2007.
[26]O'Brien K P,Remm M,Sonnhammer E L.Inparanoid:A com prehensive database of eukaryotic orthologs[J].Nucleic Acids Research,2005,33(Database issue):476-480.
[27]Wang D,Zhang Y,Zhang Z,et al.KaKs_calculator 2.0:A toolkit incorporating gamma-series methods and sliding w indow strategies[J].Genomics Proteomics Bioinformatics,2010,8(1):77-80.
[28]Trapnell C,Pachter L,Salzberg S L.TopHat:Discovering splice junctions with RNA-Seq[J].Bioinformatics,2009,25(9):1105-1111.
[29]Trapnell C,Roberts A,Goff L,et al.Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J].Nature Protocols,2012,7(3):562-578.
[30]Deng W K,Wang Y B,Liu Z X,et al.Hem I:A toolkit for illustrating heatmaps[J/OL].PLoS ONE,2014,9(11):e111988[2017-1-30].https://doi.org/10.1371/journal.pone.0111988.
[31]Quail P H.Phytochrome-regulated gene expression[J].Journal of Integrative Plant Biology,2007,49(1):11-20.
[32]Jiao Y,Lau O S,Deng X W.Light-regulated transcriptional networks in higher plants[J].Nature Reviews Genetics,2007,8(3):217-230.
[33]Ouyang X H,Li J G,Li G,et al.Genome-wide binding site analysis of FAR-RED ELONGATED HYPOCOTYL3 reveals its novel function in Arabidopsis development[J].Plant Cell,2011,23(7):2514-2535.
[34]Hurst L D.The Ka/Ksratio:Diagnosing the form of sequence evolution[J].Trends in Genetics,2002,18(9):486-487.
[35]Zhao L,Zhang N,Ma P F,et al.Phylogenomic analyses of nuclear genes reveal the evolutionary relationships within the BEP clade and the evidence of positive selection in Poaceae[J/OL].PLoS ONE,2013,8(5):e64642[2017-1-30].https://doi.org/10.1371/journal.pone.0064642.

