糯玉米opaque2基因近等基因系的创制
张晓星 朱 慧 张东民 宋丽雅 张德贵 翁建峰 郝转芳 李明顺,*
糯玉米基因近等基因系的创制
张晓星1朱 慧1张东民1宋丽雅2张德贵1翁建峰1郝转芳1李明顺1,*
1中国农业科学院作物科学研究所/ 农业部遗传育种重点实验室, 北京 100081;2北京工商大学/ 北京市植物资源研究开发重点实验室, 北京 100048
基因能够提高糯玉米赖氨酸、色氨酸等必需氨基酸的含量。用2种优质蛋白玉米(QPM) CA339和鲁2548自交系作为基因供体, 25个品质较好的糯玉米自交系作为受体, 利用回交技术和SSR分子标记辅助选择育种, 创制糯玉米近等基因系。分子标记结果表明, 不同受体以及两种供体间都存在多态性。5套创制成功的近等基因系的赖氨酸含量比其轮回亲本分别提高了59.0%、52.7%、48.5%、46.3%和61.9%, 分别由0.308%、0.313%、0.309%、0.341%、0.323%提高到0.489%、0.478%、0.458%、0.498%、0.522%。本研究表明, 可以利用此方法通过向不同遗传背景的多种受体导入, 选取赖氨酸含量提高较大、透明表型的近等基因系, 提高糯玉米的营养价值和经济价值。
糯玉米;基因;基因; 近等基因系; 赖氨酸
糯玉米起源于中国, 因其口感软糯、香甜适宜鲜食, 深受我国人民的喜爱。糯玉米的糯质主要源于隐性基因, 如果基因发生突变[1-2], 将导致颗粒紧密结合型淀粉酶I (GBSS I)表达受到抑制, 使胚乳中直链淀粉合成受阻。由于糯玉米胚乳中的淀粉绝大部分为支链淀粉, 所以糯玉米也是饲料、医药、食品、酿造、纺织、造纸等行业的重要原料, 具有很高的经济价值[3]。但是糯玉米的营养价值并不完全, 赖氨酸、蛋氨酸、色氨酸等必需氨基酸含量较少[4], 不能满足人畜对赖氨酸的需求[5], 使糯玉米的推广和应用受到了一定限制。所以在育种中提高糯玉米的赖氨酸含量, 对完善其营养品质及提高其食用价值具有重要意义。
近年来, 将()突变基因导入普通玉米是提高赖氨酸含量最有效的方法[6]。基因位于玉米第7染色体的短臂[7-8], 编码的O2蛋白属于碱性亮氨酸拉链家族[9-11], 是一个转录调节蛋白, 能够转录激活多个醇溶蛋白基因。经前人研究, 由于突变导致转录功能受到抑制, 使醇溶蛋白合成减少, 非醇溶蛋白含量相应增加, 从而使玉米籽粒的赖氨酸含量增加[12-13]。但是由于突变体籽粒的粉质、不透明、抗病性差等农艺性状, 限制了其应用和推广[14]。国际小麦玉米改良中心(CIMMYT)的科研人员培育出了优质蛋白玉米(QPM), 利用胚乳修饰基因将高赖氨酸玉米的粉质胚乳改良为硬质胚乳[15]。我国育种家也成功培育出CA335、CA339、齐205、鲁2548等QPM自交系, 既提高了玉米籽粒的赖氨酸含量, 又将玉米胚乳改良为硬质型, 籽粒产量接近或达到普通玉米杂交种[16]。
目前将基因导入糯玉米的研究还较少, 研究表明, 糯玉米中导入基因后, 籽粒赖氨酸含量会有不同程度的提高[17-18]。本研究通过供体材料CA339和鲁2548, 将导入25份口感好、品质优良、农艺性状稳定、抗病性强的糯玉米自交系, 利用SSR分子标记辅助育种技术监控在子代中的传递, 创制糯玉米基因近等基因系, 利用提高糯玉米的赖氨酸含量, 从而提高糯玉米的营养品质, 为后续研究-NILs在籽粒形态、营养成分、O2蛋白等方面的变化奠定基础。
1 材料与方法
1.1 试验材料
选取25个口感好、品质优良、农艺性状稳定、抗病性强的糯玉米自交系作为受体亲本, 优质蛋白玉米CA339和鲁2548作为基因的供体亲本。以上材料均由中国农业科学院作物科学研究所玉米优质抗逆课题组提供, 详细材料见表1。
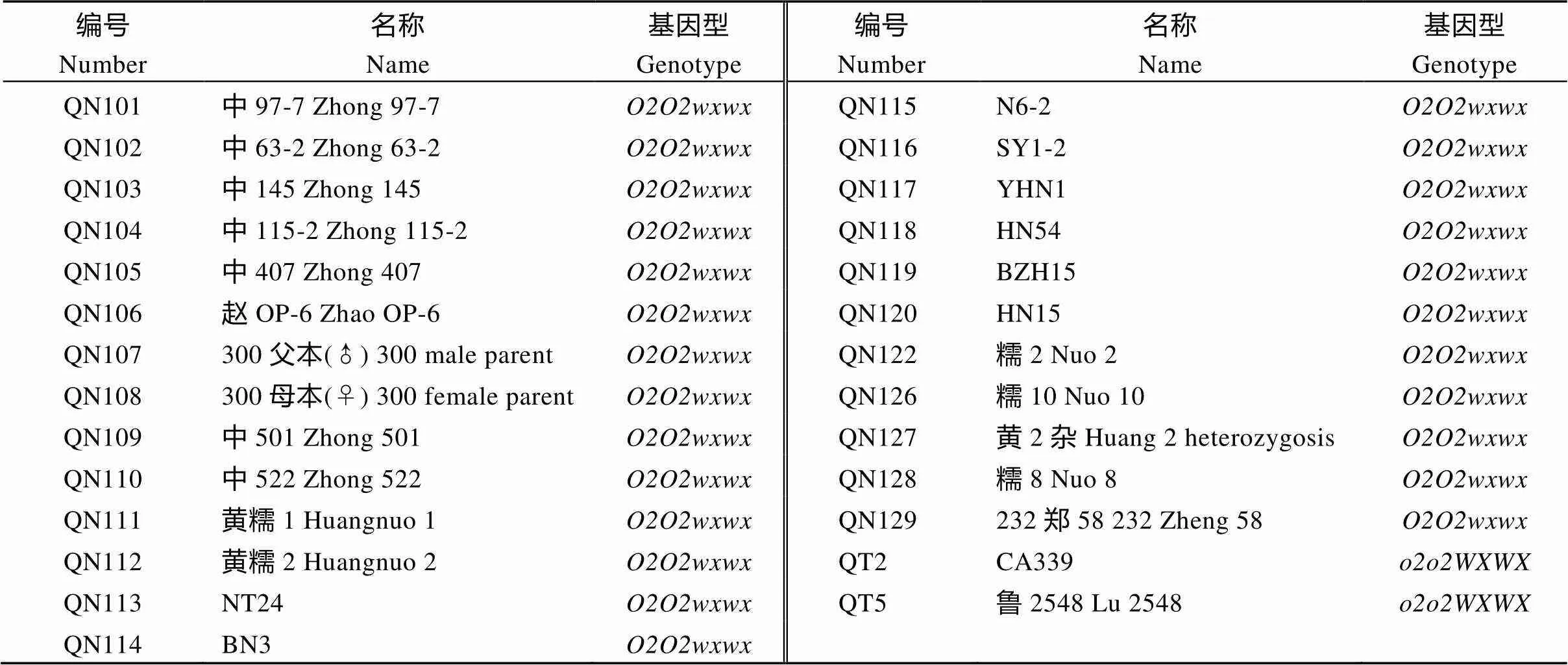
表1 试验材料
1.2 试验方法
1.2.1 田间种植与取样 试验材料于2008—2016年夏季种植于北京, 冬季种植于海南三亚,-NILs创制过程中分别种植2~5行供受体亲本, 子代群体单穗播种1~2行, 行长3 m, 株距25 cm。在-NILs创制过程中, 子代群体只保留经SSR分子标记选择基因型正确且植株表型与受体亲本植株表型相似的单株, 并严格进行杂交或自交授粉。
1.2.2-NILs创制流程 利用分子标记辅助选择创制-NILs流程如图1所示。以QPM自交系作为供体亲本, 与25个糯玉米自交系杂交, 以受体材料作为轮回亲本回交5~7代, 用SSR引物phi057、phi027选择基因型为且表型与受体亲本相似的杂合植株自交, 最后自交多代选择基因纯合且稳定遗传的植株, 直至成功创制-NILs。
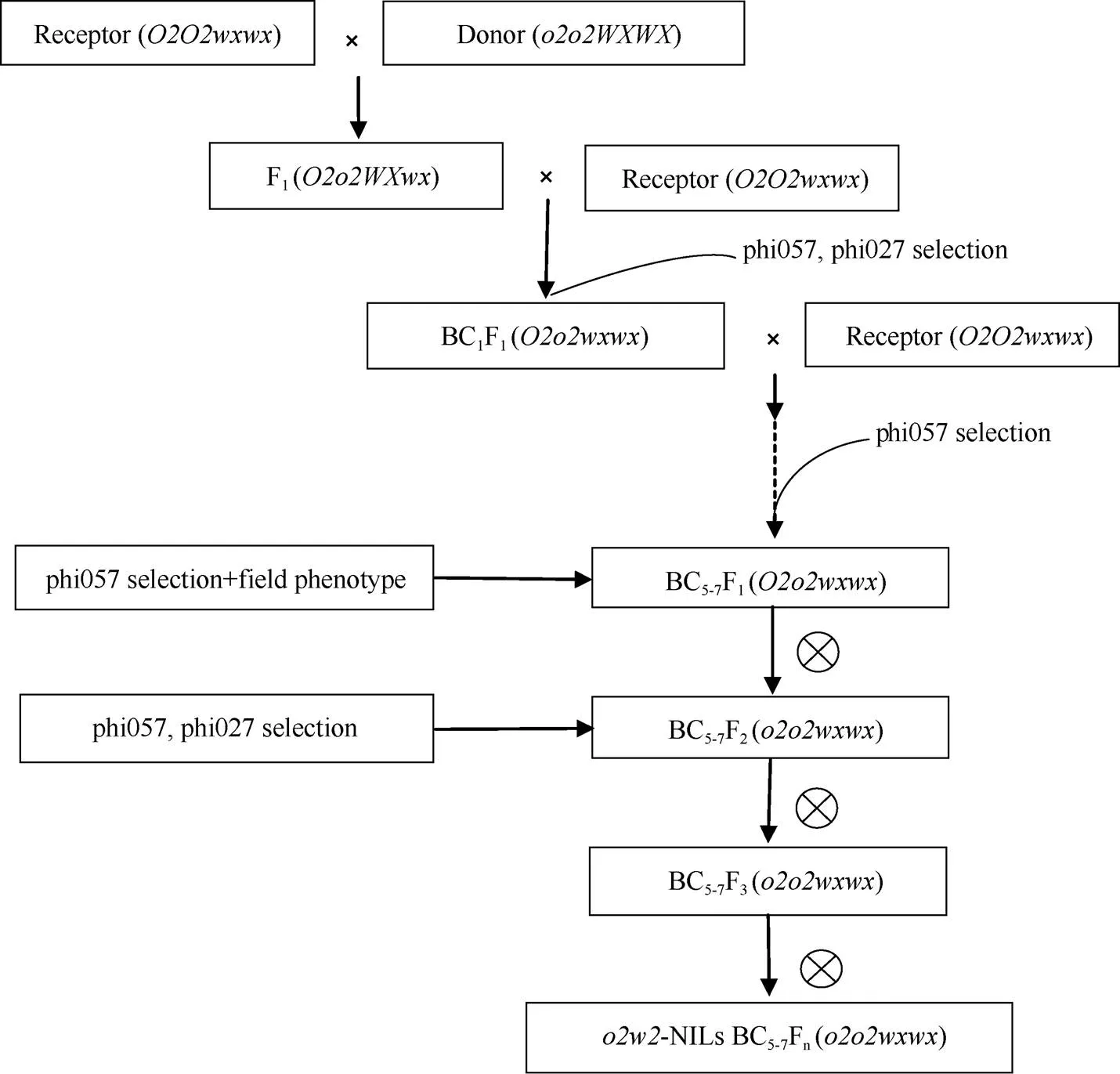
图1 o2w2-NILs创制流程图
1.2.3 玉米叶片基因组DNA的提取 植株生长到6~7叶时的单株挂牌, 取1 cm2较幼嫩的叶片装入2.0 mL离心管, 并加入2个小钢珠, 放入液氮冷冻后用研磨机将样品打碎成粉末状, 采用CTAB法[19]提取叶片基因组DNA, 用灭菌ddH2O将DNA粉末溶解。使用NanoDrop 2000检测DNA浓度, 将DNA浓度稀释到100 ng µL–1左右。
1.2.4 SSR分子标记检测基因型 引物phi057是由先锋公司开发的基因内的特异引物, 能够区分显性纯合、杂合和隐性纯合3种基因型; 引物phi027可特异性检测基因型, 在回交过程中用引物phi027检测子代的糯性。引物序列来自MaizeGDB数据库, 由北京天一辉远生物科技有限公司合成, 引物序列见表2。
PCR体系10 µL, 含1.5 µL 10×buffer、0.8 µL dNTP (25 mmol L–1)、0.3 µL引物(20 µmol L–1)、0.2 µLDNA聚合酶(2 U µL–1)、1.0 µL DNA模板和5.9 µL ddH2O。10×buffer、dNTP、DNA聚合酶购自天根生化科技(北京)有限公司。

表2 SSR引物信息
使用降落式PCR扩增引物phi057, 反应程序为94°C预变性5 min; 94°C变性30 s, 61°C退火30 s, 72°C延伸20 s, 5个循环; 94°C变性30 s, 59°C退火30 s, 72°C延伸20 s, 10个循环; 94°C变性30 s, 57°C退火30 s, 72°C延伸20 s, 20个循环; 72°C延伸10 min, 12°C保存。引物phi027使用普通式PCR扩增, 94°C预变性3 min; 94°C变性30 s, 61°C退火30 s, 72°C延伸20 s, 35个循环; 72°C延伸10 min, 12°C保存。PCR仪为伯乐公司的C2000ThermalCycler。
在PCR产物中加入1×终浓度的loading buffer, 配置8%非变性聚丙烯酰胺凝胶电泳。
1.2.5 籽粒赖氨酸含量测定 选取中97-7/、中63-2/、赵OP-6/、300父本/、中501/这5套创制成功的-NILs及其受体亲本和供体亲本CA339, 称取成熟期干燥籽粒各100 g, 送农业部谷物品质监督检验测试中心, 测定玉米籽粒赖氨酸含量。
2 结果与分析
2.1 SSR分子标记检测供体和受体亲本遗传多态性
利用引物phi057检测供受体亲本基因的遗传多态性。如图2所示, 引物phi057在25种糯玉米受体亲本间可以扩增出3类不同大小的条带, 泳道C、17、19所代表的CA339、YHN1、BZH15是一类, 泳道D、3、8、10所代表的鲁2548、中145、300母本(♀)、中522是一类, 其他是一类, 多态性较好。2个供体亲本CA339和鲁2548为隐性纯合基因型, 受体亲本都为显性纯合基因型, 为保证引物phi057在基因上的共显性以便于SSR分子标记筛选, 在创制-NILs时, 根据遗传多态性检测结果配置杂交组合, 受体亲本YHN1和BZH15与鲁2548杂交, 其余糯玉米受体亲本与CA339杂交。
2.2 回交及自交过程中利用引物phi057筛选单株
在创制-NILs的回交过程中, 用引物phi057对每一单株的基因型进行检测, 根据SSR分子标记结果筛选杂合基因型植株作为非轮回亲本, 与轮回亲本回交。图3是BC5F1世代部分群体的检测结果。从电泳结果可以看出, 泳道1、3、7、8、9、10、11、12、13、14、18、20、21、22为杂合基因型, 泳道2、4、5、6、15、16、17、19为显性纯合基因型。在田间砍除显性纯合基因型的植株, 筛选杂合基因型且表型与轮回亲本相似的植株继续回交或自交。
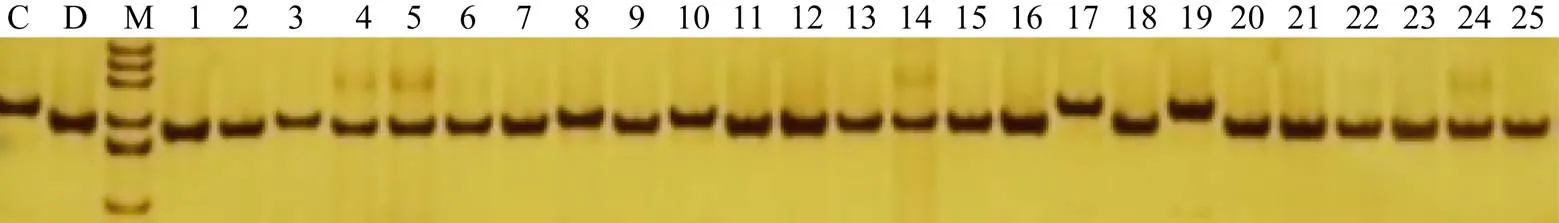
图2 引物phi057检测供体和受体亲本间的遗传多态性
C: CA339; D: 鲁2548; M: DNA marker; 1~25: 25个糯玉米自交系。
C: CA339; D: Lu 2548; M: DNA marker; 1-25: 25 waxy maize inbred lines.
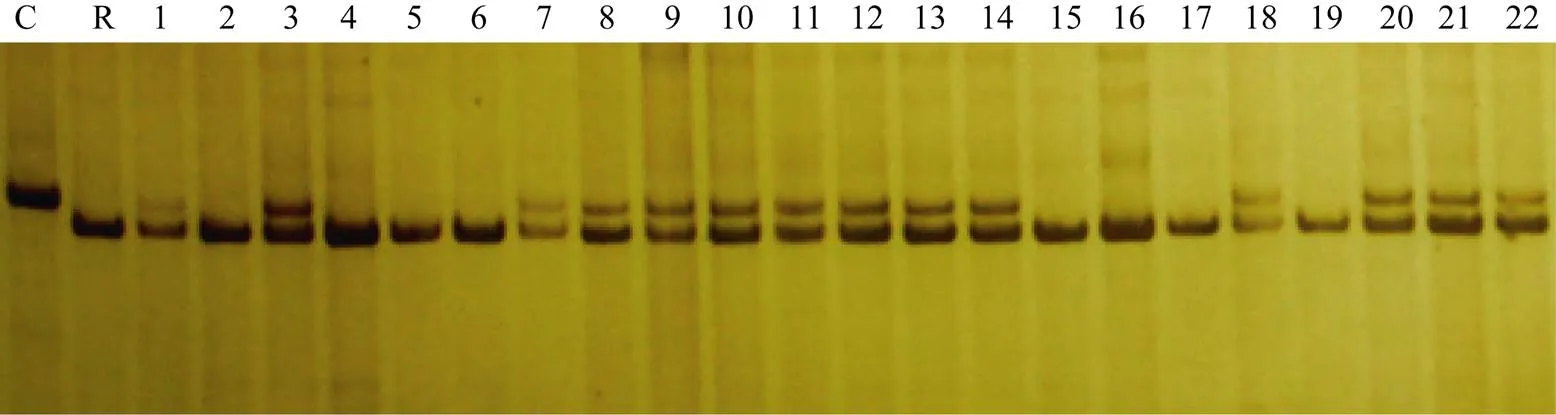
图3 回交过程中引物phi057检测糯玉米自交系BC5F1世代基因型
C: CA339; R: 轮回亲本; 1~22: BC5F1世代不同单株。
C: CA339; R: Recurrent parent; 1-22: Different individuals from BC5F1family.
将BC5F1回交世代的杂合植株进行严格的自交授粉, 将获得的BC5F2世代分离群体播种后, 用引物phi057对每一单株的基因型进行检测。图4是BC5F2世代部分群体的检测结果。可以看出, 泳道6、9、10、17、24带型与CA339带型一致, 是隐性纯合基因型, 泳道1、2、3、4、5、7、11、13、14、15、16、18、19、20、21、22、23为杂合基因型, 泳道8、12、25带型与轮回亲本带型一致, 是显性纯合基因型。筛选基因型为且田间表型与其轮回亲本相似的植株继续自交。
2.3 o2w2-NILs基因型检测
对19套创制成功的-NILs进一步用引物phi057进行SSR检测, 其带型与供体亲本带型一致, 即确保都是隐性纯合基因型。图5所示是已创制成功的-NILs部分个体引物phi057检测结果, 试验证明基因已成功导入性状优良的糯玉米自交系。用引物phi027对创制成功的-NILs的基因型进行检测, 结果如图6所示, 其带型与受体亲本带型一致, 全部为隐性纯合基因型。

图4 自交过程中引物phi057检测糯玉米自交系BC5F2世代基因型
C: CA339; R: 轮回亲本; 1~25: BC5F2世代不同单株。
C: CA339; R: Recurrent parent; 1-25: Different individuals from BC5F2family.
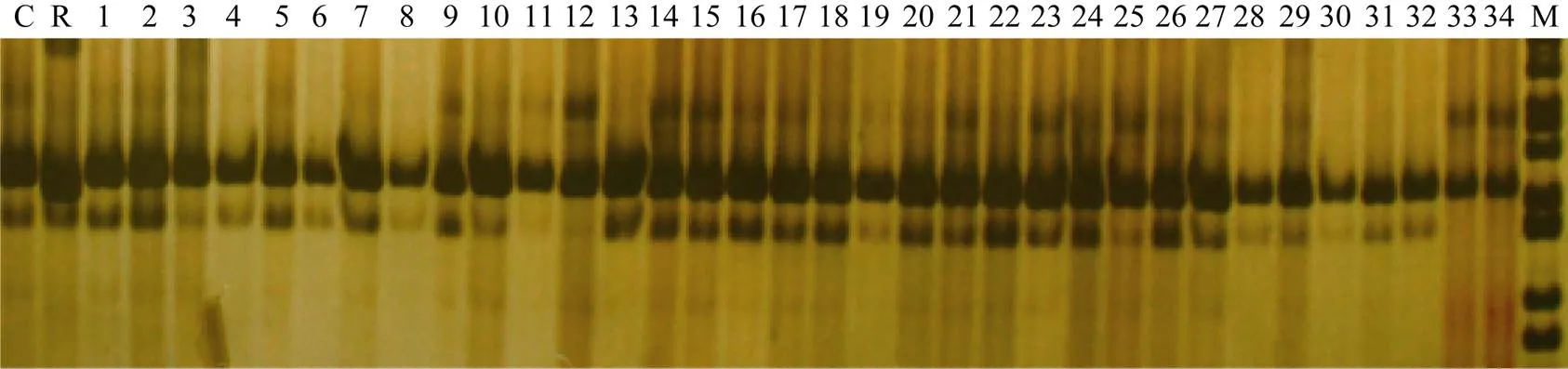
图5 引物phi057检测BC5F3世代基因型
C: CA339; R: 轮回亲本; 1~34: BC5F3世代不同单株; M: DNA marker。
C: CA339; R: Recurrent parent; 1-34: Different individuals from BC5F3family; M: DNA marker.
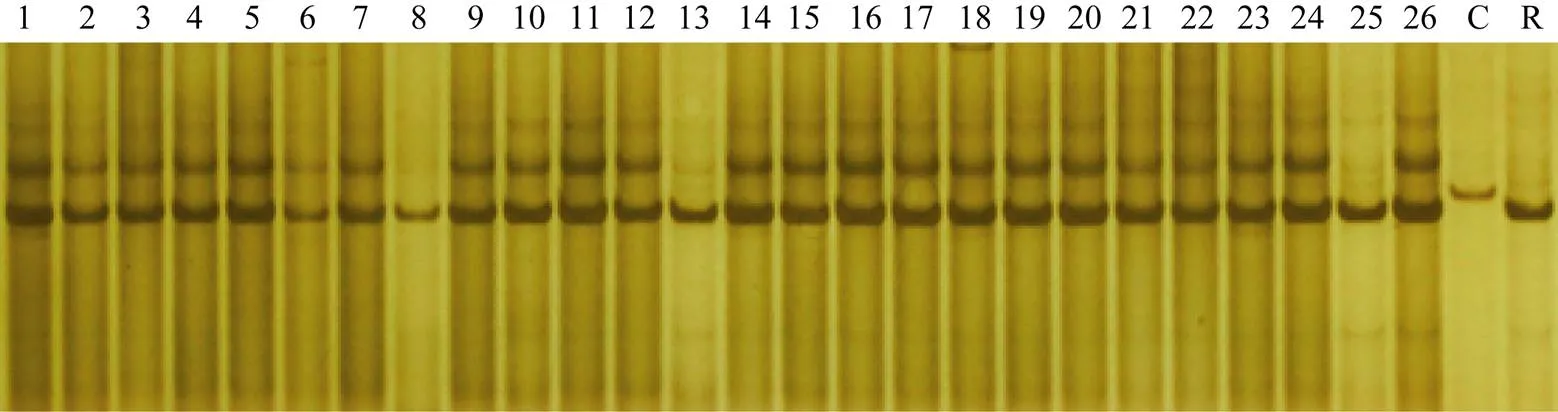
图6 引物phi027检测BC5F3世代基因型
1~26: BC5F3世代不同单株; C: CA339; R: 轮回亲本。
1-26: Different individuals from BC5F3family; C: CA339; R: Recurrent parent.
2.4 o2w2-NILs及其供受体亲本籽粒赖氨酸含量检测
对中97-7/、中63-2/、赵OP-6/、300父本/、中501/这5套创制成功的-NILs及其受体亲本和供体亲本CA339的成熟期干燥籽粒赖氨酸含量检测(图7)表明,供体亲本CA339的籽粒赖氨酸含量为0.453%, 比5个轮回亲本都高, 而且5套-NILs的籽粒赖氨酸含量比其受体亲本均显著提高, 分别由0.308%、0.313%、0.309%、0.341%、0.323%提高到0.489%、0.478%、0.458%、0.498%、0.522%, 分别提高了59.0%、52.7%、48.5%、46.3%和61.9% (<0.01)。说明基因的导入使糯玉米自交系的赖氨酸含量有了较大的提高, 增加了其营养价值。
3 讨论
玉米品质的改良一直受到育种家的重视, 糯玉米具有很高的营养价值和经济价值, 但是缺少赖氨酸、蛋氨酸、色氨酸等必需氨基酸, 使糯玉米的应用受到了限制。遗传学家在20世纪初在玉米中发现了突变基因, 能够使玉米籽粒中赖氨酸含量提高, 但是突变体产量低、抗逆性差、籽粒为粉质等农艺性状阻碍了其商业化生产。CIMMYT的科研人员利用胚乳修饰基因, 将高赖氨酸玉米的粉质胚乳改良为硬质胚乳, 育成优质蛋白玉米(QPM)[20], 促进了突变基因在玉米育种和生产中的应用。
我国的QPM育种工作也取得了一定的进展, 培育出CA335、CA339、齐205、鲁2548等QPM自交系, 可使玉米籽粒的赖氨酸含量提高到0.4%左右。利用分子标记辅助选择技术将糯玉米自交系转育为优质蛋白糯玉米自交系, 是一种高效、快速的改良糯玉米品质的方法。20世纪末, 先锋公司[21]和密苏里大学开发出基因的3对SSR特异性引物, phi057、phi112和umc1066。引物phi057是共显性标记, 能够区分显性纯合、杂合和隐性纯合基因型, 比另2种引物的标记效果更好[22-23]。在创制-NILs时, 必须先检测供体亲本和受体亲本之间的多态性, 如图2所示, 必须选择具有多态性的两个亲本进行杂交, 否则在后代进行分子标记筛选单株时无法确定基因型, 选择正确的杂交组合是创制-NILs的关键。
本试验中检测了5套-NILs的籽粒中赖氨酸含量, 其供体亲本都是CA339, 赖氨酸含量较其受体亲本都有了较大幅度的提高, 普通糯玉米自交系的赖氨酸含量平均为0.318%,-NILs的赖氨酸含量平均为0.489%。但是在不同遗传背景中提高的幅度不同, 最低的提高了46.3%, 最高的提高了61.9%, 这说明赖氨酸含量提高的不一致可能与其受体亲本有关。
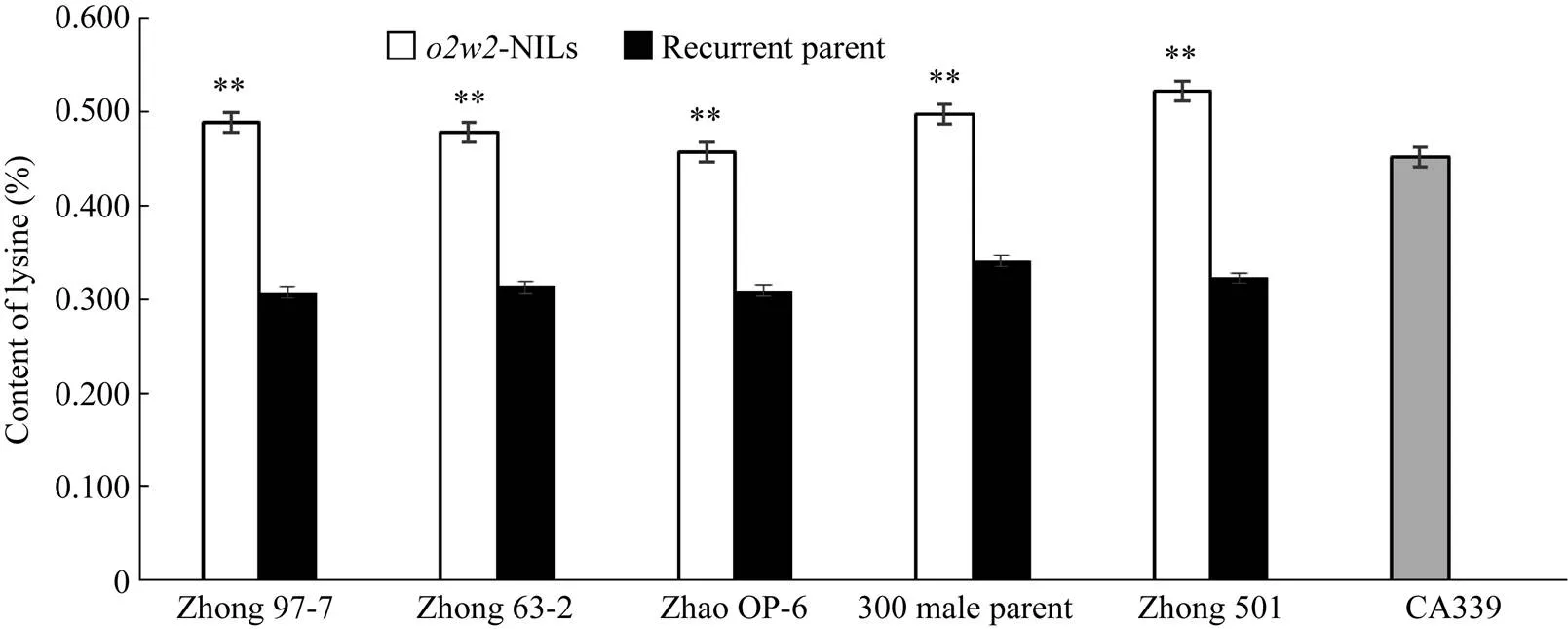
图7 赖氨酸含量检测结果
**表示差异达到0.01显著水平。
** indicates significance at the 0.01 probability level.
4 结论
通过SSR分子标记监控突变基因在子代中的传递, 把导入到口感好、品质优良、抗病性强的糯玉米自交系中, 构建玉米双隐性-NILs, 建立了一套在糯玉米中导入的分子标记辅助育种方法。通过此方法向不同遗传背景的多种受体导入, 选取赖氨酸含量提高较大、表型透明的-NILs, 提高了糯玉米的营养价值和商品价值。
[1] Briggs R W, Amano E, Smith H H. Genetic recombination with ethylmethane sulphonate inducedmutants in maize., 1965, 207: 890–891
[2] Wessler S R, Tarpley A, Purugganan M, Spell M, Okagaki R. Filler DNA is associated with spontaneous deletions in maize., 1990, 87: 8731–8735
[3] 彭泽斌, 田志国. 我国糯玉米产业现状与发展战略. 玉米科学, 2004, 12(3): 116–118 Peng Z B, Tian Z G. Industrialization present status and strategies for development of waxy corn in China., 2004, 12(3): 116–118 (in Chinese with English abstract)
[4] 杨引福, 郭强, 陈婧, 郑小亚, 蔺崇明. 中国温带糯玉米自交系遗传品质性状分析. 西北植物学报, 2009, 29: 2213–2220 Yang Y F, Guo Q, Chen J, Zheng X Y, Lin C M. Analysis of genetic and quality traits of waxy corn inbred lines in China temperate zone., 2009, 29: 2213–2220 (in Chinese with English abstract)
[5] 田清震, 李新海, 李明顺, 姜伟, 张世煌. 优质蛋白玉米的分子标记辅助选择. 玉米科学, 2004, 12(2): 108–110 Tian Q Z, Li X H, Li M S, Jiang W, Zhang S H. Molecular markers assisted selection to quality protein maize., 2004, 12(2): 108–110 (in Chinese with English abstract)
[6] Mertz E T, Bates L S, Nelson O E. Mutant gene that changes protein composition and increases lysine content of maize endosperm., 1964, 145: 270–280
[7] Hartings H, Maddaloni M, Lazzaroni N, Fonzo N D, Motto M, Salamini F, Thompson R. Thegene which regulates zein deposition in maize endosperm encodes a protein with structural homologies to transcriptional activators., 1989, 8: 2795–2801
[8] Maddaloni M, Fonzo N D, Hartings H, Lazzaroni N, Salamini F, Thompson R, Motto M. The sequence of the zein regulatory gene-() of., 1989, 17: 7532
[9] Schmidt R J, Burr F A, Aukerman M J, Burr B. Maize regulatory geneencodes a protein with a “leucine-zipper” motif that binds to zein DNA., 1990, 87: 46–50
[10] Schmidt R J, Ketudat M, Aukerman M J, Hoschek G.is a transcriptional activator that recognizes a specific target site in 22-kD zein genes., 1992, 4: 689–700
[11] Schmitz D, Lohmer S, Salamini F, Thompson R D. The activation domain of the maize transcription factorresides in a single acidic region., 1997, 25: 756–763
[12] Hunter B G, Beatty M K, Singletary G W, Hamaker B R, Dilkes B P, Larkins B A, Junq R. Maize opaque endosperm mutations create extensive changes in patterns of gene expression., 2002, 14: 2591–2612
[13] Viotti A, Sala E, Marotta R, Alberi P, Balducci C, Soave C. Genes and mRNAs coding for zein polypeptides in., 1979, 102: 211–222
[14] Crow J F, Kermicle J. Oliver Nelson and quality protein maize., 2002, 160: 819–821
[15] Vasal S K. High quality protein corn. In: Hallawer A R ed. Specialty Corns. Boca Raton: CRC Press, 2001. pp 85–121
[16] 宋敏, 张世煌. 优质蛋白玉米(QPM)育种研究进展. 新疆农业科学, 2007, 44(增刊3): 30–35 Song M, Zhang S H. Comprehensive report on study advance of breeding of high-quality protein maize (QPM)., 2007, 44(suppl-3): 30–35 (in Chinese)
[17] Zhang W, Yang W, Wang M, Wang W, Zeng G, Chen Z, Cai Y. Increasing lysine content of waxy maize through introgression ofandgenes using molecular assisted and biochemical development., 2013, 8: e56227
[18] 昂沃, 郝小琴, 吴子恺.基因导入各类隐性纯合体玉米早代籽粒赖氨酸含量的效应研究. 玉米科学, 2012, 20(2): 56–58 Ang W, Hao X Q, Wu Z K. Analysis ofgene effects on kernel lysine content into some early generations’ homozygote recessive maize., 2012, 20(2): 56–58 (in Chinese with English abstract)
[19] 卢振宇, 李明顺, 谢传晓, 李新海, 曲延英, 张世煌. 玉米叶片DNA快速提取方法改进研究. 玉米科学, 2008, 16(2): 50–53 Lu Z Y, Li M S, Xie C X, Li X H, Qu Y Y, Zhang S H. An improved study on rapid DNA extraction method from young leaves of maize., 2008, 16(2): 50–53 (in Chinese with English abstract)
[20] Vasal S K. The quality protein maize story., 2000, 21: 445–450
[21] Chin E C, Senior M L, Shu H, Smith J S. Maize simple repetitive DNA sequences: abundance and allele variation., 1996, 39: 866–873
[22] Chen Y, Zhou Z, Zhao G, Li X, Song L, Yan N, Weng J, Hao Z, Zhang D, Li M, Zhang S. Transposable elementinduces the differential expression ofmutant gene in two maizeNILs derived from the same inbred line., 2014, 9: e85159
[23] 赵刚, 吴子恺, 陈亮, 张德贵, 张世煌, 卢振宇, 白丽, 李明顺. 几个供体对优质蛋白玉米(QPM)近等基因系构建效果的比较. 玉米科学, 2010, 18(6): 11–14Zhao G, Wu Z K, Chen L, Zhang D G, Zhang S H, Lu Z Y, Bai L, Li M S. Comparison of several donor inbred lines on development of quality protein maize (QPM) near-isogenic lines., 2010, 18(6): 11–14 (in Chinese with English abstract)
Construction of Waxy MaizeNear-isogenic Lines
ZHANG Xiao-Xing1, ZHU Hui1, ZHANG Dong-Min1, SONG Li-Ya2, ZHANG De-Gui1, WENG Jian-Feng1, HAO Zhuan-Fang1, and LI Ming-Shun1,*
1Institute of Crop Science, Chinese Academy of Agricultural Sciences / Key Laboratory of Crop Genetics and Breeding, Ministry of Agriculture, Beijing 100081, China;2Beijing Technology and Business University / Beijing Key Laboratory of Plant Resources Research and Development, Beijing 100048, China
In waxy maize, thegene can increase the content of essential amino acids, such as lysine and tryptophan. Two high quality protein maize (QPM) inbred lines, CA339 and Lu 2548, were used asgene donors. Twenty-five waxy maize with advantageous traits were used as receptors. Application of backcrossing and SSR marker-assisted selection in breeding can create waxy maizenear-isogenic lines. The results of molecular marker indicated that there were polymorphisms in different receptors and between the two donors. Lysine content of five sets of successfully creatednear-isogenic lines was tested, showing that the content of lysine in-NILs was 59.0%, 52.7%, 48.5%, 46.3%, and 61.9% higher than that in their recurrent parents, which was increased from 0.308%, 0.313%, 0.309%, 0.341%, 0.323% to 0.489%, 0.478%, 0.458%, 0.498%, and 0.522%, respectively. Using this method,could be introgressed into multiple receptors with different genetic backgrounds to select near-isogenic lines with high lysine content and transparent phenotype, so as to improve nutritional and economic values of waxy maize.
Waxy maize;gene;gene; Near-isogenic lines; Lysine
10.3724/SP.J.1006.2017.01760
本研究由国家自然科学基金项目(31401390)和国家现代农业产业技术体系建设专项(CAR02-01)资助。
This study was supported by the National Natural Science Foundation of China (31401390) and the China Agriculture Research System (CAR02-01).
李明顺, E-mail: limingshun@caas.cn, Tel: 010-82108747
E-mail: zhangxiaoxing0401@163.com
2017-03-09; Accepted(接受日期): 2017-07-23; Published online(网络出版日期): 2017-08-10.
http://kns.cnki.net/kcms/detail/11.1809.S.20170810.1616.004.html

