奶牛源金黄色葡萄球菌的分离鉴定及氟苯尼考耐药基因fexA的克隆
赵素君,曹 冶,谢 晶,于吉峰,李江凌,王秋实,廖党金
(1.四川省畜牧科学研究院,四川成都 610066;2.动物遗传育种四川省重点实验室,四川成都 610066)
奶牛源金黄色葡萄球菌的分离鉴定及氟苯尼考耐药基因fexA的克隆
赵素君1,2*,曹 冶1,2,谢 晶1,2,于吉峰1,2,李江凌1,2,王秋实1,2,廖党金1,2
(1.四川省畜牧科学研究院,四川成都 610066;2.动物遗传育种四川省重点实验室,四川成都 610066)
从100个奶牛场环境及脓汁样品中分离、鉴定了9株奶牛源耐氟苯尼考金黄色葡萄球菌,分别提取质粒DNA和基因组DNA,通过特异性引物进行PCR扩增氟苯尼考耐药基因fexA,以质粒DNA为模板的PCR分别扩增出了1 446 bp特异性目的片段,而以基因组DNA为模板的PCR未扩增出该目的片段。将该目的片段克隆到pET-32(a)载体上,成功构建了pET-fexA质粒载体,将质粒载体转入BL21中,药物敏感性试验显示构建的pET-fexA/BL21基因工程菌对氯霉素和氟苯尼考高度耐药。同时,fexA阳性金黄色葡萄球菌分离株对氯霉素和氟苯尼考的耐药性显著高于质控菌株,说明fexA基因贡献细菌对氯霉素和氟苯尼考的交叉耐药。成功构建了一个基因背景清楚且对氟苯尼考耐药的细菌模型,排除了细菌中其他氟苯尼考耐药基因或泵出蛋白对fexA基因贡献细菌耐药的影响,为fexA基因编码的蛋白定位研究奠定了基础。
金黄色葡萄球菌;氟苯尼考;fexA基因;奶牛
近年来,我国奶牛业向规模化、集约化方向发展,奶牛饲养规模逐渐扩大。奶牛乳房炎是阻碍奶牛养殖业发展的最主要的疾病之一,导致严重的经济损失。金黄色葡萄球菌(Staphylococcusaureus,SA)是一种重要的人畜共患病病原,是奶牛乳房炎三大主要病原菌之一,同时也能危害奶牛的生产性能,造成子宫内膜炎及其他疾病[1-4]。氟苯尼考是氯霉素类广谱抗生素,由其同系物甲砜霉素分子结构改造而来,具有更好的安全性和抗菌效果,是氯霉素和甲砜霉素的理想替代物。20世纪90年代以来氟苯尼考开始在全球范围内使用,因其广谱、高效、吸收迅速、分布广泛、安全等特点,成为应用最广泛的兽用抗生素之一,常用于兽医临床金黄色葡萄球菌感染的治疗。但随着用药量的增多,耐药性问题也日渐严重,一方面给临床治疗带来困难,另一方面动物源耐药菌还可通过食物链感染人体或将耐药基因转移至人体病原菌,给食品安全和人体健康带来潜在危害,也给研究者提出了新的研究热点。
目前,在葡萄球菌中已发现cfr和fexA2种氟苯尼考耐药基因。2004年,Kehrenberg等[5]首次报道了从缓慢葡萄球菌的质粒pSCFS2上克隆到一种新的耐氟苯尼考fexA基因,编码的蛋白由475个氨基酸组成,有14个跨膜区。fexA基因是首个从革兰阳性球菌鉴定出的编码外排泵的基因,是E-4族成员,与其他氯霉素类药物的外排泵基因同源性很低,可同时介导对氯霉素和氟苯尼考耐药。与其他外排蛋白不同的是,在fexA基因上游存在1个类似衰减子的结构,因此fexA基因的诱导表达可通过翻译衰减来进行调节。自该基因首次发现后,动物源和人源葡萄球菌的耐氟苯尼考fexA基因相继报道。有研究在12株葡萄球菌中检测到fexA基因,其中有3株分别为猪源、人源、马源金黄色葡萄球菌[6];Argudin M A等[7]在猪、猪场的土壤、牛奶及肉品中的金黄色葡萄球菌中也发现了fexA基因。大量研究表明,除了动物源细菌,人源的氟苯尼考耐药菌株也不断被发现[8-9],可见氟苯尼考的耐药性问题日益严重,研究其耐药机制,对于食品安全和人类健康具有重要的公共卫生学意义。本研究从四川规模化奶牛场的环境样本中分离耐氟苯尼考金黄色葡萄球菌,检测氟苯尼考耐药基因fexA并对其耐药性进行分析,为金黄色葡萄球菌对氟苯尼考耐药机制的深入研究提供理论依据,从而更好地控制耐药性传播,保障养殖业健康发展。
1 材料与方法
1.1 材料
1.1.1 菌株 金黄色葡萄球菌CVCC3055购自中国兽医药品检查所中国兽医微生物菌种保存管理中心,作为质控菌株。大肠埃希菌JM109、BL21为动物遗传育种四川省重点实验室保存。
1.1.2 样品采集 采集四川省5个不同的奶牛养殖场的环境及化脓组织样品100个。
1.1.3 主要试剂TaqDNA聚合酶,dNTPs,DL2000 DNA Marker,限制性内切酶SacⅠ、BamHⅠ,为宝生物工程(大连)有限公司产品;细菌基因组DNA提取试剂盒,质粒提取试剂盒,DNA凝胶回收试剂盒,为AXYGEN公司产品;pET-32(a)载体,为Novagen公司产品;氟苯尼考、氯霉素等,为杭州微生物试剂有限公司产品。其他试剂均为国产分析纯试剂;引物合成及核酸测序由Invitrogen公司完成。
1.1.4 引物设计与合成 根据GenBank中葡萄球菌耐氟苯尼考fexA基因、金黄色葡萄球菌编码耐热核酸酶的nuc基因序列设计引物如下:fexA-F:5′-GATGAGCTCATGAAAAAGGATAGTAAATCTAAAG-3′(含SacⅠ酶切位点),fexA-R:5′-CGCGGATCCTTACCCACGCTTTTTTAAACCT-3′(含BamHⅠ酶切位点),扩增目的片段长度为1446 bp;nuc-F:5′-TTAGCCAAGCCTTGACGA- AC-3′,nuc-R:5′-AGGGCAATACGCAAAGAGGT-3′,扩增目的片段长度为480 bp。
1.2 方法
1.2.1 病原菌分离与纯化 在无菌条件下,从养殖粪污及化脓组织取样。粪污样品先用1 mL的生理盐水稀释,再吸取100 μL置于灭菌的高盐LB肉汤中,37℃培养10 h~12 h;用接种环挑取菌液划线于含8 mg/L氟苯尼考的甘露醇氯化钠琼脂培养基平板上,37℃培养18 h~24 h。将疑似葡萄球菌单菌落划线接种于血琼脂平板,于37℃恒温培养16 h,挑取单个优势菌落在血琼脂平板上再次划线,获得纯培养的菌株。
1.2.2 病原菌的形态学观察及生化鉴定试验 将菌株接种于琼脂培养基中,于37℃培养16 h后观察菌落的大小、形态,同时革兰染色,光学显微镜观察细菌形态特征,并采用全自动微生物鉴定系统(bioMerieux公司,VITEK 2 Compact System)进行生化鉴定。
1.2.3fexA基因的PCR扩增及携带fexA基因的阳性菌株的鉴定 将上述分离的疑似金黄色葡萄球菌菌株进行培养,分别提取质粒DNA和基因组DNA,通过PCR扩增目的基因fexA,以质控菌为对照。对携带fexA基因的菌株,进一步进行nuc基因的测定,来确定金黄色葡萄球菌菌株。nuc基因是耐热核酸酶的编码基因,为金黄色葡萄球菌所特有,而且在不同菌株之间具有较高的保守性,因此,很多研究以nuc基因作为扩增的靶基因[10-11]。
PCR反应体系:10×ExTaqbuffer(含MgCl2)2.5 μL,dNTP mixture(各2.5 mmol/L)2 μL,引物(20 μmol/L)各0.5 μL,模板DNA 1 μL,TaKaRa ExTaq(5 U/μL) 0.2 μL,灭菌双蒸水加至25 μL。
PCR反应条件:nuc基因94℃ 5 min;94℃ 30 s,55℃ 30 s,72℃ 45 s,28个循环;72℃延伸5 min。fexA基因94℃ 5 min;94℃ 30 s,50℃ 45 s,72℃ 45 s,28个循环;72℃延伸10 min。扩增结果经10 g/L琼脂糖凝胶电泳检测。
1.2.4 pET-fexA质粒的构建及基因工程菌pET-fexA/BL21耐药性检测 参照pET-32(a)载体说明书进行连接,将目的基因与pET-32(a)载体经SacⅠ、BamHⅠ双酶切后,用T4连接酶进行连接。经42℃热激法转入JM109中,37℃培养12 h~16 h,选择白色菌落经酶切和PCR两种方法鉴定出阳性菌,送测序公司进行测序。pET-fexA质粒成功构建后,提取质粒,经42℃热激法转入BL21中,选取氨苄阳性菌株进行培养,经氟苯尼考和氯霉素进行耐药诱导后,通过最小抑菌浓度试验检测其对氟苯尼考和氯霉素的敏感性。
1.2.5 分离菌最低抑菌浓度(MIC)测定 采用二倍琼脂稀释法,参照美国临床实验室标准化委员会(CLSI)推荐的方法进行操作[12],并参照其标准判定结果。CLSI没有规定金黄色葡萄球菌对氟苯尼考的耐药临界值,参考文献报道把MIC≥16 mg/L的葡萄球菌归为耐药菌[13]。金黄葡萄球菌CVCC3055为药物敏感性测定质控菌。
2 结果
2.1 病原菌分离与纯化结果
葡萄球菌在甘露醇氯化钠培养基琼脂平板上呈圆形,表面光滑、凸起、湿润的菌落,颜色呈金黄色或淡红色,边缘常为淡色。金黄色葡萄球菌在血平板上呈金黄色或白色菌落,大而突起,圆形,不透明,表面光滑,在菌落周围形成溶血环,培养至72 h整个菌落均变为金黄色。
2.2 病原菌的形态学及生化鉴定结果
革兰染色结果显示为革兰阳性球菌,经显微镜观察成葡萄串状。经生化鉴定仪鉴定结果为金黄色葡萄球菌。
2.3 fexA基因的PCR扩增以及携带fexA基因阳性菌株的鉴定
扩增产物经琼脂糖凝胶电泳分析,结果显示(图1和图2)以质粒DNA为模板的PCR分别扩增出了1 446 bp特异性目的条带,与预期片段大小相符;而以基因组DNA为模板的PCR未扩增出该目的片段,对照的质控菌均未扩增出目的条带。以基因组DNA为模板检测nuc基因的特异性,分别扩增出了480 bp目的条带,与预期片段大小一致。DNA测序结果经BLAST分析,与GenBank上序列同源性90%以上,在分子水平上证明了是金黄色葡萄球菌的耐药菌。

M.DNA标准DL 2 000;1~2.分离株质粒DNA的PCR产物;3~4.分离株基因组DNA的PCR产物;5.质控菌质粒DNA的PCR产物;6.质控菌基因组DNA的PCR产物
M.DNA Marker DL 2 000;1-2.PCR products of plasmid DNA of isolates;3-4.PCR products of genomic DNA of isolates; 5.PCR products of plasmid DNA of control strain; 6.PCR products of genomic DNA of control strain
图1fexA基因PCR扩增结果
Fig.1 PCR amplification result offexAgene
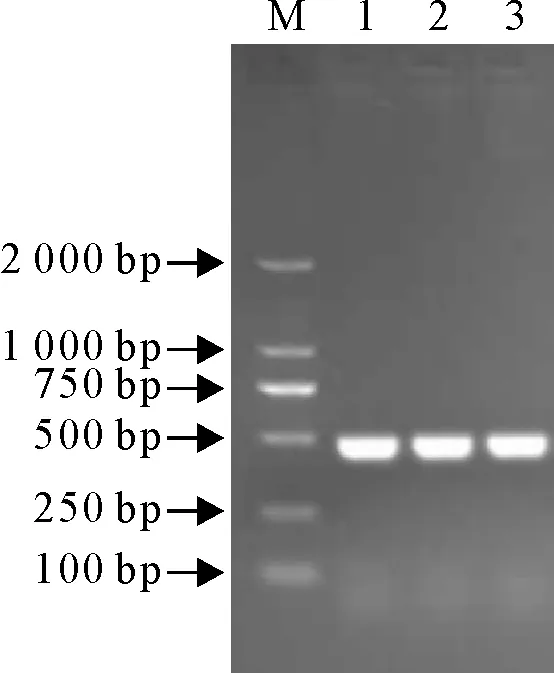
M.DNA标准DL 2 000;1~2.分离株;3.对照株
M.DNA Marker DL 2 000 ;1-2.Isolates;3.Control strain
图2nuc基因PCR扩增结果
Fig.2 PCR amplification result of the nuc gene
2.4 pET-fexA质粒的鉴定
fexA基因的PCR纯化产物与pET-32(a)载体连接后,经蓝白斑筛选阳性克隆,经PCR检测,扩增出的DNA片段大小与目的片段大小一致,说明重组质粒含有目的基因(图3)。对重组质粒进行酶切鉴定,得到了约5 900 bp和1 446 bp 的两条特异条带,说明重组质粒含有目的基因。与测序结果相符,表明原核表达质粒pET-fexA构建成功。

M.DNA标准DL 2 000;1.重组质粒pET-fexA的酶切产物;2.重组质粒pET-fexA的PCR产物
M.DNA Marker DL 2 000; 1.Enzymatic digestion products of the recombinant isolates pET-fexA; 2.PCR products of the recombinant isolates pET-fexA
图3重组质粒pET-fexA的PCR和酶切鉴定
Fig.3 Enzyme digestion identification and PCR amplification of pET-fexAplasmid
2.5 最低抑菌浓度(MIC)测定结果
pET-fexA质粒被成功构建后,转入基因工程菌E.coliBL21中,菌株命名为pET-fexA/BL21。pET-fexA/BL21和BL21以及fexA阳性金黄色葡萄球菌分离株和质控菌株分别经0.5 μg/mL终浓度的氟苯尼考和氯霉素溶液进行诱导,其对氟苯尼考和氯霉素的敏感性结果见表1,pET-fexA/BL21对氯霉素和氟苯尼考的耐药性显著增强,fexA阳性金黄色葡萄球菌分离株对氯霉素和氟苯尼考的耐药性显著高于质控菌株。
3 讨论
在广谱抗菌药物长期、广泛和不规则使用的巨大压力下,细菌通过基因突变和自然选择以及耐药基因的水平传播等途径,使得细菌耐药性越来越复杂和严重。染色体是细菌最稳定的遗传物质,遗传信息可经由亲代染色体DNA的复制直接传递给子代,当细菌长期处于抗菌药物的压力时,最终产生突变,导致细菌产生耐药性,耐药基因也可通过多种方式整合入细菌染色体,亲代获得的耐药性状能够稳定携带与遗传,从而介导细菌耐药性的垂直传播;细菌的质粒是一类能够独立于染色体外的可以进行自主复制的遗传单位,其在体外通过结合转移等方式进入到其他细菌体内,将耐药基因传递给其他同种甚至不同种属的药物敏感菌,完成了细菌耐药性的水平传播。虽然遗传物质的突变在细菌耐药中起着重要的作用,但是研究表明通过获得耐药基因而表现耐药的方式更占主导。
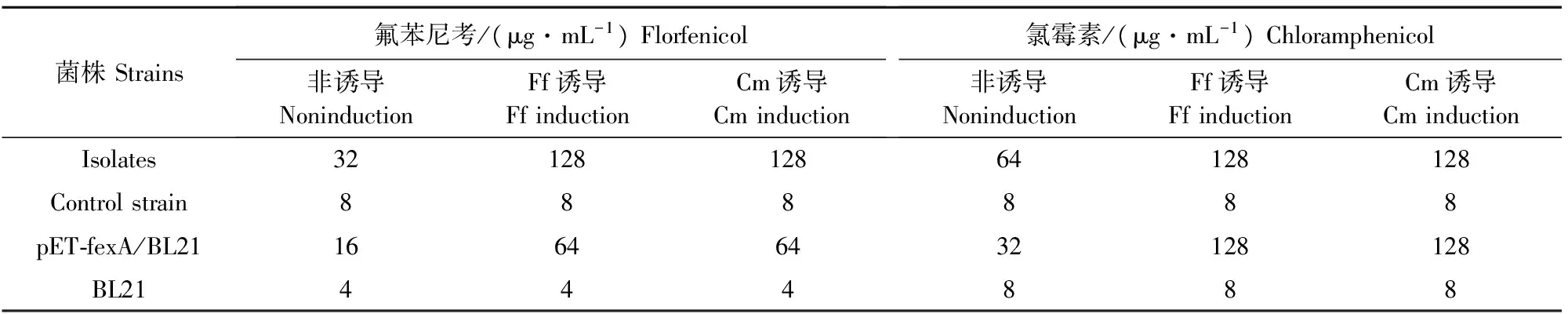
表1 最低抑菌浓度(MIC)测定结果
注:Ff.氟苯尼考;Cm.氯霉素。
Note:Ff.Florfenicol;Cm.Chloramphenicol.
近年来,研究者相继报道了动物源和人源葡萄球菌耐氟苯尼考fexA基因,研究显示fexA基因多数位于质粒上,也有少数位于染色体上的报道。Kehrenberg C等[6]在302株氯霉素耐药的葡萄球菌中发现了12株fexA阳性菌株,既有定位在质粒上的,也有定位在染色体上的。本研究中,从质粒中分离到fexA基因,说明位于质粒上的fexA基因介导了金黄色葡萄球菌氟苯尼考耐药性的传播。fexA基因的核苷酸序列分析表明,同源性非常高,这可能与其通过质粒进行水平传播的方式有关。Kehrenberg C等[14-15]研究发现该基因是新型转座子Tn558的一部分,该转座子位于质粒pSCFS2中,这在一定程度上增加了fexA传播的可能性。Kehrenberg C等通过侧翼序列的分析,对12株fexA阳性菌株进行研究,多数菌株均可以检测到完整的Tn558的结构存在。虽然该类型转座子属于非结合型转座子,但由于携带fexA基因的转座子存在于质粒中,说明fexA基因可通过质粒发生水平转移,在不同种属的细菌之间很容易传播扩散,这样动物源金黄色葡萄球菌的氟苯尼考耐药性就有可能通过接触、动物性食品加工等途径传播至人源金黄色葡萄球菌。
由于临床上的耐药菌株普遍携带多重耐药基因,为了排除细菌中其他氟苯尼考耐药基因或泵出蛋白对fexA基因贡献细菌耐药的影响,本试验成功构建了一个基因背景清楚且对氟苯尼考耐药的细菌模型,为fexA基因编码的蛋白定位研究奠定了基础。
[1] Felipe V,Morgante C A,Somale P S,et al.Evaluation of the biofilm forming ability and its associated genes inStaphylococcusspecies isolates from bovine mastitis in Argentinean dairy farms[J].Microb Pathog,2017,104:278-286.
[2] Xu J,Tan X,Zhang X,et al.The diversities of staphylococcal species,virulence and antibiotic resistance genes in the subclinical mastitis milk from a single Chinese cow herd[J].Microb Pathog,2015,88:29-38.
[3] Abebe R,Hatiya H,Abera M,et al.Bovine mastitis:prevalence,risk factors and isolation ofStaphylococcusaureusin dairy herds at Hawassa milk shed,South Ethiopia[J].BMC Vet Res,2016,12(1):270.
[4] Keefe G.Update on control of Staphylococcus aureus andStreptococcusagalactiaefor management ofmastitis[J].Vet Clin North Am Food Anim Pract,2012,28(2):203-216.
[5] Kehrenberg C,Schwarz S.FexA,a novelStaphylococcuslentusgene encoding resistance to florfenicol and chloramphenicol[J].Antimicrob Agents Chemother,2004,48(2):615-618.
[6] Kehrenberg C,Schwarz S.Distribution of florfenicol resistance genesfexAandcframong chloramphenicol-resistantStaphylococcusisolates[J].Antimicrob Agents Chemother,2006,50(4):1156-1163.
[7] Argudín M A,Tenhagen B A,Fetsch A,et al.Virulence and resistance determinants of GermanStaphylococcusaureusST398 isolates from nonhuman sources[J].Appl Environ Microbiol,2011,77(9):3052-3060.
[8] Cui L,Wang Y,Li Y,et al.Cfr-mediated linezolid-resistance among methicillin-resistant coagulase-negative staphylococci from infections of humans[J].PLoS One,2013,8(2):e57096.
[9] Zhang A,Yang Y,Wang H,et al.Prevalence of sulfonamide and florfenicol resistance genes inEscherichiacoliisolated from yaks (Bosgrunniens) and herdsmen in the Tibetan pasture[J].J Wildl Dis,2015,51(3):626-633.
[10] Pinto B,Chenoll E,Aznar R.Identification and typing of food-borneStaphylococcusaureusby PCR-based techniques[J].Syst Appl Microbiol,2005,28(4):340-352.
[11] Shittu A,Lin J,Morrison D.Molecular identification and characterization of mannitol-negative methicillin-resistantStaphylococcusaureus[J].Diagn Microbiol Infect Dis,2007,57(1):93-95.
[12] Clinical and Laboratory Standards Institute.Performance standards for antimicrobial susceptibility testing:eighteenth informational supplement,M100-S19[S].Wayne,PA,2009.
[13] Hidalgo A,Carvajal A,García-Feliz C,et al.Antimicrobial susceptibility testing of Spanish field isolates ofBrachyspirahyodysenteriae[J].Res Vet Sci,2008,87(1):7-12.
[14] Kehrenberg C, Schwarz S.Florfenicol-chloramphenicol exporter genefexAis part of the novel transposon Tn558[J].Antimicrob Agents Chemother,2005,49(2):813-815.
[15] Dai L,Wu C M,Wang M G,et al.First report of the multidrug resistance genecfrand the phenicol resistance genefexAin a Bacillus strain from swine feces[J].Antimicrob Agents Chemother,2010,54(9):3953-3955.
IsolationandIdentificationofStaphylococcusaureusfromDairyCattleandCloningofFlorfenicolResistanceGenefexA
ZHAO Su-jun1,2,CAO Ye1,2,XIE Jing1,2,YU Ji-feng1,2,LI Jiang-ling1,2, WANG Qiu-shi1,2,LIAO Dang-jin1,2
(1.SichuanAnimalScienceAcademy,Chengdu,Sichuan,610066,China; 2.AnimalBreedingandGeneticskeyLaboratoryofSichuanProvince,Chengdu,Sichuan,610066,China)
A total of 9 florfenicol-resistantStaphylococcusaureusstrains from dairy cattle were isolated and identified from the 100 dairy farm environmental samples and festering tissue samples.Plasmid DNA and genomic DNA of the 9 isolates were extracted.PCR analysis of plasmid DNA and genomic DNA with primers specific for the florfenicol resistance genefexAdemonstrated that the 1446bp amplicon was specifically detected in plasmid DNA and none in genomic DNA.The amplified fragment offexAgene was cloned into pET-32(a) vector,and a positive plasmid was obtained and named plasmid pET-fexA.The recombinant plasmids were successfully transformed intoE.coliBL21.A significant resistance to florfenicol and chloramphenicol was found in this strain.At the same time,the results of antimicrobial resistance revealed that the resistances to florfenicol and chloramphenicol of the positiveStaphylococcusaureusisolates withfexAgene fragment were significantly higher than that of quality-control strain.The research showed that thefexAgene conferred cross-resistant to florfenicol and chloramphenicol.In this study,we successfully established a florfenicol-resistant bacterial model with a clear genetic background,it ruled out the influence of other drug-resistant genes and efflux protein in bacteria on thefexAgene conferring drug-resistant bacteria,which will provide a basis for determining the localization offexAprotein.
Staphylococcusaureus; florfenicol;fexAgene; dairy cattle
2017-03-10
四川省畜牧科学研究院基本科研业务专项(SASA2014A14)
赵素君(1977- ),女(满族),辽宁锦州人,副研究员,博士,主要从事分子遗传学研究。*
S852.611
A
1007-5038(2017)12-0034-05

