神经干细胞与神经元差异表达基因的生物信息学分析
李世安,刘友,刘希光,李爱民
神经干细胞与神经元差异表达基因的生物信息学分析
李世安,刘友,刘希光,李爱民
目的通过生物信息学技术探讨神经干细胞与神经元间基因表达的差异,找出关键的差异基因及其潜在的分子调控机制;并对其所涉及的功能进行分析预测。方法从GEO表达谱数据库中下载与神经干细胞及神经元相关的表达谱基因芯片数据系列GSE70171,导入基因芯片在线分析工具morpheus,筛选出神经干细胞和神经元之间表达差异的基因,并构建聚类分析热图。采用DAVID数据库进行GO功能分析和KEGG通路富集分析,并使用STRING分析与Cytoscape软件构建PPI网络。结果筛选出表达差异的基因有4 022个,其中神经干细胞比神经元上调的基因有2 146个,下调基因1 876个。对表达差异的基因进行的生物信息学分析发现,下调的基因主要与神经元功能相关,上调基因与神经干细胞细胞再生和有肿瘤特征相关,其中神经干细胞的细胞周期通路和癌症通路存在研究价值。结论神经干细胞与神经元之间存在表达差异基因。几个关键基因CDC6、CDKN2A、CDC14A、BUB1、TTK、CHEK1、CDC25C在细胞周期通路中可能与神经干细胞再生功能相关。而KIF23、CDKN2A、TNC、CDC25C、CDCA5、BRCA1可能与神经干细胞的肿瘤特征相关。然而,这些关键基因的功能还需要今后的实验研究进一步证实。
神经干细胞;生物信息学;基因;肿瘤
在1992年,Reynolds和Weiss等[1]在中枢神经系统(central nervous system,CNS)中发现自我更新的神经干细胞(neural stem cells,NSCs)。这些细胞能增殖、迁移和分化成脑和脊髓所有细胞类型,包括神经元[2-4]。即神经干细胞(NSCs)具有在特定条件下自我更新,并分化成神经谱系(神经元)的能力。当发生神经损伤等病理改变后,处于休眠状态的NSCs进入静止状态,然后发生激活,增殖并迁移至受损部位,诱导或再重塑成新的神经细胞[5]。这些新的细胞可以代替受损的细胞,参与形成新的神经网路,并促进脑损伤的结构和功能修复[6]。但是,NSCs分化的基础机制仍然不完全清楚,尚需要进一步详细的研究。本研究通过生物信息学技术,探讨神经干细胞与神经元之间基因表达的差异,找出关键的差异基因及其潜在的分子调控机制,并分析预测其所涉及的功能;为神经干细胞及其分化的细胞可能产生有价值和稳定的人类神经细胞亚型提供参考。
1 材料与方法
1.1 材料 本研究的数据来源于美国国立生物技术信息中心(National Center of Biotechnology Information,NCBI)基因表达数据库(Gene xpression Omnibus,GEO);使用以下检索:“Neural Stem Cells”(关键词),“Mus musculus”(生物),“阵列表达分析”(研究型)和“组织”(属性名称)。系统评估后,检索了976项GSE研究。研究纳入标准如下:(1)诊断为神经干细胞和神经元的样本;(2)每组样本数均超过2例;(3)mRNA基因表达谱;(4)足够基因探针信息进行分析;(5)并不是诱导分化的神经干细胞且没有其他处理因素影响的细胞。然后,收集到基因表达谱系列号为GSE70171进行分析。最终6例样本数据(GSM1717919、GSM1717920、GSM1717921 为神经元组织,GSM1717922、GSM1717923、GSM1717924为神经干细胞组织)被纳入实验。实验使用了美国Affymetrix公司表达谱芯片(神经元与神经干细胞样本数据已经进行标准化处理)。
1.2 方法 将6例样本数据导入基因芯片在线分析工具morpheus(https://software.broadinstitute.org/morpheus/),随后对神经干细胞和神经元间差异表达基因进行筛选。根据基因名称和基因芯片信号灰度值过滤基因表达缺失数据(<5%),然后对标准的基因信号值进行fold change检验(log2FC<-2或log2FC>2,P<0.05,q<0.05),即以差异倍数为检验标准,取对数进行数据转换,用2倍差异得到明显差异表达基因,同时以P值和q值统计学筛选。最后通过信噪比(scalable profile,SNP)的差异进行排序,用pearson相关分析生成聚类分析热图。
利用DAVID数据库(https://david.ncifcrf.gov/)对差异表达基因进行GO分析(用于注释基因潜在生物现象的常见方法,包含基因产物和基因序列的注释[7-8]),从分子功能、生物过程和细胞组成的角度对其富集分析(通过P值统计学筛选和霍赫贝格校正多重检测筛选后以相关基因数排序)。同时进行KEGG分析(用生物学方法解读基因组序列和其他高通量数据的综合数据库资源[9]),从信号通路角度对其富集分析。
利用STRING数据库(http://string-db.org/)对差异基因进行蛋白质相互作用(Protein-Protein Interactions,PPI)的分析(蛋白质与蛋白质相互作用的关键评估和整合,包括直接和间接作用[10])。分析PPI网络图,找出关键节点的蛋白质和相关的蛋白及其相应的关系与稳定性。
利用Cytoscape软件(一种流行的开源软件工具,用于视觉探索由蛋白质、基因和其他类型的相互作用组成的生物分子之间相互作用网络[11])进行基因数据的整合及差异基因上下调节映射的可视化。
2 结 果
2.1 差异基因筛选及其热图 通过基因芯片在线分析工具morpheus分析,获得出现明显表达差异的基因共4 022个;其中神经干细胞比神经元上调的基因有2 146个,下调基因1 876个。通过pearson相关分析生成聚类热图,由于定量资料数据通过对数转换后服从正态分布,进行分层聚类;聚类距离用pearson计算的相关系数矩阵,取平均值。见图1。
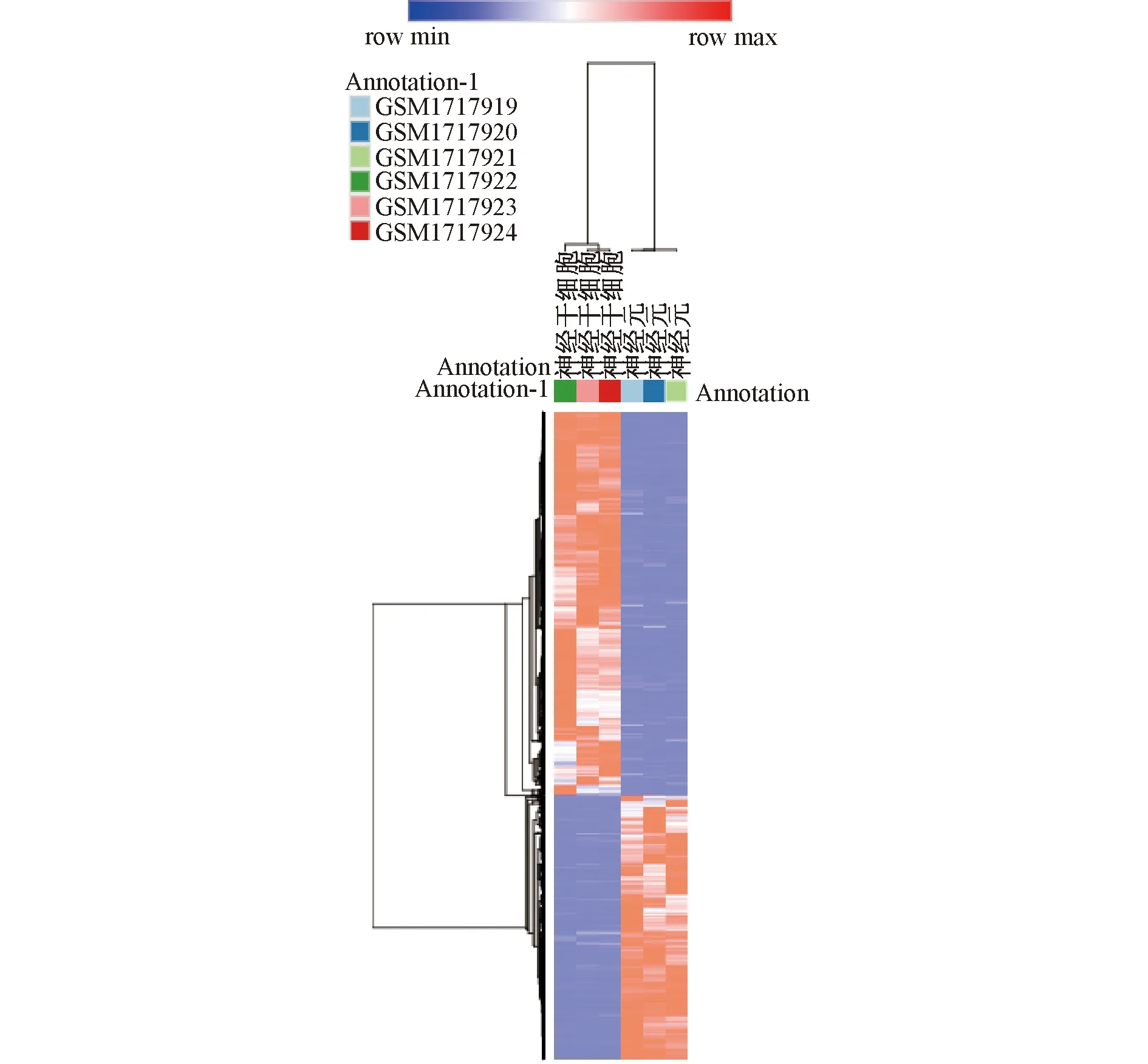
图1 mRNA表达谱数据系列集GSE70171中表达差异的基因鉴定及pearson相关聚类热图(红:上调差异基因;蓝:下调差异基因)
2.2 差异基因GO分析及KEGG分析结果 表达上调基因GO分析结果显示涉及:生物过程1 591条,细胞组成131条,分子功能245条。表达下调基因GO分析结果显示涉及:生物过程1 469条,细胞组成192条,分子功能248条。见图2。
2.3 差异表达基因进行KEGG分析的结果 表达上调基因KEGG分析结果显示涉及53条;表达下调的基因KEGG分析结果显示涉及75条。根据KEGG结果分析NSCs与神经元在代谢通路上的主要差异,见图3、图4。在NSCs与神经元差异基因中提取与肿瘤相关的信号通路,见表1。然后单独分析NSCs与神经元差异基因在癌症的途径通路上变化,见图5。
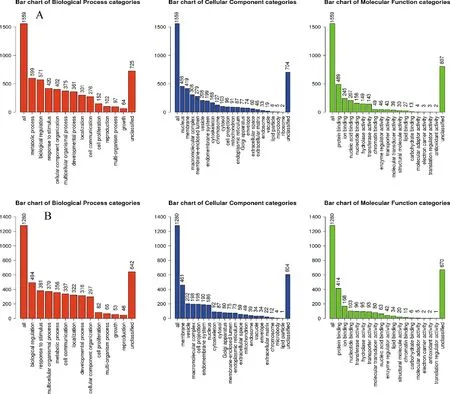
A:上调差异基因 B:下调差异基因图2 差异基因相关GO分析结果(前5位条,纵坐标:相关基因数)
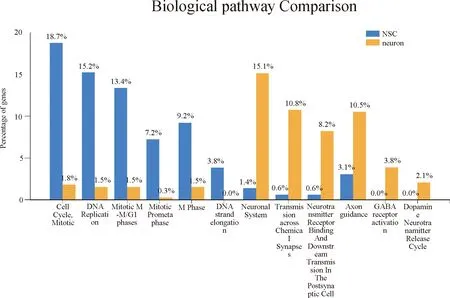
图3 上调差异基因与下调差异基因KEGG分析结果对比(前12位条)
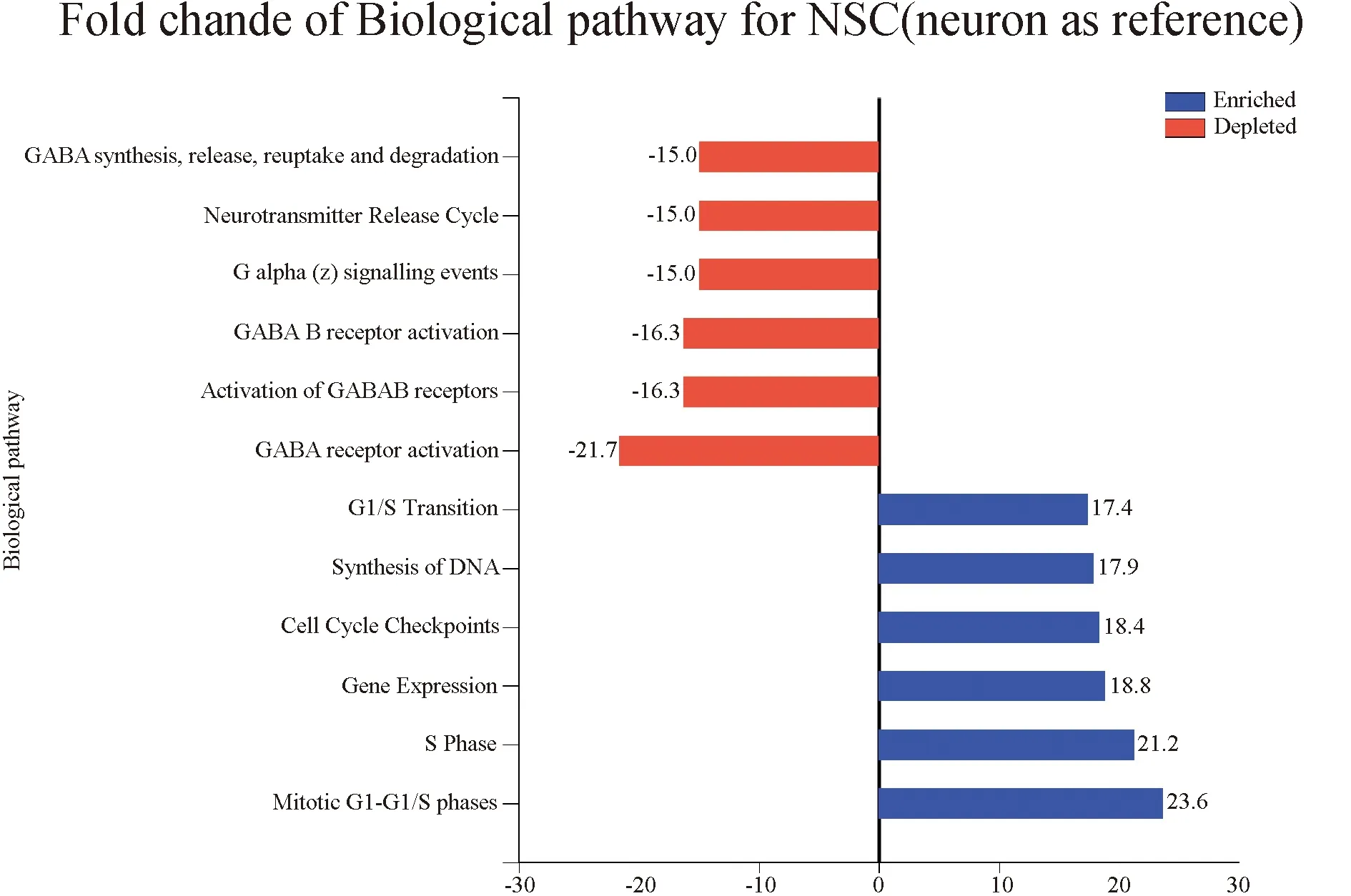
图4 神经干细胞与神经元在代谢通路中基因序列富集和缺失对比(前6位条)

表1 差异基因与肿瘤相关通路KEGG分析结果

(续表)
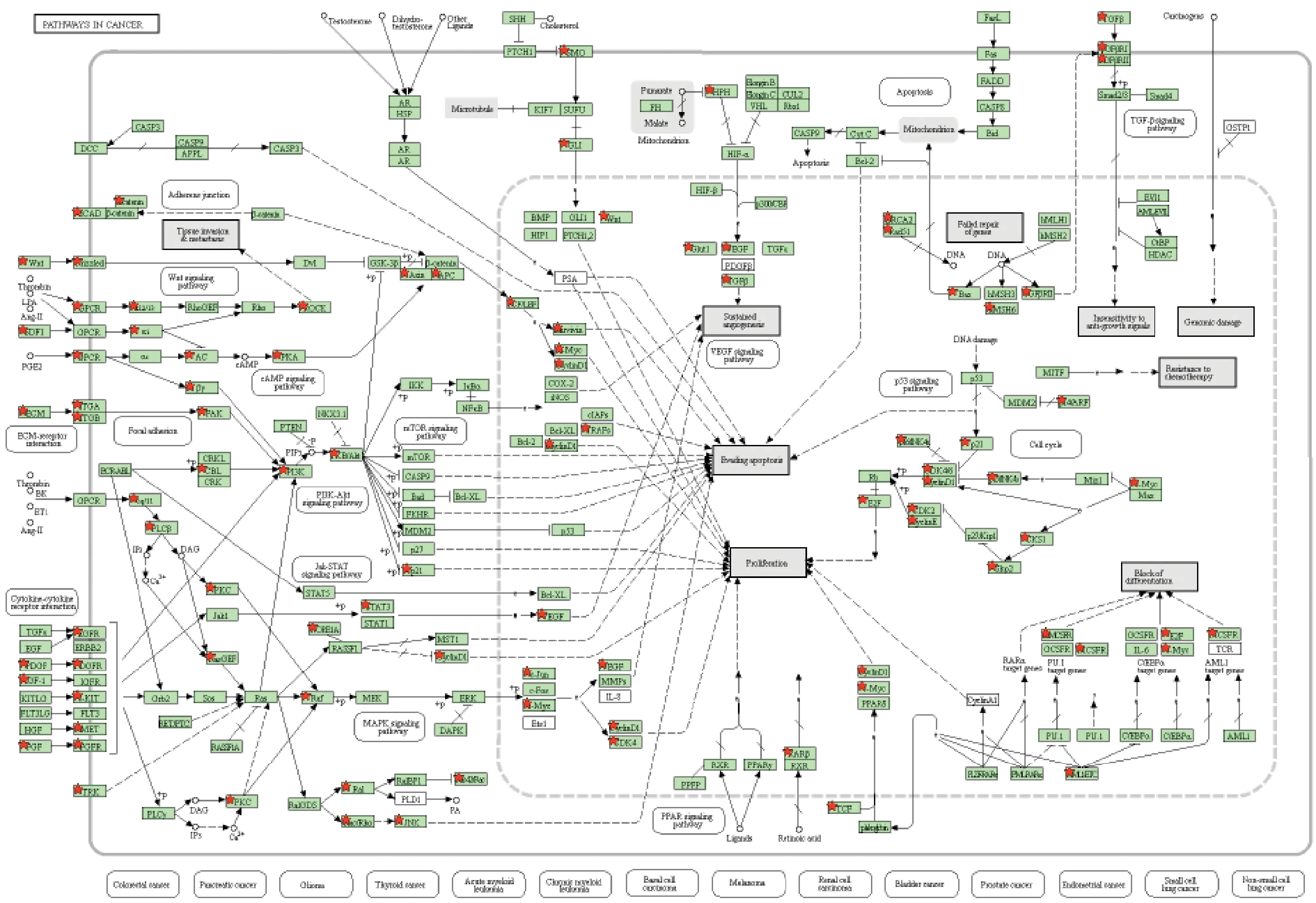
图5 差异基因涉及的癌症通路(★:差异基因)
2.4 差异表达基因的STRING分析结果 通过STRING在线工具对差异基因进行PPI网络分析,其中显示节点数101个(差异基因对应的蛋白质及其相关蛋白质),边数830个(差异基因对应的蛋白质及其相关蛋白质之间相互作用关系的标识)。图中显示有3大块PPI网络中心节点,即核心蛋白质,若删除这些节点蛋白后,网络结构的稳定性降低。见图6。
2.5 差异表达基因进行的Cytoscape分析结果 利用Cytoscape软件根据log2FC对差异基因上下调进行注释并且去除离散的差异基因。其中红色代表神经干细胞比神经元上调的基因,即NSCs表达量多的基因;蓝色代表神经干细胞比神经元下调的基因,即神经元表达量多的基因。通过结合KEGG分析结果,对在癌症中通路单独注释。其中差异基因在癌症中通路上对应的蛋白质及其相关蛋白质的相互作用关系以基因与基因间的连接线来表示。见图7。
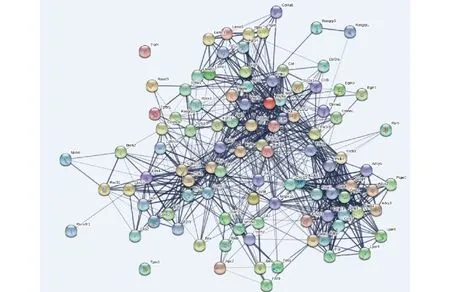
图6 差异表达基因PPI网络及核心基因
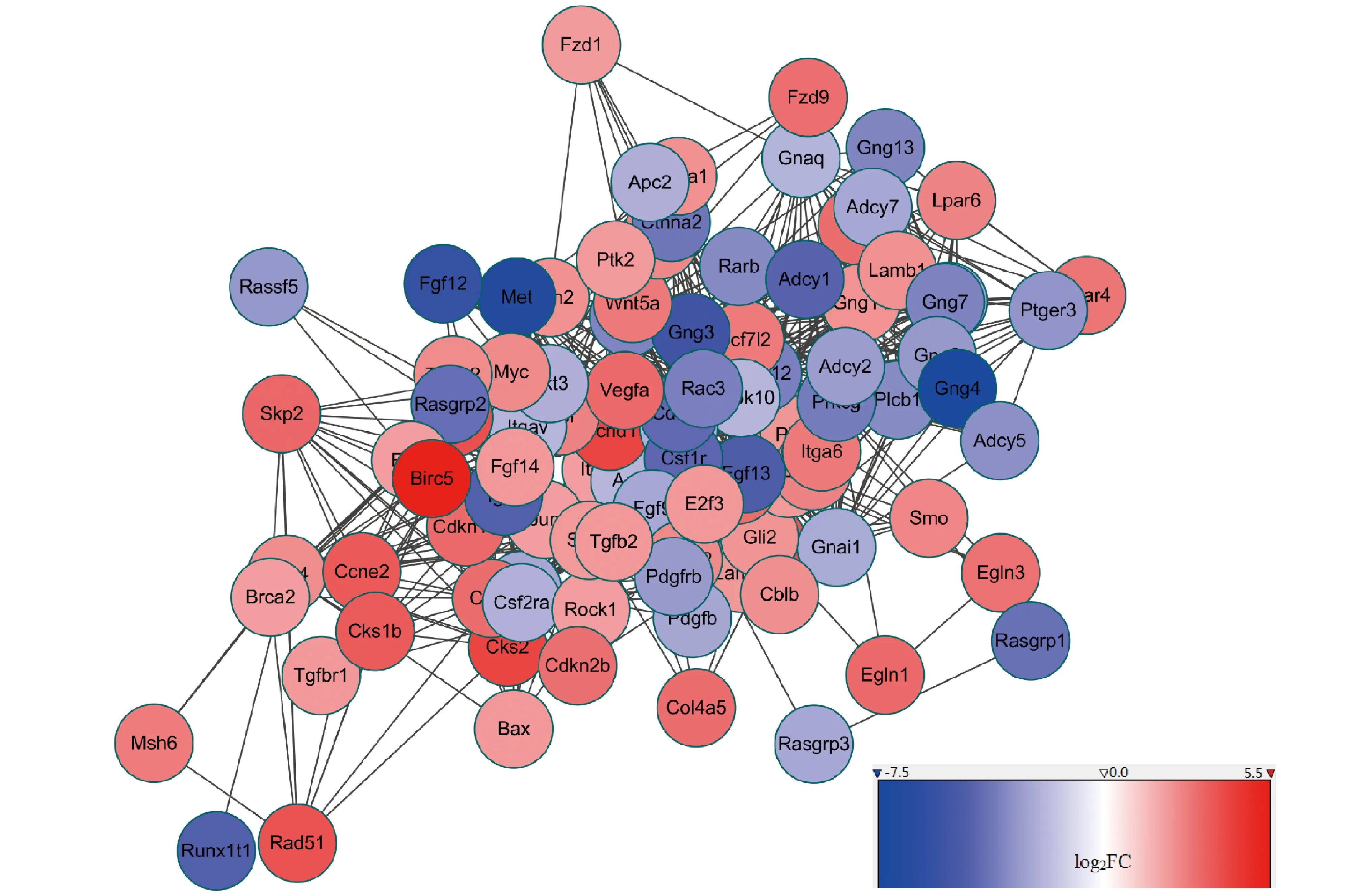
图7 差异基因PPI网络中KEGG在癌症的途径通路注释(红:上调;蓝:下调)
3 讨 论
研究表明神经干细胞在不同微环境中接收细胞周围的特异性分子信号后分化为不同类型的神经元[12]。这些新形成的神经元建立了新的神经回路或修改现有途径。在这种状况下,若大脑中的神经元发生死亡等病理改变,NSCs治疗似乎是一种有希望恢复失去的神经功能的方法。神经干细胞分化为特定类型的神经元及其形成的连接网络可以修复和替代已死亡或受损的神经元[13]。
在本研究中,筛选出正常的神经干细胞与神经元之间表达有差异的基因共4 022个,包括 2 146个上调基因和1 876个下调基因。GO分析结果显示,下调富集的基因主要参与神经系统发育、神经发生、神经元的生长、神经元分化;上调富集的基因主要参与细胞表达、细胞大分子生物合成、调节氮化合物代谢、调节含核苷酸碱基化合物的代谢过程。KEGG分析结果显示,下调的基因主要分布在癌症通路、神经配体受体相互作用、钙信号通路、谷氨酸能突触等上;上调富集的基因主要分布代谢通路、细胞周期、癌症通路、癌症微小RNA等中。结合功能和通路注释,上调基因主要展现了神经元的具体功能;在细胞周期通路上,上调的基因有CDC6、CDKN2A、CDC14A、BUB1、TTK、CHEK1、CDC25C,这表明上调的基因主要展现了神经干细胞的增殖、分裂和自我更新的功能。最后,差异基因通过PPI网络分析发现在上调的基因中核心的蛋白质相互作用稳定,并且细胞周期通路中蛋白集中。以上分析结果显示了NSCs的再生潜能和CNS中的神经细胞的可塑性,并且进一步验证了利用NSCs再生修复功能,可以帮助损伤后脑组织结构和功能的恢复;如出血性卒中[14]和创伤性脑损伤[15]等疾病。
然而,在肿瘤相关通路上,上、下调的基因都包括肿瘤相关的通路,而上调的基因更多;如癌症中的途径、HTLV-I感染、癌症中的微小RNA、病毒致癌等。单独提取得分最高癌症通路进行PPI网络分析,利用Cytoscape软件构成差异图显示基因表达量与之相关基因的富集。找出其中几个富集得分高的基因(KIF23、CDKN2A、TNC、CDC25C、 CDCA5、 BRCA1)来,在NCBI数据库中做GeneRIFs注释。在大多数人类恶性肿瘤中观察到驱动蛋白家族成员23(KIF23)的高表达;驱动蛋白家族成员23是微管依赖性分子马达的成员,负责在细胞有丝分裂期间细胞器的运输与染色体的移动。KIF23是一种核蛋白质,其定位于有丝分裂轴的区域之间,在胶质瘤组织中表达水平高于正常脑组织[16]。有研究表明与色素痣组织相比,黑素瘤组织中CDKN2A(p16INK4A)和CDKN2A(p14ARF)mRNA和蛋白表达显著降低;CDKN2A(p16INK4A)和CDKN2A(p14ARF)过表达抑制A375细胞从G0/ G1到S期的增殖、迁移、侵袭和进展,并促进细胞凋亡[17]。大量实验证明TNC(粘附调节细胞外基质分子)在许多人类恶性肿瘤中高度表达。癌细胞和基质成纤维细胞中的粘附调节细胞外基质分子TNC的过表达是食管鳞状细胞癌患者整体存活和无病生存的独立危险因素[18]。CDC25C(细胞分裂周期25同源C)是细胞周期G2期向M期发生转移必不可少的。研究显示,在细胞间期中CDC25C抑制细胞凋亡信号调节激酶1(ASK1),并介导丝裂原活化蛋白激酶激酶(MAP)家族的细胞凋亡[19]。这些研究结果提示神经干细胞有发生肿瘤的可能。
本研究采用生物信息学技术对神经干细胞与神经元相关的基因芯片数据分析,利用morpheus分析工具、DAVID数据库、STRING分析和Cytoscape软件,筛选出神经干细胞与神经元差异的表达基因,从生物学功能与蛋白质相互作用关系网络等方面进行深入研究,探讨表达的差异基因可能的分子机制,为研究神经干细胞的临床治疗等方面提供帮助。
综上所述,本研究结果显示,神经干细胞与神经元之间存在明显表达差异基因。几个关键基因CDC6、CDKN2A、CDC14A、BUB1、TTK、CHEK1、CDC25C在细胞周期通路中可能与神经干细胞再生功能相关;而KIF23、CDKN2A、TNC、CDC25C、CDCA5、BRCA1可能与神经干细胞的肿瘤性相关。但是这些关键基因的确切功能还需要今后的实验研究进一步证实。
[1] Reynolds BA,Weiss S.Generation of neurons and astrocytes from isolated cells of the adult mammalian central nervous system[J].Science,1992,255:1707.
[2] Doe CQ.Neural stem cells:balancing self-renewal with differentiation[J].Development,2008,135:1575.
[3] Gage FH.Mammalian neural stem cells[J].Science,2000,287:1433.
[4] Wang TT,Jing AH,Luo XY,etal.Neural stem cells:isolation and differentiation into cholinergic neurons[J].Neuroreport,2006,17:1433.
[5] Bifari F,Decimo I,Pino A,etal.Neurogenic radial glia-like cells in meninges migrate and differentiate into functionally integrated neurons in the neonatal cortex[J].Cell Stem Cell,2017,20:360.
[6] Saha B,Jaber M,Gaillard A.Potentials of endogenous neural stem cells in cortical repair[J].Front Cell Neurosci,2012,6:14.
[7] Bastos HP,Tavares B,Pesquita C,etal.Application of gene ontology to gene identification[J].Methods Mol Biol,2011,760:141.
[8] Gene Ontology Consortium.The Gene ontology(GO) project in 2006[J].Nucleic Acids Res,2006,34 (Database issue):D322.
[9] Kanehisa M,Sato Y,Kawashima M,etal.KEGG as a reference resource for gene and protein annotation[J].Nucleic Acids Res,2016,44 (D1):D457.
[10] Szklarczyk D,Franceschini A,Wyder S,etal.STRING v10:protein-protein interaction networks,integrated over the tree of life[J].Nucleic Acids Res,2015,43(Database issue):D447.
[11] Su G,Morris JH,Demchak B,etal.Biological network exploration with Cytoscape 3[J].Current Protoc Bioinformat,2014,47:8.13.1.
[12] Fandel TM,Trivedi A,Nicholas CR,etal.Transplanted human stem cell-derived interneuron precursors mitigate mouse bladder dysfunction and central neuropathic pain after spinal cord injury[J].Cell Stem Cell,2016,19:544.
[13] Braak H,Del Tredici K.Assessing fetal nerve cell grafts in Parkinson’s disease[J].Nature Med,2008,14:483.
[14] Jessberger S.Neural repair in the adult brain[J].F1000Res,2016,5:169.
[15] Liu YP,Lang BT,Baskaya MK,etal.The potential of neural stem cells to repair stroke-induced brain damage[J].Acta Neuropathol,2009,117:469.
[16] Takahashi S,Fusaki N,Ohta S,etal.Downregulation of KIF23 suppresses glioma proliferation[J].J Neuro-Oncol,2012,106:519.
[17] Bai M,Yu NZ,Long F,etal.Effects of CDKN2A (p16INK4A/p14ARF) over-expression on proliferation and migration of human melanoma A375 cells[J].Cell Physiol Biochem,2016,40:1367.
[18] Yang ZT,Yeo SY,Yin YX,etal.Tenascin-C,a prognostic determinant of esophageal squamous cell carcinoma[J].PloS One,2016,11:e0145807.
[19] Cho YC,Park JE,Park BC,etal.Cell cycle-dependent Cdc25C phosphatase determines cell survival by regulating apoptosis signal-regulating kinase 1[J].Cell Death Differ,2015,22:1605.
Bioinformaticsanalysisofdifferentiallyexpressedgenesinneuralstemcellsandneurons
LIShi-an,LIUYou,LIUXi-guang,etal.
DepartmentofNeurosurgery,AffiliatedLianyungangHospitalofXuzhouMedicalUniversity,Lianyungang222002,China.
Correspondingauther:LIUYou
ObjectiveThrough the bioinformatic technique,the differences in gene expression between neural stem cells and neurons were explored.The key genes and their potential molecular regulation mechanisms were identified.The functions involved were analyzed and predicted.MethodsThe gene series GSE70171 related to neural stem cells and neurons were downloaded from the GEO expression profile database.Firstly,the genes were analyzed by gene chip online analysis tool Morpheus.Then among these genes,genes that differ in expression between neural stem cells and neurons were screened out.Finally,expression differences in gene construction cluster analysis heat map.DAVID database was used to perform GO functional analysis and KEGG pathway enrichment analysis.The STRING analysis and Cytoscape software were used to build PPI network.ResultsThere were 4022 genes screened out,among which neurons had more 2 146 genes down-regulated and 1 846 genes up-regulated than neural stem cells.The bioinformatic analysis of the genes expressing the differences revealed that the down-regulated genes were mainly related to neuronal function,and the up-regulation genes were related to the regeneration of neural stem cells and the neoplasms characteristics.The cell cycle pathway and the microRNAs pathway of neural stem cells were of great value.ConclusionsThe result is a differential gene between neural stem cells and neurons.Several key genes,such as CDC6,CDKN2A,CDC14A,BUB1,TTK,CHEK1,CDC25C,may be related to the regeneration function of neural stem cells in the cell cycle pathway.And KIF23,CDKN2A,TNC,CDC25C,CDCA5,BRCA1 may be related to the neoplasms characteristics of neural stem cells.However,the function of these key genes requires further confirmation in future experimental studies.
neural stem cell;bioinformatics;gene;neoplasm
222002 连云港,徐州医科大学附属连云港医院神经外科
刘友
10.3969/j.issn.1672-7770.2017.06.002
R741.05
A
1672-7770(2017)06-0406-08
(收稿2017-05-17 修回2017-07-31)

