游离细胞催化法生产丙烯酰胺的菌株改造策略
于慧敏,焦松,沈忠耀
清华大学化学工程系,教育部工业生物催化重点实验,北京 100084
游离细胞催化法生产丙烯酰胺的菌株改造策略
于慧敏,焦松,沈忠耀
清华大学化学工程系,教育部工业生物催化重点实验,北京 100084
丙烯酰胺是一种重要的酰胺类化合物,其聚合产物——聚丙烯酰胺被广泛应用于三次采油、水处理、造纸、冶金等领域。以腈水合酶为核心的生物催化法,尤其是游离细胞催化法,具有转化效率高、产物纯度高以及环境友好等优点,是目前丙烯酰胺生产的主要方法;而腈水合酶及其生产菌株的性能在丙烯酰胺生产工艺中起着至关重要的作用。分别从细胞层次和酶层次对常用的生产菌株性能改造策略进行了简介,并重点阐述了目前用于腈水合酶生产菌株改造的基因重组策略。最后介绍了腈水合酶和产酶细胞的协同改造策略及改造效果。其中,通过对产腈水合酶红球菌R. ruber TH在酶和细胞层面的协同改造,重组游离细胞TH8实现了工业规模约500g/L高浓丙烯酰胺的三批次水合制备。
丙烯酰胺;腈水合酶;游离细胞催化;细胞和胞内酶的协同改造

于慧敏,博士,教授。2001年获清华大学化工系生物化工专业工学博士学位;2005~2006年在美国麻省理工学院化工系做访问学者,2012年任职教授。主要从事工业生物催化及生物纳米技术研究,致力于发展生物催化剂构建技术与理论、强化生物过程、构筑能够在工业环境中高效稳定发挥作用的生物催化剂和生物催化新工艺。主要研究对象为生物法生产丙烯酰胺、脂肽类生物表面活性剂、透明质酸等多个大宗和精细化学品。2003年获全国百篇优秀博士论文奖,2004年获清华大学华新杰出学者奖,2007年获北京市科技新星称号。2008年入选教育部新世纪优秀人才。2010年和2013年分别获中国石油和化学工业联合会青年科技突出贡献奖和技术发明二等奖。2016年获东营市科学技术合作奖。E-mail:yuhm@tsinghua.edu.cn
随着“绿色发展”理念越来越深入人心,以高效、低耗、绿色为典型特征的工业生物技术日益受到各国政府、研究单位和产业界的高度重视[1]。而随着生物技术的快速发展,诸如羟基乙酸、L-苯丙氨酸、D-对羟基苯甘氨酸、烟酰胺等越来越多的化学品实现了生物法绿色生产。其中,丙烯酰胺的生物法生产无疑是其中最为成功的案例之一[2]。丙烯酰胺(AM)是一种重要的化工产品,其聚合产物——聚丙烯酰胺(PAM)广泛应用于石油、水处理、造纸、冶金等领域[3]。尤其在石油开采中,PAM作为一种性能优异的絮凝剂、增稠剂、增强剂等,被广泛应用于钻井、堵水、减阻和驱油等[4]。
1 丙烯酰胺的生产方法
丙烯酰胺通常由丙烯腈水合得到,其生产工艺经历了从化学催化法到生物酶催化法的转变。1964年美国氰氨公司率先开发了硫酸水合法,实现了丙烯酰胺的工业化生产。随后,以Cu为催化剂的催化水合法被开发出来并广泛应用。1985年,日本日东化学公司开发了以腈水合酶催化为核心的微生物法生产工艺。生物酶催化法相较于传统的化学催化法具有转化效率高、产物纯度高以及环境友好等优点,目前已经逐渐占据了主导地位。
在微生物法生产工艺中,丙烯酰胺是通过腈水合酶催化丙烯腈与水发生水合反应而得到[图1(a)]。其中,腈水合酶的应用形式可以是固定化细胞、固定化酶、游离细胞乃至游离酶。目前,游离细胞催化法由于其工艺简单、操作方便、细胞可以实现多批次回用等特点,已经成为我国生物法生产丙烯酰胺的主要工艺,其简要工艺过程如图1(b)所示。
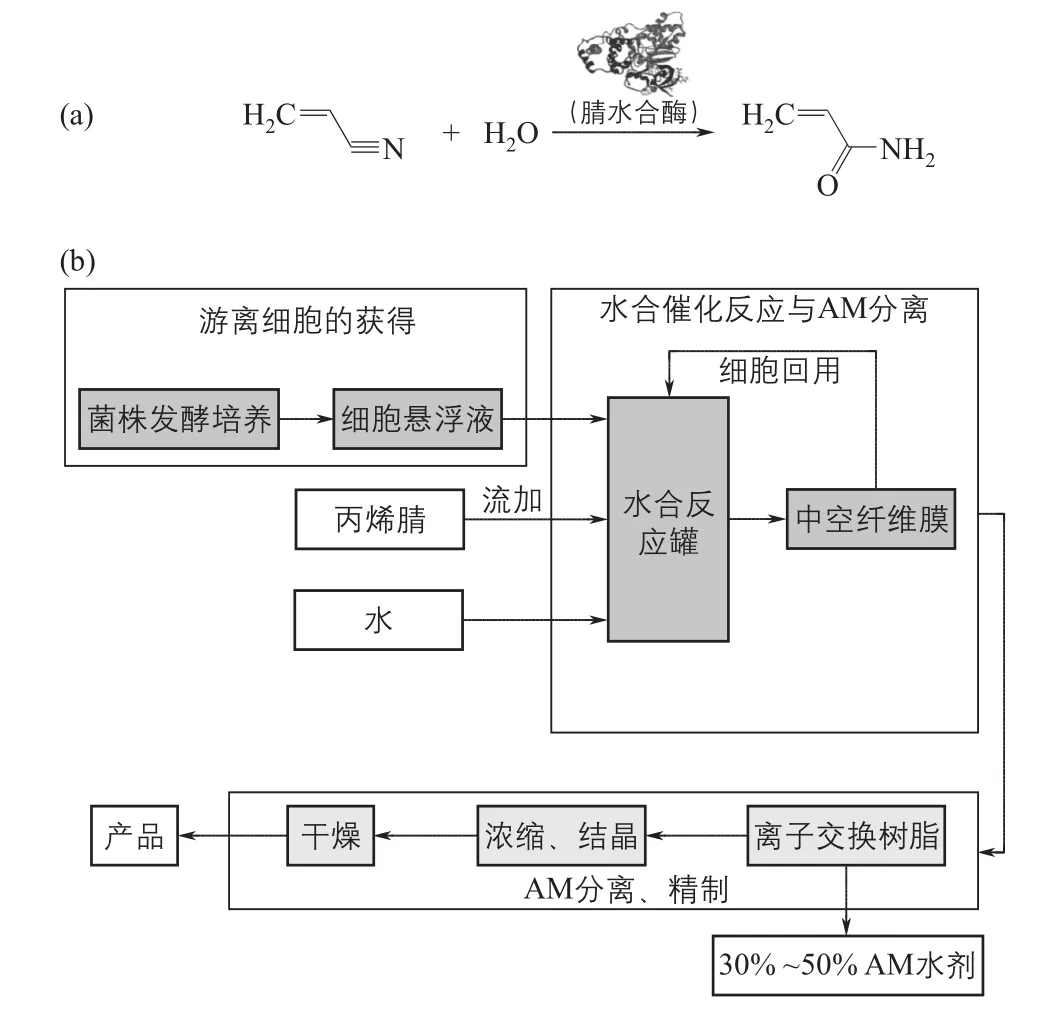
图1 游离细胞法丙烯酰胺生产工艺
整个生产过程主要包含游离细胞的获得、水合催化反应和AM分离、精制三个阶段。生产菌株通过种子罐以及发酵罐的培养,产生高活性的腈水合酶。发酵液经过离心或微滤分离获得细胞沉淀,经过洗涤后即可用于水合反应。丙烯腈的水合反应在水合反应釜中进行。其中,丙烯腈通常以流加的方式进入水合釜的细胞悬浮液,并通过搅拌与反应釜中均匀分散的细胞接触并立即发生反应生成AM产物;当AM达到一定的浓度后,所得反应液通过中空纤维超滤膜将菌体细胞与AM粗品进行分离。菌体细胞可回收利用进行下一批次水合反应,而得到的粗品AM经过阴阳离子交换树脂纯化后,可获得30%~50%的AM水剂产品。进一步经过浓缩、结晶、干燥,即得到最后的AM晶体(粉剂)产品。
2 腈水合酶生产菌株及其改造方法
2.1 腈水合酶生产菌株及其性能需求
催化丙烯腈生成丙烯酰胺的腈水合酶广泛存在于诺卡氏菌(Nocardia)、红球菌(Rhodococcus)、假单胞菌(Pseudomonas)等细菌中。自1984年以来,已经实现工业化应用的菌株包括日本日东化学公司的Rhodococcus sp. N774、京都大学山田秀明等发现的Pseudomonas Chlororaphis B23和Rhodococcus rhodochrous J1[5]、上海农药研究所的诺卡氏菌Nocardia sp. 86-163[6]以及清华大学的Rhodococcus ruber TH[7]等。
虽然以腈水合酶为核心的生物催化法生成AM已经有30多年的历史,相关的生产技术也已越来越成熟,但在生产过程中仍然存在诸多问题,包括:①部分生产菌株在发酵培养过程中会出现絮凝沉降的现象,导致所得细胞生物量低,腈水合酶酶活也明显下降,无法正常地用于后续催化水合过程;②游离细胞催化水合反应过程中会有少量的副产物丙烯酸生成,这不仅导致了丙烯腈的单耗较高,还提高了后续的精制工艺成本;③在水合反应过程中,游离细胞内的腈水合酶受到流加的丙烯腈和高浓度AM溶液抑制作用以及不稳定的反应温度的影响,其催化活性会迅速下降,因此,无法在一批次水合反应中获得较高的AM产物浓度。目前工厂水合反应后AM的浓度一般仅为30%左右。为了获得高浓度的AM水剂(40%~50%)和粉剂(60%以上AM降温结晶),必须进一步通过高温浓缩工序实现,这样便导致了生产成本的显著提高。为了解决AM产物浓度不高的问题,必须显著提升用于AM生产的游离细胞和胞内腈水合酶的稳定性(抗逆性)。
2.2 重组菌株的细胞层次性能改造策略
菌株的性能对于目标产物或者目标酶的生产起着至关重要的作用。为了得到理想的生产菌株,研究者往往通过代谢工程、基因工程和蛋白质工程等手段来改善生产菌株的性能。表1总结列出了近几年来开发的针对不同目标产物进行的各种有效的菌株性能改造策略,其中包括了针对sigma因子[8]、cAMP受体蛋白[9]等的全局转录机器工程方法、分子伴侣过表达加强对蛋白质的保护[10]、合成强化对目标产物获得具有重要作用的中间产物[11]以及敲除与目标产物代谢产生竞争作用的代谢途径[12]等。
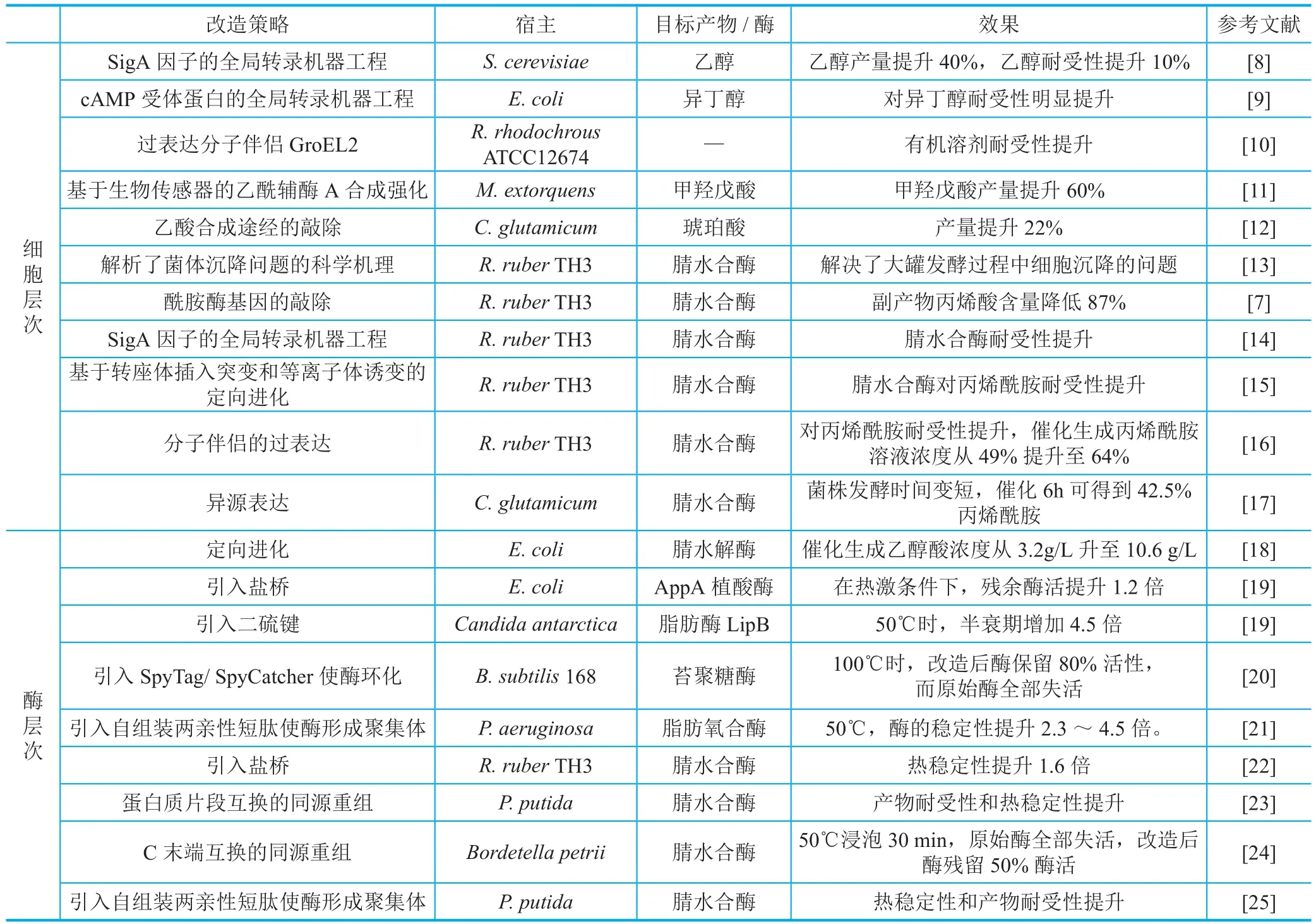
表1 近几年典型的菌株性能改造策略
其中,在丙烯酰胺生产菌株的性能改造方面,为了解决红球菌细胞在大罐发酵过程中发生絮凝沉降的问题,Jiao等[13]发现了红球菌细胞在一定条件下的光滑型-粗糙型菌落分型现象,并将这一现象与发酵过程中的菌体细胞絮凝沉降现象相关联;发现了只有光滑型菌落的菌体可用于大罐发酵,而粗糙型菌落的菌体则会在发酵过程中发生细胞沉降;关联了红球菌菌落分型与细胞表面亲疏水性的关系,发现了疏水性强的细胞具有高沉降速率;阐明了影响细胞表面亲疏水性的关键分子是胞外多糖,还解决了企业在10t及以上大罐发酵过程中不时发生细胞絮凝沉降现象、无法实现高密度培养的问题。
Ma等[7]则通过敲除R. ruber TH中主要的酰胺酶基因,解决了重组细胞在AM生产过程中的副产物丙烯酸生成的问题,得到了高酶活、低副产物的基因工程红球菌R. ruber TH3。
更多的研究关注于如何提高菌体对外界不利环境的耐受性。在细胞中,酶与其他蛋白的作用过程是一个复杂的过程,既有一些辅助蛋白负责酶的正确折叠,也有一些辅助蛋白负责酶失活后的再生。在对腈水合酶生产菌株的研究中,Ma等[14]通过全局转录机器工程的方法,克隆并随机突变了红球菌R.ruber TH3的SigA因子,通过随机突变的方法,筛选得到的突变菌在40%丙烯酰胺溶液浸泡条件下,酶活半衰期延长了1.6倍。通过转座体插入突变和等离子体诱变的方式,孙云鹏等[15]得到的改造菌在40%的丙烯酰胺浸泡时,半衰期提高30min。以上两种方式均是通过随机突变、定向进化方式从而得到稳定性更好地生产菌株。Tian等[16]则通过分子伴侣过表达的方式,利用分子伴侣对于酶失活后的保护和修复作用,在红球菌R. ruber TH3中过表达E. coli的分子伴侣GroEL-ES,不仅使得腈水合酶的酶活有了明显提升,而且当在50%的丙烯酰胺溶液中浸泡时,改造后菌体的腈水合酶剩余酶活是原始菌体的3.8倍。最后利用该改造菌体,催化生成的丙烯酰胺溶液浓度由原始的49%提升至64%。而通过在有机溶剂耐受性更强的菌株中重组表达腈水合酶则是另一种方式,通过将R. rhodochrous M33中的腈水合酶基因在Corynebacterium glutamicum中表达,丙烯酰胺溶液浓度在催化6h后达到42.5%[17]。
2.3 酶分子层次的性能改造策略
游离细胞的催化性能,归根结底取决于胞内酶自身的活性和稳定性。以酶的稳定性改造为例,酶分子层次的改造策略一般集中在定向进化和理性设计两个方面。如前文表1所示,在定向进化方法中,研究者往往通过易错PCR、DNA shuf fl ing等手段构建突变酶后,再利用高效的筛选方法得到耐受性提高的酶[18]。在理性设计方法中,研究者则往往通过改变酶的分子内或者分子间作用力来提高酶的稳定性,其中包括引入二硫键、盐桥、氢键,改变疏水作用[19],嗜热酶的同源重组[19],以及引入SpyTag/SpyCatcher使酶环化[20]、引入自组装两亲性短肽使酶形成聚集体[21]等方式。定向进化方式需要构建大量的突变库,并进行大量且高效的筛选,费时费力,往往适用于酶的结构未知时的改造;而理性设计则一般是在已知酶的三维结构的基础上进行合理的设计。
对于腈水合酶而言,由于大量的腈水合酶的三维结构已经被解析,因此更多的研究集中于理性设计的方法,如图2所示。
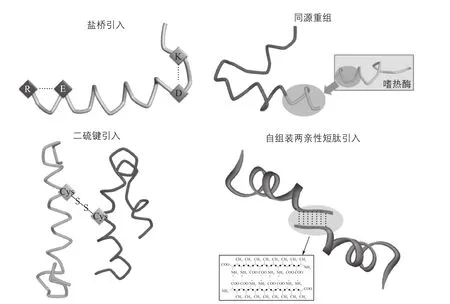
图2 腈水合酶性能改造策略
在对腈水合酶的盐桥引入改造中,Chen等[22]利用分子模拟方法,在R. ruber TH3的腈水合酶中引入了嗜热腈水合酶1UGQ和1V29的稳定区域,通过增加盐桥结构使酶稳定性有所提升。而Cui等[23]则利用位点靶定氨基酸重组技术和分子模拟来确定同源交换的蛋白片段,从而提高酶的稳定性。Sun等[24]则通过直接将嗜热腈水合酶PtNHase的C末端直接引入热敏性腈水合酶BpNHase的C末端的方式来提高腈水合酶的稳定性。在这些改造过程中,酶的稳定性与酶的刚性程度息息相关,无论是嗜热酶的同源重组,还是引入二硫键、盐桥、氢键,其往往是通过增强酶的刚性来实现酶的稳定性的提升。基于这样的原理,Yu等[19]介绍了刚性化柔性位点的方法,结合腈水合酶的三维结构,利用B-FITTER等方法找到该结构中的柔性位点,进而通过引入盐桥、二硫键等方式来增强腈水合酶的稳定性。除了在酶本身引入突变,使柔性位点的刚性增强外,Liu等[25]通过在腈水合酶柔性结构更强的β亚基N末端引入自组装两亲性短肽的方式使腈水合酶形成有序的纳米结构,进而形成水凝胶的方式来提高酶的稳定性。
2.4 酶和细胞协同改造策略
对于腈水合酶而言,酶分子一直是工业生物技术的重要对象,其活性和稳定性是其在工业环境中高效稳定运行的关键要素。然而单纯的在酶层次上的改造有时难以得到理想的效果,充分利用细胞内部环境中分子伴侣等辅助分子对于酶的保护作用,实现重组酶和细胞的协同强化,是实现腈水合酶生产菌株性能提升的有效方式。清华大学化工系进行性能改造的出发菌株是R. ruber TH,经过48h发酵培养后其腈水合酶的酶活可达到300U/mg菌干重(1440万工厂单位),表现出了优良的丙烯酰胺生产性能[7]。然而,如图3所示,该菌株细胞中的酰胺酶会催化部分丙烯酰胺转化为丙烯酸,导致了副产物的生成,造成了原料的浪费和后续的精制成本提高。因此,通过酰胺酶基因的敲除,得到的重组菌R. ruber TH3副产物生成量减少了87%[7]。而过表达分子伴侣GroEL-GroES来协助腈水合酶新生肽链折叠和腈水合酶失活后的修复,则显著提高了重组红球菌细胞的有机溶剂耐受性和热稳定性[16]。在酶分子改造层次,为了进一步提升该生产菌株的腈水合酶催化稳定性,根据“木桶原理”提出了对酶热敏感区“短板”进行搜索并在该“短板”结构区(热不稳定区域和亚基末端)引入嗜热酶盐桥模块、强化盐桥网络提高酶稳定性的新方法[4]。在此基础上,进一步采用分子模拟辅助,成功构建了腈水合酶亚基界面间的二硫键,获得了稳定性显著提高的“双桥”强化腈水合酶。将该重组酶基因采用质粒载体转化光滑型菌落的副产物基因敲除型宿主细胞TH3,获得了染色体-质粒载体“双源”高表达、性能优异的新一代产业化工程菌TH8。TH8菌株的构建策略如图3所示。
在30t水合釜中,TH8重组新菌株不仅单批次细胞催化生产AM的产物浓度可从320g/L(32%)提高到480g/L(48%)以上的高浓度水平,而且实现了重组细胞在大釜中生产高浓度AM的连续3批次回用;此外,无需后续高温浓缩(90~94℃)工序,精制后即可出售AM水剂产品(40%~50%);显著降低生产晶体产品(60%浓度降温结晶)提浓工序的能耗,产生了巨大的经济和社会效益。
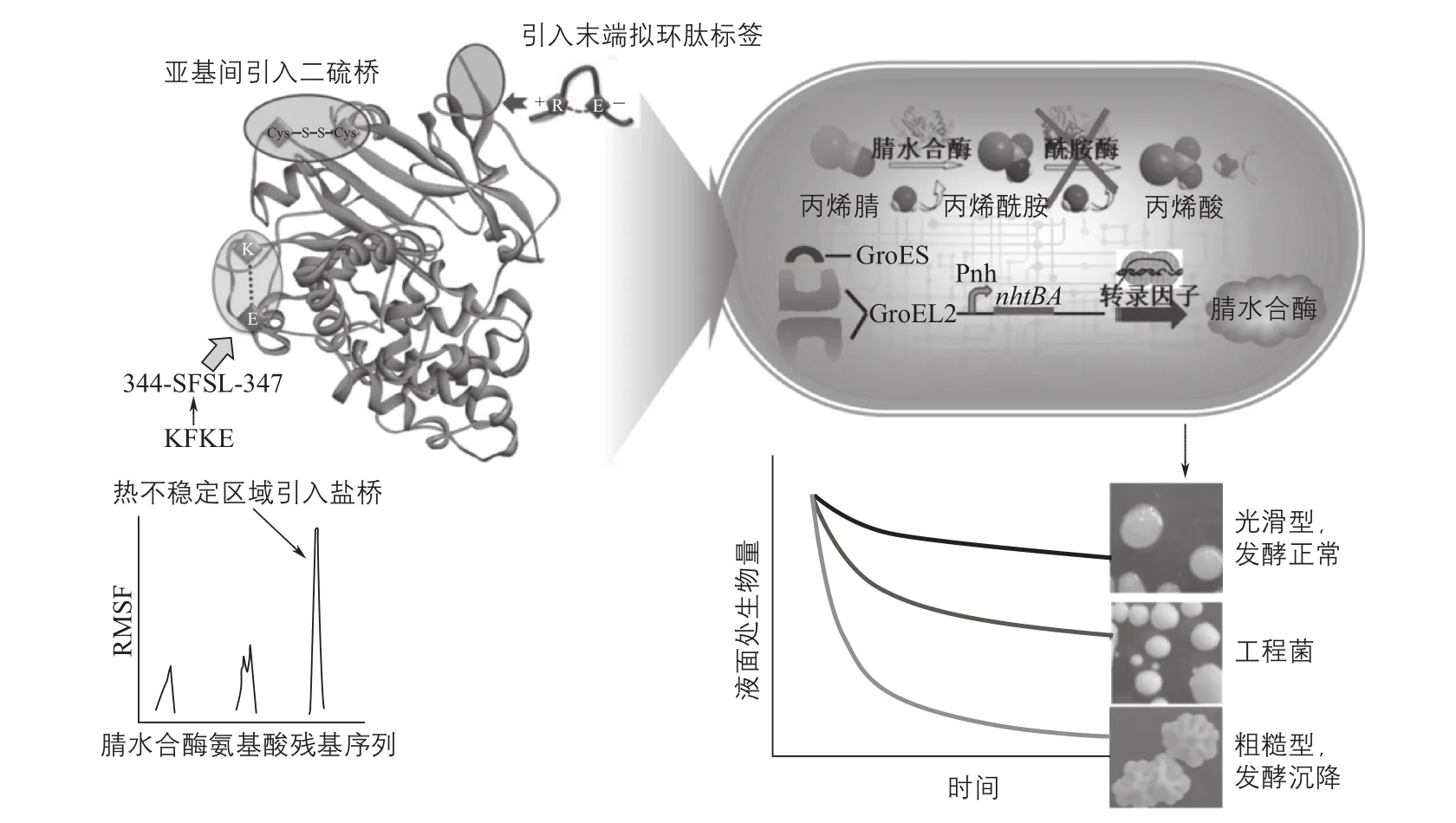
图3 腈水合酶和生产细胞协同改造策略
3 总结与展望
工业酶的活性和稳定性是其在工业环境中高效稳定运行的关键要素。对于已经或接近实现产业化的高活性工业酶,其稳定性(包括热稳定性、有机溶剂耐受性等)强化是工业生物催化的迫切需求,也是最普遍的共性科学问题之一。因此,发展胞内酶分子稳定性强化的有效方法,并实现重组酶和细胞的协同强化,是游离细胞生物催化法工业应用的迫切需求。
聚丙烯酰胺作为重要的驱油剂、水处理剂,在我国国民经济中具有重要地位,而作为其单体的丙烯酰胺的需求仍在不断增长。微生物法作为丙烯酰胺生产的主要方法,其生产菌株的性能对于生产效率的提升和节能、降耗、减排具有重要意义。而通过酶分子水平和细胞层面各种基因工程方法的协同改造,我国的腈水合酶生产菌株性能已经有了巨大的提升。
今后,全新的高活性、高稳定性目标酶的发掘与改造、高性能游离酶催化新工艺、固定化酶催化新工艺以及微反应器生物催化新工艺[26-28]等新型反应过程的开发与强化,都是生物法生产丙烯酰胺乃至其他高附加值化学品生产新工艺的重要发展方向。
[1] BORNSCHEUER U T,HUISMAN G W,KAZLAUSKAS R J,et al.Engineering the thirdwave of biocatalysis[J]. Nature,2012,485(7397):185-194.
[2] 林建平,吴坚平,杨立荣. 精细化学品的生物合成[J]. 生物产业技术,2016,5:7-16.
[3] 向晓丽. 微生物法生产丙烯酰胺技术概况[J]. 湖北化工,2000,17(6):35-36.
[4] 陈杰. 微生物法生产丙烯酰胺的红球菌细胞-腈水合酶协同改造[D].北京:清华大学,2015.
[5] YAMADA H,KOBAYASHI M. Nitrile hydratase and its application to industrial production of acrylamide[J]. Bioscience,Biotechnology,and Biochemistry,1996,60(9):1391-1400.
[6] 孙旭东. 膜生物反应器游离细胞催化体系生产丙烯酰胺的研究[D]. 北京:清华大学,2004.
[7] MA Y,YU H,PAN W,et al. Identification of nitrile hydrataseproducing Rhodococcus ruber TH and characterization of an ami E-negative mutant[J]. Bioresource Technology,2010,101(1):285-291.
[8] QIU Z,JIANG R. Improving Saccharomyces cerevisiae ethanol production and tolerance via RNA polymeraseⅡsubunit Rpb7[J].Biotechnology for Biofuels,2017,10(1):125-137.
[9] CHONG H,GENG H,ZHANG H,et al. Enhancing E. coli isobutanol tolerance through engineering its global transcription factor cAMP receptor protein(CRP)[J]. Biotechnology and Bioengineering,2014,111(4):700-708.
[10] TAKIHARA H,MATSUURA C,OGIHARA J,et al. Rhodococcus rhodochrous ATCC12674 becomes alkane-tolerant upon GroEL2 overexpression and survives in the n-octane phase in two phase culture[J]. Microbes and Environments,2014,29(4):431-433.
[11] LIANG W F,CUI L Y,CUI J Y,et al. Biosensor-assisted transcriptional regulator engineering for Methylobacterium extorquens AM1 to improve mevalonate synthesis by increasing the acetyl-CoA supply[J]. Metabolic Engineering,2017,39:159-168.
[12] ZHU N,XIA H,WANG Z,et al. Engineering of acetate recycling and citrate synthase to improve aerobic succinate production in Corynebacterium glutamicum[J]. PLoS One,2013,8(4):e60659.
[13] JIAO S,CHEN J,YU H,et al. Tuning and elucidation of the colony dimorphism in Rhodococcus ruber associated with cell fl occulation in large scale fermentation[J]. Applied Microbiology and Biotechnology,2017,101(16):6321-6332.
[14] MA Y,YU H. Engineering of Rhodococcus cell catalysts for tolerance improvement by sigma factor mutation and active plasmid partition[J]. Journal of Industrial Microbiology amp; Biotechnology,2012,39(10):1421-1430.
[15] 孙云鹏. 转座体插入突变与等离子体诱变强化红球菌耐受性的研究[D]. 北京:清华大学,2010.
[16] TIAN Y,CHEN J,YU H,et al. Overproduction of the Escherichia coli chaperones GroEL-GroES in Rhodococcus ruber improves the activity and stability of cell catalysts harboring a nitrile hydratase[J].Journal of Microbiology and Biotechnology,2016,26:337-346.
[17] KANG M S,HAN S S,KIM M Y,et al. High-level expression in Corynebacterium glutamicum of nitrile hydratase from Rhodococcus rhodochrous for acrylamide production[J]. Applied Microbiology and Biotechnology,2014,98(10):4379-4387.
[18] LUO H,MA J,CHANG Y,et al. Directed evolution and mutant characterization of Nitrilase from Rhodococcus rhodochrous tg1-A6[J]. Applied Biochemistry and Biotechnology,2016,178(8):1510-1521.
[19] YU H,HUANG H. Engineering proteins for thermostability through rigidifying fl exible sites[J]. Biotechnology Advances,2014,32(2):308-315.
[20] WANG J,WANG Y,WANG X,et al. Enhanced thermal stability of lichenase from Bacillus subtilis 168 by SpyTag/SpyCatcher-mediated spontaneous cyclization[J]. Biotechnology for Biofuels,2016,9(1):79.
[21] LU X,LIU S,ZHANG D,et al. Enhanced thermal stability and speci fi c activity of Pseudomonas aeruginosa lipoxygenase by fusing with self-assembling amphipathic peptides[J]. Applied Microbiology and Biotechnology,2013,97(21):9419-9427.
[22] CHEN J,YU H,LIU C,et al. Improving stability of nitrile hydratase by bridging the salt-bridges in speci fi c thermal-sensitive regions[J].Journal of Biotechnology,2013,164(2):354-362.
[23] CUI Y,CUI W,LIU Z,et al. Improvement of stability of nitrile hydratase via protein fragment swapping[J]. Biochemical and Biophysical Research Communications,2014,450(1):401-408.
[24] SUN W,ZHU L,CHEN X,et al. The stability enhancement of nitrile hydratase from Bordetella petrii by swapping the C-terminal domain of β subunit[J]. Applied Biochemistry and Biotechnology,2016,178(8):1481-1487.
[25] LIU Y,CUI W,LIU Z,et al. Enhancement of thermo-stability and product tolerance of Pseudomonas putida nitrile hydratase by fusing with self-assembling peptide[J]. Journal of Bioscience and Bioengineering,2014,118(3):249-252.
[26] LI J,CHEN J,WANG Y,et al. Hydration of acrylonitrile to produce acrylamide using biocatalyst in a membrane dispersion microreactor[J]. Bioresource Technology,2014,169:416-420.
[27] LI J,LIU J,CHEN J,et al. Multiple reuses of Rhodococcus ruber TH3 free cells to produce acrylamide in a membrane dispersion microreactor[J]. Bioresource Technology,2015,187:198-204.
[28] LI J,GUO M,JIAO S,et al. A kinetic study of the biological catalytic hydration of acrylonitrile to acrylamide[J]. Chemical Engineering Journal,2017,317:699-706.
Genetic strategies to engineer recombinant strains for acrylamide production using free resting cells as biocatalysts
YU Huimin,JIAO Song,SHEN Zhongyao
Department of Chemical Engineering, Tsinghua University, Key Laboratory of Industrial Biocatalysis(Ministry of Education),Beijing 100084, China
Acrylamide is one kind of important amide compounds. Polyacrylamide, the polymerized product of acrylamide, has been widely used in enhanced oil recovery, water treatment, papermaking industry, metallurgy and so on.Because of the advantages such as high productivity, high product purity and environmental-friendly, biocatalysis approach using the free resting cells harboring nitrile hydratase as the biocatalysts is the main method to produce acrylamide so far. As a result, the performances of nitrile hydratase and its producing strains play a vital role in the whole process. In this paper,we summarized the strategies commonly used to evolve the engineered strains from both the cell level and the enzyme level;and especially highlighted the superior engineered strains harboring nitrile hydratase for acrylamide production. Finally,we introduced a synergistic evolution strategy for the cells of Rhodococcus and in-cell nitrile hydratases. With this method,we obtained the engineered strain R. ruber TH8. During the industrial scale bioproduction of acrylamide through free-cell catalysis of TH8, about 500 g/L acrylamide were obtained and the recombinant cells could be reused for three batches.
acrylamide; nitrile hydratase; free-cell catalysis; synergistic evolution of cells and in-cell enzymes
10.3969/j.issn.1674-0319.2017.06.007
国家973计划(2013CB733600),国家自然科学基金(21476126,21776157)

