甘油原料转化生产手性环氧氯丙烷关键酶的开发
张晓健 ,郑裕国
1.浙江工业大学生物工程学院,杭州 310014 2.浙江省生物有机合成技术研究重点实验室,杭州 310014
甘油原料转化生产手性环氧氯丙烷关键酶的开发
张晓健1,2,郑裕国1,2
1.浙江工业大学生物工程学院,杭州 310014 2.浙江省生物有机合成技术研究重点实验室,杭州 310014
手性环氧氯丙烷是一种重要的三碳手性合成子,在医药、农药、化工、材料等领域有着广泛的应用。开发以甘油替代石油基原料合成手性环氧氯丙烷的绿色合成工艺具有重要的开发价值。生物催化技术可有效提高过程安全性与原子经济性,降低“三废”排放,提升产品质量。阐述了生物催化合成手性环氧氯丙烷关键酶技术的研究进展,进行了生物合成路线设计、卤化酶酶库构建、卤醇脱卤酶与环氧化物水解酶的筛选与改造、卤醇脱卤酶/环氧化物水解酶双酶串联合成手性环氧氯丙烷工艺构建等技术开发,为手性环氧氯丙烷绿色生物合成技术的研究与应用提供理论基础与技术支持。
甘油;手性环氧氯丙烷;1,3-二氯-2-丙醇;生物催化;卤醇脱卤酶;环氧化物水解酶
新世纪以来,作为化石燃料的重要替代产品,生物柴油备受青睐,产量与市场迅速扩张。在生物柴油生产过程中,会产生大量的副产物甘油[1-2]。随着生物柴油产业的飞速发展,甘油在全球范围内过剩,市场价格一路下跌。相较于石油基原料,从甘油出发,合成具有高附加值的甘油衍生产品不但具有原料可再生、绿色环保等优点,而且在生产成本上逐渐显现优势,成为绿色化工的重要新兴领域[3-5]。
手性环氧氯丙烷(epichlorohydrin,ECH)是一种重要的三碳手性合成子,广泛应用于手性制药与精细化工产业,是包括新型他汀类降血脂药物、芳氧丙胺醇类β-肾上腺素拮抗剂、抗生素海藻唑啉、左旋肉碱、维生素等多种重要手性药物的合成前体(图1)[6-7]。随着 手性制药与精细化工产业的不断扩张,对手性环氧氯丙烷的需求也随之不断扩大。
传统的手性环氧氯丙烷合成方法是以石油基原料丙烯出发合成手性环氧氯丙烷,存在着原料价格高、副产物多、原子经济性差、“三废”排放大等严重问题。近年来,甘油出发的合成路线得到发展,主要是通过甘油氯化合成1,3-二氯-2-丙醇,再经皂化反应生产环氧氯丙烷,最后采用salen催化剂拆分获得(R)-或者(S)-环氧氯丙烷。该技术虽已在工业生产中得到应用推广,但仍存在一些严重缺陷:生产过程中需加温加压,属于高耗能过程;存在大量强酸强碱的使用,环保问题突出;手性拆分制备手性环氧氯丙烷,理论产率不超过50%,催化剂价格昂贵。当前的手性环氧氯丙烷生产工艺不能满足现代绿色化学的发展要求[6-8]。
生物催化具有高活性、高立体选择性、反应条件温和等优势,成为可持续发展过程中替代和拓展传统有机化学合成的重要方法。探索开发手性环氧氯丙烷的生物催化合成途径是近年来科学家研究的热点[6]。本文结合国内外研究进展,基于笔者团队研究成果,探索开发甘油出发的手性环氧氯丙烷生物合成技术,开展了酶法合成路线设计、关键酶的开发及生物催化体系的构建优化等工作,为手性环氧氯丙烷的绿色合成技术开发提供理论指导与技术支持。
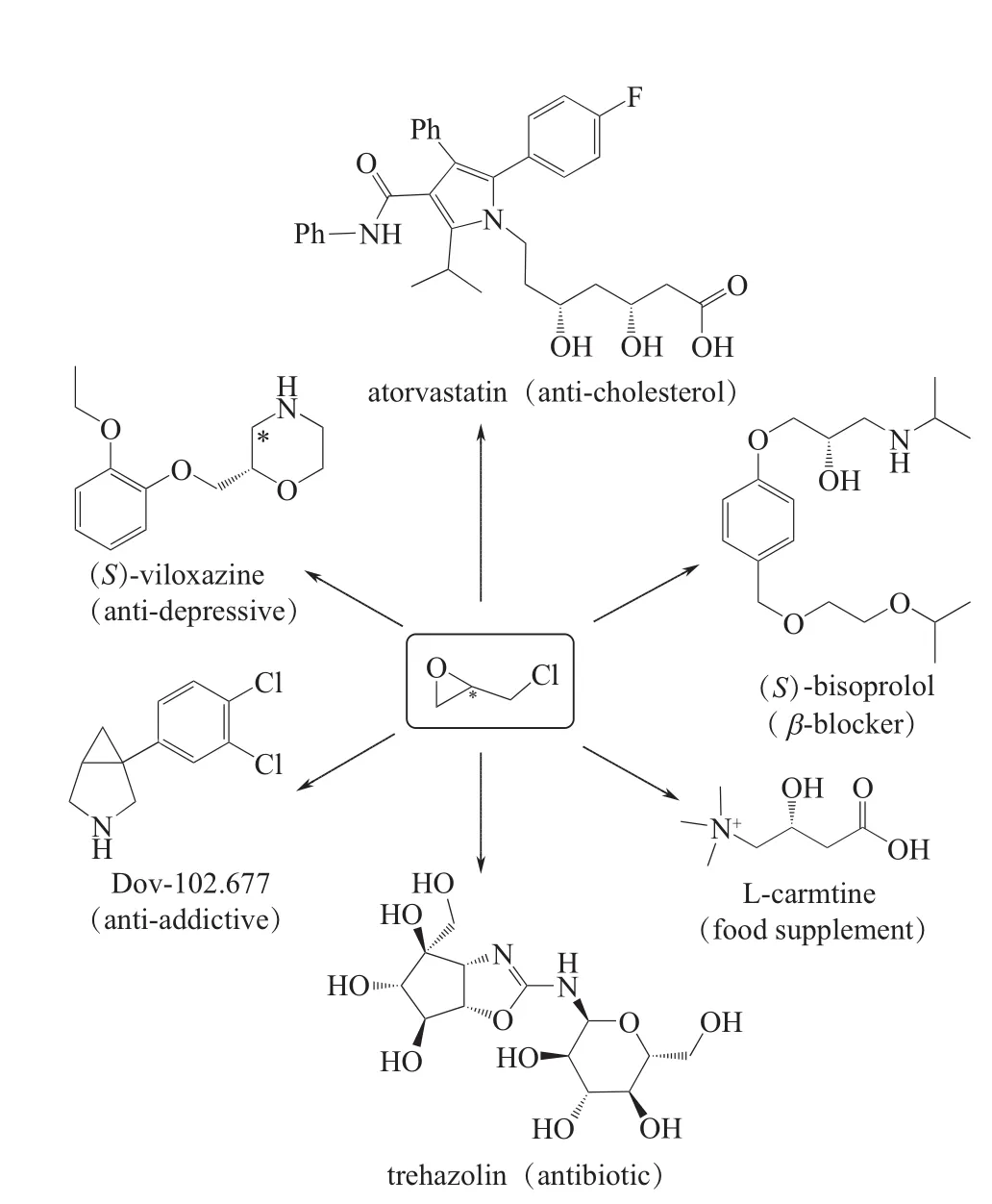
图1 手性环氧氯丙烷的用途
1 三酶催化合成手性环氧氯丙烷路线分析
针对当前手性环氧氯丙烷合成工艺反应条件苛刻、“三废”排放严重、催化剂价格昂贵等问题,笔者团队以甘油为出发原料,深入分析合成路径、生物催化剂介入可行性与原子经济性等,引入三步生物催化步骤,构建手性环氧氯丙烷的生物合成过程(图2)。首先设计采用卤化酶催化甘油氯化合成1,3-二氯-2-丙醇,之后利用卤醇脱卤酶脱卤环化制备环氧氯丙烷,最后在立体选择性环氧化物水解酶的作用下,水解拆分获得光学纯的R或S型环氧氯丙烷。该三酶组合催化合成工艺彻底革除了原有化学合成工艺中强酸高温氯化、皂化、贵金属催化剂拆分等过程,产物光学纯度高,催化剂成本明显降低,酸碱用量大幅压缩,副产物、溶剂用量、“三废”排放量和能耗大幅度降低,具有显著的优越性。
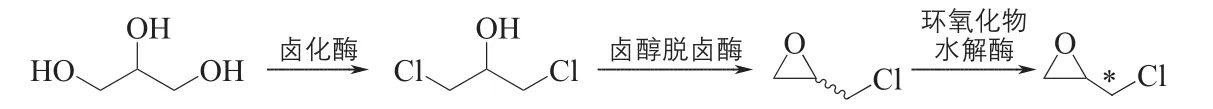
图2 以甘油为原料合成手性环氧氯丙烷的生物合成工艺线路
2 卤化酶催化甘油氯化的探索
生物催化卤化在21世纪逐渐成为新的研究热点,不断有新的含卤生物物质被发现,相关的生物卤化过程被探讨,新型的卤化酶被发掘研究。目前已经报道的卤化酶主要有四类:过氧化物卤化酶(haloperoxidase)、黄素依赖卤化酶( fl avin-dependent halogenase)、无血红素铁依赖卤化酶(non-haem irondependent halogenase)以及SA M为底物卤化酶(halogenases using S- adenosylmethionine)等[9-12]。Wu等[13]利用氯过氧化物酶催化3-氯丙烯氧化反应合成(R)-环氧氯丙烷,产物e.e.值为97.5%,产率达67.3%。然而,对生物卤化反应的研究尚处于起步阶段,已经报道具有卤化能力的微生物仍较少,卤化酶数量和种类更是屈指可数,生物卤化机制的研究尚处于起步阶段,大大制约了生物催化卤化技术的开发与应用。目前国内外尚无甘油的生物催化氯化的报道,相关催化机制也不明确。
笔者团队通过基因挖掘等生物信息学手段,在GenBank、EMBL、DDBJ等公共DNA数据库快速筛选、挖掘潜在的卤化酶基因序列,并加以功能验证。共获得42条过氧化物卤化酶序列、32条黄素依赖卤化酶序列、26条无血红素铁依赖卤化酶序列以及潜在的3条SAM为底物卤化酶序列。合成以上基因并构建重组载体,在工程菌中进行卤化酶的高效可溶表达,建立了大容量、多样性的卤化酶酶库。以此为基础,并跟踪生物卤化领域的国内外最新进展,综合利用结构模拟、定向进化、基因编辑等多种手段进行潜在甘油氯化酶的筛选、改造及生物催化甘油氯化机制的研究。
3 卤醇脱卤酶催化1,3-二氯丙醇合成环氧氯丙烷
卤醇脱卤酶(halohydrin dehalogenase,Hhe,EC 4.5.1.X)属于短链脱氢酶/还原酶家族,可通过分子内亲核取代机制催化邻卤醇转化为环氧化物和卤化氢。近年来的研究发现,卤醇脱卤酶在生物催化与转化领域发挥着重要的作用,利用其合成光学纯的手性环氧化物中间体具有巨大的应用价值。国内外已经开展卤醇脱卤酶催化合成手性环氧氯丙烷的研究,并取得了一定的成果[14-15]。
3.1 卤醇脱卤酶的筛选与酶库构建
长期以来,有报道的卤醇脱卤酶的数量非常有限。2013年,科学家通过宏基因组筛选、数据发掘等方法,筛选获得多株卤醇脱卤酶,并根据同源性把这些新发现的酶分为四个亚类,拓展了卤醇脱卤酶的种类和多样性[16]。在现有的卤醇脱卤酶中,对手性环氧氯丙烷合成具有立体选择性催化能力的酶株十分有限,且立体选择性往往不理想[6,17]。来源于Arthrobacter erithii H10a的卤醇脱卤酶DehA可催化1,3-二氯-2-丙醇合成(R)-环氧氯丙烷,e.e.值达89.3%。 来 源 于Corynebacterium sp. N-1074的HheB,选择性催化合成(R)-环氧氯丙烷的e.e.值达90%[18]。
笔者团队基于不同pH下酚酞溶液的吸光度变化,建立了产卤醇脱卤酶微生物高通量筛选模型,从环境样品中筛选对1,3-二氯-2-丙醇具有较强脱卤环氧化能力的菌株,并通过基因挖掘等生物信息学手段,在公共DNA数据库快速筛选、挖掘潜在的卤醇脱卤酶并进行功能验证。目前已获得32条卤醇脱卤酶序列,并在大肠杆菌中进行重组表达,构建了多株高活性、高立体选择性的卤醇脱卤酶重组工程菌。以此为基础,建立用于环氧氯丙烷合成的卤醇脱卤酶酶库。其中,来源于Tistrella mobilis ZJB1405的卤醇脱卤酶HHDHTm催化合成(S)-环氧氯丙烷的e.e.值>60%,反应2 min,底物转化率达95.2%[19]。
3.2 卤醇脱卤酶的立体选择性催化机制与分子改造
研究显示卤醇脱卤酶的立体选择性受到酶的底物口袋尺寸、底物口袋和卤素结合域相关氨基酸残基等因素的影响。Tang等[20]对来源于Arthrobacter sp. AD2的卤醇脱卤酶HheAAD2的定点突变研究显示,改变酶的卤素离子结合域关键氨基酸残基可显著影响酶对底物2-氯-1-苯乙醇的立体选择性。对来源于Arthrobacter sp. AD1的HheC进行迭代饱和突变,改变底物口袋相关的氨基酸残基并进而改变底物p-硝基-2-溴-1-苯乙醇在活性中心的定位与构象,产生的突变酶具有极高的S型底物选择性[21-22]。环氧氯丙烷分子小,在酶活中心空间位阻小,手性识别困难,目前关于卤醇脱卤酶立体选择性催化手性环氧氯丙烷的分子机制尚未得到深入揭示。
笔者团队对HheC的三维蛋白结构分析发现,175~188位氨基酸残基形成的环参与卤素离子的结合,推测以上位点在催化过程中的构象变化参与了酶的立体选择性调节。Phe12和Trp249位氨基酸残基也参与卤素离子的稳定与释放,对酶的立体选择性与活性发挥着重要作用(图3)。选择以上位点进行定点突变和联合突变,经高通量筛选获得高活性HheC突变体P175S/W249P,催化1,3-二氯-2-丙醇合成(S)-环氧氯丙烷的e.e.值由野生型HheC的5.7%提高至95.3%。
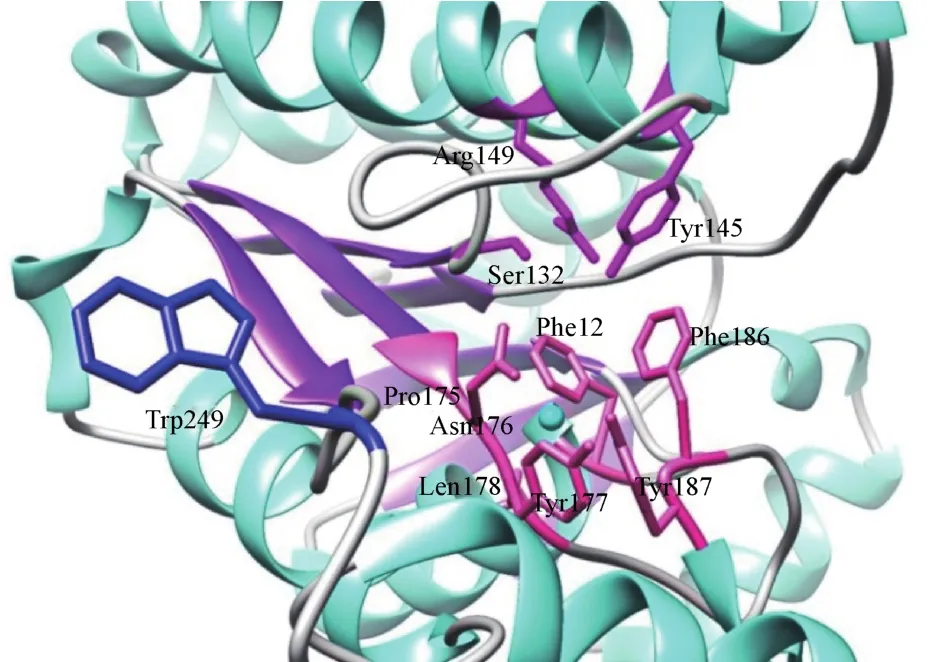
图3 卤醇脱卤酶HheC的三维结构
3.3 卤醇脱卤酶催化手性环氧氯丙烷反应体系优化
将HheC突变体P175S/W249P在大肠杆菌工程菌中重组表达,以重组工程菌为全细胞催化剂,催化1,3-二氯-2-丙醇合成手性环氧氯丙烷。通过反应体系优化与反应过程控制,构建并优化卤醇脱卤酶催化手性环氧氯丙烷反应体系。底物浓度为20mmol/L、pH10.0时,反应5min,(S)-环氧氯丙烷的收率达93.7%,e.e.值达95.3%。当底物浓度提升至40mmol/L时,(S)-环氧氯丙烷的产率与e.e.值分别为90.2%和92.1%(图4)。产物e.e.值与产率等主要指标优于国内外文献报道水平[18-19,23]。
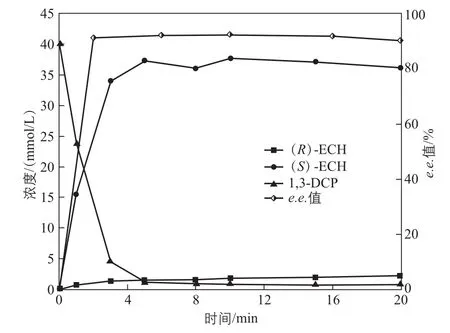
图4 HheC突变体P175S/W249P不对称催化(S)-环氧氯丙烷合成反应过程
4 环氧水解酶拆分制备手性环氧氯丙烷
4.1 环 氧水解酶的筛选
环氧化物水解酶(epoxide hydrolase,EH,EC 3.3.2.3)能够催化外消旋环氧化物选择性水解,制备高光学纯度的环氧化物和相应的邻位二醇。微生物来源的环氧化物水解酶来源广,获得方便,且具有很高的立体选择性,在手性环氧化物生物合成中的应用已成为研究热点[24-25]。利用环氧化物水解酶可对外消旋的环氧氯丙烷进行高效拆分,获得光学纯的手性环氧氯丙烷。
笔者团队以环氧氯丙烷为目标底物,从环境样品中筛选或在公共DNA数据库中进行基因挖掘,获得37条环氧化物水解酶序列,以此为基础,建立了环氧化物水解酶酶库。其中来源于Parvibaculum lavamentivorans ZJB14001的PlEH2催化拆分制备(S)-环氧氯丙烷的e.e.值>99%,产率为12.8%。来源于Agromyces mediolanus的AmEH水解拆分消旋环氧氯丙烷制备(S)-环氧氯丙烷的e.e.值>99%,产率为21.5%[26]。
4.2 环氧化物水解酶的立体选择性催化机制与分子改造
近年来,环氧化物水解酶的分子机制逐渐得到揭示,通过理性、半理性设计,定向进化等手段进行分子改造获得高活性、高立体选择性环氧化物水解酶逐渐引起科学家们的兴趣。Spelberg等[27]对来源于Agrobacterium radiobacter AD1 的ArEH进行定点突变,将参与底物结合和环氧开环的Tyr突变为Phe后,突变酶对R型芳香类环氧化物的对映选择性提高了2~5倍。Thomaeus等[28]基于土豆环氧化物水解酶StEH的晶体结构与稳态动力学分析,对Tyr149位点氨基酸残基进行突变,破坏了质子传递链氨基酸残基与水的氢键,显著提高了酶对反式二苯乙烯氧化物的对映选择性。
笔者团队选择对环氧化物水解酶AmEH进行定点饱和突变与联合突变,构建大容量突变文库。基于4-对硝基苄基嘧啶与环氧化物反应在600nm处吸光值发生变化的原理,建立环氧化物水解酶高通量筛选模型。筛选获得AmEH突变体W182F/S207V/N240D。突变体改变了关键亲核氨基酸残基Asp181的氧原子与被进攻的(R)-环氧氯丙烷或(S)-环氧氯丙烷的环氧碳原子之间的距离与夹角(图5),催化(R)-环氧氯丙烷水解的酶活及对映体选择率分别是原始酶的约1.7倍和7.0倍。
4.3 环氧化物水解酶促手性拆分反应条件优化
20世纪90年代,科学家即开始了对环氧水解酶拆分环氧氯丙烷的研究。Woo等[29]利用来自Novosphingobium aromaticivorans的环氧化物水解酶催化拆分环氧氯丙烷,底物浓度500mmol/L,获得(S)-环氧氯丙烷的e.e.值>99%,产率为10.7%。Yildirim等[30]利用来自Aspergillus niger的环氧化物水解酶拆分外消旋环氧氯丙烷,产物(S)-环氧氯丙烷的e.e.值为99%,产率为26%。
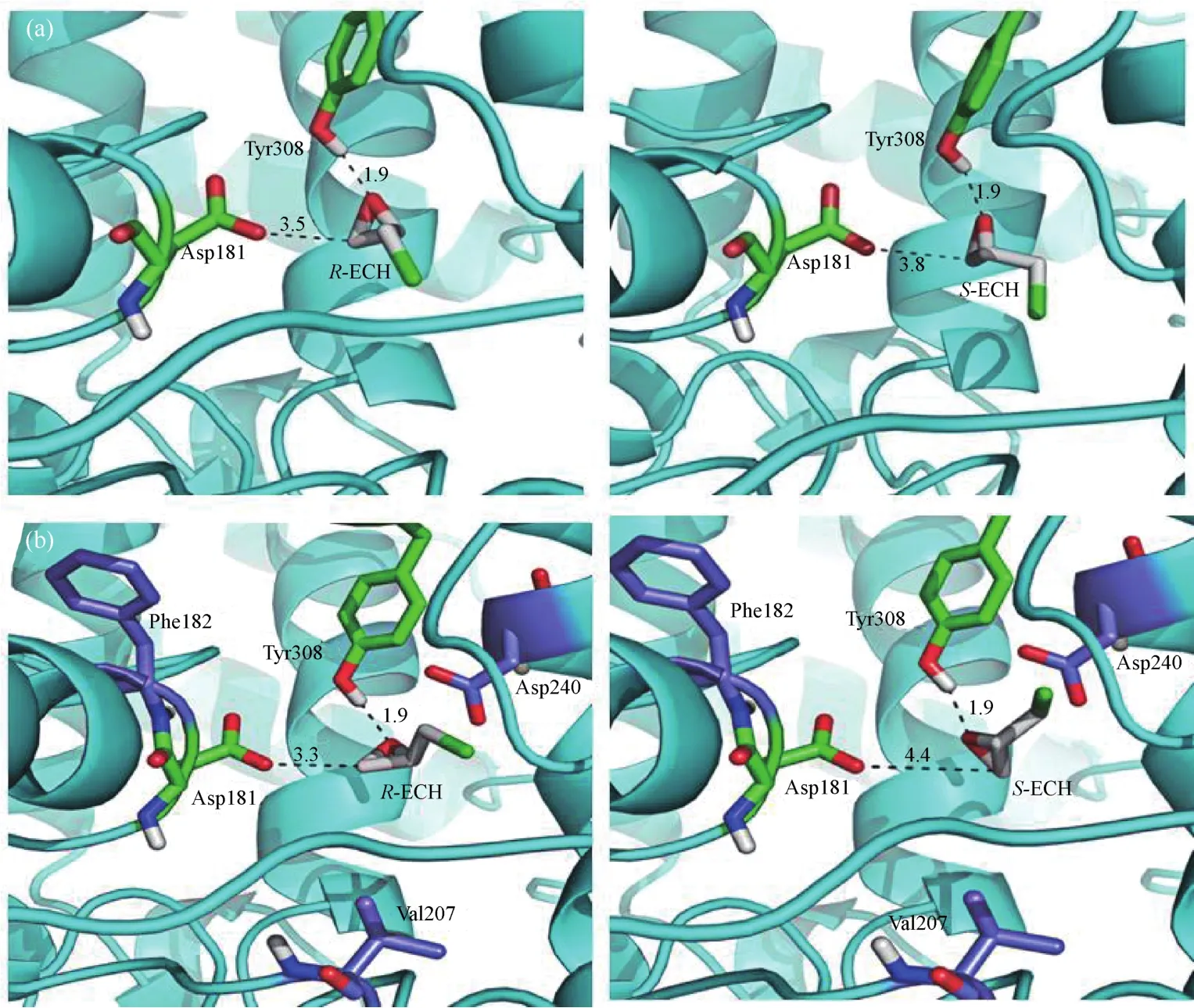
图5 AmEH(a)或突变体(b)分别与(R)-或(S)-环氧氯丙烷的分子对接模拟
笔者团队在前期分子改造研究的基础上,以表达AmEH突变体W182F/S207V/N240D重组大肠杆菌为全细胞催化剂,通过反应体系优化,构建了环氧化物水解酶手性拆分环氧氯丙烷技术。底物浓度75mmol/L,反应35min,拆分获得(S)-环氧氯丙烷的e.e.值>99.9%,产率达45.8%;底物浓度进一步提高至450mmol/L,反应90min,(S)-环氧氯丙烷的e.e.值>99.9%,产率为40.5% (图6)[31]。与目前已报道的环氧化物水解酶相比,AmEH突变体的对映选择性高,并且具有较高的底物耐受性与产物产率,工业应用开发潜力巨大。
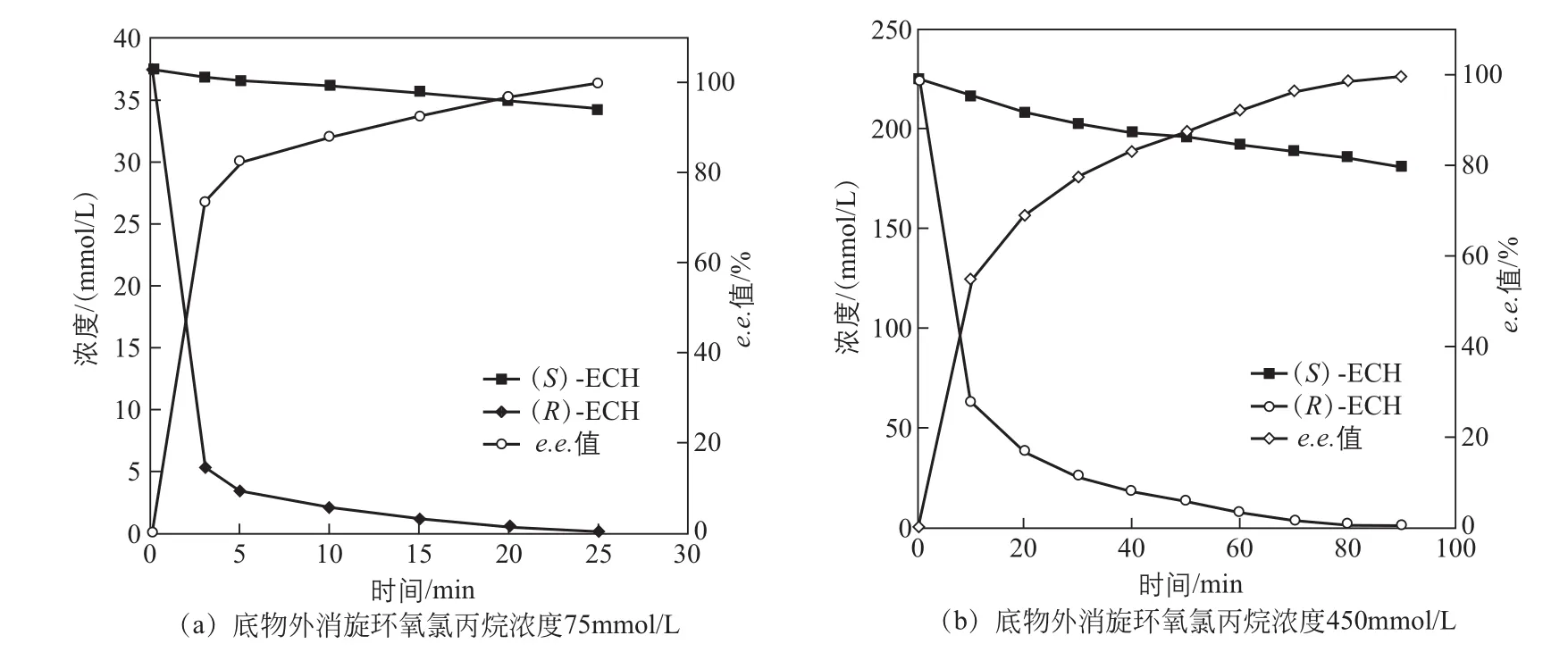
图6 环氧化物水解酶AmEH突变体催化不对称拆分外消旋环氧氯丙烷的反应进程
5 双酶偶联催化制备手性环氧氯丙烷
卤醇脱卤酶催化1,3-二氯-2-丙醇不对称合成手性环氧氯丙烷,理论产率可达100%,但由于目前已有卤醇脱卤酶的立体选择性尚不理想,产物的光学纯度还不能达到工业生产要求。环氧化物水解酶拆分制备手性环氧氯丙烷具有高度立体选择性,但理论产率不超过50%。笔者团队构建了卤醇脱卤酶/环氧化物水解酶双酶串联催化合成(S)-环氧氯丙烷技术,利用立体选择性卤醇脱卤酶催化1,3-二氯-2-丙醇高效合成(S)-环氧氯丙烷,利用环氧化物水解酶选择性水解体系中少量的(R)-环氧氯丙烷,实现手性环氧氯丙烷的高产率、高光学纯制备。
利用含卤醇脱卤酶HheC突变体P175S/W249P重组工程菌与含环氧化物水解酶AmEH突变体W182F/S207V/N240D重组工程菌为催化剂,通过反应体系优化与过程控制,构建双酶串联催化合成(S)- 环氧 氯丙烷技术。底物浓度20mmol/L,经双酶串联催化,反应10~15min,产物(S)-环氧氯丙烷的e.e.值>99.9%,产率达91.2%。底物浓度提高至100mmol/L,产物e.e.值>99%,产率为54.2%[18]。该技术克服了手性环氧氯丙烷合成中,使用单一生物催化剂的不足,在原子经济性、产品光学纯度与产率方面优势明显,开发潜力巨大。
6 总结与展望
采用生物催化技术,由甘油出发合成手性环氧氯丙烷,具有成本低廉、条件温和、环境友好等突出特点,符合当代绿色经济的发展趋势。笔者团队通过生物催化路线设计、关键酶的筛选与改造、双酶串联催化工艺构建等技术开发,探索建立手性环氧氯丙烷的生物合成技术。笔者课题的研究对于开发甘油出发生产手性环氧氯丙烷的绿色合成技术具有重要参考意义。
甘油的生物催化氯化与高光学纯度、高产率的手性环氧氯丙烷的生物制备是该领域研究的难点所在,相关研究方兴未艾。相信在不久的未来,随着生物信息学、基因组与蛋白质组学、基因编辑技术的快速发展,生物资源开发与生物催化剂改造技术进入更高层次,高活性甘油氯化酶与高立体选择性卤醇脱卤酶、环氧化物水解酶等关键酶将得到进一步深入研发,进而推动手性环氧氯丙烷生物合成技术的发展与应用,助力我国传统化工产业的改造升级。
[1] OKOYE P U,HAMEED B H. Review on recent progress in catalytic carboxylation and acetylation of glycerol as a byproduct of biodiesel production[J]. Renewable amp; Sustainable Energy Reviews,2016,53:558-574.
[2] SINGH D,GANESH A,MAHAJANI S. Heterogeneous catalysis for biodiesel synthesis and valorization of glycerol[J]. Clean Technologiesamp; Environmental Policy,2015,17(4):1103-1110.
[3] SUN S,HE M,DAI Y,et al. Catalytic acetalization:an efficient strategy for high-value utilization of biodiesel-derived glycerol[J].Catalysts,2017,7(6):184-195.
[4] GONZALEZGARAY A,GONZALEZMIQUEL M,GOSALBEZ G G. High-value propylene glycol from low-value biodiesel glycerol:a techno-economic and environmental assessment under uncertainty[J]. ACS Sustainable Chemistry amp; Engineering,2017,5(7):5723-5732.
[5] PRADIMA J,KULKARNI M R,ARCHNA. Review on enzymatic synthesis of value added products of glycerol,a by-product derived from biodiesel production[J/OL]. Resource-Efficient Technologies,2017[2017-11-21]. https://doi.org/10.1016/j.reffit.2017.02.009. DOI:10.1016/j.ref fi t.2017.02.009.
[6] JIN H X,OUYANG X K. Enzymatic approaches to the preparation of chiral epichlorohydrin[J]. RSC Advances,2015,5(113):92988-92994.
[7] GILBEAU P,KRAFFT P. Process for manufacturing epichlorohydrin:EP20080709018[P]. 2008-02-15.
[8] ALMENA A,MARTÍN M. Techno-economic analysis of the production of epichlorohydrin from glycerol[J]. Computer Aided Chemical Engineering,2016,38(12):49-54.
[9] STEPHENS J T,PETE M J. Bioactive heme-haloperoxidase compositions and methods of their use:US2015/011435[P]. 2015-01-09.
[10] SHEPHERD S A,KARTHIKEYAN C,LATHAM J,et al.Extend ing the biocatalytic scope of regiocomplementary flavindependent halogenase enzymes[J]. Chemical Science,2015,6(6):3454-3460.
[11] SMITH J L,KHARE D. Recent advances in the structural and mechanistic biology of non-haem Fe(ii),2-oxoglutarate and O2-dependent halogenases[M]//BOLLINGER M J,MYLLYHARJU J,TRIEVEL R,et al. 2-Oxoglutarate-Dependent Oxygenases.Cambridge UK:Royal Society of Chemistry,2015:401-413.
[12] EUSTÁQUIO A S,HÄRLE J,NOEL J P,et al. S-Adenosyl-L-methionine hydrolase(adenosine-forming),a conserved bacterial and archaeal protein related to SAM-dependent halogenases[J].ChemBioChem,2008,9(14):2215-2219.
[13] WU J,LIU C,JIANG Y,et al. Synthesis of chiral epichlorohydrin by chloroperoxidase-catalyzed epoxidation of 3-chloropropene in the presence of an ionic liquid as co-solvent[J]. Catalysis Communications,2010,11(8):727-731.
[14] SCHALLMEY A,SCHALLMEY M. Recent advances on halohydrin dehalogenases-from enzyme identification to novel biocatalytic applications[J]. Applied Microbiology and Biotechnology,2016,57(18):351-360.
[15] YOU Z Y,LIU Z Q,ZHENG Y G. Properties and biotechnological applications of halohydrin dehalogenases:current state and future perspectives[J]. Applied Microbiology and Biotechnology,2013,97(1):9-21.
[16] SCHALLMEY M,KOOPMEINERS J,WELLS E,et al. Expand ing the halohydrin dehalogenase enzyme family:Identification of novel enzymes by database mining[J]. Applied and Environmental Microbiology,2014,80(23):7303-7315.
[17] KOOPMEINERS J,HALMSCHLAG B,SCHALLMEY M,et al.Biochemical and biocatalytic characterization of 17 novel halohydrin dehalogenases[J]. Applied Microbiology and Biotechnology,2016,100(17):1-11.
[18] XUE F,LIU Z Q,WANG Y J,et al. Efficient synthesis of (S)-epichlorohydrin in high yield by cascade biocatalysis with halohydrin dehalogenase and epoxide hydrolase mutants[J]. Catalysis Communications,2015,72(5):147-149.
[19] XUE F,LIU Z Q,WANG Y J,et al. Biochemical characterization and biosynthetic application of a halohydrin dehalogenase from Tistre lla mobilis,ZJB1405[J]. Journal of Molecular Catalysis B:Enzymatic,2015,115:105-112.
[20] TANG L,ZHU X,ZHENG H,et al. Key residues for controlling enantioselectivity of halohydrin dehalogenase from Arthrobacter sp. Strain AD2,revealed by structure-guided directed evolution[J].Applied and Environmental Microbiology,2012,78(8):2631-2637.
[21] TANG L,VAN MERODE A E J,LUTJE SPELBERG J H,et al.Steady -state kinetics and tryptophan fluorescence properties of halohydrin dehalogenase from Agrobacterium radiobacter. Roles of W139 and W249 in the active site and halide-induced conformational change[J]. Bioche mistry,2003,42(47):14057-14065.
[22] CHAO G,CHENY,ZHENG Y,et al. Explor ing the enantioselective mechanism of halohydrin dehalogenase from Agrobacterium radiobacter AD1 by iterative saturation mutagenesis[J]. Applied and Environmental Microbiology,2015,81(8):2919-2926.
[23] XUE F,LIU Z Q,WAN N W,et al. Purification,gene cloning,and characterization of a novel halohydrin dehalogenase from Agromyces mediolanus ZJB120203[J]. Applied Biochemistry and Biotechnology,2014,174(1):352-364.
[24] NELLAIAH H,MORISSEAU C,ARCHELAS A,et al.Enantioselective hydrolysis of p-nitrostyrene oxide by an epoxide hydrolase preparation from Aspergillus niger[J]. Biotechnology amp;Bioengineering,2015,49(1):70-77.
[25] KONG X D,MA Q,ZHOU J,et al. A smar t library of epoxide hydrolase variants and the top hits for synthesis of (S)-β-blocker precursors[J]. Angewandte Chemie,2014,53(26):6641-6644.
[26] XUE F,LIU Z Q,ZOU S P,et al. A novel enantioselective epoxide hydrolase from Agromyces mediolanus ZJB120203:cloning,characterization and application[J]. Process Biochemistry,2014,49(3):409-417.
[27] SPELBERG J H L,RINK R,ARCHELAS A,et al. Biocatalytic potential of the epoxide hydrolase from Agrobacterium radiobacter AD1 and a mutant with enhanced enantioselectivity [J]. Advanced Synthesis amp; Catalysis,2002,344(9):980-985.
[28] THOMAEUS A,NAWORYTA A,MOWBRAY S L,et al. Remova l of distal protein-water hydrogen bonds in a plant epoxide hydrolase increases catalytic turnover but decreases thermostability[J]. Protein Science,2010,17(7):1275-1284.
[29] WOO J H,HWANG Y O,KANG J H,et al. Enantioselective hydrolysis of racemic epichlorohydrin using an epoxide hydrolase from Novosphingobium aromaticivorans[J]. Journal of Bioscience And Bioengineering,2010,110(3):295-297.
[30] YILDIRIM D,TUKEL S S,ALPTEKIN O,et al. Immobi lized Aspergillus niger epoxide hydrolases:cost-effective biocatalysts for the prepation of enantiopure styrene oxide,propylene oxide and epichlorohydrin [J]. Journal of Molecular Catalysis B:Enzymatic,2013,88(2):84-90.
[31] XUE F,LIU Z Q,WAN N W,et al. Engineering the epoxide hydrolase from Agromyces mediolanus for enhanced enantioselectivity and activity in the kinetic resolution of racemic epichlorohydrin[J]. RSC Advances,2015,5(40):31525-31532.
Research and development on key enzymes for biosynthesis of chiral epichlorohydrin using glycerol
ZHANG Xiaojian1,2,ZHENG Yuguo1,2
1. College of Biotechnology and Bioengineering, Zhejiang University of Technology, Hangzhou 310014, China 2. Key Laboratory of Bioorganic Synthesis of Zhejiang Province, Hangzhou 310014, China
Chiral epichlorohydrin is an important three-carbon chiral building block for applications in pharmacy, pesticide,chemical, material and many other industries. It is of great value to develop green process for chiral epichlorohydrin synthesis from glycerol. Technologies of biocatalytic synthesis were applied to improve process safety, to reduce waste discharge,and to upgrade product quality. The biosynthesis chiral epichlorohydrin was developed, and the related technologies were described including design of synthetic route, construction of halogenase library, screening and modi fi cation of halohydrin dehalogenase and epoxide hydrolase, as well as production of chiral epichlorohydrin using halohydrin dehalogenase/epoxide hydrolase cascade biocatalysis. Our studies contribute to the exploration and development of chiral epichlorohydrin synthesis.
glycerol; chiral epichlorohydrin; 1,3-dichloro-2-propanol; biocatalysis; halohydrin dehalogenase; epoxide hydrolase
10.3969/j.issn.1674-0319.2017.06.009

郑裕国,教授,博士生导师。浙江工业大学生物工程学院院长、生物转化与生物净化教育部工程研究中心主任、浙江省生物有机合成技术研究重点实验室主任、浙江省生物催化与微生物发酵重点科技创新团队负责人等。长期从事生物化工研究和产业化技术开发,以第一完成人获国家技术发明二等奖2项、国家科技进步二等奖1项、省部级科学技术一等奖6项、中国专利优秀奖1项。发表SCI论文200余篇,授权发明专利90多件。主持开发的产业化技术建成工业化生产装置10余套,近3年新增销售50多亿元。先后获得全国优秀教师、全国优秀科技工作者、浙江省功勋教师等称号。E-mail:zhengyg@zjut.edu.cn

