454高通量技术比较两种不同臭豆腐卤水中微生物多样性
贺 静, 谢 靓, 曾玉伦, 陈淼芬, 蒋立文*
(1.湖南农业大学 食品科技学院,湖南 长沙 410128;2.食品科学与生物技术湖南省重点实验室,湖南 长沙 410128;3.兽用中药资源与中兽药创制国家地方联合工程研究中心,湖南 长沙 410128)
454高通量技术比较两种不同臭豆腐卤水中微生物多样性
贺 静1,2, 谢 靓1,2, 曾玉伦1,2, 陈淼芬3, 蒋立文1,2*
(1.湖南农业大学 食品科技学院,湖南 长沙 410128;2.食品科学与生物技术湖南省重点实验室,湖南 长沙 410128;3.兽用中药资源与中兽药创制国家地方联合工程研究中心,湖南 长沙 410128)
利用454高通量技术对两种不同臭豆腐卤水中微生物不同分类水平的多样性进行了评估和统计分析。经过与SILVA数据库对比,发现了7个门、10个纲、17个目、26个科、24个菌属。经分析发现两个样品的微生物不同分类学差异显著,主要集中在属水平上,分布都非常不均一。在样品A中厌氧球菌属和卟啉单胞菌属所占的比例最高,分别为40.22%、37.54%,成为优势菌属。两个样品之间相同的微生物属只有6个,比例差别极大。在样品B中氨基杆菌属的比例很大,为86.94%,占绝对优势。说明卤水原料来源不同对微生物的差异显著,同时发现臭豆腐卤水的发酵过程只与少数主要菌种有关。
臭豆腐卤水; 微生物; 高通量技术; 分析
臭豆腐是一种极具特色的臭味食品[1-2]。其特殊风味的形成,主要是豆腐在卤水制作过程中豆豉、冬笋、香菇等在自身内源酶和微生物分泌酶系作用下形成大量包括吲哚、苯酚类风味成分使得豆腐具有特殊的气味。由于其发酵周期长,不同品牌卤水差异较大,因此研究卤水制作过程中微生物多样性和群落构成具有重要意义,例如徐睿烜[3]、孙贵朋等[4]、何理等[5]利用传统方法从臭豆腐卤水中分离得到了优势微生物。但基于传统分离培养得到的微生物只能代表目前培养条件可能获得的微生物,无法预测所有微生物的信息。目前研究表明可培养微生物在整个微生物群落中不超过1%,所以结果具有一定的局限性[6]。相比之下,采用PCR-DGGE等分子生物学手段为研究微生物多样性和群落结果带来了革命性的突破[7-8]。454高通量测序技术是一种可以在一次测序试验中完成多个样本的检测,能获得海量序列且灵敏度高,不仅能检测出优势微生物,对于低丰度微生物也能检出,能全面、准确地揭示环境中微生物的多样性[9-10]。高通量测序技术已被广泛用于发酵食品中,如发酵蔬菜[11-12]、发酵乳[13-17]、酒精饮料[18]、发酵豆腐[19-20]等。454高通量测序技术研究避开了微生物分离培养的过程,扩展了微生物资源的利用空间,为微生物的研究提供了有效工具[21-22]。本研究通过454高通量测序技术对两种不同臭豆腐卤水中微生物信息进行比较,以探讨微生物与臭豆腐卤水风味成分关系,为全面掌握臭豆腐卤水中细菌的组成和改进臭豆腐卤水的制作工艺提供有效的参考。
1 材料与方法
1.1材料
1.1.1 样品来源 两份臭豆腐卤水样品A(CDH_1)、B(CDQ_2)来自湖南两个不同的臭豆腐生产企业。
1.1.2 试剂及仪器 电泳仪(DYY-12,北京六一仪器厂);凝胶成像仪(GDS-8000,美国UVP 公司); PCR仪(ABI GeneAmp®9700型);454 Titanium FLX+测序仪,瑞士Roche;QuantiFluorTM-ST蓝色荧光定量系统,Promega公司;TransStart Fastpfu DNA Polymerase,TransGen AP221-02;AxyPrepDNA 凝胶回收试剂盒,AXYGEN公司。
1.2方法
1.2.1 PCR扩增 采用破碎法提取两份卤水样品中基因组DNA[11],完成基因组DNA抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA并进行DNA扩增,454测序由上海美吉公司完成。DNA扩增采用细菌 V1-V3 区通用引物。具体PCR扩增引物,正向引物为27F: 5′-AGAGTTTGATCCTGGCT-CAG-3′;反向引物为533R: 5′-TTACCGCGGCTGCTG-GCAC-3′(表1)。PCR扩增程序:①95 ℃预变性2 min;②95 ℃变性30 s;③55 ℃退火30 s;④72 ℃延伸30 s;⑤重复②~④ 25次;⑤72 ℃延伸5 min。PCR 反应产物用 2%琼脂糖凝胶电泳检测合格后用于测序,焦磷酸测序按照 454 Roche GS-FLX 的标准流程进行。
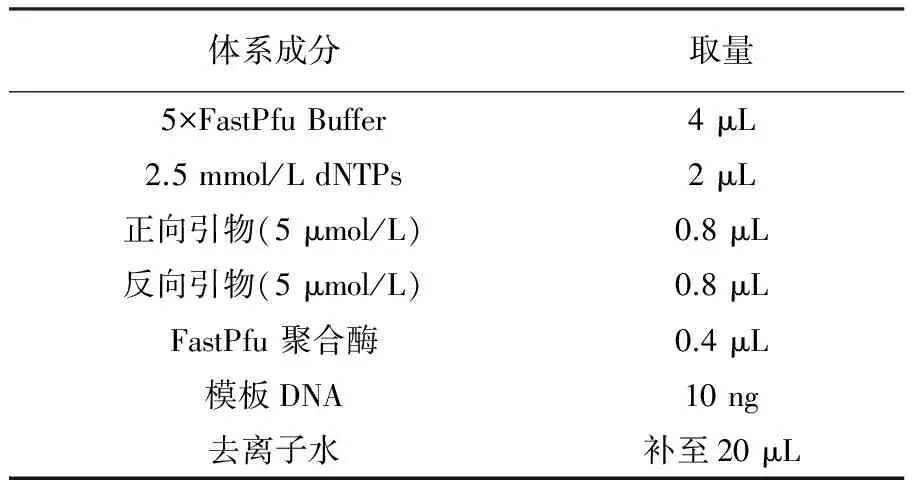
表1 PCR 反应体系(20 μL)
1.2.2 数据分析 对454序列处理及后续数据进行分析采用一定的控制标准。为了得到每个OTU对应的物种分类信息,采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并在各个水平(门、纲、目、科、属)统计每个样品的群落组成,采用SILVA的核糖体数据库进行对比。同时对OTU数据进行进一步的分析,利用Rarefaction曲线、Rank-Abundance曲线进行了多样性评估;利用Venn、PCA图进行统计分析。
2 结果与分析
2.1微生物在门的水平进行分类及比较
由图1和表2可知,从A(CDH_1)、B(CDQ_2)两个样品中共分离得到7个微生物菌门,其中都含有的有5个。在样品A中厚壁菌门、拟杆菌门、放线菌门为优势菌门,所占比例分别为49.25%、38.08%、11.07%,都超过10%;在样品B中互养菌门为优势菌门,所占比例为87.21%。
2.2微生物在纲的水平进行分类比较
由图2和表3可知,样品A中属于厚壁菌门的梭菌纲、属于拟杆菌门的拟杆菌纲、属于放线菌门放线菌纲也是高居前3位,所占比例分别为42.90%、38.07%、11.07%。样品B中属于互养菌门的互养菌纲所占比例达到87.21%,成为优势菌纲。其他微生物菌纲,如属于厚壁菌门芽胞杆菌菌纲、属于变形菌门的γ-变形菌纲和α-变形菌纲在两个样品中所占的比例都相对较低。同时发现属于拟杆菌门的黄杆菌纲只在样品A卤水中少量出现了,样品B卤水中没有出现。
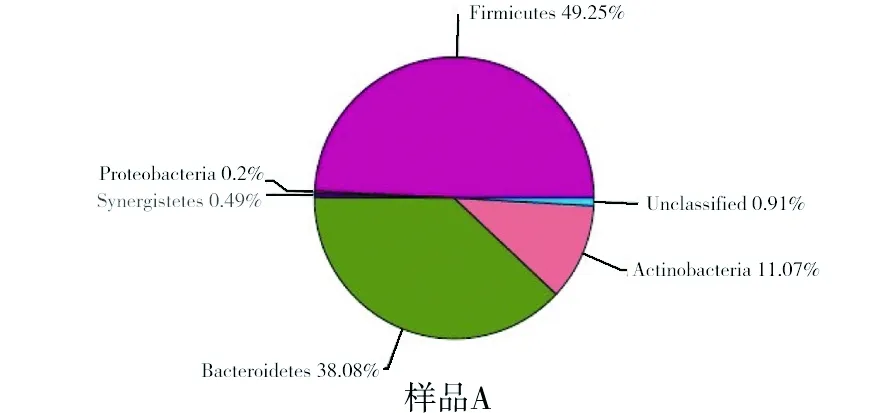
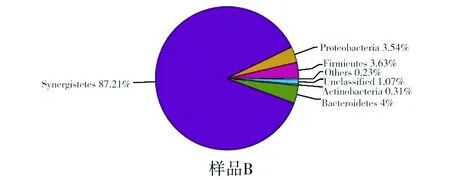
图1 不同卤水样品门分类Fig.1 Phyla classification of different brine
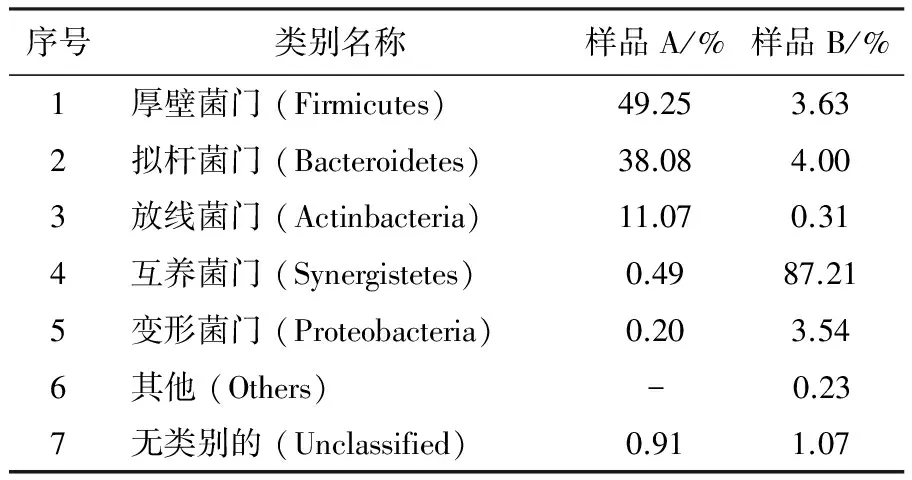
序号类别名称样品A/%样品B/%1厚壁菌门(Firmicutes)49.253.632拟杆菌门(Bacteroidetes)38.084.003放线菌门(Actinbacteria)11.070.314互养菌门(Synergistetes)0.4987.215变形菌门(Proteobacteria)0.203.546其他(Others)-0.237无类别的(Unclassified)0.911.07

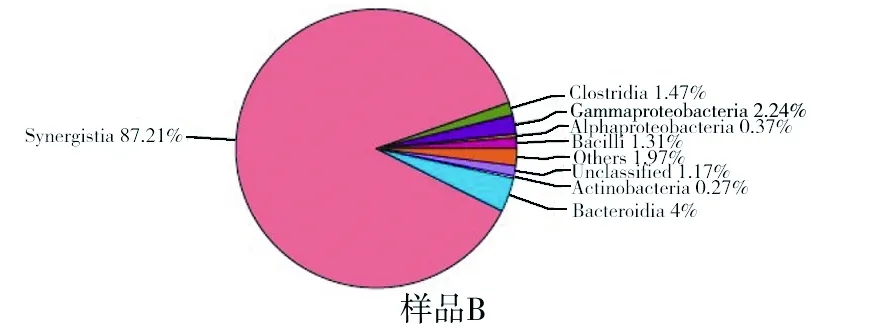
图2 不同卤水样品纲分类Fig.2 Class classification of different brine

序号类别名称样品A/%样品B/%1梭菌纲(Clostridia)42.901.472拟杆菌纲(Bacteroidia)38.074.003放线菌纲(Actinobacteria)11.070.274芽胞杆菌纲(Bacilli)6.241.315互养菌纲(Synergistia)0.4987.216γ⁃变形菌纲(Gammaproteobacteria)0.090.377α⁃变形菌纲(Alphaproteobacteria)0.052.248黄杆菌纲(Flavobacteria)0.01-9其他(Others)0.101.9710无类别的(Unclassified)0.991.17
2.3微生物在目的水平进行分类比较
由图3和表4可知,两个样品中共24个微生物菌纲,相同的只占9种,在样品A中分列前3位的是属于梭菌纲的梭菌目、属于拟杆菌纲的拟杆菌目、属于放线菌纲的放线菌目,所占比例分别为42.89%、38.07%、10.09%。在样品B中属于互养菌纲的互养菌目的比例为87.21%,所占比例最高。其他微生物菌目,如黄杆菌目、棒状杆菌目、盐厌氧菌目、海洋螺菌目都只在样品A卤水中出现,但是所占比例极少;芽胞杆菌目、鞘脂单胞菌目、微球菌目只在样品B卤水中出现,所占比例也是极少的。两个样品微生物菌群变化不明显。
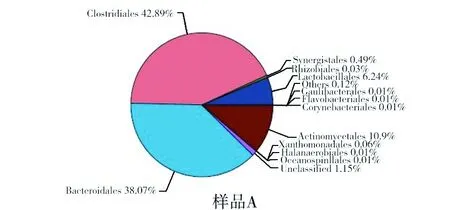
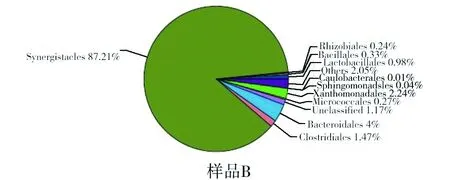
图3 不同卤水样品目分类Fig.3 Order classification of different brine
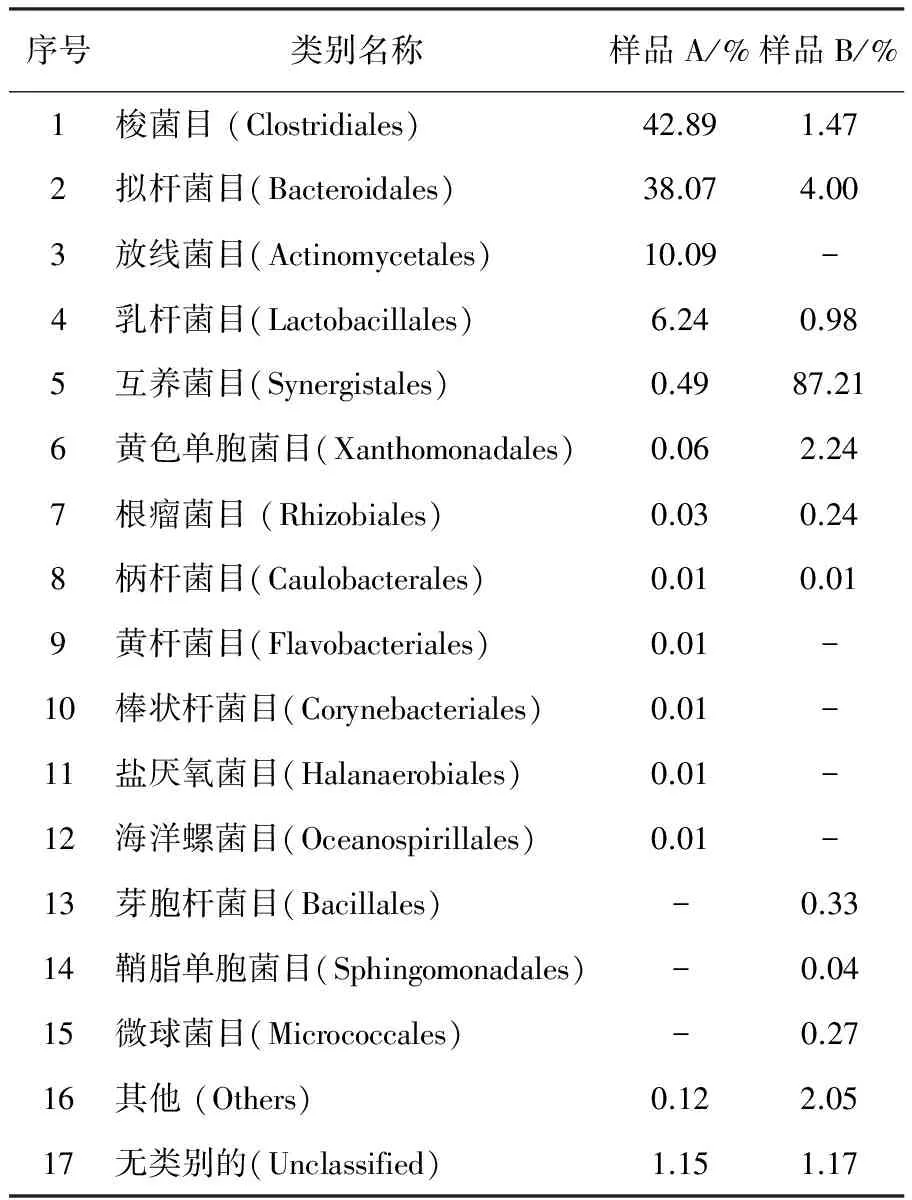
序号类别名称样品A/%样品B/%1梭菌目(Clostridiales)42.891.472拟杆菌目(Bacteroidales)38.074.003放线菌目(Actinomycetales)10.09-4乳杆菌目(Lactobacillales)6.240.985互养菌目(Synergistales)0.4987.216黄色单胞菌目(Xanthomonadales)0.062.247根瘤菌目(Rhizobiales)0.030.248柄杆菌目(Caulobacterales)0.010.019黄杆菌目(Flavobacteriales)0.01-10棒状杆菌目(Corynebacteriales)0.01-11盐厌氧菌目(Halanaerobiales)0.01-12海洋螺菌目(Oceanospirillales)0.01-13芽胞杆菌目(Bacillales)-0.3314鞘脂单胞菌目(Sphingomonadales)-0.0415微球菌目(Micrococcales)-0.2716其他(Others)0.122.0517无类别的(Unclassified)1.151.17
2.4微生物在科的水平进行分类比较
由图4和表5可以看出,两个样品中共26个微生物菌科。
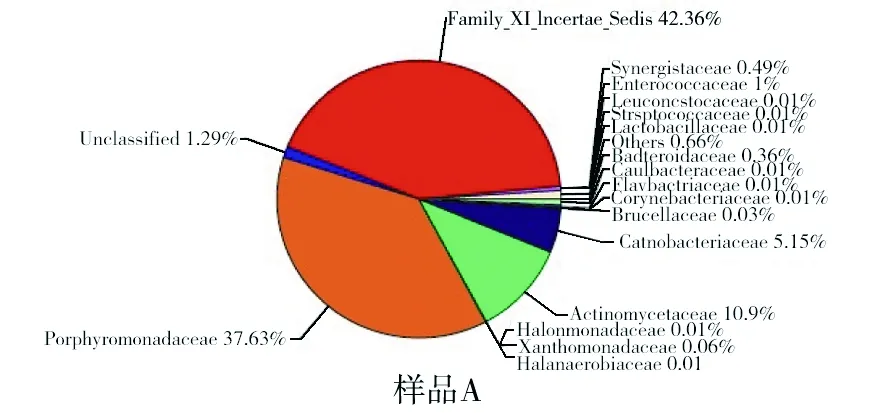
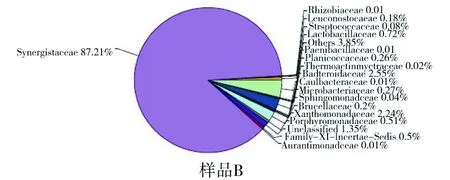
图4 不同卤水样品科分类Fig.4 Family classificationof different brine
样品A中有很大一部分是科未定的,其比例占到42.36%;其次是属于拟杆菌目的紫单胞菌科、属于放线菌目的放线菌科,所占比例分别为37.63%、10.09%。而在样品B中属于互养目的互养菌科所占比例为87.21%,优势菌群几乎没有变化。两个样品中出现了11个不同的菌科,根瘤菌科、类芽胞杆菌科、球菌科、高温放线菌科、微杆菌科、鞘脂单胞菌科、橙单胞菌科在样品A中都没有出现,在B卤水中没有发现黄杆菌科、棒状杆菌科、盐单胞菌科、盐厌氧菌科。总之,在科水平上,两个卤水样品的主要菌群变化不大,但是两个卤水的差异明显。
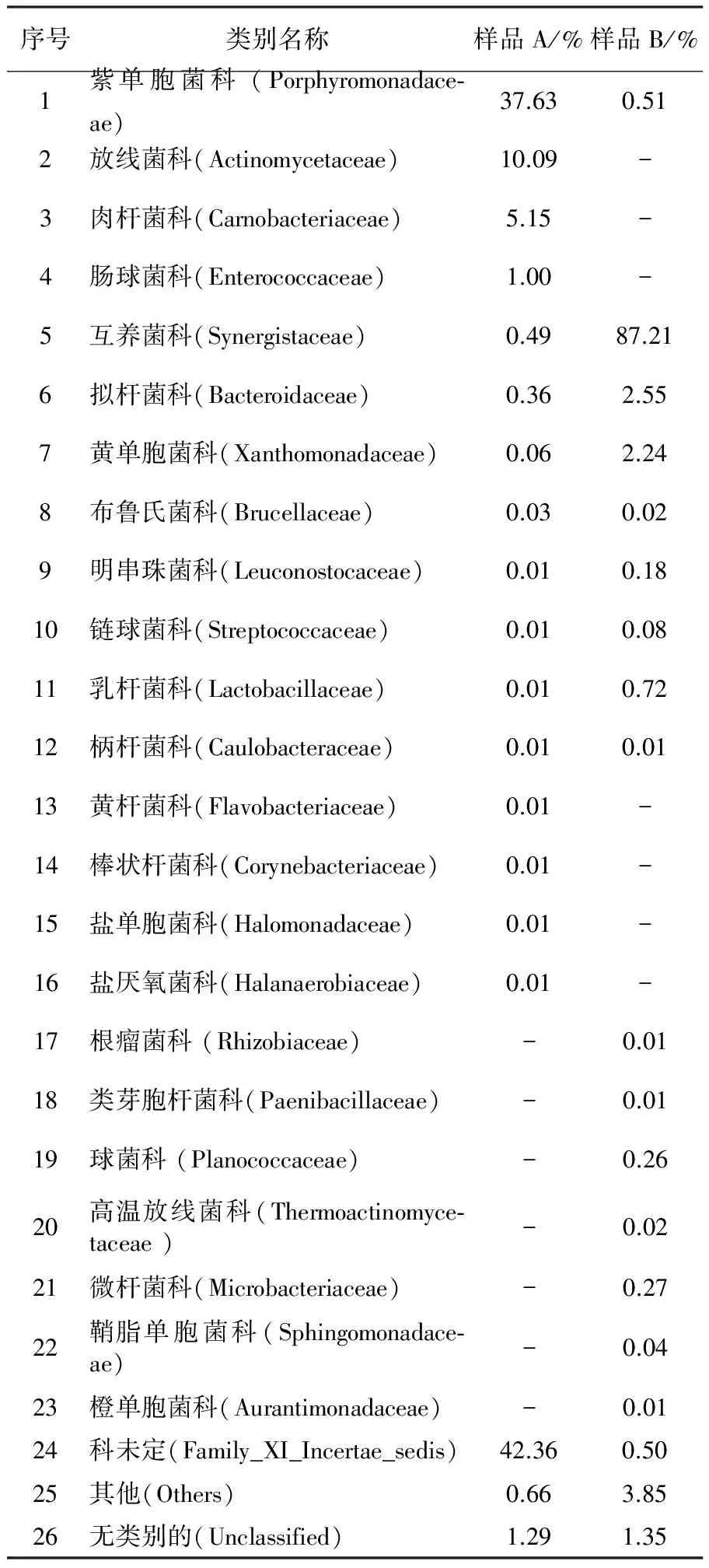
表5 不同卤水微生物科分类之间的差异
2.5微生物在属的水平进行分类比较
由图5和表6可知,厌氧球菌属和卟啉单胞菌属,在样品A卤水中为优势菌属,所占比例分别为40.22%、37.54%,这两种菌属在样品B卤水中完全没有出现;氨基杆菌属在样品B卤水中的含量达到了86.94%,占绝对优势,而在样品A卤水的比例为0.49%。两个样品在24个菌属中,仅有6个相同的菌属,且所占比例差异较大,所以两个样品差异显著。


图5 不同卤水样品属分类Fig.5 Genus classification of different brine

序号类别名称样品A/%样品B/%1厌氧球菌属(Anaerococcus)40.22-2卟啉单胞菌属(Porphyromonas)37.54-3放线杆菌属(Actinobaculum)6.99-4肉杆菌属(Atopostipes)5.05-5消化链球菌属(Peptoniphilus)2.060.446四体球菌属(Tetragenococcus)1.00-7氨基杆菌属(Aminobacterium)0.4986.948拟杆菌属(Bacteroides)0.362.559依格纳季氏菌属(Ignatzschineria)0.062.2410乳球菌属(Lactococcus)0.010.0411乳杆菌属(Lactobacillus)0.010.7212短波单胞菌属(Brevundimonas)0.01-13盐单胞菌属(Halomonas)0.01-14棒状杆菌属(Corynebacterium)0.01-15假单胞菌属(Kroppenstedtia)-0.0216微杆菌属(Microbacterium)-0.1217链球菌属(Streptococcus)-0.0118苍白杆菌属(Ochrobactrum)-0.0319根瘤菌属(Rhizobium)-0.0120芽胞杆菌属(Brevibacillus)-0.0121未分级的(Norank)0.010.0222无法培养(Uncultured)0.030.5123其他(Others)0.874.4324未分类的(Unclassified)5.291.92
经过上面不同类水平的分析并结合图6,还发现两个样品的微生物分布都高度集中在属的水平上,且主要微生物菌群的变化都不明显。
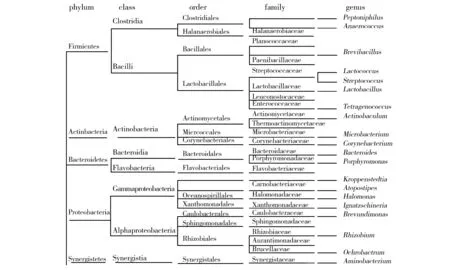
图6 两个样品中细菌组成图Fig.6 Compositions of bacteria in two samples
2.6Rarefaction曲线
Rarefaction曲线是从样本中抽取一定量的个体,统计这些个体代表的物种数目,并以个体数与物种数来构建曲线。图7横坐标表示的是OTU数目,纵坐标表示的是样本序列数,样品A和B的曲线趋于平坦,说明测序数据足以覆盖所有的微生物,表明了样品中微生物的多样性。对两个样品的丰度进行比较,可知样品B(CDQ_2)中微生物的多样性程度明显比样品A(CDH_1)要丰富。

图7 Number of Reads Sampled label:0.97曲线Fig.7 Number of Reads Sampled label:0.97 curve
2.7Rank-Abundance曲线
Rank-Abundance曲线是分析多样性的一种方式,可用来解释多样性的两个方面,即物种丰度和物种均匀度。图8中横坐标表示OTU等级,纵坐标对应的是OTU中序列数的相对百分含量。样品B(CDQ_2)的曲线比样品A(CDH_1)的曲线稍缓,说明样品B(CDQ_2)中的微生物分布更均一。
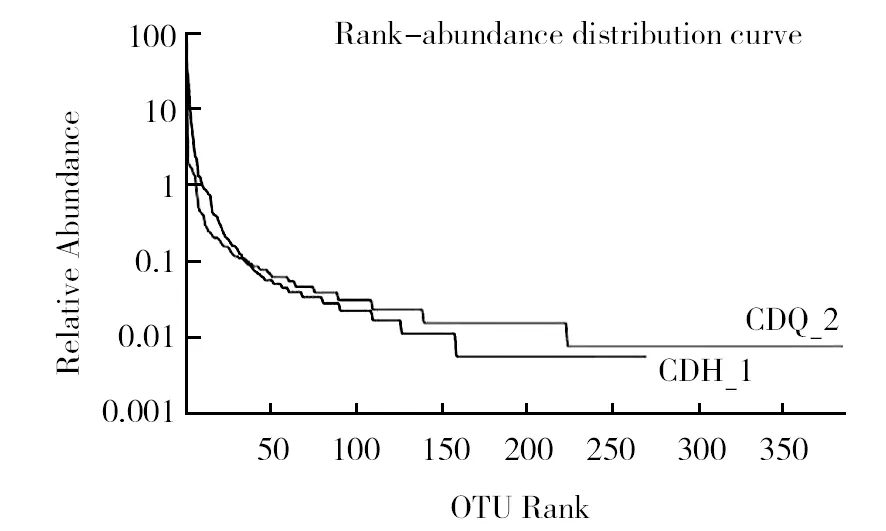
图8 相对丰度图Fig.8 Rank-Abundance curve
2.8OTU分布Venn图
经过454高通量测序,在97%相似度水平上划分 OTU,在样品A(CDH_1)和B(CDQ_2)分别发现243个和360个高质量的OTU。图9直观地统计了两个样品中所共有和独有的OTU数目及重叠情况。从图9中可分析出重叠部分OTU占总OTU的4%左右,说明两个样品间微生物的差异性显著。
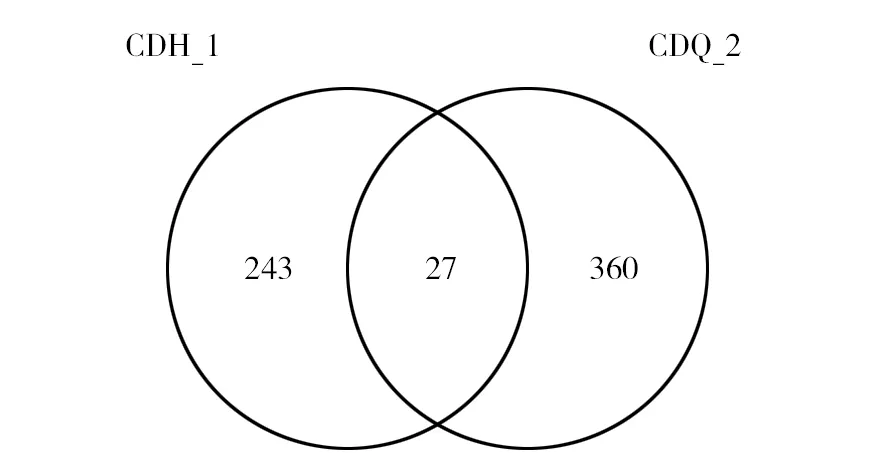
图9 维恩图Fig.9 Venn diagram
2.9PCA分析
图10中以样本A(CDH_1)和样本B(CDQ_2)中所有菌群做样品,进行PCA分析,主成分1(PC1)和主成分2(PC2)是造成两个样品的两个最大差异特征,贡献率分别为100% 和0%。 两个样品出现明显的分离状态且距离比较远,经分析可以推断两个样品的菌落结构差异比较大。这种现象说明,原料来源不同,微生物的情况差异显著。

图10 主成分分析图Fig.10 Principal Component Analysis
3 讨 论
本研究通过454高通量技术,对两个不同卤水中的微生物多样性和菌落结构进行了研究。Rarefaction曲线得出样品B中微生物多样性程度比样品A高;Rank-Abundance曲线中得出样品B曲线比样品A平缓。从微生物的门、纲、目、科、属的水平进行分类比较,测出了7个菌门、10个菌纲、17个菌目、26个菌科、24个菌属。同时通过OTU分布Venn图和PCA图分析也发现两个样品中微生物的相似度不高,差异显著。样品A和样品B中的微生物分布都非常不均一,都高度集中到属的水平上。例如,样品B中86.94%的细菌都是互养门、互养纲、互养目、互养科的氨基杆菌属。经过以上研究发现臭豆腐卤水的发酵过程只与少数主要菌种有关,为后续臭豆腐的工业化发展提供一定的依据。由于微生物发酵自然进行,样品没有办法重复,有一定的偶然性,同类别的发酵食品品质指标有差异,如风味物质、感官等实际上还是原料、微生物、工艺等差异性造成的,所以下一步有必要对臭豆腐卤水的原料在发酵过程中微生物群变化规律、生理生化反应、气味物质形成及变化进行研究,以期待为传统产业升级提供参考。
[1] 蒋立文,陈晨,吴跃飞, 等.臭味食品的种类及臭味食品的化学成分研究现状[J]. 中国酿造, 2015, 34(2):6-9.
[2] 虞镇溦,胡会萍,李里,等.浙江绍兴臭豆腐卤液中微生物菌群的分析[J]. 食品工业科技, 2012, 33(14):183-87.
[3] 徐睿烜. 长沙臭豆腐制作过程中主要指标的分析[D]. 长沙: 湖南农业大学, 2015.
[4] 孙贵朋,张雪娇,王妍,等.臭豆腐卤液中细菌多样性研究[J]. 现代食品科技, 2010, 26(10):1087-1091.
[5] 何理, 吴晖.市售臭豆腐中菌种的分离及生化鉴定[J]. 现代食品科技, 2012, 28(8):925-955.
[6] Amann R I,Ludwig W,Schleifer K H. Phylogenetic iden-tification and in situ detection of individual microbial cells without cultivation[J].Microbiological Reviews,1995,59(1):143-169.
[7] 李桥,王龙龙.454高通量测序技术在土壤微生物中的应用[J].绿色科技, 2013, (8): 203-205.
[8] Schuster S C. Next-genetation sequenceing transforms today’s biology[J].Nat Methods, 2008, 5(1): 16-18.
[9] 黄勇.基于高通量测序的微生物基因组学研究[D]. 北京:中国人民解放军军事医学科学院, 2013.
[10] 蔡元锋,贾仲君.基于新一代高通量测序的环境微生物转录组学研究进展[J]. 生物多样性, 2013, 21(4):401-410.
[11] 侯强川, 郭壮, 张家超, 等. 俄罗斯卡尔梅克共和国发酵蔬菜中细菌多样性研究[J]. 食品与发酵工业, 2014, 40(7): 16-22.
[12] Jung J Y,Lee S H,Kim J M,et al. Metagenomic analy-sis of kimchi,a traditional Korean fermented food[J].Applied and Environmental Microbiology,2011,77(7):2264-2274.
[13] 张和平.自然发酵乳制品中乳酸菌的生物多样性[J]. 生命科学, 2015, 27(7):837-846.
[14] Masoud W,Takamiya M,Vogensen F K,et al. Character-ization of bacterial populations in Danish raw milk cheesesmade with different starter cultures by denaturating gradientgel electrophoresis and pyrosequencing[J]. InternationalDairy Journal,2011,21(3):142-148.
[15] Quigley L,O′Sullivan O,Beresford T P,et al. High-throughput sequencing for detection of subpopulations ofbacteria not previously associated with artisanal cheeses[J]. Applied and Environmental Microbiology,2012,78(16):5717-5723.
[16] Dobson A,O′Sullivan O,Cotter P D,et al. High-through-put sequence-based analysis of the bacterial composition of kefir and an associated kefir grain[J]. FEMS Microbiology Letters,2011,320(1):56-62.
[17] Leite A M O,Mayo B,Rachid C,et al. Assessment of the microbial diversity of Brazilian kefir grains by PCR-DGGE and pyrosequencing analysis[J].Food Microbiology,2012,31(2):215-221.
[18] Jung M J,Nam Y D,Roh S W,et al. Unexpected con-vergence of fungal and bacterial communities during fer-mentation of traditional Korean alcoholic beverages inocu-lated with various natural starters[J].Food Microbiology,2012,30(1):112-123.
[19] 李里. 臭豆腐风味产生菌的分离、鉴定及风味产生机理初探[D]. 武汉:华中农业大学,2010.
[20] 金林毅,黄飞,田浩,等. 高通量测序法对7种发酵豆腐细菌组成的比较研究[J]. 食品卫生杂志,2015,27(2):114-119.
[21] 段曌,肖炜,王永霞,等.454 测序技术在微生物生态学研究中的应用[J]. 微生物学杂志,2011,31(5): 76-81.
[22] 周雪雁,刘翊中,陈轶霞, 等. 动物肠道菌群结构分析方法进展[J]. 微生物学杂志,2013,33(5): 81-85.
ComparisonofTwoDifferentStinkyTofuBrinesMicrobialDiversitywith454High-ThroughputTechniques
HE Jing1, 2, XIE Jing1, 2, ZENG Yu-lun1, 2, CHEN Miao-fen3, JIANG Li-wen1, 2
(1.Coll.ofFoodSci. &Tech.,HunanAgric.Uni.,Changsha410128; 2.HunanProv.KeyLab.ofFoodSci. &Biotech.,Changsha410128; 3.Nat′l&Prov.UnionEngin.Res.Ctr.fortheVet.HerbalMed.Res. &Initiative,Coll.ofFoodSci. &Tech.,Changsha410128)
454 high-throughput techniques were used to evaluate and statistically analyze the microbial diversity assessment in two different stinky tofu brines. It was found 7 phyla, 10 classes, 17 orders, 26 families and 24 genera after comparing with SILVA database. Larger differences of microbial species and concentrated at the genus level, and the distribution was very heterogeneous were found in these two samples. In the sample AAnaerococcusandPorphyromonasowe the highest proportion, 40.22%, 37.54% respectively. Only six of the same genus in the two samples, and the proportion of great difference was very heterogeneous were found. In the sample B,Aminobacteriumhave accounted for the largest proportion for 86.94%, become the absolute dominant bacteria. The experiment showed that the original material of the stinky tofu brine had a great influence on microbial differences. And found in stinky tofu brine during the fermentation only with a few major strains related.
stinky tofu brine; microbial; high-throughput technique; analysis
国家自然科学基金项目(31571819)
贺静 女,硕士研究生。研究方向为食品加工与安全。E-mail:1242447550@qq.com
* 通讯作者。男,教授,博士。研究方向为食品生物技术。E-mail:hnndjlw@163.com
2016-09-05;
2016-11-02
Q938;TS214.2
A
1005-7021(2017)04-0046-07
10.3969/j.issn.1005-7021.2017.04.008

