沙门菌tcpS基因的血清型分布及其在特定血清型鉴定中的应用
熊 丹,宋 丽,陶 静,郑慧娟,周子皓,潘志明,焦新安
DOI:10.3969/j.issn.1002-2694.2017.09.001
沙门菌tcpS基因的血清型分布及其在特定血清型鉴定中的应用
熊 丹,宋 丽,陶 静,郑慧娟,周子皓,潘志明,焦新安
目的建立基于tcpS基因的PCR鉴定方法,同时筛选肠炎沙门菌、鸡白痢/伤寒沙门菌和都柏林沙门菌,为流行病学调查提供方便快速的实验室技术支持,并为PCR鉴定沙门菌方法的发展提供新的基因候选。方法利用生物信息学的方法分析沙门菌tcpS基因的血清型分布特点,选择27种不同血清型沙门菌以及10株非沙门菌菌株,对所建立PCR体系的特异性和灵敏性进行检测,并将该方法应用于江苏省分离的48株猪场分离株、22株鸡场分离株和11株牛场分离株的检测。结果生物信息学分析表明tcpS基因仅存在于肠炎沙门菌、鸡白痢/伤寒沙门菌和都柏林沙门菌中,该PCR方法具有高度特异性和灵敏性,可准确快速地从临床样本中筛选这3种血清型沙门菌,且对实际样本检测符合率达100%。结论基于tcpS的PCR检测方法可准确筛选肠炎沙门菌、鸡白痢/伤寒沙门菌和都柏林沙门菌,能作为传统血清学分型的辅助方法,为沙门菌PCR鉴定方法的发展提供了优良的基因候选。
沙门菌;tcpS基因;PCR;血清型
沙门菌病是公共卫生学上有重要意义的人兽共患病之一,其病原沙门菌属于肠杆菌科,蛋、家畜和肉制品是主要传播媒介,它不但可引起多种畜禽疾病,造成全身性败血症和肠炎,还可以作为食源性疾病的病原体,引起人类胃肠炎和食物中毒[1-2]。在我国细菌性食物中毒中,约有70%~80%是由沙门菌引起的[3]。
根据Kauffman-White血清型分型方法,依据沙门菌菌体抗原及鞭毛抗原的不同,沙门菌目前的血清型有2 600种以上[4],中国已报道了292种不同的血清型,分属于35个O群[5]。大多数的动物感染是由几种主要的血清型沙门菌引起的,其中含有肠炎沙门菌、鸡白痢/伤寒沙门菌和都柏林沙门菌[6-8]。肠炎沙门菌是人感染食源性病原菌的主要致病菌,并且是农产品和感染动物中最主要的沙门菌血清型[9]。鸡白痢/伤寒沙门菌能特异性地感染家禽,可分别引起白痢和禽伤寒,且鸡伤寒垂直传播所带来的危害已引起广泛的重视[10]。都柏林沙门菌可增加奶牛的流产、发病率和致死率,并且降低牛奶的产量,引起奶牛业的重大经济损失,已引起世界范围内的高度关注[11-12]。因此,快速检测这三种重要的血清型沙门菌显得尤为重要。
传统的检测方法即非选择性和选择性增菌、生化特性及血清学鉴定,传统方法费力耗时,需4~7 d才能完成,其它如抗体检测法,虽然快速,但其高假阳性使之不适于常规检测。此外,低水平的病原菌污染,食品加工后导致沙门菌的“致伤”及食品其它成分的干扰等因素,使得沙门菌的检测受到一些限制。因此,急需一些快速、特异、灵敏的检测方法,以及时发现致病菌,控制污染及其可能对人体健康产生的危害[13]。分子生物学技术的发展使得许多食品微生物学者寻求方便的方法如PCR技术来检测病原菌,以期增加敏感性同时减少检测时间。
聚合酶链式反应(Polymerase chain reaction, PCR)是一种体外核酸扩增技术,具有敏感性强、特异性高、快速、简便等优点,在医学、生物学等领域中得到了广泛应用。由于该方法快速、特异、敏感等优点,使之在沙门菌的检测中已得到广泛的应用[14]。PCR方法尤其适用于大样本沙门菌的血清型快速判断,可大大缩短检测时间。如果样本不属于特定血清型沙门菌,也可根据PCR结果,选择性的使用多价抗血清进行进一步判断,可节约血清分型的时间以及血清的使用[15]。
沙门菌tcpS基因全长882 bp,目前关于其功能还未有研究报道,NCBI数据库注释其为一种假定蛋白,结构预测表明其4-36位氨基酸能够形成coiled-coil结构域。本研究通过生物信息学方法首次发现tcpS基因仅存在于肠炎、鸡白痢/伤寒和都柏林这3种血清型沙门菌中,以特异性引物通过PCR技术验证了tcpS基因在不同血清型沙门菌及其它细菌中的分布。该PCR体系能快速准确鉴定肠炎、鸡白痢/伤寒和都柏林血清型沙门菌,可作为沙门菌传统血清学分型的辅助方法,并为这3种特定血清型沙门菌的监测与实验室诊断提供了一种简单快速、重复性好的新方法。
1 材料和方法
1.1 材料
1.1.1 菌株 评价PCR方法特异性和灵敏性的沙门菌和非沙门菌菌株信息见表1。
1.1.2 主要试剂 沙门菌诊断血清购自宁波天润生物药业有限公司,Taq DNA 聚合酶、dNTPs、DL2000 DNA Marker购自大连宝生物工程有限公司,琼脂糖购自西班牙Biowest公司。
1.1.3 主要仪器 PCR扩增仪、电泳仪TY2795、凝胶成像系统GelDoc XR+购自Bio-RAD公司,离心机、biophotometer分光光度计购自德国Eppendorf公司。
1.2 方法
1.2.1 生物信息学分析tcpS基因的血清型分布 在NCBI中使用Blastn在线比对软件(http://blast.ncbi.nlm.nih.gov/Blast.cgi)在全基因组数据库中搜索tcpS基因序列(GenBank accession number KM408432),初步分析tcpS基因的血清型分布特点。
表1 评价建立的PCR方法特异性和灵敏性的沙门菌和非沙门菌菌株
Tab. 1 Salmonella and non-Salmonella strains used to evaluate the specificity and sensitivity of the developed PCR method
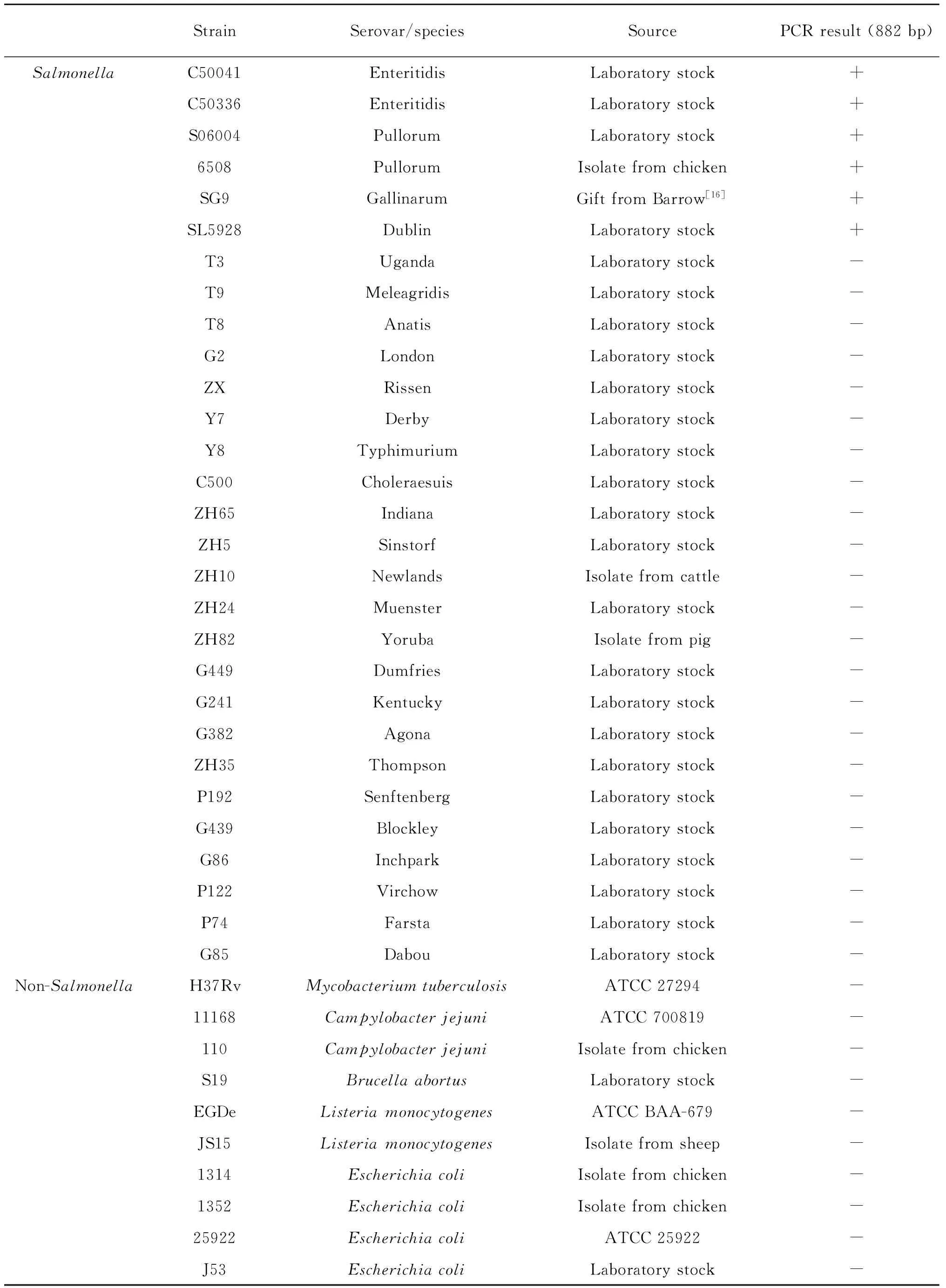
StrainSerovar/speciesSourcePCRresult(882bp)SalmonellaC50041EnteritidisLaboratorystock+C50336EnteritidisLaboratorystock+S06004PullorumLaboratorystock+6508PullorumIsolatefromchicken+SG9GallinarumGiftfromBarrow[16]+SL5928DublinLaboratorystock+T3UgandaLaboratorystock-T9MeleagridisLaboratorystock-T8AnatisLaboratorystock-G2LondonLaboratorystock-ZXRissenLaboratorystock-Y7DerbyLaboratorystock-Y8TyphimuriumLaboratorystock-C500CholeraesuisLaboratorystock-ZH65IndianaLaboratorystock-ZH5SinstorfLaboratorystock-ZH10NewlandsIsolatefromcattle-ZH24MuensterLaboratorystock-ZH82YorubaIsolatefrompig-G449DumfriesLaboratorystock-G241KentuckyLaboratorystock-G382AgonaLaboratorystock-ZH35ThompsonLaboratorystock-P192SenftenbergLaboratorystock-G439BlockleyLaboratorystock-G86InchparkLaboratorystock-P122VirchowLaboratorystock-P74FarstaLaboratorystock-G85DabouLaboratorystock-Non-SalmonellaH37RvMycobacteriumtuberculosisATCC27294-11168CampylobacterjejuniATCC700819-110CampylobacterjejuniIsolatefromchicken-S19BrucellaabortusLaboratorystock-EGDeListeriamonocytogenesATCCBAA-679-JS15ListeriamonocytogenesIsolatefromsheep-1314EscherichiacoliIsolatefromchicken-1352EscherichiacoliIsolatefromchicken-25922EscherichiacoliATCC25922-J53EscherichiacoliLaboratorystock-
1.2.2 引物设计与合成 以tcpS基因为模板,设计并分析引物,上游引物tcpS-F:ATGTCTATAAGCACCACAATG,下游引物tcpS-R:TCATTTCAATAATGATTCAAGC,引物由南京金斯瑞生物科技有限公司合成。
1.2.3 模板制备 取过夜培养的菌液,PBS洗3遍,采用煮沸法进行细菌DNA的提取,100°C,10 min,12 000 r/min离心5 min,取上清液作为DNA模板,4 ℃备用。
1.2.4 PCR反应 PCR反应体系为(25 μL):ddH2O 16.25 μL,dNTP 2 μL,10×PCR buffer 2.5 μL,tcpS-F 1 μL,tcpS-R 1 μL,模板2 μL,rTaq酶0.25 μL。PCR程序为94 ℃ 5 min;94 ℃ 45 s,59 ℃ 45 s,72 ℃ 1 min,30个循环;72 ℃ 10 min。PCR产物进行1%琼脂糖凝胶电泳。
1.2.5 特异性评价 用已建立的PCR方法分别对27种不同血清型的沙门菌和10株非沙门菌菌株进行检测(表1),验证该方法的特异性。
1.2.6 灵敏性评价 将沙门菌C50041菌株的基因组和菌液分别倍比稀释,用已建立的PCR方法分别对不同稀释度进行检测,以分析该方法的检测灵敏性。
1.2.7 临床样本检测 将所建立的PCR 方法应用于本实验室在江苏省某猪场、鸡场和牛场分离的待检沙门菌,与传统血清型鉴定方法进行比较。
2 结 果
2.1 生物信息学鉴定tcpS基因的分布 Blastn结果表明tcpS基因仅存在于肠炎沙门菌、鸡白痢/伤寒沙门菌和都柏林沙门菌中,且核苷酸序列相似性为100%,而沙门菌其它血清型和其它物种均没有这个基因。
2.2 PCR检测方法的特异性分析 PCR电泳结果表明仅在以肠炎、鸡白痢/伤寒和都柏林沙门菌基因组为模板的泳道中有目的条带(图1),胶回收后测序结果表明所有的tcpS基因的核苷酸序列相似性均为100%。
2.3 PCR检测方法的灵敏性分析 以不同浓度的肠炎沙门菌C50041基因组和菌液为模板分别进行PCR检测,电泳结果显示该PCR方法可最低检测到4.35 pg/μL的基因组DNA和10 CFU的沙门菌(图2)。
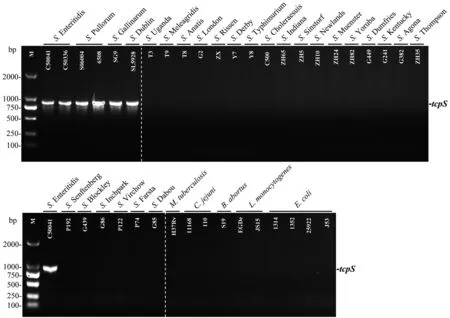
图1 PCR方法检测肠炎、鸡白痢/伤寒和都柏林沙门菌的特异性分析Fig.1 Specificity of the PCR method for detection of Salmonella serovars Enteritidis, Pullorum/Gallinarum, and Dublin
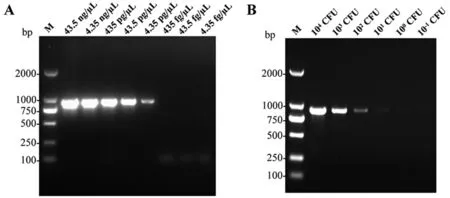
图2 PCR方法检测肠炎沙门菌C50041基因组DNA(A)和细菌数(B)的灵敏性分析Fig. 2 Sensitivity of the PCR method for detection of genomic DNA (A) and cells (B) from S. Enteritidis (strain C50041)
2.4 PCR检测临床样本 用建立的PCR方法对江苏省某猪场(48株)、鸡场(22株)和牛场(11株)分离的沙门菌进行检测,以验证所建立方法的准确性。PCR结果显示,48株猪场分离的沙门菌、22株鸡场分离的沙门菌和11株牛场分离的沙门菌完全符合其相应的传统血清学分型结果(表2)。
3 讨 论
血清型鉴定作为沙门菌分型的分类方法已有80多年历史,至今仍然是国际微生物检测机构认定的进行微生物学初筛的标准方法,对病原微生物研究的首要步骤也是血清学鉴定[17]。
表2 临床样本中传统血清学分型方法与建立的基于tcpS的PCR方法的比较
Tab. 2 Comparison between traditional serotyping method and the developed tcpS-based PCR method on clinical Salmonella isolates
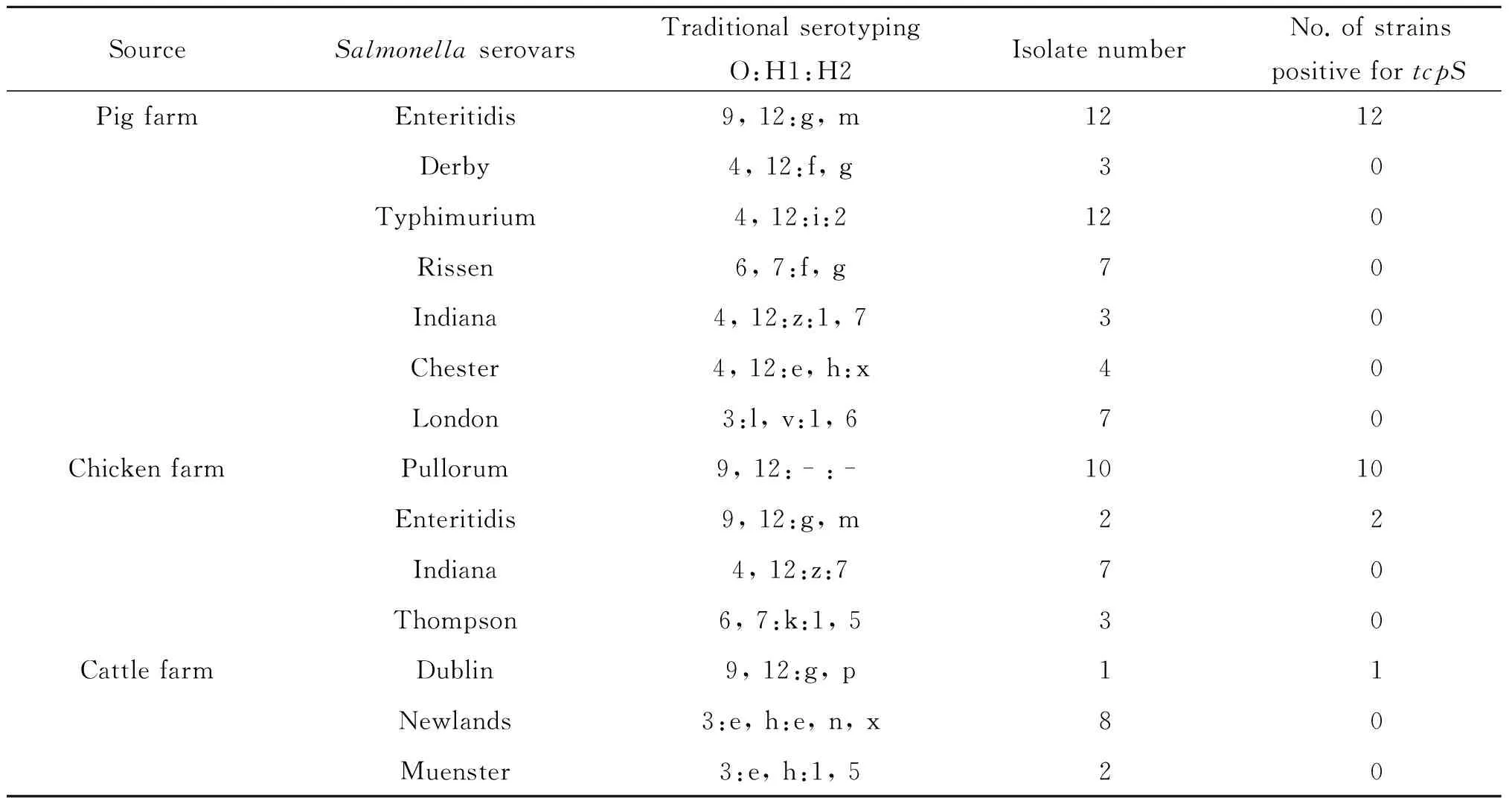
SourceSalmonellaserovarsTraditionalserotypingO:H1:H2IsolatenumberNo.ofstrainspositivefortcpSPigfarmEnteritidis9,12:g,m1212Derby4,12:f,g30Typhimurium4,12:i:2120Rissen6,7:f,g70Indiana4,12:z:1,730Chester4,12:e,h:x40London3:l,v:1,670ChickenfarmPullorum9,12:-:-1010Enteritidis9,12:g,m22Indiana4,12:z:770Thompson6,7:k:1,530CattlefarmDublin9,12:g,p11Newlands3:e,h:e,n,x80Muenster3:e,h:1,520
沙门菌可分为2 000多种血清型,鉴定时需质量保证且种类齐全的上百种血清,同时,也要求具备一定的经验及准确判断能力的实验者。传统血清型鉴定需购买特定的沙门菌血清型鉴定试剂盒,价格昂贵,步骤繁琐,有效期短,包含的血清型也不齐全,且货源紧缺,尤其在大样本中分离特定的血清型沙门菌(如肠炎沙门菌、鸡白痢/伤寒沙门菌或者都柏林沙门菌)时,需耗费数天时间,且工作量庞大,其结果是由肉眼进行判断,因而可能存在人为误差。目前,一些基于DNA和抗原的检测方法已经应用在沙门菌的检测上,如PCR和ELISA[18-19]。本文建立的基于tcpS基因的PCR检测方法操作简单,成本非常低,在3 h内即可完成对这3种血清型沙门菌的筛选,且准确率达到100%。
本研究所建立的PCR方法作为传统血清学方法的一种补充,可用于大样本中肠炎沙门菌、鸡白痢/伤寒沙门菌和都柏林沙门菌的快速判断,可大大缩短检测时间。如果样本不属于这3种血清型,也可根据PCR结果,选择性的使用多价抗血清进行进一步判断,可节约血清分型的时间以及血清的使用。本研究为PCR鉴定沙门菌方法的发展提供新的基因候选,然而,更多的血清型特异性基因仍需探索,这些基因和tcpS的联合使用将有助于沙门菌血清型鉴定方法的进一步发展。
本研究首次发现tcpS基因仅存在于肠炎、鸡白痢/伤寒和都柏林这3种血清型沙门菌中,以特异性引物通过PCR技术验证了tcpS基因在不同沙门菌血清型及其它细菌中的分布。基于tcpS的PCR方法能快速高通量筛选肠炎、鸡白痢/伤寒和都柏林血清型沙门菌,可作为沙门菌传统血清学分型的辅助方法,并为这3种特定血清型沙门菌的监测与实验室诊断提供了一种简单快速、重复性好的新方法。
[1] Nesbitt A, Ravel A, Murray R, et al. Integrated surveillance and potential sources ofSalmonellaenteritidisin human cases in Canada from 2003 to 2009[J]. Epidemiol Infect, 2012, 140(10): 1757-1772. DOI:10.1017/S0950268811002548
[2] Severi E, Booth L, Johnson S, et al. Large outbreak ofSalmonellaenteritidisPT8 in Portsmouth, UK, associated with a restaurant[J]. Epidemiol Infect, 2012, 140(10): 1748-1756. DOI: 10.1017/S0950268811002615
[3] Zhan L, Ye JL, Mei LL, et al. Development of a multiplex PCR assay for the identification ofSalmonellaentericaserovarenteritidis[J]. Chin J Zoonoses, 2012, 28(3): 244-247. (in Chinese)
占利, 叶菊莲, 梅玲玲, 等. 肠炎血清型沙门菌多重PCR检测方法的建立及应用[J]. 中国人兽共患病学报, 2012, 28(3): 244-247.
[4] Ranieri ML, Shi C, Moreno Switt AI, et al. Comparison of typing methods with a new procedure based on sequence characterization forSalmonellaserovar prediction[J]. J Clin Microbiol, 2013, 51(6): 1786-1797. DOI: 10.1128/JCM.03201-12
[5] Sun HL, Jiang Y, Li SC, et al. Analysis of serotype, biofilm-forming ability and antimicrobial resistance ofSalmonellastrains isolated from animals[J]. Chin J Anim Vet Sci, 2012, 43(10): 1630-1638. (in Chinese)
孙化露, 姜逸, 李树纯, 等. 动物源性沙门菌的血清型、生物被膜形成能力和耐药性分析[J]. 畜牧兽医学报, 2012, 43(10): 1630-1638.
[6] Saeki EK, Alves J, Bonfante RC, et al. Multiplex PCR (mPCR) for the detection ofSalmonellaspp. and the differentiation of the Typhimurium and Enteritidis serovars in chicken meat[J]. J Food Safety, 2013, 33(1): 25-29. DOI: 10.1111/jfs.12019
[7] Nielsen LR. Review of pathogenesis and diagnostic methods of immediate relevance for epidemiology and control ofSalmonellaDublin in cattle[J]. Vet Microbiol, 2013, 162(1): 1-9. DOI: 10.1016/j.vetmic.2012.08.003
[8] Zhu C, Yue M, Rankin S, et al. One-step identification of five prominent chickenSalmonellaserovars and biotypes[J]. J Clin Microbiol, 2015, 53(12): 3881-3883. DOI: 10.1128/JCM.01976-15
[9] Paião FG, Arisitides LG, Murate LS, et al. Detection ofSalmonellaspp.SalmonellaEnteritidis and Typhimurium in naturally infected broiler chickens by a multiplex PCR-based assay[J]. Braz J Microbiol, 2013, 44(1): 37-41. DOI: 10.1590/S1517-83822013005000002
[10] Soria MC, Soria MA, Bueno DJ. Comparison of 2 culture methods and PCR assays forSalmonelladetection in poultry feces[J]. Poult Sci, 2012, 91(3): 616-626. DOI: 10.3382/ps.2011-01831
[11] Carrique-Mas JJ, Willmington JA, Papadopoulou C, et al.Salmonellainfection in cattle in Great Britain, 2003 to 2008[J]. Vet Rec, 2010, 167(15): 560-565. DOI: 10.1136/vr.c4943
[12] Nielsen LR, Dohoo I. Time-to-event analysis of predictors for recovery fromSalmonellaDublin infection in Danish dairy herds between 2002 and 2012[J]. Prev Vet Med, 2013, 110(3-4): 370-378. DOI: 10.1016/j.prevetmed.2013.02.014
[13] Kumar A, Arora V, Bashamboo A, et al. Detection ofSalmonellatyphi by polymerase chain reaction: implications in diagnosis of typhoid fever[J]. Infect Genet Evol, 2002, 2(2): 107-110. DOI: 10.1016/S1567-1348(02)00090-4
[14] Qing LT, Qu XL. Application of nuclear acid probe and PCR techniques in food detection[J]. Prog Vet Med, 2000, 21(1): 22-24. DOI: 10.16437/j.cnki.1007-5038.2000.01.006. (in Chinese)
卿柳庭, 屈小玲. 核酸探针和PCR技术在食品检验中的应用[J]. 动物医学进展, 2000, 21(1): 22-24.
[15] Herrera-León S, Ramiro R, Arroyo M, et al. Blind comparison of traditional serotyping with three multiplex PCRs for the identification ofSalmonellaserotypes[J]. Res Microbiol, 2007, 158(2): 122-127. DOI: 10.1016/j.resmic.2006.09.009
[16] Wigley P, Hulme S, Powers C, et al. Oral infection with theSalmonellaentericaserovar Gallinarum 9R attenuated live vaccine as a model to characterise immunity to fowl typhoid in the chicken[J]. BMC Vet Res, 2005,1:2. DOI: 10.1186/1746-6148-1-2
[17] Wattiau P, Boland C, Bertrand S. Methodologies forSalmonellaentericasubsp.entericasubtyping: gold standards and alternatives[J]. Appl Environ Microbiol, 2011, 77(22): 7877-7885. DOI: 10.1128/AEM.05527-11
[18] Ricke SC, Khatiwara A, Kwon YM. Application of microarray analysis of foodborneSalmonellain poultry production: a review[J]. Poult Sci, 2013, 92(9): 2243-2250. DOI: 10.3382/ps.2012-02740
[19] Park SH, Aydin M, Khatiwara A, et al. Current and emerging technologies for rapid detection and characterization ofSalmonellain poultry and poultry products[J]. Food Microbiol, 2014, 38: 250-262. DOI: 10.1016/j.fm.2013.10.002
SerotypedistributionofSalmonellatcpSgeneanditsapplicationinidentificationofspecificserovars
XIONG Dan, SONG Li, TAO Jing, ZHENG Hui-juan, ZHOU Zi-hao, PAN Zhi-ming, JIAO Xin-an
(JiangsuKeyLaboratoryofZoonosis,JiangsuCo-innovationCenterforPreventionandControlofImportantAnimalInfectiousDiseasesandZoonoses,JointInternationalResearchLaboratoryofAgricultureandAgri-productSafetyoftheMinistryofEducation,KeyLaboratoryofPreventionandControlofBiologicalHazardFactors(AnimalOrigin)forAgrifoodSafetyandQuality,MinistryofAgricultureofChina,Yangzhou225009,China)
AtcpS-based PCR method was established to simultaneously screenSalmonellaentericaserovars Enteritidis, Pullorum/Gallinarum, and Dublin. The developed PCR method provides laboratorial support as a convenient and rapid approach for epidemiological investigation, andtcpScan be a potential candidate gene for the development of PCR-basedSalmonellaidentification. The serotype distribution ofSalmonellatcpSgene was analyzed by bioinformatic approach. The specificity and sensitivity of the PCR method were determined based on 27 differentSalmonellaserovars and 10 non-Salmonellastrains. The PCR method was applied to clinicalSalmonellaisolates from one pig farm (48 isolates), one chicken farm (22 isolates) and one cattle farm (11 isolates) from Jiangsu Province.Insilicoanalysis showed thattcpSexisted only inSalmonellaEnteritidis, Pullorum/Gallinarum, and Dublin. The developed PCR method had potent specificity and sensitivity, and could screen the three specificSalmonellaserovars accurately. The coincidence rate of the clinical sample detection was up to 100%. ThetcpS-based PCR detection method could screenSalmonellaEnteritidis, Pullorum/Gallinarum, and Dublin accurately, and could be an assistant method to the traditional serotyping method. Furthermore, the noveltcpSgene can be a potent gene candidate for the development of PCR method for the identification ofSalmonellaserovars.
Salmonella;tcpSgene; PCR; serotype
Pan Zhi-ming, Email: zmpan@yzu.edu.cn
国家重点研发计划课题(2016YFD0501607);公益性行业(农业)科研专项(201403054);国家自然科学基金(31320103907, 31230070);江苏省重点研发项目(BE2015343);江苏省第9批“六大人才高峰”高层次人才项目(NY-028);扬州大学科技创新团队;江苏省高校优势学科建设工程项目联合资助
潘志明,Email: zmpan@yzu.edu.cn
江苏省人兽共患病学重点实验室/江苏省动物重要疫病与人兽共患病防控协同创新中心/农业与农产品安全国际合作联合实验室/农业部农产品质量安全生物性危害因子(动物源)控制重点实验室,扬州 225009
R378.2
:A
:1002-2694(2017)09-0757-06
2017-03-29编辑:刘岱伟
Funded by the National Key Research and Development Program Special Project (No. 2016YFD0501607), the Special Fund for Agroscientific Research in the Public Interest (No. 201403054), the National Natural Science Foundation of China (Nos. 31320103907 and 31230070), the Research and Development Program of Jiangsu (No. BE2015343), the “Six Talent Peaks Program” of Jiangsu Province (No. NY-028), the Yangzhou University Science and Technology Innovation Team, and the Priority Academic Program Development of Jiangsu Higher Education Institutions (PAPD)

