诺如病毒新重组株GII.P16/ GII.2引起福建省2016年冬季病毒性胃肠炎暴发
吴冰珊,黄枝妙,欧剑鸣,齐孝旗,黄业伟,翁育伟
DOI:10.3969/j.issn.1002-2694.2017.09.010
诺如病毒新重组株GII.P16/ GII.2引起福建省2016年冬季病毒性胃肠炎暴发
吴冰珊1,2,黄枝妙1,欧剑鸣1,齐孝旗1,黄业伟3,翁育伟1,2
目的本文旨在鉴定福建省2016年冬季导致病毒性胃肠炎暴发的病原体,并对病原体进行分子特征研究。方法对疫情暴发地上送的福建省2016年胃肠炎暴发急性病例标本,采用荧光PCR初步判定病原体,常规RT-PCR检测诺如病毒RNA聚合酶和衣壳蛋白基因片段,并进行序列测定和分子特征分析。结果在3起暴发疫情中18份标本经荧光PCR检测均为诺如病毒核酸阳性,其中15份标本RT-PCR检测判为GII型诺如病毒,9份标本成功测序。分析测序结果,证实引起本轮疫情为诺如病毒新重组株,该重组株有别于本地散发流行及全球暴发流行的优势基因型GII.4。毒株RNA聚合酶核苷酸序列与2016年日本的GII.4悉尼变异株Kawasaki194毒株的同源性最高,达98%,为GII.P16亚型;衣壳蛋白核苷酸序列与2008 年比利时的IPH2161-08VG06毒株同源性最高(97.7%~98.8%),为GII.2亚型。结论这是福建省首次报道诺如病毒重组株GII.P16/ GII.2的检出,并引起病毒性胃肠炎暴发。
病毒性胃肠炎;暴发;诺如病毒;GII.P16/ GII.2;分子特征
Supported by the National High Technology Research and Development project (No. 2011AA02A114) Corresponding author: Wen Yu-wei, Email: wengywfjcdc@aliyun.com
诺如病毒 (Norovirus, NV)自1968年被发现以来,已成为引起人类急性非细菌性胃肠炎流行和暴发的主要病原,容易引起学校、养老院、医院及邮轮等环境的疫情集中暴发,病毒感染力强,主要是通过污染的水、食物和环境直接或间接造成人传人感染。福建省地处我国东南沿海,横跨中亚热带和南亚热带两个自然地理带,属亚热带湿润季风气候,全省共有9个设区市。2006年我省首次出现诺如病毒暴发[1],之后全省各地发生多次暴发,引起业内和社会的关注。2016年12月厦门、永泰及漳州三所学校相继发生疑似病毒引起的胃肠炎暴发事件,综合流行病学调查、临床表现及实验室检测结果,证实这3起事件为诺如病毒重组株引起的感染事件。本文拟通过对阳性标本进行分析,并与本省散发流行的诺如病毒地方株及相关序列进行比对,以探讨该重组株的分子特征。
1 材料和方法
1.1 材料 2016年冬季福建省多地市相继发生多起疑似病毒性腹泻暴发疫情,其中3起疫情暴发地所在市疾控中心共送检18份标本,包括漳州某幼儿园学生病例2份肛拭子、1份呕吐物、1份粪便;永泰某小学5份发病学生肛拭子、1份粪便;厦门某幼儿园8份发病学生肛拭子。送检标本均以生理盐水处理成10%悬液,高速离心后取上清液检测。
1.2 暴发定义 2015年中国疾病预防控制中心下发的《诺如病毒感染暴发调查和预防控制技术指南》规定,病毒性腹泻暴发是指7 d内同一学校、托幼机构、医疗机构、养老院、工厂、建筑工地、游轮、社区/村庄等集体单位或场所,发生20例及以上有流行病学关联的诺如病毒感染病例,其中至少2例是实验室诊断病例。
1.3 主要试剂 病毒核酸提取试剂盒购自天隆公司,诺如病毒核酸荧光检测试剂盒购自上海之江和江苏硕世公司,胶回收纯化试剂盒购自QIAGEN公司,逆转录酶购自Invitrogen公司,Taq DNA聚合酶购自Takara公司,引物序列由铂尚公司合成,其他试剂均为分析纯试剂。
1.4 实验室方法
1.4.1 病毒RNA提取 取离心后的悬液上清200 μL,按照天隆公司的病毒核酸提取试剂盒说明书操作,NP968-S全自动病毒核酸提取仪提取病毒核酸,吸取核酸置于1.5 mL的离心管,-20 ℃冻存备用。
1.4.2 诺如病毒的检测 按照商品试剂盒说明书采用荧光RT-PCR检测诺如病毒核酸。采用逆转录、多重PCR扩增杯状病毒RNA多聚酶(RdRp)/衣壳蛋白基因片段,引物组合可同时鉴定诺如病毒(I型和II型)和扎如病毒[2],目的片段分别为330 bp、380 bp和435 bp,扩增产物经琼脂糖凝胶电泳鉴定。
1.4.3 基因片段序列测定 为对诺如病毒进行基因亚型鉴定,采用JV12[3]/G2SKR引物对扩增诺如病毒多聚酶/衣壳蛋白基因片段,产物片段为1 110 bp。扩增产物经琼脂糖凝胶电泳鉴定,阳性产物胶回收纯化后送铂尚公司测序。
1.4.4 序列分析 采用DNAstar软件对扩增产物核苷酸测序结果进行编辑、推导相应的氨基酸序列,选择本省散发流行地方株及GenBank中收录的参考毒株序列进行分析,分别针对诺如病毒RNA聚合酶3′端780 bp和衣壳蛋白5′端250 bp核苷酸序列,采用Mega 6.06版本构建进化树,选择邻位相连法(Neighbor joining,NJ),bootstrap值设置为1 000次,核酸替代模型Kimura 2-parameter+G,比较序列同源性,分析毒株系统进化特征。
1.4.5 重组分析 采用Simplot软件分析毒株重组位点,采用200 bp的滑动窗口, 20 bp的滑动步伐。
2 结 果
2.1 流行概况 2016年12月福建省厦门市某幼儿园、漳州市某幼儿园、永泰县某小学分别发 生病毒胃肠炎暴发,发病例数共计53例(厦门11例、漳州21例、永泰21例),病例年龄组较小(4~6岁)。发病时间均在48 h内,临床表现以呕吐为主及不同程度的腹泻和腹痛。这3起暴发疫情均发生于学校,传播方式为人传人,发病人群年龄距离较小,症状较轻以呕吐为主,无重症和死亡病例。由于疫情早期迅速采取干预措施,暴发局限于小范围的人群,未造成大面积扩散。综合流行病学调查和实验室检测结果认为这3起疫情均排除食源性和水源性传播的可能性,判定为诺如病毒感染引起的人传人聚集性/暴发事件。
2.2 诺如病毒检测 送检的18份标本经荧光PCR检测均含GII型诺如病毒核酸;采用杯状病毒特异分型引物进行扩增,产物经琼脂糖凝胶电泳分析,15份标本在380 bp处有特异条带,判定为诺如病毒GII型(表1)。
2.3 序列测定 对阳性标本核苷酸进行测序,有9份成功测序, NCBI在线提交测序结果进行同源性比对搜索,结果表明为诺如病毒核苷酸序列片段。
2.4 序列分析结果 在线软件(http://www.rivm.nl/mpf/norovirus/typingtool)证实[4]本轮暴发毒株为诺如病毒重组株GII.P16/GII.2,毒株间序列高度同源,相似性介于96.9%~100%暴发毒株的RNA聚合酶核苷酸序列与2016年日本的GII.4悉尼变异株Kawasaki194毒株的同源性最高,达98%,属GII.P16;而衣壳蛋白与2008 年比利时IPH2161-08VG06毒株同源性最高(97.7%-98.8%),属GII.2。比对分析本轮暴发毒株与我省散发流行毒株,我省流行的诺如病毒以GII型为主(图1-2)。2016年哨点监测结果表明福建省诺如病毒散发毒株主要为GII.4、GII.6和 GII.3,仍以2012 Sydney变异株GII.Pe/GII.4为优势流行型别,并检测到GII.17型,未检测到GII.2型。采用Simplot软件分析毒株重组位点,位于ORF1/ORF2交界处735-737bp处(图3)。
表1 暴发性胃肠炎标本诺如病毒检测结果
Tab.1 Detection results of norovirus collected from viral gastroenteritis outbreaks

来源source送检数量number送检发生时间dateofinspection荧光PCR检测结果resultsofrealtimeRT-PCRRT-PCR检测结果resultsofRT-PCR厦门Xiamen82017-1-68/8NVGII(+)7/8NVGII(+)漳州Zhangzhou42016-12-274/4NVGII(+)4/4NVGII(+)永泰Yongtai62016-12-306/6NVGII(+)4/6NVGII(+)
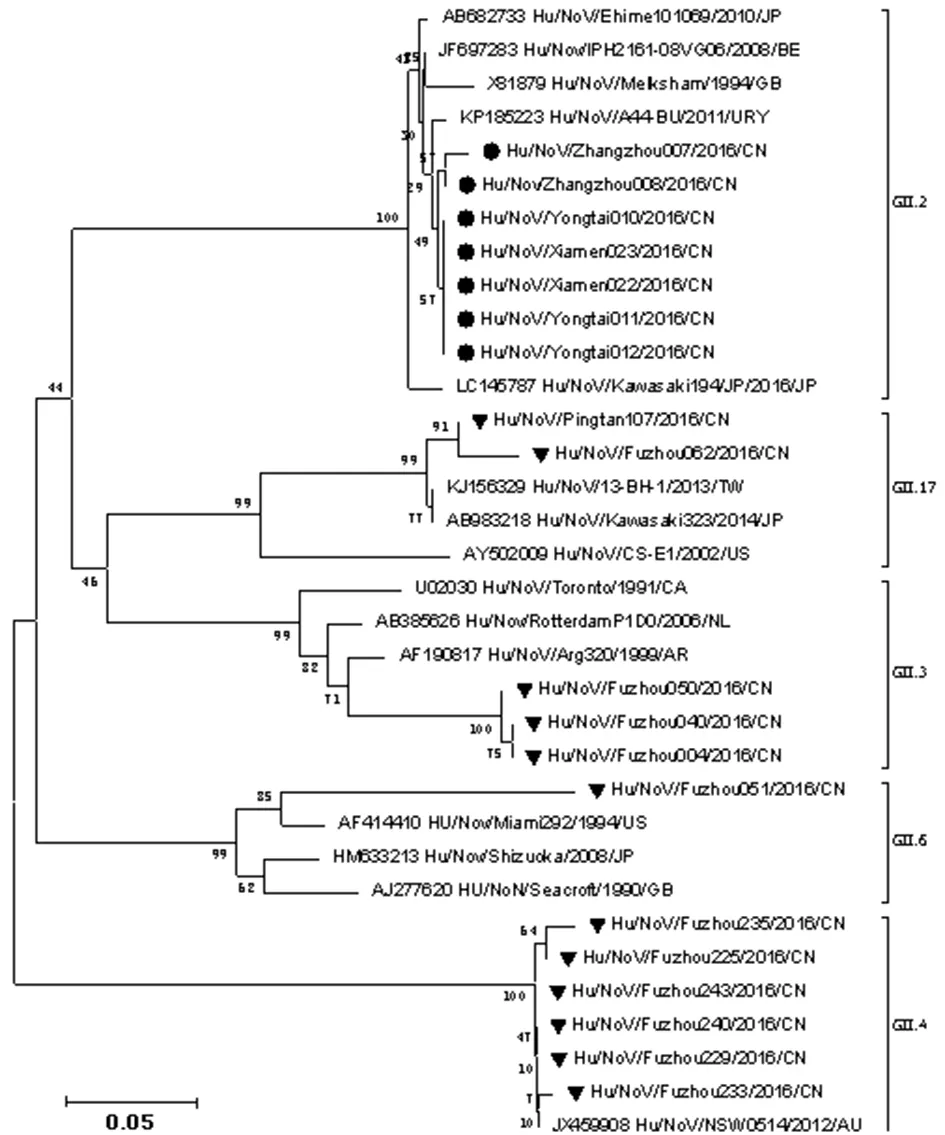
●Outbreak strains of norovirus in 2016;▼ Sporadic strains of norovirus in 2016图1 诺如病毒衣壳蛋白核苷酸片段进化分析(250 bp)Fig.1 Phylogenetic analyses based on partial VP1 genes of norovirus (250 bp)

●Outbreak strains of norovirus in 2016;▼ Sporadic strains of norovirus in 2016图2 诺如病毒RNA聚合酶核苷酸片段进化分析(780 bp)Fig.2 Phylogenetic analyses based on partial RNA polymerase genes of norovirus (780 bp)
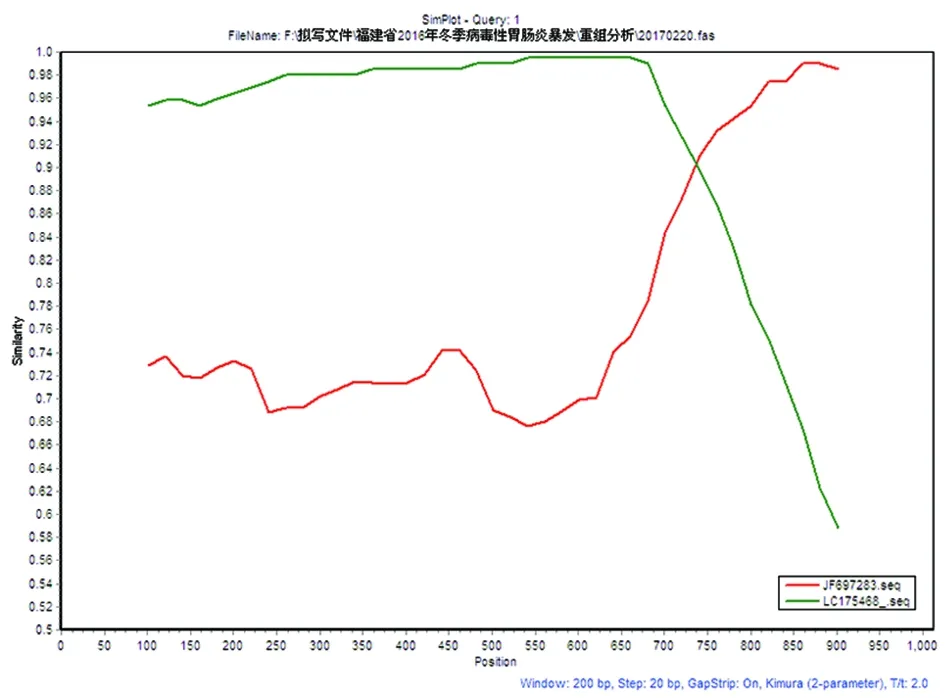
图3 Simplot 分析2016年诺如病毒GII.P16/GII.2重组株Fig.3 Simplot analyses of the NoV recombinant GII.P16/GII.2 detected in 2016
3 讨 论
根据衣壳蛋白区序列差异,诺如病毒分为 7个基因组(GI ~ GVII),各基因组可细分为不同的基因型[5],造成人类感染的主要是基因组I和II (GI和GII),全球约50%急性胃肠炎暴发是由感染诺如病毒引起的[6]。诺如病毒变异率极高,每隔2~3年出现新的变异/重组株,人群普遍缺少对新变异株的免疫能力,导致新变异株极易引起急性胃肠炎暴发。既往福建省从未在散发或暴发病例中检测到诺如病毒GII.P16/GII.2,这是福建省首次检测到诺如病毒GII.P16/GII.2,也是首次证实由GII.P16/GII.2引起的病毒性腹泻暴发疫情。在这次暴发中,疫情均发生在学校,传播方式为人传人,易感人群年龄距离较小,症状较轻以呕吐为主,无重症和死亡病例。由于及时采取控制措施,未造成大范围的扩散,认为疫情流行特征和GII.4引起的暴发相类似,提示这两个型别的病毒感染特征基本一致,诺如病毒GII.P16/GII.2具有在局部人群的传播流行能力。
与流感病毒相类似,不同基因亚型的诺如病毒可同时感染同一个宿主;另一方面诺如病毒复制过程中存在亚基因组的产生,因此不同型别的病毒容易在ORF1/ORF2 重叠交界区发生型间或型内重组,自然环境下可检测到不同G/P组合的毒株,重组株的产生是诺如病毒进化的主要机制之一[7-8]。诺如病毒GII. P16在自然流行过程不断发生进化,近年来重组株不断被发现。目前已检测到的组合包括GIIP.16/GII.16.和GII.P16/GII.2、GII.P16/GII.3、GII.P16/GII.13、及新近发现的GII.P16/GII.4重组株。2011年孟加拉在一名婴儿检出GII.P16/ GII.3[9],2010-2011年GII.P16/GII.3在巴西造成4起暴发[10],随后西班牙也报道了GII.P16/GII.3的检出[11],2010年欧洲首先在意大利发现GII.P16/GII.13[12]。2016年日本在1起暴发中首次检出9株GII.P16/GII.4_Sydney2012新重组株[13],并对基因组进行全长测序。GII.P16/GII.4毒株的出现改变GII.Pe/GII.4_Sydney2012在日本的流行状况。
2009-2010年诺如病毒引起日本胃肠炎暴发,其中 GII.2亚型占44.6%,首次报道GII.P16/GII.2引起的暴发[14]。引起福建省本轮暴发的诺如病毒GII.P16/GII.2的RdRp序列与新重组株Kawasaki194毒株(GII.P16/GII.4)的同源性最高,提示诺如病毒GII.P16型RdRp节段已进化能结合更多型别的衣壳蛋白。根据目前文献报道,造成全球范围病毒性腹泻暴发的主要型别为GII.4,并非GII.P16/GII.2诺如病毒。GII.P16/GII.2引起散发病毒胃肠炎感染所占比例也甚低,世界各地对该型诺如病毒认识有限,尚未彻底掌握病毒传染性、致病力及致病机制等病原体特征。GenBank中收录的GII.P16/GII.2参考毒株序列不够多,不足以从系统发生树分析的角度对新出现的变异株的来源进行更为细致的溯源分析。本实验室监测资料结果显示,截至2016年尚未在福州地区5岁以下散发腹泻儿童检出GII.P16/GII.2诺如病毒。由于病毒性腹泻日常监测地理范围仅局限于福州地区,本次暴发诺如病毒GII.P16/GII.2的地理范围超出监测区,不排除福州以外的人群存在GII.P16/GII.2散发感染。
2014年12月长沙市某厂区暴发了一起由新型诺如病毒GII.P17/GII.17引起的急性胃肠炎疫情[15],本研究也提示福建省内不仅存在多种型别的诺如病毒同时流行,并且新型诺如病毒不断出现引起暴发,因此在监测工作中应同时开展诺如病毒的RdRp和衣壳蛋白检测,以期及时发现新的毒株。为明确诺如病毒GII.P16/GII.2在本省的流行消长情况,长期监测、扩大监测对象包括地区与全人群监测是必须的。
[1] Wu BS, Xie JF, Shen XN, et al. Epidemiological research of noroviruses causing Viral gastroenteritis outbreaks in Fujian[J]. Chin J Epidemiol, 2008, 29(4): 382. (in Chinese)
吴冰珊,谢剑锋,沈晓娜.福建省诺如病毒腹泻暴发的流行病学研究. [J].中华流行病学杂志,2008,29(4):382.
[2] Honma S, Nakata S, Kinoshita-Numata K, et al. Evaluation of nine sets of PCR primers in the RNA dependent RNA polymerase region for detection and differentiation of members of the family Caliciviridae, Norwalk virus and Sapporo virus[J]. Microbiol Immunol, 2000, 44: 411-419.
[3] Vinjé J,Vennema H,Maunula L,et al. International collaborative study to compare reverse transcriptase PCR assays for detection and genotyping of noroviruses[J]. J Clin Microbiol,2003, 41(4): 1423-1433.
[4] Kroneman A, Vennema H, Deforche K, et al. An automated genotyping tool for enteroviruses and noroviruses[J]. J Clin Virol, 2011, 51(2):121-125. DOI: 10.1016/j.jcv.2011.03.006
[5] Vinjé J.Advances in laboratory methods for detection and typing of norovirus[J]. J Clin Microbiol,2015, 53: 373-381. DOI: 10.1128/JCM.01535-14
[6] Zheng DP, Widdowson MR. Molecular epidemiology of genogroup II-genotype 4 noroviruses in the United States between 1994 and 2006[J]. J Clinical Microbiol, 2010, 48(1): 168-177.
[7] Bull RA, Hansman GS, Clancy LE, et al. Norovirus recombination in ORF1/ORF2 overlap[J]. Emerg Infect Dis, 2005, 11(7): 1079-1085.
[8] Eden JS, Tanaka MM, Boni MF, et al. Recombination within the pandemic norovirus GII.4 lineage[J]. J Virol, 2013, 87(11): 6270-6282. DOI: 10.1128/JVI.03464-12
[9] Nahar S,Afrad MH,Matthijnssens J, et al. Novel intergenotype human norovirus recombinant GII.16/GII.3 in Bangladesh[J]. Infect Genet Evol,2013, 20: 325-329. DOI: 10.1016/j.meegid.2013.09.021
[10] Fumian TM,da Silva Ribeiro de Andrade J,Leite JP, et al. Norovirus recombinant strains isolated from gastroenteritis outbreaks in Southern Brazil, 2004-2011[J]. PLoS One,2016, 11(4): e0145391. DOI: 10.1371/journal.pone.0145391
[11] Arana A,Cilla G,Montes M,et al. Genotypes, recombinant forms, and variants of norovirus GII.4 in Gipuzkoa (Basque Country, Spain), 2009-2012[J]. PLoS One,2014, 9(6): e98875. DOI: 10.1371/journal.pone.0098875
[12] Medici MC,Tummolo F,Martella V, et al. Novel recombinant GII.P16_GII.13 and GII.P16_GII.3 norovirus strains in Italy[J]. Virus Res,2014, 88: 14214-14215. DOI: 10.1016/j.virusres.2014.04.005
[13] Matsushima Y,Shimizu T,Ishikawa M, et al. Complete genome sequence of a recombinant GII.P16-GII.4 norovirus detected in Kawasaki City,Japan, in 2016[J]. Genome Announc,2016, 4(5). pii: e01099-16. DOI: 10.1128/genomeA.01099-16
[14] Iritani N,Kaida A,Abe N,et al. Increase of GII.2 norovirus infections during the 2009-2010 season in Osaka City, Japan[J]. J Med Virol,2012, 84(3): 517-525. DOI: 10.1002/jmv.23211
[15] Yao D, Chen JF, Ye W, et al. Etiology and genotype features analysis of an acute gastroenteritis outbreak associated with Norovirus GII.17[J]. Chin J Zoonoses, 2016, 32(7): 641-643.DOI: 10.3969/j.issn.1002-2694.2016.07.010 (in Chinese)
姚栋, 陈静芳, 叶文, 等. 一起诺如病毒GⅡ.17型引起的急性胃肠炎病原学诊断及基因特征分析[J]. 中国人兽共患病学报, 2016, 32(7): 641-643. DOI: 10.3969/j.issn.1002-2694.2016.07.010
ViralgastroenteritisoutbreaksassociatedwithnewrecombinantstrainGII.P16/GII.2ofnorovirusinFujian,2016
WU Bing-shan1,2, HUANG Zhi-miao1, OU Jian-ming1, QI Xiao-qi1, HUANG Ye-wei3, WENG Yu-wei1,2
(1.FujianCenterforDiseaseControlandPrevention,Fuzhou350001,China; 2.FujianKeyLaboratoryofZoonoses,Fuzhou350001,China; 3.PublicHealthSchoolofFujianMedicalUniversity,Fuzhou350001,China)
We delineated the molecular characteristic of recombinant strain GII.P16/GII.2 of norovirus associated with acute viral gastroenteritis outbreaks in Fujian Province in winter of 2016. Norovirus were detected in specimens of patients collected from the gastroenteritis outbreaks by real-time reverse transcription-PCR and reverse transcription-PCR (RT-PCR). The PCR products of the positive samples were purified, and partial RNA dependent RNA polymerase (RdRp) gene and partial capsid gene were sequenced. The sequences were analyzed using bioinformatics software and online database,and phylogenetic tree were also constructed. Norovirus were detected in all 18 stools. Analysis of 9 positive sequences indicated an emergence of norovirus GII. P16/ GII.2 and confirmed being the cause of gastroenteritis outbreaks. All the strains shared homology of 98% with strains of Kawasaki 194 of Japan detected in 2016 and 97.7%-98.8% with IPH2161-08VG06 of Belgium detected in 2008, RdRp and capsid separately. These outbreak strains showed some degree of differences from the predominant strain, 2012 Sydney GII.4 variant. This is the first time to have found norovirus GII.P16/ GII.2 causing viral gastroenteritis outbreaks in Fujian. More in-depth analysis of the Norovirus GII.P16/ GII.2 could be useful to optimize preventative strategies and develop new and more effective therapeutic measure.
viral gastroenteritis; outbreaks; norovirus; GII.P16/GII.2; molecular characteristic
翁育伟,Email:wengywfjcdc@aliyun.com
1.福建省疾病预防控制中心,福州 350001; 2.福建省疾病预防控制中心人兽共患病重点实验室,福州 350001; 3.福建医科大学公共卫生学院,福州 350001
R373.2
:A
:1002-2694(2017)09-0805-04
2017-02-28编辑:刘岱伟
国家高技术研究发展计划(2011AA02A114)

