四川省嗜肺军团菌血清Ⅰ型(LP1)基因分型研究
曾林子,廖虹瑜,祁 腾,刘 谊,刘莉莉,罗隆泽
DOI:10.3969/j.issn.1002-2694.2017.09.006
四川省嗜肺军团菌血清Ⅰ型(LP1)基因分型研究
曾林子1,廖虹瑜1,祁 腾1,刘 谊2,刘莉莉1,罗隆泽1
目的为了解四川省嗜肺军团菌(Legionellapneumophila,LP)基因特征,采用多位点串联重复序列分析(MLVA)和基因序列分型(SBT)两种方法对四川省1989—2016年分离到的42株嗜肺军团菌血清I型(LP1)进行基因分型研究。方法根据文献报道的军团菌8个VNTR位点和SBT 7个管家基因对42株嗜肺军团1型菌(LP1)分别进行PCR扩增。VNTR结果经毛细管电泳分析后,得到MLVA分型。SBT结果测序后上传至欧洲军团菌感染组(EWGLI)数据库得到ST分型。结果42株嗜肺军团菌血清I型(LP1)分为8个MLVA型,优势型别为M08型(47.6%)和M07型(23.8%)。SBT分为 12个ST型,其中有2个ST型(ST2359,ST2360)为首次发现。ST1(52.3%)、ST630(14.2%)为优势型别。12个ST型分为3个克隆群(Clonal Complex,CCs)和2个singleton。结论MLVA方法和SBT方法均能用于LP1型分子流行病学监测及分子遗传学特征的研究。四川省LP1型具备较高的遗传多样性和地区特异性,有感染人的风险,应加强对公共卫生环境中军团菌的监测工作。
四川省;嗜肺军团菌血清Ⅰ型;基因分型
军团菌是一种兼性胞内寄生的革兰阴性杆菌,广泛存在于各种水环境中,尤其是空调冷却塔、供水系统和温泉水等人工水环境中。它能通过呼吸道传播,引起以肺部感染为主要症状的军团菌病。军团菌目前大约有 50 个种, 72 个血清型,90%以上的军团菌病都是由嗜肺军团菌引起的,其中嗜肺军团菌血清Ⅰ型菌株(Lp1) 为主要的致病血清型[1]。为了解四川省嗜肺军团菌血清Ⅰ型菌株的分子特征,丰富嗜肺军团菌基因库数据,本研究采用多位点串联重复序列分析(MLVA)方法和多位点测序方法(SBT)对四川省1989—2016年分离到的嗜肺军团菌血清I型(LP1)进行了基因分型研究。
1 材料和方法
1.1 菌株来源 42株LP1来源于1989-2016年期间四川省6个城市的监测样本,其中29株分离自酒店集中空调冷却塔水,2株分离自酒店淋浴热水,4株来源于成都市内喷泉水,6株来源于雅安市、康定县的温泉水,1株来源于四川省第1例军团菌病人。
1.2 菌株培养与鉴定 菌株复苏后接种于炭粉酵母浸出物培养基(BCYE)(北京路桥生物试剂有限公司),置于37 ℃,5% CO2环境下培养48~60h后,采用文献[2]的军团菌属特异性16S rRNA基因进行PCR扩增。对确定为嗜肺军团菌的菌株用血清凝集方法进行血清学分型鉴定(天津生物芯片)。
1.3 军团菌DNA制备 挑取分离纯化后的军团菌于1 mL生理盐水中制备成菌悬液,DNA提取按照试剂盒(QIAam pDNA MiniKit)说明进行操作,提取的DNA 置于-20 ℃保存备用。
1.4 MLVA 参考文献[3-4]引物,选取其中8个可变数目串联重复(Variable Number Tanden Repeat,VNTR)位点进行多重PCR扩增,引物用荧光标记。扩增产物用毛细管电泳仪读取片段长度,确定串联重复序列重复数。将8对引物分为3个VNTR组进行扩增(VNTR1、VNTR2、 VNTR3),PCR反应体系均为25 μL,其中2×PCR Mixture(擎科生物限公司) 12.5 μL,上下游引物(10 mmol/L)各1 μL,去离子水5.5 μL(VNTR1、VNTR2)或者7.5 μL(VNTR3),军团菌DNA模板1 μL。PCR反应条件为94 ℃预变性5 min,94 ℃变性 30 s,60 ℃退火30 s,延伸72 ℃ 45 s,共35个循环,72 ℃终末延伸10 min。
1.5 SBT 参考文献[5-6]引物,对7个基因位点flaA、pilE、asd、mip、momps、proA和neuA(neuA*)进行PCR 扩增、测序。将得到的序列信息与欧洲军团菌感染工作组(EWGLI)数据库中的序列进行比对,获得相应的ST 型。PCR反应体系均为25 μL,其中1×PCR Mixture(擎科生物有限公司) 22 μL,上下游引物(10 mmol/L)各1 μL,军团菌DNA模板1μL。PCR反应条件为95 ℃预变性5 min,95 ℃变性 30 s,55 ℃退火30 s,延伸72 ℃ 45 s,共35个循环,72 ℃终末延伸10 min。
1.6 聚类分析 采用BioNumerics Version 7.6软件对MLVA和SBT分型结果进行聚类分析。
2 结 果
2.1 16S rRNA及血清学鉴定 42株实验菌株军团菌属16S rRNA 检测均为阳性,扩增条带为650 bp。血清凝集实验显示均为嗜肺军团菌血清Ⅰ型。
2.2 MLVA聚类分析 42株LP1的8个MLVA位点均能扩增出产物,通过BioNumerics Version 7.6聚类分析分为8个MLVA型,聚类分析见图1,其中M08型菌株最多(n=20),占总数量的47.6%,其次为M07型(n=10),比例为23.8%,M01型和M04型也包含2种以上的菌株,比例分别为14.2%(n=6)和4.7%(n=2)。M02型、M03型、M05型、M06型仅有1株菌。MLVA型跟菌株来源有相关性,20株M08型菌株,除1株来源于喷泉水以外其余19株均分离自空调冷却水。10株M07型菌株,除1株来源于喷泉水以外其余9株均分离自温泉水。
2.3 SBT聚类分析 42株LP1的7个管家基因扩增均为阳性,分为12个ST型,其中2个ST型(ST2359,ST2360)为新ST型,剩余10个ST型(ST1,ST36,ST578,ST579,ST630,ST971,ST1011,ST1279,ST1562,ST1777)在欧洲军团菌感染工作组(EWGLI)数据库中有报道。ST1,ST36,ST630,ST2359型包含2株以上的菌株,以ST1型最常见,比例分别为52.3%(n=22)、4.7%(n=2)、14.2%(n=6)、9.5%(n=4),其余8个型均只有1株菌。聚类分析见图2。
为了解各ST型间的相互关系,同样通过BioNumerics Version 7.6软件,绘制了四川省LP1的克隆群分布图,见图3。12个ST型分为3个克隆群(Clonal Complex,CCs)和2个singleton。其中CC1群为四川省优势克隆群,包括30株菌,涵盖了ST1, ST630, ST1011, ST1777 四个型别,CC3群有7株菌,包含了ST2359, ST2360, ST971、ST1562四个型,CC2群有3株菌,包含两个ST型,ST36和ST579。3个克隆群菌株来源不同,CC1的菌株主要来源于人造水环境,包括空调冷却水、酒店淋浴热水及城市喷泉水,CC3的菌株主要来源于天然水环境——温泉水,CC2菌株来源于病人和人造水环境。

图2 42株LP1的 SBT型聚类分析Fig.2 Clustering results of data obtained by SBT analysis of 42 LP1 isolates
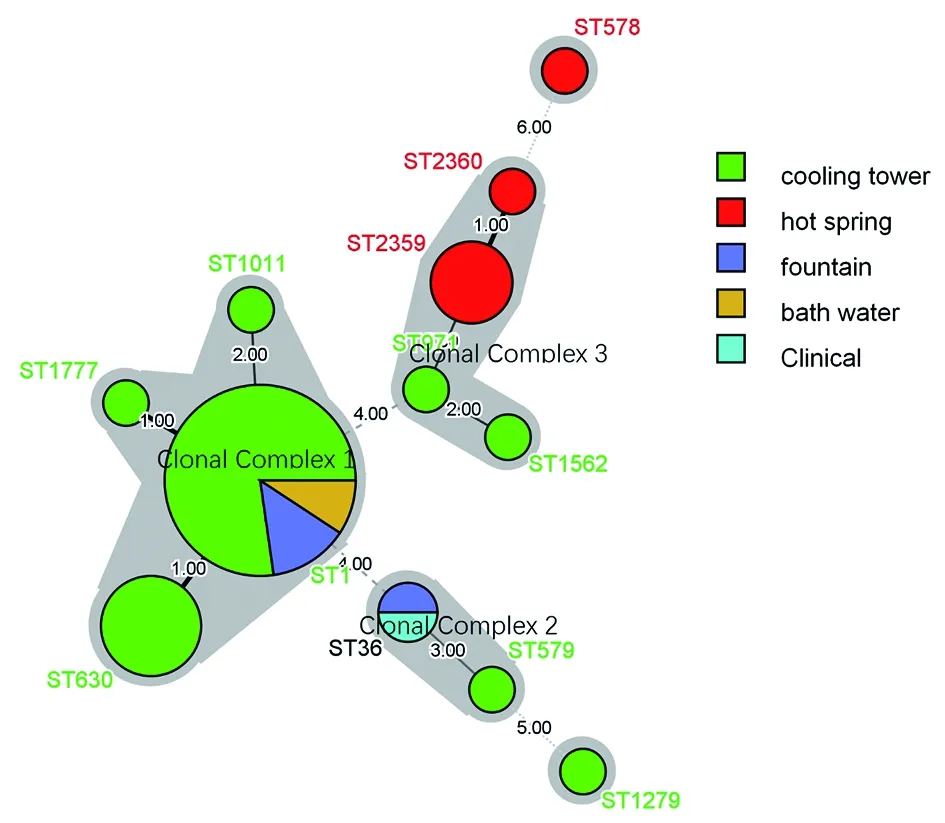
图3 42株LP1的克隆群分析结果Fig. 3 Minimum spanning tree analysis of 42 LP1
3 讨 论
目前,用于嗜肺军团菌研究的分子分型方法很多,常见的有基因序列分型(Sequence based typing,SBT) 、多位点可变数目串联重复序列多态性分析(Multiple Loci VNTR Analysis,MLVA) 、脉冲场凝胶电泳分型(Pulsed Field Gel Electrophoresis,PFGE) 、扩增片断长度多态性分型(Amplified Fragment LengthPolymorphism,AFLP) 等[7-9]。本研究选用MLVA和SBT方法对四川省LP1型菌株进行分子分型,这两种方法都具有重复性好、实验操作简便、实验结果数字化、易于标准化等优点,结果可与中国其他地区及其他国家作比较。
军团菌VNTR分型方法可选择不同的位点,C.Pourcel[3,10-11]报道了选用9个组合位点(Lpms1、Lpms4、Lpms4b、Lpms5、Lpms11、Lpms13、Lpms17、Lpms19、Lpms25)和8个组合(Lpms1_b、Lpms3 、Lpms13、Lpms17、Lpms19_b、Lpms33、Lpms34、Lpms35)位点对军团菌进行VNTR分型研究,结果发现8-MLVA方法更适用于嗜肺军团菌血清1型的分子分型,本研究最终选择8-MLVA方法对四川省分离的嗜肺军团菌进行分子分型。
C. Pourcel建立了嗜肺军团菌的MLVA基因库(http://mlva.upsud.fr/ml vav4/genotyping),截止到2016年9月,基因库里一共收录了1 140株嗜肺军团菌的MLVA数据,其中包括338株LP1。本研究发现,四川省优势MLVA型为M07和M08,分别占总数量的23.8% (10/42)和47.6% (20/42),它们均属于VACC1,全部分离自人工水环境(空调冷却水、酒店淋浴热水以及喷泉水)。这两种MLVA型同样广泛存在于世界其他国家,英国、法国、瑞士、德国、以色列、希腊、瑞典,西班牙均有报道[7]。除了分离自环境外,还有临床病人株,表明四川省分离到的M07、M08菌株有感染人的风险。M04属于VACC2,包括两株菌,分别来源于1989年四川省第1例军团菌病病人和2011年成都市区喷泉水,苏格兰、法国、英国、瑞士、德国等地也有过此型别的报道[5],报道的大部分M04菌株都来源于临床病人,因此,M04型别的菌株容易引起人的感染。来源于喷泉水的菌株分离自成都市闹市区的喷泉中,长年有大量游客和本地人到此处观光游览,有关卫生部门应该加强此地人工喷泉的监测,做好消毒工作,阻断传染源。M06型属于VACC6,于2007年分离自成都酒店空调冷却水中,美国和瑞士有相关型别报道,其中瑞士的菌株来源于临床病人。M01、M02、M03、M05属于四川省特有的MLVA型,未见到有文献报道,这些菌株均来源于环境样本,包括空调冷却水和温泉水,其中M01型菌株除1株来源于空调水,其余都来源于温泉水,温泉水温度在39~42 ℃。此型别的菌株是否具有致病力,还需要进一步的研究。
基因序列分型(SBT)是欧洲军团菌工作组推荐的一种军团菌分子分型的方法,选用以下7个管家基因flaA、pilE、asd、mip、momps、proA和neuA(neuA*)用于PCR扩增,经研究发现,某一些菌株的neuA基因无法扩增,故用neuA*代替[12],本研究中有1株菌选用了neuA*,这株菌来源于成都的空调冷却水,ST型别为ST1777。本研究第一次应用SBT方法建立了四川省LP1型菌株的数据库,42株LP1菌分成12个ST型别,显示四川省LP1的具有较高的遗传多样性。
据欧洲军团菌工作组报道,目前数据库里收录了11 563株军团菌,有7 675株军团菌分离自临床病人,3 792株菌株来源于环境样本,未知来源的菌株有96株(截止到2017年4月5日)。数据库里一共有2 355种ST型,优势型别是ST1型,四川省的优势型同样也是ST1,占总数量的53.3%(22/42),所有的ST1型菌株均来源于人工水环境,包括空调冷却水(77.3%)、喷泉水(13.6%)和酒店淋浴热水(9.1%),这与秦天[13]的调查报道一致。E.van Heijnsbergena[14]曾做过荷兰土壤中军团菌的研究,结果未从土壤中分离到ST1型的军团菌。ST1型菌株广泛存在于世界各地,除了来自于环境样本,还有分离自临床病人,目前四川省尚没有从临床病人身上分离到。
ST630是四川省仅次于ST1型的(14.2%)型别,均来源于空调冷却水,分布于四川省境内的四个城市:成都市、乐山市、内江市、雅安市。此型在中国广州市和北京市也见到报道,同样也分离自环境样本,国外报道较少,法国有1例病人报道。其他的型别如ST579、ST1011、ST578在欧洲、亚洲、北美洲都曾经从环境样本和病人身上分离到,而ST971、ST1562、ST1777存在于广东省和四川省环境样本中。ST2359、ST2360是四川省发现的新型,分离自雅安市境内某温泉。温泉水里的军团菌污染是引起人类感染军团菌病的主要来源之一,ST36包括两株菌,用8-MLVA方法将它们聚为M04型,两种方法都成功地把这两株菌聚为一类,表明它们之间有着高度的一致性。
本研究通过用8-MLVA和SBT的方法,对四川省1989-2016年分离到的LP1分别进行基因分型,结果表明这两种方法均能用于嗜肺军团菌分子流行病学监测及分子遗传学特征的研究,展示了四川省LP1优势基因型和优势菌群的分布特征,也表明了四川省的军团菌有感染人的风险。在今后的监测工作中,可把两种方法结合起来,可以更加准确地迅速找到军团菌病的传染源及暴发流行的趋势。
[1] Keller BW, Hajjeh R, Maria AD, et al. Community outbreak of Legionnaires disease: an investigation confirming the potential for cooling tower to transmitLegionellaspecies[J]. Clin Infect Dis, 1996, 22: 257-261.
[2] Lebeau I, Lammertyn E, DeBuck E, et al. First proteomic analysisLegionellapneumophilabased on its developing genome sequence[J]. Res Microbiol, 2005, 156(1): 119-129. DOI:10.1016/j.resmic.2004.08.010
[3] Pourcel C, Visca P, Afshar B, et al. Identification of variable-number tandem-repeat (VNTR) sequences inLegionellapneumophilaand development of an optimized multiple-locus VNTR analysis typing scheme[J]. J Clin Microbiol,2007, 45(4): 1190-1199. DOI: 10.1128/JCM.02078-06
[4] Zhu BQ, Ren HY, Zhou HJ, et al. Assessment on the application of multiple-locus variable-number tandem repeat analysis forLegionellasubtyping[J]. Chin J Zoonoses, 2012, (03): 256-260, 283. DOI: 10.3969/j.issn.1002-2694.2012.03.015 (in Chinese)
朱兵清,任红宇,周海健,等. MLVA方法在军团菌分子分型中的应用评价[J].中国人兽共患病学报,2012,(03):256-260,283.
[5] Gaia V, Fry NK, Afshar B, et al. Consensus sequence-based scheme for epidemiological typing of clinical and environmental isolates ofLegionellapneumophila[J]. J Clin Microbiol,2005, 43(5): 2047-2052. DOI: 10.1128/JCM.43.5.2047-2052.2005
[6] Yang M, Cheng HJ, Xiong CH, et al. Genetic characteristics ofLegionellapneumophilaserotype 1 strains[J]. Chin J Zoonoses, 2010, (12): 1151-1153, 1156. DOI: 1002-2694(2010)12-1151-03 (in Chinese)
杨梦,程慧键,熊长辉,等.嗜肺军团菌血清1型基因检测及分子分型研究[J].中国人兽共患病学报,2010,(12):1151-1153,1156.
[7] Zhang LY, Li Y, Zheng WL, et al. Sequence-based typing ofLegionellapneumophilaisolates from the water of public cooling tower[J]. Chin J Zoonoses, 2013, (03): 262-266, 273. DOI: 10.3969/cjz.j.issn.1002-2694.2013.03.011 (in Chinese)
章乐怡,李毅,郑文力,等.公共场所冷却塔水嗜肺军团菌的基因序列分型研究[J].中国人兽共患病学报,2013,(03):262-266,273.
[8] Zhang LY, Li Y, Shangguan ZH, et al. Contamination status and molecular typing ofLegionellapneumophilain spring environments[J]. Chin J Zoonoses, 2016, (12): 1096-1101. DOI: 10.3969/j.issn.1002-2694.2016.012.010 (in Chinese)
章乐怡,李毅,上官智慧,等. 温泉水中嗜肺军团菌污染调查及分子分型研究[J]. 中国人兽共患病学报,2016,(12):1096-1101.
[9] Pourcel C,Vidgop Y,Ramisse F,et al. Characterization of a tandem repeat polymorphism inLegionellapneumophilaand its use for genotypin[J]. J Clin Microbiol,2003, 41(5): 1819-1826. DOI: 10.1128/JCM.41.5.1819-1826
[10] Visca P,D’Arezzo S,Ramisse F,et al. Investigation of the population structure ofLegionellapneumophilaby analysis of tandem repeat copy number and internal sequence variation[J]. Microbiology,2011, 157(Pt 9): 2582-2594. DOI: 10.1099/mic.0.047258-0
[11] Ratzow S, Gaia V, Helbig JH, et al. Addition of neuA, the gene encoding N-acylneuraminate cytidylyl transferase, increases the discriminatory ability of the consensus sequence-based scheme for typingLegionellapneumophilaserogroup 1 strains[J]. J Clin Microbiol, 2007, 45(6): 1965-1968. DOI: 10.1128/JCM.00261-07
[12] Qin T, Yan GB, Ren HY, et al. High prevalence, genetic diversity and intracellular growth ability ofLegionellain hot spring environments[J]. PLoS One,2013, 8(3): e59018. DOI: 10.1371/Journal .pone.0059018
[13] Qin T, Zhou HJ, Ren HY, et al. Distribution of sequence-based types ofLegionellapneumophilaserogroup 1 strains isolated from cooling towers, hot springs, and potable water systems in China[J]. Appl Environ Microbiol,2014, 80(7): 2150-2157. DOI: 10.1128/AEM.03844-13
[14] van Heijnsbergen E, van Deursen A,Bouwknegt M, et al. Presence and persistence of viable, clinically relevantLegionellapneumophilabacteria in garden soil in the Netherlands[J]. Appl Environ Microbiol,2016, 82(17): 5125-5131. DOI: 10.1128/AEM.00595-16
GenetypingofLegionellapneumophilaserogroup1inSichuanProvince,China
ZENG Lin-zi1, LIAO Hong-yu1, QI Teng1, LIU Yi2, LIU Li-li1, LUO Long-ze1
(1.SichuanCenterforDiseaseControlandPrevention,Chengdu610041,China;2.ShenzhenHongcaiTestingTechnologyCO.,LTD,Shenzhen518172,China)
The aim of the research is to investigate the genetic characteristics ofLegionellapneumophilaserogroup1 (LP1) in Sichuan Province. The sequence-based typing (SBT) and multiple-locus VNTR analysis (MLVA) were used to describe the genetic polymorphism of 42 strains which were isolated from 1989-2016 in Sichuan Province, China. According to the reference, PCR was used to detect the 8-VNTR loci and 7 housekeeping genes respectively. The VNTR results were determined by using capillary electrophoresis, and the SBT results were sequenced and compared with the database of EWGLI. Results showed that totally 42 stains were divided into 8 MLVA types with the advantage types were M08(47.6%) and M07(23.8%). Twelve ST types were obtained with 3 main clonal complex and 2 singleton, including 2 novel ST types, among those, ST1 was the predominant type, accounting for 52.3%,following by ST630(14.2%). In conclusion, our results demonstrated MLVA and SBT were both applied to the research for molecular epidemiological investigation of LP1 and showed the high genetic polymorphism and the regional specificity. The results also suggest that the isolates are a potential threat to the public, effective control and prevention strategies are urgently needed.
Sichuan Province;Legionellapneumophilaserogroup1; gene typing
Luo Long-ze, Email: luoscdc@126.com
罗隆泽:Email:luoscdc@126.com
1.四川省疾病预防控制中心,成都 610041; 2.深圳市虹彩检测技术有限公司,深圳 518172
R378
:A
:1002-2694(2017)09-0784-05
2017-04-06编辑:梁小洁
四川省医学会青年创新课题(No.Q15081)
Supported by the Youth Innovation Project of Sichuan Medical Association (No.Q15081)

