结核分枝杆菌耐药相关基因扩增多重PCR体系的建立与评估
李自慧 潘丽萍 孙琦 吕翎娜 杜博平 张洪静 孙照刚 张宗德
·论著·
结核分枝杆菌耐药相关基因扩增多重PCR体系的建立与评估
李自慧 潘丽萍 孙琦 吕翎娜 杜博平 张洪静 孙照刚 张宗德
目的 建立结核分枝杆菌耐药相关基因多重PCR反应体系,评估该体系以临床分离株DNA和痰菌DNA为模板扩增耐药相关基因的成功率。方法 常规提取结核分枝杆菌临床分离株(47株)及涂阳肺结核患者(21例)痰菌DNA。根据目前研究已发现的结核分枝杆菌耐药相关基因合成引物,建立2个多重PCR体系以扩增目的基因。扩增片段经测序证实后,计算该体系用于两种样本的扩增成功率。结果 针对临床常用8种抗结核药物,选取结核分枝杆菌rpoB、katG、inhA、embB、gyrA、rpsL、rrs、eis共8个耐药相关基因设计合成引物。建立2个多重PCR体系扩增8个耐药基因,该体系扩增47株结核分枝杆菌临床分离株DNA和21例菌阳肺结核患者痰菌DNA的成功率分别为100.0%(47/47)和76.2%(16/21)。结论 本研究建立了高效扩增8个结核分枝杆菌耐药基因的多重PCR体系,结果对进一步研发结核病耐药分子诊断产品具有重要意义。
结核, 抗多种药物性; 聚合酶链反应; 基因测定; 核酸扩增技术; 技术评估, 生物医学
结核病(tuberculosis,TB)是由结核分枝杆菌感染引起的一种严重威胁人类健康的传染性疾病。近年来耐药结核病的出现和传播使得全球结核病的防控进一步受到威胁。据估计全球约20%的结核分枝杆菌分离株至少耐一种主要的抗结核药物[1],更严重的是耐多药结核病(multidrug-resistant tuberculosis,MDR-TB)、广泛耐药结核病(extensively drug-resistant tuberculosis,XDR-TB)甚至全耐药结核病(totally drug-resistant tuberculosis,TDR-TB)的出现和增多,使得患者病程延长,死亡风险增高,一旦治疗失败会导致耐药结核分枝杆菌的长期传染。我国不仅是结核病高负担国家,耐药结核病疫情也十分严峻。据统计,我国新发和复发结核病患者中MDR-TB分别占5.7%和25.6%,其中约8.0%为XDR-TB患者,均高于全球平均水平[2-3]。
研究发现,结核分枝杆菌耐药基因突变(包括药物作用靶标基因、修饰或分解药物的酶类基因等)、药物外排泵、药代动力学差异(包括药物代谢快慢、不同病变组织药物渗透力等)均有可能导致耐药的发生,其中耐药基因突变是导致结核病耐药的一个重要原因。例如:约95%的利福平耐药菌株rpoB基因发生突变,50%~95%的异烟肼耐药菌株katG基因发生突变[4]。近年来应用全基因组测序技术也发现了大量可能与耐药相关的基因位点[5-7]。在耐药检测方面,基于细菌培养技术的传统表型药物敏感性试验(简称“药敏试验”)需要2~3个月才能得到结果,而基于分子生物学技术的耐药基因突变检测技术可以快速检出耐药基因,指导临床及时选择有效药物,尽早治疗和控制耐药结核病。
目前,可用的耐药结核病分子检测方法仍十分缺乏,世界卫生组织推荐使用Xpert MTB/RIF检测和线性探针检测(line probe assay,LPA)技术。前者是由美国赛沛(Cepheid)公司研发、基于聚合酶链式反应(polymerase chain reaction,PCR)的技术,能快速、简便地检测利福平耐药,其主要缺点是费用十分昂贵,无法普及推广[8]。LPA主要基于PCR扩增和反向杂交技术,其代表产品GenoType MTBDRplus可以检测利福平和异烟肼耐药,GenoType MTBDRsl可以检测氟喹诺酮类药物及二线注射类抗结核药物耐药,但目前总体价格昂贵,需要专业技术人员操作,敏感度也略欠缺[9-11]。PCR技术目前仍然是分子检测的基础,但单一PCR反应每次只能扩增1个基因,实际工作要完成多样本的多个耐药基因检测,工作量巨大。多重PCR(multiplex PCR)通过在同一反应体系里加入一对以上的引物,可以在同一反应管中实现对多个核酸片段的扩增,在提高通量、保持高特异度和高敏感度的同时,能大大降低成本、节省样本,更加简单、快速和高效。本研究拟针对目前比较明确的结核分枝杆菌耐药基因,建立多重PCR反应体系,并评估其扩增临床分离株DNA及痰菌DNA的成功率,为进一步研发结核病耐药基因检测产品奠定基础。
材料和方法
1. 菌株及痰标本来源:收集结核分枝杆菌临床分离株47株,以及菌阳肺结核患者痰样本21例,所有样本来源于首都医科大学附属北京胸科医院。
2. 临床分离株DNA的提取:用TE缓冲液[pH=8.0,含三羟甲基氨基甲烷(Tris)-Cl 0.1 mol/L,乙二胺四乙酸(EDTA) 0.5 mol/L]重悬细菌,加入溶菌酶至5 mg/ml,37 ℃过夜振荡消化,加入蛋白酶K至终浓度为1 mg/ml,加入10%十二烷基硫酸钠(SDS)至终浓度为1%,37 ℃振荡1 h。用DNA小量提取试剂盒(51304,Qiagen公司)按操作说明提取菌株DNA,溶解于适量无菌去离子水,用NanoDrop 2000(美国Thermo Scientific)进行定量检测后,贮存于-20 ℃。
3. 痰菌DNA的提取:在生物安全柜中对痰标本常规采用N-乙酰-L-半胱氨酸氢氧化钠液化,取500 μl液体离心收集沉淀,用TE缓冲液洗一遍,重悬后加入玻璃珠,室温剧烈涡旋,然后95 ℃热灭活20 min,离心取上清,加1/10体积氯化钠和2倍体积预冷无水乙醇沉淀DNA,最后离心收集沉淀,用70%乙醇洗一遍,将DNA重新溶解于20 μl TE缓冲液,贮存于-20 ℃。
4. 耐药相关基因的引物设计及合成:参考文献[12],同时采用Primer premier 5.0软件设计和评价引物,由生工生物工程(上海)股份有限公司进行引物合成。
5. PCR扩增及测序:PCR扩增采用的水生栖热菌(thermus aquaticus,Taq)酶购于北京康为世纪生物科技有限公司(CW0654A),脱氧核糖核苷三磷酸(deoxy-ribonucleoside triphosphate,dNTP)购于天根生化科技(北京)有限公司(CD111-02)。由生工生物工程(上海)股份有限公司完成PCR产物的双向测序。
6. 主要仪器:生物安全柜(NU-425-300E,美国Nuaire公司)、基因扩增仪(My Cycler,美国Bio-rad公司)、凝胶成像系统(GelDoc XR,美国Bio-rad公司)、核酸分析仪(Nanodrop2000,美国Thermo公司)。
结 果
1. 耐药相关基因扩增所用引物序列:本研究选取目前发现的8个结核分枝杆菌耐药相关基因序列,分别为rpoB、katG、inhA、embB、gyrA、rpsL、rrs和eis,设计合成的引物序列见表1。

表1 结核分枝杆菌耐药基因扩增引物序列
注 F为上游引物;R为下游引物
2. 耐药基因扩增多重PCR体系的建立:本研究根据不同耐药基因扩增片段大小,尝试不同组合和扩增体系,最终通过优化可以实现利用2个多重PCR体系扩增8个耐药基因,扩增体系见表2。PCR扩增条件为:95 ℃ 10 min;95 ℃ 30 s,62 ℃ 30 s,72 ℃ 40 s,30个循环,72 ℃ 5 min。2%琼脂糖凝胶电泳显示,8个条带对应基因大小与预期一致(图1)。
3. 测序结果:将8个条带切胶测序,测序序列与结核分枝杆菌标准菌株H37Rv相应基因逐一进行比对,结果证明扩增序列是预期的8个耐药基因序列。以katG基因测序结果为例,比对结果证明扩增条带为katG基因,并且该菌株在第1355碱基位置由碱基T突变为C(图2)。
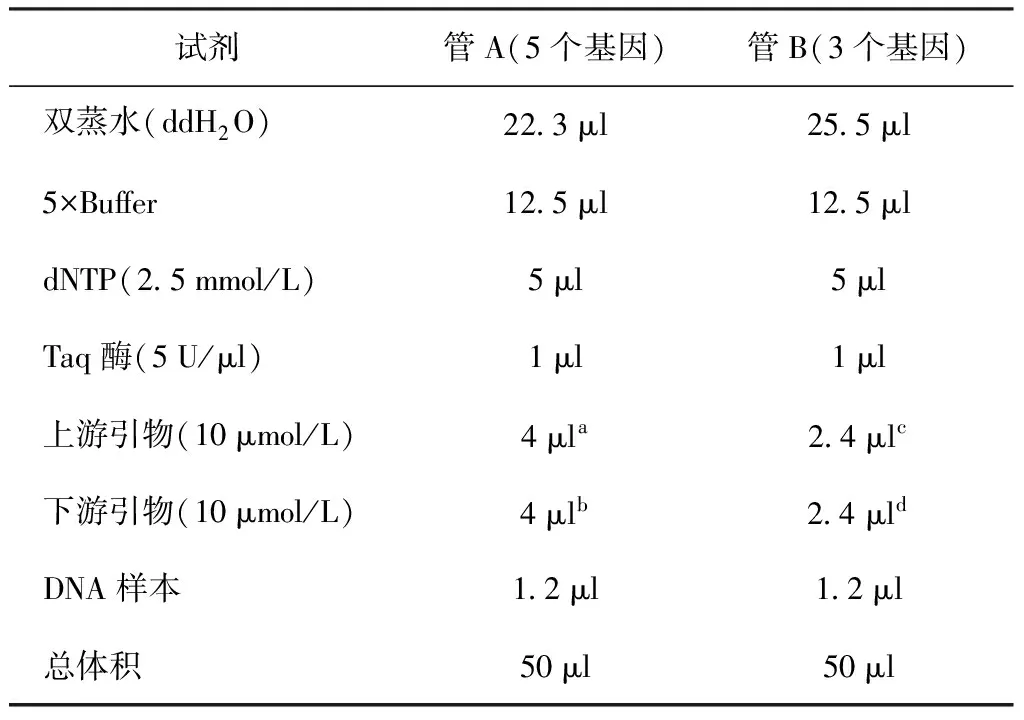
表2 结核分枝杆菌耐药基因扩增多重PCR体系
注 管A包含5对基因引物:embB、katG、rrs、rpoB和inhA;管B包含3对基因引物:eis、gyrA和rpsL。a:指管A中5条上游引物各0.8 μl;b:指管A中5条下游引物各0.8 μl;c:指管B中3条上游引物各0.8 μl;d:指管B中3条下游引物各0.8 μl
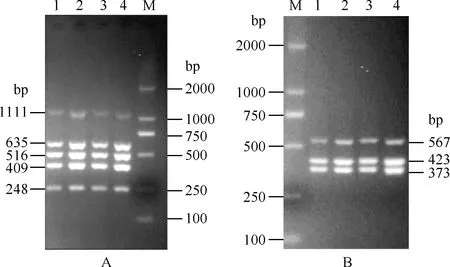
1~4为4个结核分枝杆菌临床分离株DNA样本;M为DNA分子量标准,条带从大至小分别为2000、1000、750、500、250和100 bp。A图为A管扩增产物电泳图,5个条带从大到小分别为embB、katG、rrs、rpoB和inhA;B图为B管扩增产物电泳图,3个条带从大到小分别为eis、gyrA和rpsL图1 PCR产物2%琼脂糖凝胶电泳图
4. 多重PCR体系扩增临床分离株DNA的成功率:提取47株结核分枝杆菌临床分离株DNA,以菌株DNA为模板,采用已建立的多重PCR体系进行扩增,结果47株的8个条带全部成功扩增(图3),扩增成功率为100.0%。
5. 多重PCR体系扩增痰菌DNA的成功率:提取21例痰涂片染色阳性的患者痰菌DNA,以其为模板,采用已建立的多重PCR体系进行扩增,结果有16例痰样本提取的DNA 8个条带全部成功扩增(图4),扩增成功率为76.2%。另外3例痰样本提取的DNA仅扩增出了部分条带,2例痰样本提取的DNA无任何条带扩增。
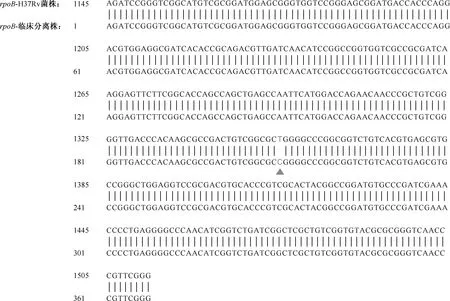
图2 katG基因PCR产物测序结果与基因序列比对结果
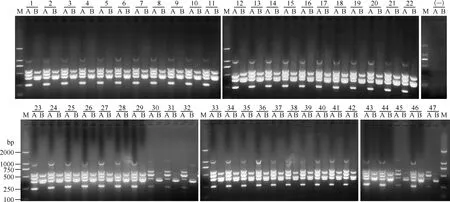
1~47为结核分枝杆菌临床分离株DNA样本;A代表A管,包含5对基因扩增引物(embB、katG、rrs、rpoB和inhA);B代表B管,包含3对基因扩增引物(eis、gyrA和rpsL); M为DNA分子量标准,条带从大至小分别为2000、1000、750、500、250和100 bp;(-)为阴性对照图3 以临床分离株DNA为模板的PCR产物2%琼脂糖凝胶电泳图
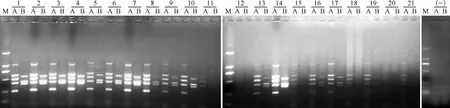
1~21为痰样本提取的DNA;A代表A管,包含5对基因扩增引物(embB、katG、rrs、rpoB和inhA);B代表B管,包含3对基因扩增引物(eis、gyrA和rpsL);M为DNA分子量标准,条带从大至小分别为2000、1000、750、500、250和100 bp;(-)为阴性对照图4 以痰菌DNA为模板的PCR产物2%琼脂糖凝胶电泳图
讨 论
本研究针对目前结核病治疗最常用的一线口服抗结核药物(异烟肼、利福平、乙胺丁醇、吡嗪酰胺)、注射类抗结核药物(链霉素、卡那霉素、阿米卡星、卷曲霉素)和氟喹诺酮类药物,检索文献、选取较确切的8个耐药基因,合成引物,扩增包含常见突变位点的基因片段。其中rpoB基因涉及利福平耐药,本实验扩增片段包含1123~1531区域内突变位点;katG基因和inhA启动子区涉及异烟肼耐药。本实验扩增片段包含katG基因438~1072区域和mabA-inhA启动子区常见突变位点,但inhA结构基因本身突变涉及异烟肼耐药所占比例较少[13-14],本研究暂不考虑;embB基因涉及乙胺丁醇耐药,扩增片段包含852~1962区域内突变位点;rpsL基因涉及链霉素耐药,扩增片段包含目前发现的全部突变位点。rrs基因涉及链霉素、阿米卡星、卡那霉素、卷曲霉素耐药,扩增片段包含1158位点后的所有突变位点;eis基因涉及卡那霉素耐药,扩增片段包含该基因启动子区内突变位点;gyrA基因涉及氟喹诺酮类药物耐药,扩增片段包含-29~394区域内突变位点。另外,pncA基因突变是吡嗪酰胺耐药的主要机制(约85%吡嗪酰胺耐药菌株存在pncA突变),但其突变位点繁多且分散,几乎涵盖整个基因,有的表型耐药和基因型耐药存在不一致[4,15-17]。研究发现,ahpC启动子区涉及异烟肼耐药,但也有研究认为其更像是因过氧化氢酶和(或)过氧化物酶活性缺失而引起的补偿性突变,并不是引起异烟肼耐药的原因[18]。本研究暂不纳入这些基因。具体耐药基因突变位点可参见数据库网站https://tbdreamdb.ki.se/Info/。
笔者采用2个多重PCR体系扩增8个包含常见突变位点的结核分枝杆菌耐药基因,以临床分离株提取的DNA为模板时成功率为100.0%,以肺结核患者痰样本提取的DNA为模板时成功率为76.2%。分析后者成功率低的原因,第一有可能是痰样本提取的结核分枝杆菌DNA含量相对较少,以宿主DNA为主,对扩增过程有干扰;更主要的原因可能是痰样本含菌量不同,提取出的细菌DNA含量存在差异,含量低的样本有可能扩增阴性。尽管本研究提取的痰样本均来自于痰涂片抗酸染色阳性的患者,但可能因为痰液黏稠、细菌在痰液中分布不均,部分提取的痰液含菌量较少,使得扩增未能成功。因此,采取措施提高痰菌量,如1例患者提取多个痰样本DNA,同时进一步优化痰样本结核分枝杆菌DNA的提取方法,如优化裂解液,增加研磨时间,以提高破菌效率、提高细菌DNA含量,均有可能会大大提高多重PCR扩增的成功率。
另外,虽然多重PCR能够同时扩增目的基因的数量在理论上没有限制,但实践中常常因为多对引物同时存在,容易产生非特异性扩增、扩增效率不同、扩增结果判读困难等原因而受到限制。因而在设计引物时要综合考虑引物的特异性、长度、退火温度等因素,并通过调整镁离子浓度、引物浓度等优化反应体系,以最终获得靶基因片段的有效扩增[19]。本研究也曾尝试将8对引物放在1个多重PCR体系扩增,但干扰明显,多数样本无法实现对所有目的基因的有效扩增,还需进一步调整和优化。
总之,本研究成功实现了利用2个多重PCR体系扩增8个结核分枝杆菌耐药基因,扩增基因包含常见的耐药突变位点,研究结果可为进一步应用测序、芯片杂交等技术确定突变形式奠定基础,这对研发相关诊断产品,快速预测耐药类型、正确选择抗结核药物进行治疗、控制耐药结核病疫情具有重要意义。
[1] Dheda K, Gumbo T, Maartens G, et al. The epidemiology, pathogenesis, transmission, diagnosis, and management of multidrug-resistant, extensively drug-resistant, and incurable tuberculosis. Lancet Respir Med, 2017. pii: S2213-2600(17)30079-6. [Epub ahead of print].
[2] Zhao Y, Xu S, Wang L, et al. National survey of drug-resis-tant tuberculosis in China. N Engl J Med, 2012,366(23):2161-2170.
[3] World Health Organization. Multidrug and extensively drug-resistant TB (M/XDR-TB): 2010 global report on surveillance and response. Geneva: World Health Organization, 2010.
[4] Zhang Y, Yew WW. Mechanisms of drug resistance inMycobacteriumtuberculosis: update 2015. Int J Tuberc Lung Dis, 2015, 19(11):1276-1289.
[5] Farhat MR, Shapiro BJ, Kieser KJ, et al. Genomic analysis identifies targets of convergent positive selection in drug-resis-tantMycobacteriumtuberculosis. Nat Genet, 2013, 45(10):1183-1189.
[6] Zhang H, Li D, Zhao L, et al. Genome sequencing of 161Mycobacteriumtuberculosisisolates from China identifies genes and intergenic regions associated with drug resistance. Nat Genet, 2013,45(10):1255-1260.
[7] Casali N, Nikolayevskyy V, Balabanova Y, et al. Evolution and transmission of drug-resistant tuberculosis in a Russian population. Nat Genet, 2014, 46(3):279-286.
[8] World Health Organization. WHO Strategic and technical advisory group for tuberculosis (STAG-TB) [R/OL]. Geneva: World Health Organization, 2010[2017-06-26]. http://www. who.int/tb/advisory_bodies/stag_tb_report_2010.pdf.
[9] Gardee Y, Dreyer AW, Koornhof HJ, et al. Evaluation of the GenoType MTBDRsl Version 2.0 Assay for Second-Line Drug Resistance Detection ofMycobacteriumtuberculosisIsolates in South Africa. J Clin Microbiol, 2017, 55(3):791-800.
[10] Tagliani E, Cabibbe AM, Miotto P, et al. Diagnostic Performance of the New Version (v2.0) of GenoType MTBDRsl Assay for Detection of Resistance to Fluoroquinolones and Second-Line Injectable Drugs: a Multicenter Study. J Clin Microbiol, 2015,53(9):2961-2969.
[11] Theron G, Peter J, Richardson M, et al. The diagnostic accuracy of the GenoType®MTBDRsl assay for the detection of resistance to second-line anti-tuberculosis drugs. Cochrane Database Syst Rev, 2014,(10):CD010705.
[12] Lipin MY, Stepanshina VN, Shemyakin IG, et al. Association of specific mutations inkatG,rpoB,rpsLandrrsgenes with spoligotypes of multidrug-resistantMycobacteriumtuberculosisisolates in Russia. Clin Microbiol Infect, 2007,13(6):620-626.
[13] Dalla Costa ER, Ribeiro MO, Silva MS, et al. Correlations of mutations inkatG,oxyR-ahpCandinhAgenes and in vitro susceptibility inMycobacteriumtuberculosisclinical strains segregated by spoligotype families from tuberculosis prevalent countries in South America. BMC Microbiol, 2009,9:39.
[14] 陈曦,马玙,金奇,等. 耐异烟肼结核分枝杆菌临床分离株耐药相关基因突变研究. 中华结核和呼吸杂志, 2005,28(4):250-253.
[15] Morlock GP, Crawford JT, Butler WR, et al. Phenotypic characterization ofpncAmutants ofMycobacteriumtuberculosis. Antimicrob Agents Chemother, 2000,44(9):2291-2295.
[16] Scorpio A, Zhang Y. Mutations inpncA, a gene encoding pyrazinamidase/nicotinamidase, cause resistance to the antituberculous drug pyrazinamide in tubercle bacillus. Nat Med, 1996, 2(6):662-667.
[17] 谷蕴婷,黄海荣. 结核分枝杆菌对吡嗪酰胺耐药相关研究进展. 中华结核和呼吸杂志, 2016,39(10):814-817.
[18] Almeida Da Silva PE, Palomino JC. Molecular basis and mecha-nisms of drug resistance inMycobacteriumtuberculosis: classical and new drugs. J Antimicrob Chemother, 2011,66(7):1417-1430.
[19] 刘宁伟,刘威,黄留玉. 多重核酸检测技术研究进展. 生物技术通讯, 2016,27(4):596-600.
(本文编辑:薛爱华)
Establishment and evaluation of the multiplex PCR systems for amplifyingMycobacteriumtuberculosisgenes involved in drug resistance
LIZi-hui,PANLi-ping,SUNQi,LYÜLing-na,DUBo-ping,ZHANGHong-jing,SUNZhao-gang,ZHANGZong-de.
BeijingChestHospital,CapitalMedicalUniversity,BeijingTuberculosisandThoracicTumorResearchInstitute,BeijingKeyLaboratoryforDrugResistantTuberculosisResearch,Beijing101149,China
ZHANGZong-de,Email:zzd417@163.com;SUNZhao-gang,Email:sunzg75@163.com
Objective To establish multiplex PCR systems for amplifyingMycobacteriumtuberculosisgenes involved in drug resistance and evaluate the success rates of amplification using DNA templates from clinical isolates and sputum samples of pulmonary tuberculosis patients. Methods DNA was routinely extracted from 47M.tuberculosisclinical isolates and 21 sputum samples of smear positive pulmonary tuberculosis patients. Primers for amplifying genes involved in drug resistance were designed and synthesized, and 2 multiplex PCR systems were established. The amplified fragments were confirmed by sequencing, and the success rates of amplification by multiplex PCR were analyzed. Results We selected 8 gene sequences includingrpoB,katG,inhA,embB,gyrA,rpsL,rrsandeis, which were involved in resistance to 8 commonly used anti-tuberculosis drugs. Two multiplex PCR systems were established to amplify 8 sequences, and the success rates of amplification were 100.0% (47/47) for DNA from 47 clinical isolates and 76.2% (16/21) for DNA from 21 sputum samples of smear positive pulmonary tuberculosis patients. Conclusion Two multiplex PCR systems are established for amplifying eightM.tuberculosisgene sequences involved in resistance to anti-tuberculosis drugs used in clinical practice. The result has important value for developing new genotypic tests to diagnose drug-resistant tuberculosis.
Tuberculosis, multidrug-resistant; Polymerase chain reaction; Genetic testing; Nucleic acid amplification techniques; Technology assessment, biomedical
10.3969/j.issn.1000-6621.2017.08.002
“十二五”国家科技重大专项(2015ZX10004801-003);重大传染病防治协同创新中心项目(PXM2016_014226_000052);北京市自然科学基金(7164245);北京市科技新星计划(Z161100004916080);北京市医院管理局“青苗”计划专项(QML20151501)
101149 首都医科大学附属北京胸科医院 北京市结核病胸部肿瘤研究所 耐药结核病研究北京市重点实验室
张宗德,Email:zzd417@163.com;孙照刚,Email:sunzg75@163.com
2017-06-30)

