基于转录组的沙棘木蠹蛾简单重复序列特征分析
崔明明,陶 静,宗世祥
基于转录组的沙棘木蠹蛾简单重复序列特征分析
崔明明,陶 静,宗世祥*
(北京林业大学林木有害生物防治北京市重点实验室,北京 100083)
为开发沙棘木蠹蛾微卫星信息,利用已获得的转录组数据,对其EST-SSR位点进行发掘,进而分析其特征。结果发现含SSR的序列5126条,识别的SSR总数为7499个,SSR出现频率为51.41%。微卫星序列主要以单碱基重复为主,发生频率为39.52%。研究共发现77种碱基重复基元,所占比例最高的为(A/T)n(73.74%),其次是(AT/AT)n(3.37%)。微卫星多为重复次数为10且长度为10 bp的短序列。研究结果为沙棘木蠹蛾的SSR分子标记研究,遗传多样性分析,种群遗传结构以及关键性状基因的发掘等研究奠定基础。
沙棘木蠹蛾;转录组;微卫星
简单重复序列(simple sequence repeat,SSR)又称作微卫星(microsatellites),普遍存在于真核生物基因组,是由若干个碱基组成的简单串联重复序列。SSR标记是分子标记手段的一种。现有常用的分子标记手段,包括RFLP,RAPD,ISSR,AFLP,SSR,SNPs等,都各有其优缺点(闫华超等,2006),相比之下,SSR标记具有数量丰富、分布广泛、共显性遗传、多态性丰富等特点,且试验成本低,结果相对稳定,操作简单,是遗传学背景研究非常有效的工具,被公认为是遗传学研究中最理想的分子标记手段之一(Liuetal., 2013),已被广泛应用于种群遗传多样性分析、动植物分类和进化、遗传图谱的构建、基因定位和克隆、分子标记辅助育种、品种鉴定等领域(Lietal., 2002;Varshneyetal., 2005)。
SSR按来源分,有基因组SSR(g-SSR)和转录组来源的SSR(EST-SSR)(王东等,2014)。与g-SSR相比,EST-SSR标记无需构建基因组文库、杂交、测序,避免了大量人力、物力和时间的投入,同时EST反映了基因组的编码区域,直接获得生物个体基因表达信息,因此EST-SSR多态性可能与基因功能直接相关(Eujayletal., 2002)。目前,昆虫中已有黑翅土白蚁Odontotermesformosanus(Huangetal., 2012)、烟粉虱Bemisiatabaci(Xieetal., 2012),细梢小卷蛾Rhyacionialeptotubula(Zhuetal., 2013)、黄粉虫Tenebriomolitor(Zhuetal., 2013)、扶桑绵粉蚧Phenacoccussolenopsis(罗梅等,2014)、云南切梢小蠹Tomicusyunnanensis(袁远等,2014)、绿豆象Callosobruchuschinensis(Duanetal., 2014)、粘虫Mythimnaseparate(胡艳华等,2015)、沟眶象Eucryptorrhynchuschinensis(武政梅等,2016)等借助现有的转录组数据成功开发了EST-SSR。
沙棘木蠹蛾Eogystiahippophaecolus(Huaetal., 1990)是我国重要的钻蛀性害虫,主要分布在内蒙、辽宁、山西、陕西、宁夏和甘肃等地,以幼虫钻蛀并取食沙棘Hippophaerhamnoides的根部和干部进行危害,可导致沙棘整株枯死,严重影响了沙棘林的经济和生态效益(路常宽等,2004;宗世祥等,2005a,2005b,2005c)。目前,有关沙棘木蠹蛾的研究主要在生物学生态学特性,灾害监测和控制技术策略上,关于该昆虫遗传信息的研究仅见陶静等利用AFLP分子标记分析其种群遗传多样性和遗传结构的报道(Taoetal., 2012)。沙棘木蠹蛾SSR分子标记的开发,能进一步揭示沙棘木蠹蛾的遗传背景。此外,本课题组已经完成了对沙棘木蠹蛾的转录组测序及组装,得到了质量较高的转录组数据,为开发EST-SSR提供了丰富的资源。为此,本研究基于沙棘木蠹蛾转录组数据发掘SSR位点,分析其组成及分布特征,为进一步利用SSR分子标记分析其种群间的遗传结构和遗传分化以及构建遗传图谱奠定基础,也将为其功能基因的开发利用、比较基因组学的研究等提供依据。
1 材料与方法
1.1 材料来源
1.1.1 供试虫源与处理
沙棘木蠹蛾幼虫采自中国东北部辽宁省建平县。经过72 h的饥饿处理,用无菌水清洗干净虫体,液氮速冻后存于-80℃冰箱。利用液氮研磨法磨碎昆虫样本,用TRIzol法提取RNA,使用RNeasy Plus Mini Kit(No.74134;Qiagen,Hilden,Germany)试剂盒。提取的RNA用Nanodrop 8000(Thermo,Waltham,MA,USA)检测浓度和A260/A280的值,经检测合格的样品进行下一步的测序工作。
1.1.2 转录组数据来源
测序工作由北京百迈克生物科技有限公司完成。原始数据已上传到Sequence Read Archive(SRA)数据库,登录号为SRR4409152。
1.1.3 沙棘木蠹蛾EST-SSR的筛选
利用MISA软件(MicroSAtellite identification tool, http://www.pgrc.Ipk-gatersleben.de/misa/),对筛选得到的1 kb以上的Unigene序列进行SSR位点搜索和分析。所检测SSR位点包括单碱基重复、二碱基重复、三碱基重复、四碱基重复、五碱基重复和六碱基重复6类。筛选标准为单碱基重复至少10次,二碱基重复至少6次,三碱基至六碱基的最少重复均为5次。复合SSR两个位点间最大间隔碱基数为100。
2 结果与分析
2.1 沙棘木蠹蛾EST-SSR位点的数量与分布
对筛选得到的1 kb以上的Unigene共14587条序列进行SSR分析。结果,包含SSR的序列有5126条,识别的SSR总数为7499个,其中3497条 Unigene只包含单个SSR位点,有1629条序列包含1个以上的SSR的序列。SSR发生频率(含有SSR的Unigene数目与总Unigene的数目之比)为35.14%,SSR出现频率(检出SSR个数与总Unigene数目之比)为51.41%。平均每隔2568.01 bp就含有1个SSR位点。沙棘木蠹蛾转录组中SSR位点的序列总长度达到81383.15 bp,SSR 位点平均长度为20.86 bp,一至六碱基重复的SSR位点的平均长度分别为9.80、13.05、15.33、19.33、25.67、42.00 bp(表1)。

表1 沙棘木蠹蛾SSR 不同重复基元分布情况Table 1 Distribution of different repeat motifs in Eogystia hippophaecolus transcriptome
2.2 沙棘木蠹蛾EST-SSR基元类型和比例
沙棘木蠹蛾EST-SSR重复类型丰富,单核苷酸至六核苷酸重复都可以发现。从SSR位点数量上看,出现最多的为单核苷酸重复,占总SSR位点数量的76.88%,其次是二、三核苷酸重复,分别占11.64%和10.68%,四、五、六核苷酸重复类型的数量很少,总计不足1% (表1)。
转录组SSR中共观察到77种重复基元,单核苷酸至六核苷酸种类分别有2、9、30、32、5、1种。在这77种重复基元中,以单碱基重复基元A/T数量占绝对优势,占总SSR的73.74%;其次是二碱基重复AT/AT以及单碱基重复G/C所占比例较高,分别为3.37%和3.13%;二碱基重复基元中,以AT/AT和TA/TA较多,占二碱基重复SSR总数的54.80%。在三碱基重复基元中GCG/CGC出现次数多占三碱基重复SSR的15.07%,其次是ATT/AAT、GGC/GCC和TAT/ATA,分别为12.08%、10.96%和8.72%;其他四碱基至六碱基重复基元类型虽多,但数量均较少,出现频率均较低。不同类型的SSR重复基元的分布见图1。
2.3 EST-SSR基元长度和重复次数
在识别的SSR的长度分布上,由于单碱基重复序列占的比重较大,基元长度为10 bp的占50.45%,长度为11 bp的占15.43%,其次是长度为12 bp(10.46%)和15 bp(8.01)的基元(见图2)。基元的重复次数上,以重复10次的基元数量最多(54.02%),其次是重复11次(16.73%),重复6次(7.20%)和12次(5.26%)的基元。不同重复次数的位点个数分布见图3。
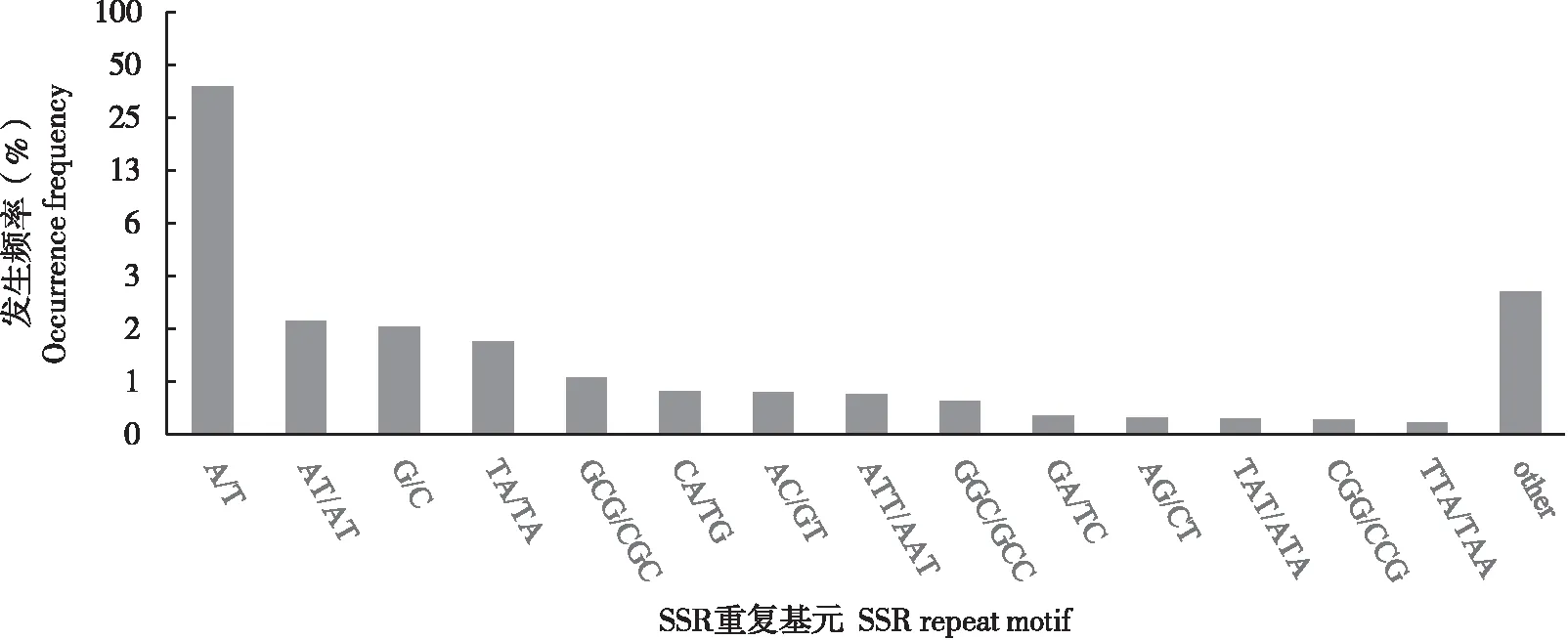
图1 基于重复基元类型的微卫星分布Fig.1 Microsatellites distribution on different repeat motifs

图2 沙棘木蠹蛾EST-SSR的长度分布图Fig.2 Length distribution of EST-SSR in Eogystia hippophaecolus transcriptome

图3 沙棘木蠹蛾EST-SSR重复次数分布图Fig.3 Distribution of EST-SSR repeat frequency in Eogystia hippophaecolus transcriptome
3 结论与讨论
随着二代测序的发展,SSR的发掘不再局限于构建SSR富集文库方法以及高通量发掘已测得物种基因组的方法,越来越多的基于转录组数据库高通量筛选微卫星的研究结果表明,开发转录组数据是一种高通量发掘SSR的有效方法(Lovinetal., 2009;Arthoferetal., 2011;Baietal., 2011)。
本研究通过对沙棘木蠹蛾转录组数据的筛选,从14587条Unigene中共识别了7499个SSR位点,分布在5126条Unigene序列上。SSR发生频率为35.14%,SSR出现频率为51.41%。平均每隔2568.01 bp就含有1个SSR位点,SSR位点平均长度为20.86 bp。沙棘木蠹蛾的转录组SSR出现频率相较于其他昆虫差异较大,如黑翅土白蚁转录组SSR出现频率为9.98%(Huangetal., 2012),烟粉虱为5.07%(Xieetal., 2012),细梢小卷蛾为3.09%(Zhuetal., 2013),粘虫为1.93%(胡艳华等,2015),沟眶象的10.36%(武政梅等,2016)。出现该现象的可能原因为:一是物种的特异性,可能沙棘木蠹蛾SSR的数量较为丰富;二是构建转录组的方法不同造成转录组数据的差异,比如,构建转录组选用的昆虫虫态和数量上、测序和组装所设置的参数的不同,都将造成转录组本身的Unigene数量上的差异;三是SSR位点的搜索方法或计算标准不同造成统计结果的差异,比如,本研究对转录组数据中长度在1 kb以上的Unigene进行的SSR高通量的发掘,筛选标准为单碱基重复至少10次,即SSR长度最小设置为10 bp。相对于沟眶象的类似研究中,同样是长度在1 kb以上的Unigene进行的搜索,筛选标准为单碱基重复至少12次,但SSR长度最小设置为12 bp,而单碱基重复的数量又相对较多,造成了结果的较大差异。而对于扶桑绵粉蚧(罗梅等,2014)、云南切梢小蠹(袁远等,2014)的研究,均是搜索转录组数据中所有的Unigene进行SSR的发掘,且SSR长度最小设置为12 bp,这就引起了差异较大的比较结果。综上三个原因,在进行类似研究的比较分析时,要基于参数设置基本相同的前提下,或者将参数列出以作为参考。
在发掘得到的沙棘木蠹蛾转录组SSR中,单碱基重复类型的数量最高,二、三碱基的数量相当。而绿豆象(Duanetal., 2014)、云南切梢小蠹(袁远等,2014)、沟眶象(武政梅等,2016)等昆虫的 EST-SSR主要以三碱基重复为主。这是由于本研究筛选标准为单碱基重复至少10次,本研究中SSR长度为10 bp的总数占到50%以上,因此单碱基重复SSR数量在本研究中占到绝对优势。本研究中SSR重复基元中所占比例最高的为A/T,所占比为73.74%。即使将筛选标准设置为单碱基重复至少12次,A/T所占比例达30.34%,在此筛选标准下占比依然最高,这与黄粉虫(Zhuetal., 2013)、云南切梢小蠹(袁远等,2014)、扶桑绵粉蚧(罗梅等,2014)的转录组中SSR的分析结果近似。
SSR标记作为一种高效的分子标记手段,其缺点是SSR标记具有特异性,必须进行PCR检测进行验证(李明芳和郑学勤,2004)。因此,在后期开发沙棘木蠹蛾SSR分子标记上,要对SSR重复单元前后的序列设计引物,并对其稳定性和多态性进行验证。
本研究基于转录组数据进行沙棘木蠹蛾EST-SSR的高通量发掘,并对发掘到的EST-SSR的特征进行了分析,结果得到了在种类和数量上均较为丰富的EST-SSR,进一步证明了利用转录组数据发掘SSR是一种高效可行的方法。研究结果丰富了沙棘木蠹蛾的分子标记,对其种群遗传结构、种群遗传多样性以及功能基因的开发利用、比较基因组学的研究都具有重要的参考价值。
References)
Arthofer W, Steiner FM, Schlick-Steiner BC.Rapid and cost-effective screening of newly identified microsatellite loci by high-resolution melting analysis [J].MolecularGeneticandGenomics, 2011, 286 (3): 225-235.
Bai X, Mamidala P, Rajarapu SP,etal.Transcriptomics of the bed bug (Cimexlectularius) [J].PLoSONE, 2011, 6 (1): e16336.
Duan CX, Li DD, Sun SL,etal.Rapid development of microsatellite markers forCallosobruchuschinensisusing Illumina paired-end sequencing [J].PLoSONE, 2014, 9 (5): e95458.
Eujayl I, Sorrells M, Banm M,etal.Isolation of EST-derived microsatellite markers for genotyping the A and B genomes of wheat [J].TheoreticalandAppliedGenetics, 2002, 104 (2): 399-407.
Hu YH, Li M, Zhang HF,etal.The information analysis of SSR loci in theMythimnaseparate(Walker) transcriptome [J].JournalofShanxiAgriculturalUniversity, 2015, 35 (5): 484-489.[胡艳华, 李敏, 张虎芳, 等.粘虫转录组中SSR位点的信息分析[J].山西农业大学学报, 2015, 35 (5): 484-489]
Huang Q, Sun P, Zhou X,etal.Characterization of head transcriptome and analysis of gene expression involved in caste differentiation and aggression inOdontotermesformosanus(Shiraki) [J].PLoSONE, 2012, 7 (11): e50383.
Li MF, Zheng XQ.Research progress of methods of SSR primers development [J].Hereditas, 2004, 26 (5):769-776.[李明芳, 郑学勤.开发SSR引物方法之研究动态[J].遗传, 2004, 26 (5): 769-776.]
Li YC, Korol AB, Fahima T,etal.Microsatellites: Genomic distribution, putative functions and mutational mechanisms: A review [J].MolecularEcology, 2002, 11 (12): 2453-2465.
Liu T, Zhu S, Fu L,etal.Development and characterization of 1827 expressed Sequence tag-derived simple sequence repeat markers for ramie (BoehmerianiveaL.Gaud) [J].PLoSONE, 2013, 8 (4): e60346.
Lovin DD, Washington KO, Bruyn B,etal.Genome-based polymorphic microsatellite development and validation in the mosquitoAedesaegyptiand application to population genetics in Haiti [J].BMCGenomics, 2009, 10: s590.
Lu CK, Luo YQ, Zong SX,etal.Adult behaviors and sex attraction ofHolcocerushippophaecolus[J].JournalofBeijingForestryUniversity, 2004, 26 (2): 79-83.[路常宽, 宗世祥, 骆有庆, 等.沙棘木蠹蛾成虫行为学特征及性诱效果研究[J].北京林业大学学报, 2004, 26 (2): 79-83.]
Tao J, Chen M, Zong SX,etal.Genetic structure in the seabuckthorn carpenter moth (Holcocerushippophaecolus) in China: The role of outbreak events, geographical and host factors [J].PLoSONE, 2012, 7 (1): e30544.
Varshney RK, Graner A, Sorrells ME.Genic microsatellite markers in plants: Features and applications [J].TrendsinBiotechnology, 2005, 23 (1): 48-55.
Wang D, Cao LY, Gao JP.Data mining of simple sequence repeats inCodonopsispilosulatranscriptome [J].ChineseTraditionalandHerbalDrugs, 2014, 46 (8): 2390-2394.[王东, 曹玲亚, 高建平.党参转录组中SSR位点信息分析[J].中草药, 2014, 46 (8): 2390-2394]
Wu ZM, Gao P, Wen JB.Analysis of microsatellites’ characteristics inEucryptorrhynchuschinensis[J].JournalofEnvironmentalEntomology, 2016, 38 (5): 979-983.[武政梅, 高朋, 温俊宝.沟眶象转录组微卫星特征分析[J].环境昆虫学报, 2016, 38 (5): 979-983]
Xie W, Meng QS, Wu QJ,etal.Pyrosequencing theBemisiatabacitranscriptome reveals a highly diverse bacterial community and a robust system for insecticide resistance [J].PLoSONE, 2012, 7 (4): e35181.
Yan HC, Gao L, Li GL.Application and development of molecular markers [J].BiologyBulletin, 2006, 41 (2): 17-19.[闫华超, 高岚, 李桂兰.分子标记技术的发展及应用[J].生物学通报, 2006, 41 (2): 17-19.]
Yuan Y, Zhang LF, Wu GX,etal.High-throughput discovery microsatellites inTomicusyunnanenss(Coleoptera: Scolytonae) [J].JournalofEnvironmentalEntomology, 2014, 36 (2): 166-170.[袁远, 张丽芳, 吴国星, 等.云南切梢小蠹微卫星的高通量发掘[J].环境昆虫学报, 2014, 36 (2): 166-170]
Zhu JY, Li YH, Yang S,etal.De novo assembly and characterization of the global transcriptome forRhyacionialeptotubulausing Illumina paired-end sequencing [J].PLoSONE, 2013, 8 (11): e81096.
Zhu JY, Wu GX, Yang B.High-throughput discovery of SSR genetic markers in the yellow mealworm beetle,Tenebriomolitor(Coleoptera: Tenebrionidae), from its transcriptome database [J].ActaEntomologicaSinica, 2013, 56 (7): 724-728.
Zong SX, Jia FY, Luo YQ,etal.Harm characteristics and population dynamics ofHolcocerushippophaecolus[J].JournalofBeijingForestryUniversity, 2005, 27 (1): 70-74.[宗世祥, 贾峰勇, 骆有庆, 等.沙棘木蠹蛾危害特性与种群数量的时空动态的研究[J].北京林业大学学报, 2005, 27 (1): 70-74]
Zong SX, Luo YQ, Xu ZC,etal.Geostatistical analysis on spatial distribution ofHolcocerushippophaecoluseggs and larvae [J].ActaEcologicaSinica, 2005, 25 (4): 831-836.[宗世祥, 骆有庆, 许志春, 等.沙棘木蠹蛾卵和幼虫空间分布的地统计学分析[J].生态学报, 2005, 25 (4): 831-836]
Zong SX, Yao GL, Luo YQ,etal.Niche of main boring pests inHippophaerhamnoidea[J].ActaEcologicaSinica, 2005, 25 (12): 3264-3270.[宗世祥, 姚国龙, 骆有庆, 等.沙棘主要蛀干害虫种群生态位[J].生态学报, 2005, 25 (12): 3264-3270]
Feature analysis of simple sequence repeats inEogystiahippophaecolustranscriptome
CUI Ming-Ming, TAO Jing, ZONG Shi-Xiang*
(Beijing Key Laboratory for Forest Pest Control, Beijing Forestry University, Beijing 100083, China)
In order to exploit SSR information ofEogystiahippophaecolus, we identify EST-SSR loci and analyze their features according to the transcriptome ofE.hippophaecolus.Results show 5126 unigenes contain 7499 SSR loci in total (51.41%).Mononucleotide repeats predominated with an occurrence frequency of 39.52%.There are 77 kinds of repeat motifs existing inE.hippophaecolustranscriptome.(A/T)n (73.74%) is most frequent in all the repeat types, and next is (AT/AT)n (3.37%).Most of the SSR are less than ten times of repetition and are 10 bp in length.The results should contribute to researches in SSR marker, genetic diversity, population genetic structure and genomic signatures identification inE.hippophaecolus.
Eogystiahippophaecolus; transcriptome; microsatellites
崔明明,陶静,宗世祥.基于转录组的沙棘木蠹蛾简单重复序列特征分析[J].环境昆虫学报,2017,39(3):605-610.
国家自然基金面上项目(31470651)
崔明明,女,硕士研究生,研究方向为昆虫分子生物学
*通讯作者Author for correspondence,E-mail: zongsx@126.com
Received: 2017-01-01;接受日期Accepted: 2017-02-23
Q963;S433.3
A
1674-0858(2017)03-065-06

