陕西及周边地区PRRSV Nsp2与GP5基因变异分析
吴旭锦,朱小甫
(咸阳职业技术学院 畜牧兽医研究所 动物疫病分子生物学诊断实验室,陕西 咸阳 712000)
陕西及周边地区PRRSVNsp2与GP5基因变异分析
吴旭锦,朱小甫
(咸阳职业技术学院 畜牧兽医研究所 动物疫病分子生物学诊断实验室,陕西 咸阳 712000)
【目的】 了解陕西及周边省份部分地区猪繁殖与呼吸综合征病毒(PRRSV)Nsp2和GP5的变异情况。【方法】 采用套式RT-PCR方法,从肝、脾、肺和血清中直接扩增Nsp2、GP5基因并克隆测序,将获得的PRRSV流行毒株序列与GenBank中30个PRRSV参考毒株进行比对分析,构建进化树。【结果】 获得了13株PRRSV的Nsp2基因序列(GenBank登录号 KM233849~KM233861)和12株GP5基因序列(GenBank登录号 KM233862~KM233873)。基因进化树分析显示所有测定PRRSV流行毒株均属北美洲型。对Nsp2基因序列分析发现,12株PRRSV为氨基酸缺失毒株,1株PRRSV为经典毒株。发现GSXF毒株有可能是一株重组毒株。PRRSV变异株与经典株相比,GP5蛋白中存在多处氨基酸点突变。【结论】 陕西猪场PRRSV流行毒株以变异株为主,同时存在经典毒株和重组毒株。
猪繁殖与呼吸综合征病毒;Nsp2基因;GP5基因;陕西及周边省份
猪繁殖与呼吸综合征(Porcine Reproductive and Respiratory Syndrome,PRRS)最早发生在20世纪80年代的美国[1],随后欧洲也爆发了该病。直到1991年,荷兰中央兽医局分离到了新病毒[2],随后美国和加拿大也分离到该病毒,至此确定了病原为猪繁殖与呼吸综合征病毒(Porcine Reproductive and Respiratory Syndrome Virus,PRRSV)。本病传播迅速,很快蔓延到全世界。我国于1996年首次在北京地区猪群中分离到了PRRSV[3]。2006年,我国南方爆发的猪无名高热引起了巨大损失,最终农业部确定病原为高致病性猪繁殖与呼吸综合征病毒(Highly-pathogentic PRRSV,HP-PRRSV),PRRSV的变异株引起学者们的高度关注[4-6]。
PRRSV分为2个基因型,即北美洲型(以VR-2332为代表株)和欧洲型(以LV为代表株),两个类型毒株之间核苷酸同源性差异较大。PRRSV基因组全长约15 kb,含8个开放阅读框(ORF),编码2个非结构蛋白和6个结构蛋白[7],其中Nsp2属非结构蛋白,由ORF1a编码,与PRRSV对细胞或组织的嗜性有关。美洲型PRRSVGP5基因长度为603 bp,其编码200个氨基酸,而欧洲型PRRSVGP5基因长度为606 bp,编码201个氨基酸。GP5蛋白是PRRSV的主要中和抗原蛋白,免疫保护作用突出。国内外研究表明,Nsp2和GP5的变异程度较大,且其变异会导致病毒致病力的改变,因而是研究PRRSV遗传变异的热点区域[8-9]。本课题组对陕西省部分地区2013-2014年PRRSVNsp2和GP5基因变异情况进行了调查,以期了解陕西省PRRSV的变异特点,为PRRS防控提供理论参考。
1 材料与方法
1.1 材 料
1.1.1 病料与血清 所有组织病料由本课题组采集或由猪场送检,主要包括病死猪肺脏、脾脏、肝脏和淋巴结,将各组织研磨处理,12 000 r/min离心10 min,收集上清液,共计65份。另采集病猪血清42份,将组织病料上清液和血清置-70 ℃保存备用。
1.1.2 试 剂 TRIzol Reagent、pGEM-T Easy载体购自Invitrogen公司;AMV反转录酶(10 U/μL)、RNA酶抑制剂(40 U/μL)、DEPC处理水、rTaqDNA酶(5 U/μL)、dNTP(各成分均为10 mmol/L)、UNIQ-10柱式DNA胶回收试剂盒等均购自生工生物工程(上海)有限公司。DL2000 DNA分子质量标准为宝生物工程(大连)有限公司产品。DH5α感受态细胞为动物疫病分子生物学诊断实验室保存。
1.1.3 引物设计与合成 根据GenBank上公布的PRRSV CH-1a(GenBank登录号:AY032626)、HUN4(GenBank登录号:EF635006)以及JXA1(GenBank登录号:EF112445)全基因组序列,利用Primer Premier 5.0软件,设计了4对引物,由生工生物工程(上海)有限公司合成,用DEPC处理水稀释到20 μmol/L备用。引物序列与预期扩增片段长度见表1。

表 1 供试引物的相关信息
1.2 方 法
1.2.1 总RNA的提取 取病料上清液或血清250 μL,放入1.5 mL无菌无RNA酶EP管中,加入750 μL TRIzol Reagent裂解10 min,加入200 μL预冷的氯仿,剧烈振摇,充分乳化,静置10 min后于4 ℃、12 000 r/min离心10 min;吸取上清液600 μL转移至另一EP管中,加入750 μL冰冷的异丙醇颠倒混匀,置-20 ℃沉淀30 min;4 ℃、12 000 r/min再次离心10 min,弃去上清液,加入1 mL冰冷的体积分数75%酒精洗涤1次,弃去酒精,倒置离心管自然干燥即得总RNA。
1.2.2 第一链cDNA的合成 在核酸干燥过程中,按照以下体系配制反转录反应液: DEPC处理水12.5 μL,5×AMV Buffer 4.0 μL, dNTP 2.0 μL,下游引物Nsp2-1R或GP5-1R 1.0 μL,AMV 反转录酶0.25 μL,RNA酶抑制剂反转录酶0.25 μL,总体积20.0 μL。核酸干燥后用配制好的反转录反应液充分溶解核酸,置42 ℃水浴反转录90 min,取出即可作为模板用于PCR扩增。
1.2.3Nsp2、GP5基因的套式RT-PCR扩增Nsp2、GP5基因扩增体系和条件一致,第1次扩增反应体系为:cDNA 2.0 μL,10×PCR Buffer 2.5 μL,Mg2+2.0 μL,dNTP 1.0 μL,上、下游引物Nsp2-1F/Nsp2-1R或GP5-1F/GP5-1R各0.5 μL,rTaqDNA聚合酶0.5 μL,用超纯水补足总体积25.0 μL。反应条件设定为:95 ℃预变性5 min;94 ℃ 变性50 s, 55 ℃退火60 s,72 ℃延伸60 s,共进行35个循环;最后72 ℃延伸10 min。取2.0 μL第1次扩增产物作为模板进行第2次扩增,体系同上,引物选用第2次扩增引物Nsp2-2F/Nsp2-2R或GP5-2F/GP5-2R。反应条件设定为:95 ℃预变性5 min;进入循环后94 ℃变性 40 s, 58 ℃退火50 s,72 ℃ 延伸50 s,共35个循环;最后72 ℃延伸10 min。取5.0 μL第2次扩增产物,进行10 g/L琼脂糖凝胶电泳,凝胶成像系统中照相观察。
1.2.4Nsp2、GP5基因序列测定与分析 将扩增得到的Nsp2、GP5基因PCR产物用UNIQ-10柱式DNA胶回收试剂盒回收,连接pGEM-T Easy载体,转化DH5α感受态细胞,菌液PCR鉴定为阳性后,取样送生工生物工程(上海)有限公司进行测序。选取GenBank中登录的参考序列(表2),利用DNAStar软件进行序列比对分析,绘制系统发生树,并选取部分代表毒株比较相应的氨基酸序列。

表 2 用于序列比较的PRRSV参考毒株基本信息
2 结果与分析
2.1 PRRSVNsp2、GP5基因的套式RT-PCR扩增结果
从部分送检病料和血清中成功扩增出了Nsp2、GP5基因片段,扩增的PRRSV变异株Nsp2基因长约562 bp,经典株约为649 bp(图1);扩增的GP5基因长约927 bp(图2),包含了完整的GP5基因603 bp,片段长度与预期大小相符。
将不同地域来源的毒株阳性样品测序后共获得了13株PRRSV的Nsp2基因和12株PRRSV的GP5基因序列,将序列上传GenBank获得了相应的登录号,序列基本信息见表3。
2.2 PRRSVNsp2基因序列比对
将30个PRRSV参考毒株全基因序列截短至与本研究测定的Nsp2长度一致后,将其与本试验测定的13株PRRSVNsp2基因片段进行比对分析,应用DNAStar软件绘制进化树。从进化树(图3)可见,参比Nsp2基因序列分为2大群,即欧洲型和北美洲型。其中需要注意的是,我国参考毒株NVDC-NM1-2011为欧洲型毒株,提示我国存在欧洲型PRRSV。本研究测定的13株病毒均属于北美洲型,而北美洲型又可细分为5个亚群,其中仅有SXLC01-Nsp2株属于第2亚群,其余12株均属于第1亚群。

M.DL2000 DNA分子质量标准;1~6.部分PRRSV Nsp2基因扩增产物(1~5为部分Nsp2基因变异株,6为Nsp2基因经典株)
图 1 部分PRRSVNsp2基因扩增结果
Fig.1 Amplification of partial PRRSVNsp2 gene
M.DL2000 DNA分子质量标准;1~8.GP5基因扩增产物
图 2 部分PRRSVGP5基因扩增结果
Fig.2 Amplification of partial PRRSVGP5 gene
为便于分析,选取参考毒株CH-1a、HUN4、JXA1、VR-2332、TJ与本研究测定毒株进行核苷酸序列同源性比对分析。结果发现,所测定的13个毒株间Nsp2基因同源性在74.7%~99.8%,其与JXA1强毒株同源性在66.4%~98.7%,与2001年的CH-1a相比,同源性为78.4%~90.0%,与经典北美洲型VR-2332毒株同源性为75.1%~94.3%,与HUN4强毒株同源性在66.0%~98.6%,与TJ株同源性在66.0%~98.0%。

表 3 基于Nsp2和GP5基因序列的陕西及周边省份PRRSV毒株基本信息
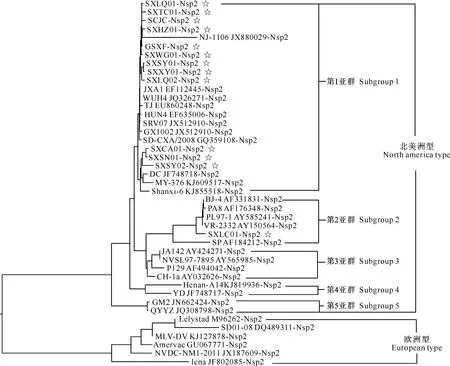
☆.本研究测定毒株 Isolates used in the research
图 3 陕西及周边省份PRRSVNsp2基因进化树
Fig.3 Phylogenetic tree of PRRSV based onNsp2 sequence in Shaanxi and neighboring provinces
推导氨基酸序列比较表明,本研究测定的13个PRRSV毒株间同源性为56.5%~97.3%,其与JXA1强毒株同源性为56.0%~97.8%,与CH-1a同源性为69.4%~86.0%,与VR-2332毒株同源性为62.9%~90.7%。从图4可见,本研究测定的13个毒株中,仅SXLC01-Nsp2未发生氨基酸缺失现象,其他毒株均存在30个氨基酸的不连续缺失现象。
2.3 PRRSVGP5基因序列比对
将PRRSV参考毒株全基因序列截短至与本研究测定的PRRSVGP5长度一致,将其与获得的12株PRRSV流行毒株GP5基因比对,绘制进化树。结果(图5)显示,PRRSV可分为北美洲型和欧洲型两大群,其中欧洲型的6个毒株分群上没有变化;北美洲型毒株亚群分布上有一定变化,用GP5基因比对可将其分为4个亚群,其中GSXF-ORF5属于第3亚群,而GSXF-Nsp2则属于第1亚群;其余11个毒株仍属于第1亚群。
与参考毒株CH-1a、HUN4、JXA1、VR-2332、TJ进行同源性比较,结果发现所测定的12个PRRSV毒株间GP5基因同源性为89.6%~99.5%;其与经典PRRSV毒株VR-2332同源性在89.0%~97.2%,其中GSXF-ORF5与VR-2332同源性高达97.2%;与经典疫苗株CH-1a同源性为92.1%~95.6%;与高致病性猪蓝耳病灭活苗JXA1株同源性为90.2%~99.0%;与HUN4株同源性为90.7%~99.5%;与TJ株同源性为90.5%~99.1%。
本研究测定的12株PRRSV GP5蛋白比较重要的位点有R13、R151、S137,这些位点变化与PRRSV毒力有关。此外,变异株和经典株GP5相比存在多处点突变,如E3→G3,G9→C9,S16→F16,C24→Y24,F25→L25,N58→Q58,S66→T/I66,A92→(G/S)92,V94→A94,F101→Y101,T121→I121,F127→L127,R164→G164,V185→A185,I189→L189,Q196→(L/R)196。

图中方框表示与经典株相比,变异株Nsp2缺失30个不连续氨基酸
图 4 陕西及周边省份Nsp2推导氨基酸序列缺失现象
Fig.4 Absence of Nsp2 deduced amino acid sequence in Shaanxi and neighboring provinces
3 讨 论
自从20世纪80年代美国报道发生PRRS以来,该病迅速蔓延至世界各地。2006年我国爆发高致病性PRRS,造成了巨大的经济损失,随后国内许多研究都证实PRRSV变异株普遍存在于我国猪场,成为严重威胁养猪业的重大疫病之一[4,10]。
Nsp2基因编码PRRSV非结构蛋白,易发生变异。研究认为,PRRSV变异株与经典毒株相比,Nsp2推导氨基酸序列存在不连续的30个氨基酸的缺失[11-12]。以前认为,氨基酸缺失的特征是高致病性PRRSV的重要标志,但后来有学者研究证明该缺失与毒力无直接联系[9]。张民泽等[13]研究认为,不同毒株间Nsp2编码区存在较大差异,可能与病毒对细胞或组织的嗜性有关。本研究设计了套式RT-PCR方法,为分析2013-2014年陕西省猪场PRRSV流行特点,成功扩增出了涵盖变异株缺失区域的Nsp2基因片段,获得了13株PRRSV流行毒株序列。本研究PRRSVNsp2基因进化树分析表明,13株PRRSV均属于北美洲型,未发现欧洲型毒株。24个PRRSV参考毒株和13个测定PRRSV流行毒株中的北美洲型可细分为5个亚群,其中仅有1株本研究测定的流行毒株属于第2亚群,其余12株均属于第1亚群,表明第1亚群为优势亚群。分析显示,获得的13株PRRSV Nsp2氨基酸序列中,12株存在不连续的30个氨基酸缺失,为PRRSV变异株;陕西洛川毒株SXLC01-Nsp2不存在氨基酸缺失现象,为PRRSV经典毒株,与VR-2332核苷酸序列同源性高达94.3%,氨基酸序列同源性为90.7%。结果证实陕西猪场PRRSV以变异株为主,同时存在经典毒株。
GP5蛋白是PRRSV主要的保护性抗原,且GP5蛋白在不同的PRRSV中变异较大,这种多变性会直接影响疫苗的交叉保护率[14]。本研究中PRRSVGP5基因进化树分析表明,用GP5基因分群和用Nsp2基因分群结果在大群上一致,个别毒株在亚群分布上有一定差异。获得的12个PRRSVGP5基因毒株中,其与经典株VR-2332基因同源性达89.0%~97.2%,其中GSXF-ORF5株与经典株VR-2332同源性高达97.2%,遗传上和经典VR-2332株更为相似。但从Nsp2基因对比分析看,GSXF株与VR-2332同源性仅为77.6%,而与国内变异株HUN4、JXA1、TJ株的同源性高达98.0%以上,提示GSXF毒株有可能是一株重组毒株。目前已有研究认为,PRRSV不同毒株之间会发生重组[15],Liu等[16]通过动物试验证实早在感染后1周即可发现PRRSVGP5和Nsp2基因重组现象。本研究进一步印证了在猪场的流行毒株中存在重组毒株,表明猪群中的PRRSV流行毒株存在多样性和复杂性。
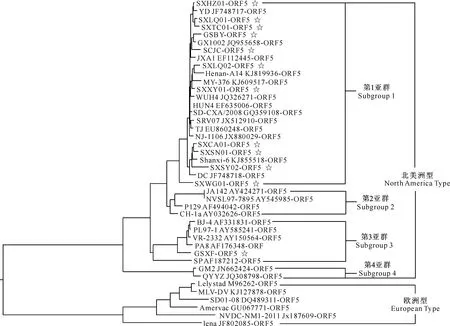
☆为本研究测定毒株 Isolates used in the research
图 5 陕西及周边省份PRRSVGP5基因进化树
Fig.5 Phylogenetic tree of PRRSV based onGP5 sequence in Shaanxi and neighboring provinces
Allende等[17]研究认为,GP5蛋白中13和151位氨基酸与毒力相关,本研究测定的12株均为R13、R151,符合强毒株的特征。流行毒株与经典疫苗株的137位氨基酸上均为S137,提示获得的流行毒株均是野毒。Ostrowski等[18]采用噬菌体表面展示确定了PRRSV ORF5的2个最主要的抗原表位,分别为位于第27-30位氨基酸 (A/L)27L28V29N30和37-39位氨基酸S37H38(L/I)39。比较发现,在这2个抗原表位中,测定毒株和参考毒株CH-1a、HUN4、JXA1、VR-2332、TJ的27位均为V,29位差异较大,测定毒株有6株为V29,6株为A29,这一差异对抗原性有无影响,有待于进一步研究。测定毒株和参考株S37H38无变化,39位上2个测定毒株为L,10株为I,提示第37-39位氨基酸抗原表位遗传稳定。此外,PRRSV变异株和经典株GP5相比存在多处点突变,这些突变对PRRSV的致病性、抗原性有无影响,需要深入研究。
近些年来,由于面临PRRS的巨大压力,猪场越来越倾向于使用弱毒疫苗控制PRRS,市场上经典弱毒株和变异株致弱毒株共存,猪场频繁使用疫苗和一个猪场使用多个疫苗毒株加剧了PRRSV的变异和重组,为PRRS的防控带来了更大的困难,继续研究PRRSV流行毒株关键基因的遗传变异特点,把握PRRSV的变异规律,有助于人们进一步了解PRRS的流行规律,为防控PRRS提供参考。
[1]Keffaber K K.Reproductive failure of unknown etiology [J].Am Assoc Swine Pract Newsl,1989(1):1-9 .
[2]Han J,Rutherford M S,Faaberg K S.The porcine reproductive and respiratory syndrome virus nsp2 cysteine protease domain possesses both trans and cis-cleavage activities [J].J Virol,2009,83(18):9449-9463.
[3]郭宝清,陈章水,刘文兴,等.从疑似PRRS流产胎儿分离PRRSV的研究 [J].中国畜禽传染病,1996(2):1-4.
Guo B Q,Chen Z S,Liu W X,et al.Isolation and identification of porcine reproductory and respiratory syndrome(PRRS) virus from aborted fetuses suspected of PRRS [J].China Animal Infectious Diseases,1996(2):1-4.
[4]Tian K G,Yu X I,Zhao T Z,et al.Emergence of fatal PRRSV variants:unparalleled outbreaks of a typical PRRS in China and molecular dissection of the unique hallmark [J].PLoS One,2007,2(6):526.
[5]Zhou I,Yang H C.Porcine reproductive and respiratory syndrome in China [J].Virus Res,2010,154:31-37.
[6]马静云,李浩波,王玲玲,等.高致病性PRRSV的分离及其Nsp2和GP5基因的序列分析 [J].中国兽医科学,2008,38(8):650-657.
Ma J Y,Li H B,Wang L L,et al.Isolation of highly pathogenic porcine reproductive and respiratory syndrome virus and sequence analysis of itsNsp2 andORF5 genes [J].Chinese Veterinary Science,2008,38(8):650-657.
[7]Gao Z,Guo X,Yang H.Genomic characterization of two Chinese isolates of porcine respiratory and reproductive syndrome virus [J].Archives of Virology,2004,149(7):1341-1351.
[8]Yin G,Gao L,Shu X,et al.Genetic diversity of the GP5 gene of porcine reproductive and respiratory syndrome virus isolates in southwest China from 2007 to 2009 [J].PLoS One,2012,7(3):33756.
[9]Zhou L,Zhang J I,Zeng J W,et al.The 30-amino-acid deletion in the Nsp2 of highly pathogenic porcine reproductive and respiratory syndrome virus emerging in China is not related to its virulence [J].J Virol,2009,83:5156-5167.
[10]冷 雪,李真光,王凤雪,等.高致病性猪繁殖与呼吸综合征病毒VM1株全基因测序及致病性分析 [J].中国兽医学报,2012,32(12):1752-1757.
Leng X,Li Z G,Wang F X,et al.Genomic sequencing and pathogenicity analysis of highly pathogenic isolate NM1 of porcine reproductive and respiratory syndrome virus [J].China J Vet Sci,2012,32(12):1752-1757.
[11]Tong G Z,Zhou Y J,Hao X F,et al.Highly pathogenic porcine reproductive and respiratory syndrome,China [J].Emerging Infectious Diseases,2007,13(9):1434-1436.
[12]韦祖樟,孙 志,袁世山.猪繁殖与呼吸综合症病毒分子生物学研究进展 [J].微生物学通报,2008,35(3):408-413.
Wei Z Z,Sun Z,Yuan S S.Current advances on molecular biology of porcine reproductive and respiratory syndrome virus [J].Microbiology,2008,35(3):408-413.
[13]张民泽,谢杰雄,郭泗虎,等.PRRSV XH-GD株Nsp2部分缺失感染性克隆的构建 [J].中国预防兽医学报,2013,35(1):10-14.
Zhang M Z,Xie J X,Guo S H,et al.Construction and identification of infectious clone of partial deletion in Nsp2 for porcine reproductive and respiratory syndrome virus XH-GD strain [J].Chinese Journal of Preventive Veterinary Medicine,2013,35(1):10-14.
[14]Tjeerd G K,Lisette A C,Rob J M,et al.Challenges for porcine reproductive and respiratory syndrome virus(PRRSV) vaccinology [J].Vaccine,2009,27(28):3704-3718.
[15]Susan I,Brockmeier,Crystal I,et al.Uenomic sequence and virulence comparison of four type 2 porcine reproductive and respiratory syndrome virus strains [J].Virus Res,2012,169:212-221.
[16]Liu D, Zhou R,Zhang J L,et al.Recombination analyses between two strains of porcine reproductive and respiratory syndrome virusinvivo[J].Virus Research,2011,155(2):473-486.
[17]Allende R,Iewis T I,Lu Z,et al.North American and European porcine reproductive and respiratory syndrome viruses differ in non-structural protein coding regions [J].J Gen Virol,1999,80(2):307-315.
[18]Ostrowski M,Galeota J A,Jar A M,et al.Identification of neutralizing and no neutralizing epitopes in the porcine reproductive and respiratory syndrome virus GP5 ectodomain [J].J Virol,2002,76(9):4241-4250.
Variation of Nsp2 and GP5 genes of porcine reproductive and respiratory syndrome virus in Shaanxi and neighboring provinces
WU Xujin,ZHU Xiaofu
(Animal Epidemic Disease Diagnostic Laboratory of Molecular Biology,Institute of Animal Husbandry and Veterinary Medicine,XianyangVocationalTechnicalCollege,Xianyang,Shaanxi712000,China)
【Objective】 This study aimed to understand the variation of reproductive and respiratory syndrome virus (PRRSV)Nsp2 andGP5 genes in Shaanxi and neighboring provinces. 【Method】Nsp2 andGP5 genes were cloned and sequenced from liver,spleen,lung,and serum by RT-PCR method.Sequences of 30 PRRSV
trains were obtained in GenBank and compared with prevalent strains,and phylogenetic trees were constructed.【Result】 A total of 13 PRRSVNsp2 gene sequences (GenBank Accession No.KM233849-KM233861) and 12GP5 gene sequences (GenBank Accession No.KM233862-KM233873) were obtained.Phylogenetic trees showed that all prevalent PRRSV strains belonged to North American-type.Nsp2 gene sequence analysis revealed that 12 prevalent PRRSV strains were gene deletion strain and 1 was classical strain.GSXF may be a recombinant strain.Compared with classical strains,GP5 protein variants existed more mutations at amino acid points.【Conclusion】 Mutant PRRSV strains were dominant in Shaanxi farms,and the existence of classical strains and recombinant strains was confirmed.GP5 protein amino acid point mutations occurred with high frequency.
porcine reproductive and respiratory syndrome virus;Nsp2 gene;GP5 gene;Shaanxi and neighboring provinces
时间:2016-10-20 16:36
10.13207/j.cnki.jnwafu.2016.12.001
2015-06-15
咸阳市科学技术研究发展计划项目(2014K02-21);咸阳职业技术学院重点科研项目(2013KYA01)
吴旭锦(1979-),女,陕西西安人,副教授,博士,主要从事动物疫病分子病原学与免疫学研究。 E-mail:yaoyuanwxj@163.com
S852.65+1
A
1671-9387(2016)12-0001-08
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20161020.1636.002.html

