牛MYOZ1基因启动子活性分析
王明明,李安宁,赵志东,张亚冉,段美艳,王亚宁,李世军,昝林森,2
(1 西北农林科技大学 动物科技学院,陕西 杨凌 712100;2 国家肉牛改良中心,陕西 杨凌 712100)
牛MYOZ1基因启动子活性分析
王明明1,李安宁1,赵志东1,张亚冉1,段美艳1,王亚宁1,李世军1,昝林森1,2
(1 西北农林科技大学 动物科技学院,陕西 杨凌 712100;2 国家肉牛改良中心,陕西 杨凌 712100)
【目的】 鉴定牛MYOZ1基因转录起始位点,确定牛MYOZ1基因核心启动子区域,为进一步研究牛MYOZ1基因的转录调控机制奠定基础。【方法】 以秦川牛肌肉5′ RACE准备cDNA为模板,设计5′ RACE扩增试验,确定牛MYOZ1基因转录起始位点。以秦川牛外周血基因组DNA为模板,通过PCR克隆获得牛MYOZ1基因转录调控区-1 628/+61目的片段。通过生物信息学分析软件预测可能包含的转录因子结合位点,设计逐段缺失引物,获得7个亚克隆,将其分别与pGL3-Basic载体连接,得到牛MYOZ1基因启动子双荧光素酶报告基因重组质粒,通过脂质体法转染C2C12细胞系,检测7个重组质粒的荧光素酶活性,分析启动子活性。【结果】 确定了牛MYOZ1基因的转录起始位点,成功克隆获得7个系列缺失的牛MYOZ1基因启动子双荧光素酶报告基因重组质粒:pMYOZ1-1 628/+61、pMYOZ1-1 430/+61、pMYOZ1-1 179/+61、pMYOZ1-932/+61、pMYOZ1-676/+61、pMYOZ1-437/+61和pMYOZ1-116/+61,其中重组质粒pMYOZ1-116/+61启动子活性极显著高于pGL3-Basic,推测牛MYOZ1基因-116/+61区域可能包含核心启动子;重组质粒pMYOZ1-1 628/+61启动子活性极显著高于pMYOZ1-1 430/+61片段活性 (P<0.01) ,表明牛MYOZ1基因启动子区域-1 628/-1 430片段可能包含启动子活性增强元件。生物信息学分析发现,牛MYOZ1基因启动子-116/+61片段可能包含SP1、GC Box、CAAT等多个重要转录因子结合位点;-1 628/-1 430片段可能包含SP1、MyoD等多个重要转录因子结合位点。【结论】 成功构建了7个系列缺失的牛MYOZ1基因启动子双荧光素酶报告基因重组质粒,且初步确定了牛MYOZ1基因的核心启动子区域位于-116/+61。
牛;MYOZ1;5′ RACE;启动子;双荧光素酶报告载体
嫩度是评价肉品质的重要指标。肌肉嫩度主要受肌纤维类型影响,与肌肉中氧化型慢肌纤维含量呈正相关[1-2]。研究发现,肌原调节蛋白1(Myozenin1,MYOZ1)基因可以促进快肌纤维向慢肌纤维的转化[3]。
MYOZ1蛋白参与肌纤维Z线形成以及快肌纤维相关生理过程[4],而肌纤维Z线在肌小节组装和不同类型肌纤维形成等过程中起着广泛的调控作用[5]。当小鼠的MYOZ1基因被敲除后,肌小节Z线上无MYOZ1蛋白结合,编码慢肌纤维的基因开始转录和翻译,导致肌肉中慢肌纤维的比例增加[6]。另外,MYOZ1基因的表达可能与动物初生体质量相关。研究表明,绵羊胚胎发育后期,MYOZ1基因的表达可能与胎羊初生体质量呈正相关[7]。目前,猪MYOZ1基因核心启动子已经开始被应用于转基因技术。过氧化物酶体增殖物激活受体γ2(Peroxisome proliferator-activated receptor γ2,PPAR-γ2)是调控脂肪细胞分化及其摄取和贮存脂肪酸的关键因子。在转猪PPAR-γ2基因小鼠体内,可以通过在猪PPAR-γ2基因调控区插入猪MYOZ1基因核心启动子来上调PPAR-γ2基因在RNA和蛋白水平上的表达[8]。这表明可以通过转基因方法利用猪MYOZ1基因核心启动子改善猪肉品质。
综上所述,MYOZ1基因在骨骼肌生长发育、肌纤维类型调控以及动物的形体和肉质研究方面均具有重要价值。基因的表达受启动子调控,目前对于MYOZ1基因启动子的研究主要集中在猪[9]和小鼠[10]上,在牛上尚未见相关研究报道,而牛MYOZ1基因与小鼠和猪的MYOZ1基因差异较大,因此对牛MYOZ1基因启动子的研究具有重要的现实意义。本试验以秦川肉牛为研究对象,对其MYOZ1基因的转录起始位点和启动子核心区进行了研究,以期为进一步探讨牛MYOZ1基因的转录调控奠定基础。
1 材料与方法
1.1材料
双荧光素酶报告基因载体pGL3-Basic(内含萤火虫荧光素酶基因)、pGL3-Control及其内参质粒pRL-TK(含海肾荧光素酶基因)、DNA胶快速回收试剂盒、质粒提取试剂盒,均购自Promega公司;大肠杆菌感受态细胞DH5α,购自北京全式金生物公司;PMD19-T(simple)载体、BglⅡ、HindⅢ限制性内切酶,购自TaKaRa公司;T4 DNA连接酶,购自Neb公司;SMARTTMRACE Kit试剂盒,购自Clonetech公司;PBS、DMEM培养基及胎牛血清,购自Hyclone公司;LipofectamineTMReagent 2000脂质体转染试剂盒,购自Invitrogen公司;无血清OPTI-MEM培养基,购自GIBCO公司。秦川牛外周血基因组DNA及其肌肉组织 5′ RACE 准备cDNA、小鼠成肌细胞系(C2C12细胞系),均由西北农林科技大学国家肉牛改良中心保存。
1.2MYOZ1系统进化树的构建
在NCBI上搜索牛(登录号:AAI22690.1)、人(登录号:NP_067068.1)、小鼠(登录号:NP_067483.1)、猪(登录号:NP_001020393.1)、绵羊(登录号:XP_004021522.1)、山羊(登录号:XP_005699272.1)的MYOZ1氨基酸序列,应用SECentral软件比对不同物种间MYOZ1氨基酸序列的相似性,用MEAG 6.0软件构建系统进化树。
1.3牛MYOZ1基因转录起始位点的确定
以秦川牛肌肉组织5′RACE准备cDNA为模板,采用软件Primer 5.0设计5′ RACE特异引物MYOZ1-GSP1和MYOZ1-GSP2,交由南京金斯瑞公司合成,其相关信息见表1。用特异引物MYOZ1-GSP1、MYOZ1-GSP2及SMARTTMRACE Kit试剂盒提供的通用引物Universal Primer A Mix (UPM),根据试剂盒提供的方法,进行5′ RACE PCR扩增。降落PCR第1轮扩增程序为:94 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,70 ℃ 30 s,72 ℃ 3 min,5个循环;94 ℃ 30 s,68 ℃ 30 s,72 ℃ 3 min,30个循环。将第1轮扩增产物用ddH2O稀释20倍,以之为模板进行第2轮扩增,反应程序为:95 ℃ 5 min;94 ℃ 30 s,62 ℃ 30 s,72 ℃ 130 s,35个循环;72 ℃ 10 min。PCR产物用1.2%的琼脂糖凝胶电泳检测。将第2轮PCR产物连接PMD19-T(simple)载体,挑取10个单克隆送南京金斯瑞公司测序,分析确定牛MYOZ1基因转录起始位点。

表 1 试验所用的PCR引物Table 1 PCR primers used in the experiment
注:MYOZ1-F1~MYOZ1-F7序列中斜体部分为BglⅡ酶切位点,MYOZ1-R序列中斜体部分为Hind Ⅲ酶切位点。
Note:Italicized letters in MYOZ1-F1-MYOZ1-F7 presentedBglⅡ binding sites,in MYOZ1-R presentedHind Ⅲ binding sites.
1.4牛MYOZ1基因启动子及其7个逐段缺失片段的PCR扩增
1.4.1启动子5′侧翼区-1 628/+61序列克隆根据GenBank中牛MYOZ1基因上游启动区约2 kb DNA序列(GenBank登录号为:281939),应用引物设计软件Primer 5.0设计1对用于扩增牛MYOZ1基因启动子区-1 628/+61的引物M1qdzF和M1qdzR,其信息见表1,引物由南京金斯瑞公司合成。
以牛外周血基因组DNA为模板,用引物M1qdzF和M1qdzR PCR扩增牛MYOZ1基因启动子5′侧翼区-1 628/+61序列,反应体系:10×buffer 2 μL,dNTP 2 μL,MgSO41 μL,引物M1qdzF 0.6 μL,引物M1qdzR 0.6 μL,模板DNA 1.2 μL,ddH2O 12.2 μL,KOD 0.4 μL,共20 μL。PCR扩增程序为:95 ℃ 5 min;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 130 s,35个循环;72 ℃ 10 min。将扩增产物用1%琼脂糖凝胶电泳检测,切胶回收纯化后,与PMD19-T(simple) 于16 ℃连接过夜,连接产物转化感受态细胞DH5α,涂布于含氨苄青霉素的LB固体培养基上,37 ℃过夜培养。挑选阳性单克隆菌落,摇菌12 h,进行菌液PCR检测后,送南京金斯瑞公司测序。
1.4.2启动子7个逐段缺失片段的PCR扩增通过GenBank检索并下载牛MYOZ1基因上游启动区2 kb DNA序列,应用在线软件Genomatix(http://www.genomatix.de/cgibin//matinspector)、TFSEARCH(http://mbs.cbrc.jp/research/db/TFSEARCH.html)预测牛MYOZ1基因启动子区域可能的转录因子结合位点。根据软件预测的结合位点,设计7条逐段缺失启动子序列的5′端包含BglⅡ酶切位点(AGATCT)的上游引物MYOZ1-F1、MYOZ1-F2、MYOZ1-F3、MYOZ1-F4、MYOZ1-F5、MYOZ1-F6、MYOZ1-F7和1条5′端包含Hind Ⅲ酶切位点(AAGCTT)的下游引物MYOZ1-R,引物相关信息见表1,由南京金斯瑞公司合成。
以已转化连接产物的感受态细胞DH5α菌液为模板,分别用上游引物MYOZ1-F1、MYOZ1-F2、MYOZ1-F3、MYOZ1-F4、MYOZ1-F5、MYOZ1-F6、MYOZ1-F7和下游引物MYOZ1-R进行PCR反应。PCR反应体系:10×buffer 2 μL,dNTP 2 μL,MgSO41.2 μL,上游引物 0.6 μL,下游引物 0.6 μL,模板DNA 1.2 μL,ddH2O 12 μL,KOD 0.4 μL,共20 μL。PCR扩增程序为:95 ℃ 5 min;94 ℃ 30 s,65 ℃ 30 s,72 ℃ 130 s,35个循环;72 ℃ 10 min。将PCR产物用1%琼脂糖电泳检测,胶回收后,送南京金斯瑞公司测序。
1.5双荧光素酶报告基因载体的构建
将纯化的7个牛MYOZ1基因逐段缺失启动子目的片段和pGL3-Basic载体,分别用HindⅢ和BglⅡ于37 ℃双酶切3 h,琼脂糖凝胶电泳检测,并切胶回收目的片段。将回收的7个牛MYOZ1基因逐段缺失片段分别与pGL3-Basic载体用T4 DNA连接酶连接,16 ℃恒温仪上孵育2 h。将连接产物转化感受态细胞DH5α,涂布在含氨苄青霉素的LB固体培养基(氨苄质量浓度为100 mg/mL)上,37 ℃过夜培养,挑选阳性单克隆菌落,37 ℃下200 r/min摇菌培养20 h。取菌液提取重组质粒,进行HindⅢ和BglⅡ双酶切鉴定(同时设pGL3-Basic双酶切对照),送南京金斯瑞公司测序。
1.6C2C12细胞的培养和转染
用含体积分数10%FBS的DMEM培养基培养C2C12细胞系,在转染前一天将生长状态良好的C2C12细胞系按每孔1×105个细胞的量接种至24孔板,每孔加入500 μL不含抗生素的细胞培养基,设3个复孔,置于37 ℃、体积分数5% CO2培养箱中培养,待细胞单层长至70%~80%融合时进行质粒转染。
为研究牛MYOZ1基因启动子不同区域的启动活性,将1.5节构建好的7个重组质粒各800 ng分别转染C2C12细胞,同时转入内参质粒pRL-TK 10 ng,按照LipofectamineTMReagent 2000脂质体转染试剂盒说明进行试验操作。以转染pGL3-Basic质粒的C2C12细胞为阴性对照,以转染pGL3-Control质粒的C2C12细胞为阳性对照,转染24 h后收集细胞。每组试验重复3次。
1.7启动子相对活性的测定
按照Promega公司双荧光检测试剂盒说明书,用酶标仪检测萤火虫荧光素酶活性(F值)与海肾荧光素酶活性(R值),计算其比值来反映不同启动子片段的相对活性。每次转染均设pGL3-Basic阴性对照组。应用SPSS 16.0软件进行单因素方差分析,确定影响启动子活性变化的片段区域,设定显著性水平为P=0.05,极显著水平为P=0.01。
2 结果与分析
2.1不同物种MYOZ1氨基酸序列的比对
牛和绵羊的MYOZ1包含306个氨基酸,山羊、人、小鼠、猪分别包含302,299,296和298个氨基酸。用SECentral软件比对上述物种间MYOZ1基因编码的氨基酸序列,结果显示,牛MYOZ1基因编码的氨基酸序列与山羊、绵羊的相似性都高达96%,与猪的相似性为91%,与人的相似性为89%,与小鼠的相似性为85%。系统进化分析结果(图1)表明,牛与山羊、绵羊的亲缘关系较近,与猪、人、小鼠的亲缘关系较远。
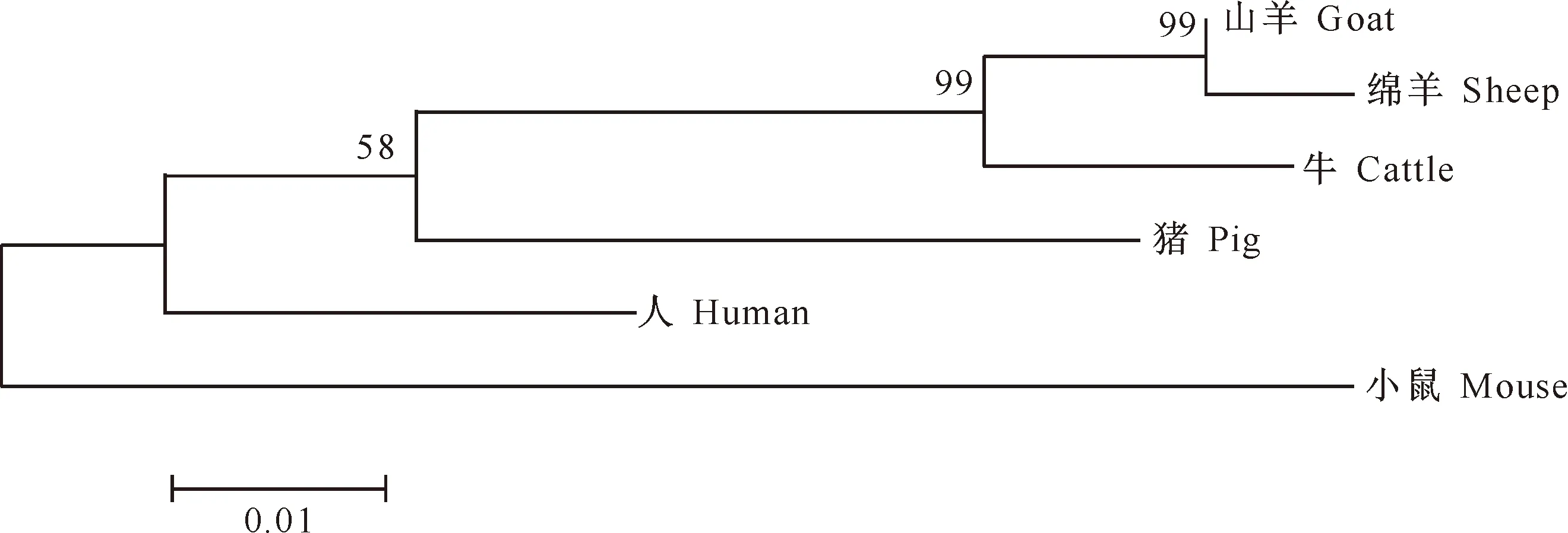
图 1不同物种MYOZ1蛋白氨基酸序列系统进化树
Fig.1Amino acid phylogram tree of MYOZ1 in different species
2.2牛MYOZ1基因转录起始位点的确定
以秦川肉牛肌肉组织5′ RACE cDNA为模板,用5′ RACE准备特异引物MYOZ1-GSP1与通用引物UPM进行第1轮5′ RACE降落PCR扩增,通过1.2%的琼脂糖凝胶电泳检测,结果(图2)显示条带并不单一。将第1轮PCR产物用ddH2O稀释20倍后作模板,用引物MYOZ1-GSP2与通用引物UPM进行第2轮5′ RACE PCR扩增,结果获得一条长度约250 bp的DNA片段(图2)。将目的片段MYOZ1连接PMD19-T(simple)载体,测序证实目的片段实际大小为274 bp;经序列比对发现,牛MYOZ1基因的转录起始位点为碱基C(图3)。
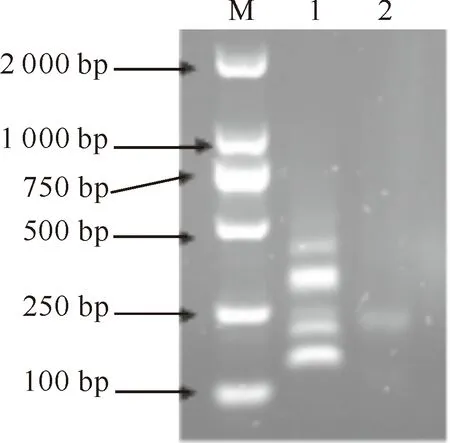
图 2牛MYOZ1基因5′ RACE的扩增
M.DL2000 DNA Marker;1.5′ RACE第1轮PCR产物;
2.5′ RACE第2轮PCR产物
Fig.2Amplification of 5′ RACE for bovineMYOZ1 gene
M.DL2000 DNA Marker;1.The first 5′ RACE PCR result;
2.The second 5′ RACE PCR result

图 3牛MYOZ1基因转录起始位点
下划线表示5′ RACE试验确定的牛MYOZ1基因转录起始位点;大写字母代表外显子;小写字母代表启动子和内含子;方框表示翻译起始位点
Fig.3Identification of bovineMYOZ1 transcription initiation sites by 5′ RACE Letters underline indicate transcription start site of cattleMYOZ1 gene by 5′ RACE.
Capital letters stand for exons.Lowercase letters stand for introns and promoter.Letters in box present translation initiation site
2.3牛MYOZ1基因启动子及其逐段缺失片段的PCR扩增
PCR扩增牛MYOZ1基因5′侧翼区-1 628/+61片段,结果(图4)获得了长度为1 689 bp的片段,与预期结果一致,测序结果证实克隆得到了目的片段(图5)。
以牛MYOZ1基因5′侧翼区-1 628/+61片段为基础,用逐段缺失5′调控区引物分别进行PCR亚克隆,结果得到177,498,737,993,1 240,1 491和 1 689 bp共7个逐段缺失的5′调控区片段(图6),测序结果证实得到了-116/+61,-437/+61,-676/+61,-932/+61,-1 179/+61,-1 430/+61,-1 628/+61共7个亚克隆片段。

图 4牛MYOZ1基因5′侧翼区PCR扩增
M.DL2000 DNA Marker;1.5′侧翼区-1 628/+61片段
Fig.4PCR result of 5′ flank region in bovineMYOZ1
M.DL2000 DNA Marker;1.5′ flank area

图 5牛MYOZ1基因5′侧翼区-1 628/+61序列
下划线表示设计的逐段缺失引物的结合位点,大写字母表示5′ UTR区碱基序列
Fig.5Sequence of bovineMYOZ1 promoter
Letters underline present the sub-clones primer binding sites.Capital letters present 5′ UTR region sequence

图 67个牛MYOZ1基因启动子逐段缺失片段的PCR扩增
M.DL2000 DNA Marker;1~7.牛MYOZ1基因启动子
逐段缺失片段,分别为 -116/+61,-437/+61,-676/+61,932/+61,-1 179/+61,-1 430/+61,-1 628/+61
Fig.67 sub-clones of bovineMYOZ1 promoter
M.DL2000 DNA Marker;1-7.The 7 sub-clones of bovine
MYOZ1 promoter are -116/+61,-437/+61,-676/+61,-932/+61,-1 179/+61,-1 430/+61,and -1 628/+61,respectively
2.4MYOZ1双荧光素酶报告基因载体的鉴定
将7个牛MYOZ1基因启动子逐段缺失片段PCR扩增产物连接pGL3-Basic载体构建重组质粒,按照扩增片段在牛MYOZ1基因中的位置, 将重组质粒分别命名为pMYOZ1-1 628/+61、pMYOZ1-1 430/+61、pMYOZ1-1 179/+61、pMYOZ1-932/+61、pMYOZ1-676/+61、pMYOZ1-437/+61和pMYOZ1-116/+61,其经Hind Ⅲ和BglⅡ双酶切分别得到1 689,1 491,1 240,993,737,498和177 bp片段(图7)。测序结果证实,牛MYOZ1基因启动子双荧光素酶报告基因载体构建成功。

图 7牛MYOZ1双荧光素酶报告基因载体的
HindⅢ和BglⅡ双酶切鉴定
M.DL2000 DNA Marker;P.pGL3-Basic双酶切对照;1~7.分别为
重组质粒pMYOZ1-1 628/+61、pMYOZ1-1 430/+61、pMYOZ1-1 179/+61、pMYOZ1-932/+61、pMYOZ1-676/+61、pMYOZ1-437/+61、pMYOZ1-116/+61的双酶切鉴定结果
Fig.7Hind Ⅲ andBglⅡ enzyme identification of dual-luciferase reporter gene vectors
M.DL2000 DNA Marker;P.pGL3-Basic digested byHindⅢ and
BglⅡ;1-7.The recombinant plasmid pMYOZ1-1 628/+61,pMYOZ1-1 430/+61,pMYOZ1-1 179/+61,pMYOZ1-932/+61,pMYOZ1-676/+61,pMYOZ1-437/+61,pMYOZ1-116/+61 digested byHindⅢ andBglⅡ
2.5牛MYOZ1启动子的活性分析
牛MYOZ1基因7个逐段缺失片段重组质粒pMYOZ1-1 628/+61、pMYOZ1-1 430/+61、pMYOZ1-1 179/+61、pMYOZ1-932/+61、pMYOZ1-676/+61、pMYOZ1-437/+61和pMYOZ1-116/+61启动子活性测定结果见图8。图8显示,7个重组质粒的启动子活性均极显著高于pGL3-Basic阴性对照(P<0.01),均极显著低于pGL3-Control阳性对照(荧光素酶相对活性极高,图8中未显示)。重组质粒pMYOZ1-116/+61的启动子活性极显著高于pGL3-Basic阴性对照(P<0.01),说明该片段上游序列的缺失并没有引起启动子活性的显著变化,推测牛MYOZ1基因-116/+61区域很可能包含核心启动子。重组质粒pMYOZ1-1 628/+61启动子活性极显著高于pMYOZ1-1 430/+61 片段活性(P<0.01),表明牛MYOZ1基因启动子区域-1 628/-1 430片段可能包含启动子活性增强元件。
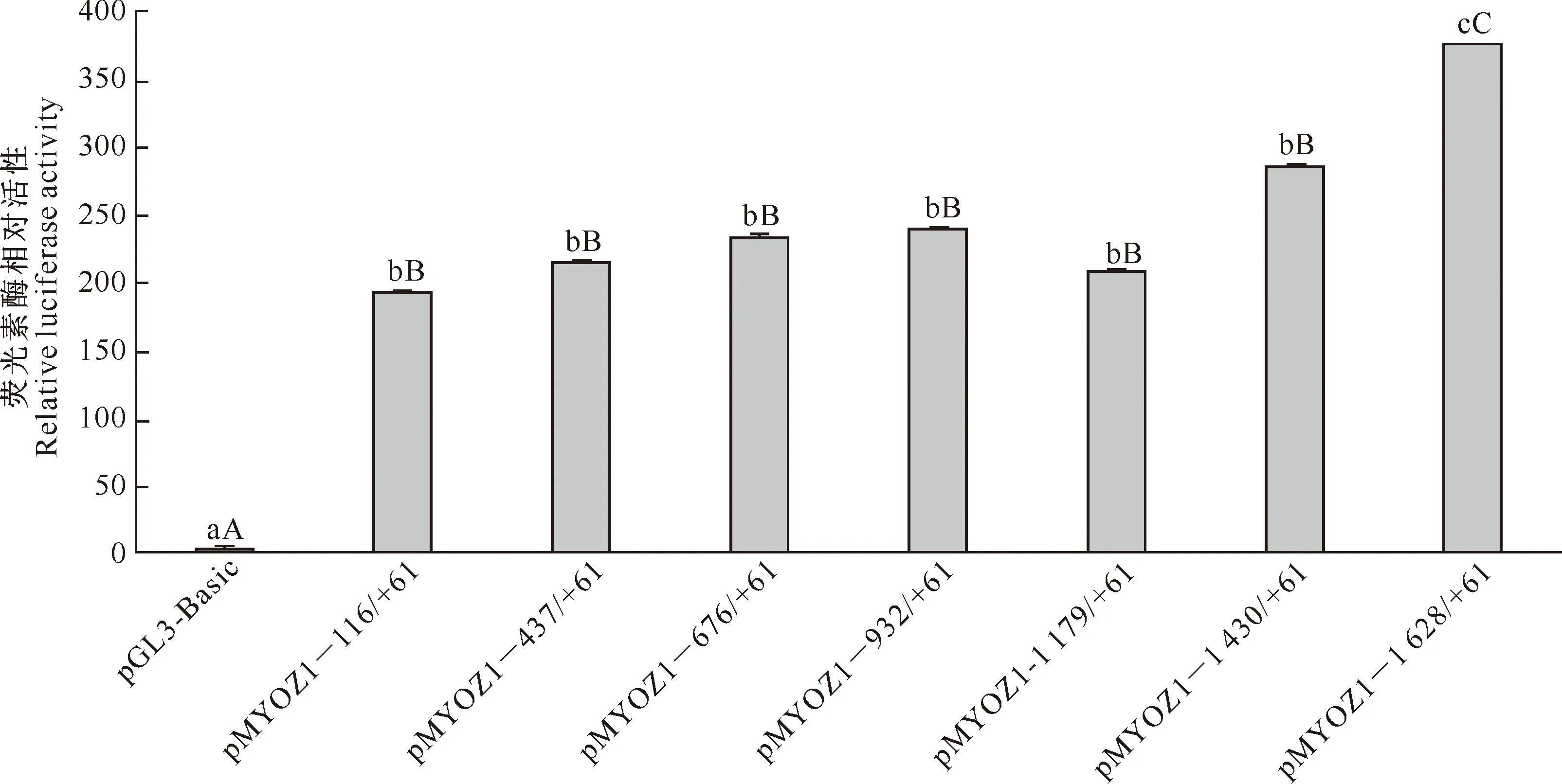
图 8牛MYOZ1 5′调控序列缺失片段在C2C12细胞中的启动子活性
图柱上标不同小写字母表示差异显著(P<0.05),标不同大写字母表示差异极显著(P<0.01)
Fig.8Promoter activities of bovineMYOZ1 5′ deleted regulatory regions in C2C12 cells
Different lowercase letters mean significant difference (P<0.05) and different uppercase letters mean extremely significant difference (P<0.01)
2.6牛MYOZ1基因启动子序列的生物信息学分析
经Genomatix、TFSEARCH软件预测发现,牛MYOZ1基因启动子区-116/+61片段可能包含SP1、GC Box、CAAT等多个重要转录因子结合位点(图9);牛MYOZ1基因启动子区-1 628/-1 430片段可能包含SRF、MyoD等多个重要转录因子结合位点(图10)。
3 讨 论
通过SECentral软件比对不同物种间MYOZ1基因编码的氨基酸序列,结果显示,牛MYOZ1基因编码的氨基酸序列与山羊、绵羊的相似性为96%,与猪、人和小鼠的相似性相对较低。通过构建系统进化树可以看出,牛与山羊和绵羊的亲缘关系较近,与人、猪和小鼠的亲缘关系相对较远。笔者推测,MYOZ1基因可能在反刍动物中相对保守,与单胃动物差异较大。
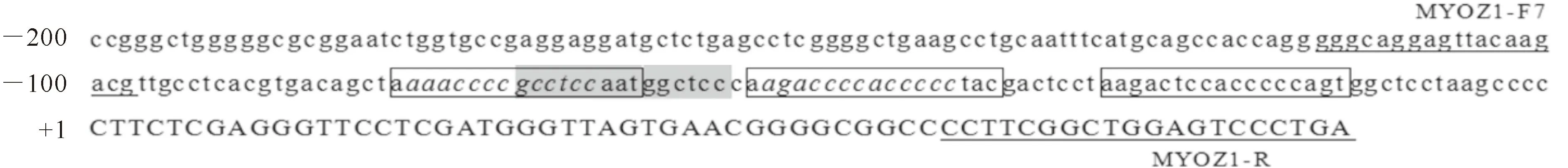
图 9牛MYOZ1基因启动子区-116/+61片段转录因子结合位点的预测
下划线表示引物结合位点,斜体字母表示GC Box区,阴影区域表示CAAT区,方框表示转录因子结合位点;下图同
Fig.9Prediction of transcription factor binding sites in the -116/+61 region of bovineMYOZ1 gene promoter
Letters underline present primer binding sites.Italicized letters present GC Box.Letters in grey areas present CAAT district.Letters in box present transcription factor binding sites.The same below

图 10牛MYOZ1基因启动子区-1 628/-1 430片段转录因子结合位点的预测
Fig.10Prediction of transcription factor binding sites in the -1 628/-1 430 region of bovineMYOZ1 gene promoter
本研究发现,构建的7个报告基因重组质粒转染组较pGL3-Basic转染组的荧光素酶活性显著提高,且差异极显著(P<0.01),说明克隆得到的各片段均包含转录因子结合位点。其中,牛MYOZ1基因片段-116/+61的启动子活性是pGL3-Basic的42倍,说明牛MYOZ1基因启动子区-116/+61可能包含某些重要的关键转录因子结合位点。生物信息学分析发现,牛MYOZ1基因启动子区片段-116/+61可能包含SP1、GC Box、CAAT等多个重要转录因子结合位点,其中SP1具有多种生理活性[11-17]。
本研究还发现,pMYOZ1-1 628/+61的荧光素酶活性与pMYOZ1-1 430/+61相比极显著提高(P<0.01),表明牛MYOZ1基因片段-1 628/-1 430可能包含启动子活性增强元件。生物信息学分析发现,该片段可能包含SRF、MyoD等多个重要转录因子结合位点,其中MyoD具有多种生理活性[18-20]。
本研究显示,牛MYOZ1基因片段-116/+61的启动子活性是pGL3-Basic的42倍。而尚杨杨[9]研究表明,猪MYOZ1基因缺失片段活性与pGL3-Basic相差不大。除活性外,由于不同物种间基因保守性的不同,MYOZ1基因的核心启动子区域可能也存在差异。如王恒[10]研究发现,小鼠MYOZ1基因-561/-185片段为核心启动子区域,用TESS预测,在MYOZ1基因启动子区域-561/-185存在有NF-κB因子的结合位点。而本研究结果表明,MYOZ1基因核心启动子区域-116/+61可能包含SP1、GC Box、CAAT等多个重要转录因子结合位点。据此,笔者推测牛MYOZ1启动子的活性很可能受转录因子SP1的调节。为了探究本试验所预测到的秦川牛MYOZ1基因启动子区转录因子对MYOZ1及其下游基因的转录调控作用,还需要通过定点突变、EMSA、ChIP等试验进行深入研究。
本研究初步确定了与牛MYOZ1转录相关的关键转录因子可能位于-116/+61之间。此结果有助于进一步分析并确定发挥重要作用的顺式作用元件及其所在位置,为牛MYOZ1基因的转录调控机制研究奠定了基础。
[1]刘居鹏,单安山,彭济昌.猪肌纤维发育及其与肉质的关系 [J].中国饲料,2006,4(16):11-14.
Liu J P,Shan A S,Peng J C.Development of pig muscle fiber and the relationship with the meat quality [J].China Feed,2006,4(16):11-14.
[2]陈代文,张克英,胡祖禹. 猪肉品质特征的形成原理 [J].四川农业大学学报,2002,15(1):60-66.
Chen D W,Zhang K Y,Hu Z Y.The forming principle of pork quality traits [J].Journal of Sichuan Agricultural University,2002,15(1):60-66.
[3]Frey N,Frank D,Lippl S,et al.Calsarcin-2 deficiency increases exercise capacity in mice through calcineurin/NFAT activation [J].J Clin Invest,2008,118(11):3598-3608.
[4]von Nandelstadh P,Ismail M,Gardin C,et al.A class Ⅲ PDZ binding motif in the myotilin and FATZ families binds enigma family proteins:a common link for Z-disc myopathies [J].Mol Cell Biol,2009,29(3):822-834.
[5]Luther P K.The vertebrate muscle Z-disc:sarcomere anchor for structure and signalling [J].J Muscle Res Cell Motil,2009,30(5/6):171-185.
[6]Frey N,Frank D,Lippl S,et al.Calsarcin-2 deficiency increases exercise capacity in mice through calcineurin/NFAT activation [J].J Clin Invest,2008,118(11):3598-3608.
[7]Xu L,Zhao F,Ren H,et al.Co-expression analysis of fetal weight-related genes in ovine skeletal muscle during mid and late fetal development stages [J].Int J Biol Sci,2014,10(9):1039-1050.
[8]Ma J,Chai J,Shang Y,et al.Swine PPAR-gamma2 expression upregulated in skeletal muscle of transgenic mice via the swine Myozenin-1 gene promoter [J].Transgenic Res,2014,24(3):409-420.
[9]尚杨杨.猪骨骼肌特异性表达基因myoz1/tnnc2启动子的克隆及功能验证 [D].武汉:华中农业大学,2013.
Shang Y Y.Cloning and function analysis of porcine skeletal muscle-specific gene myoz1/tnnc2 promoter [D].Wuhan:Huazhong Agricultural University,2013.
[10]王恒.肌肉中calsarcin基因的差异表达和转录调控研究 [D].武汉:华中农业大学,2006.
Wang H.Characterization and transcriptional regulation analysis of calsarcin gene in muscle [D].Wuhan:Huazhong Agricultural University,2006.
[11]Bessereau J L,Mendelzon D,Lepoupon C,et al.Muscle-specific expression of the acetylcholine receptor alpha-subunit gene requires both positive and negative interactions between myogenic factors,Sp1 and GBF factors [J].EMBO J,1993,12(2):443-449.
[12]Sartorelli V,Webster K A,Kedes L.Muscle-specific expression of the cardiac alpha-actin gene requires MyoD1,CArG-box binding factor,and Sp1 [J].Genes Dev,1990,4(10):1811-1822.
[13]Lin H,Yutzey K E,Konieczny S F.Muscle-specific expression of the troponin Ⅰ gene requires interactions between helix-loop-helix muscle regulatory factors and ubiquitous transcription factors [J].Mol Cell Biol,1991,11(1):267-280.
[14]Zhang L,Jin M,Margariti A,et al.Sp1-dependent activation of HDAC7 is required for platelet-derived growth factor-BB-induced smooth muscle cell differentiation from stem cells [J].J Biol Chem,2010,285(49):38463-38472.
[15]Sorianello E,Soriano F X,Fernandez-Pascual S,et al.The pr-omoter activity of human Mfn2 depends on Sp1 in vascular smooth muscle cells [J].Cardiovasc Res,2012,94(1):38-47.
[16]Yuan J,Tang Z,Yang S,et al.CRABP2 promotes myoblast di-fferentiation and is modulated by the transcription factors MyoD and Sp1 in C2C12 cells [J].PLoS One,2013,8(1):e55479.
[17]Parakati R,Dimario J X.Repression of myoblast proliferation and fibroblast growth factor receptor 1 promoter activity by KLF10 protein [J].J Biol Chem,2013,288(19):13876-13884.
[18]Weintraub H,Tapscott S J,Davis R L,et al.Activation of mu-scle-specific genes in pigment,nerve,fat,liver,and fibroblast cell lines by forced expression of MyoD [J].Proc Natl Acad Sci USA,1989,86(14):5434-5438.
[19]Kocaefe Y C,Israeli D,Ozguc M,et al.Myogenic program induction in mature fat tissue (with MyoD expression) [J].Exp Cell Res,2005,308(2):300-308.
[20]Zhang X,Chai J,Azhar G,et al.Early postnatal cardiac changes and premature death in transgenic mice overexpressing a mutant form of serum response factor [J].J Biol Chem,2001,276(43):40033-40040.
Activity of bovineMYOZ1 gene promoter
WANG Mingming1,LI Anning1,ZHAO Zhidong1,ZHANG Yaran1,DUAN Meiyan1,WANG Yaning1,LI Shijun1,ZAN Linsen1,2
(1CollegeofAnimalScienceandTechnology,NorthwestA&FUniversity,Yangling,Shaanxi712100,China;2NationalBeefCattleImprovementCenterinChina,Yangling,Shaanxi712100,China)
【Objective】 The research determined the transcription initiation site and core promoter region of bovineMYOZ1 gene by detecting the activities ofMYOZ1 promoter constructs in C2C12 cell lines to lay foundation for further research on transcriptional regulation of bovineMYOZ1 gene.【Method】 The 5′ Rapid amplification of cDNA ends (5′ RACE) was used to determine the transcription initiation site ofMYOZ1 gene,and the 5′ flanking region (-1 628/+61) of bovineMYOZ1 gene was obtained by PCR.By bioinformatics prediction of transcription factor binding sites of bovineMYOZ1 gene promoter,7 sub-clones were amplified by PCR before being inserted into pGL3-Basic vectors.The recombinant plasmids were transfected into the C2C12 cells,and the relative transcriptional activities were measured using Dual-Luciferase Reporter Assay System. 【Result】 The translational start site was determined,and seven recombinant plasmids were constructed successfully by restriction enzyme digestion and sequence analysis including pMYOZ1-1 628/+61,pMYOZ1-1 430/+61,pMYOZ1-1 179/+61,pMYOZ1-932/+61,pMYOZ1-676/+61,pMYOZ1-437/+61,and pMYOZ1-116/+61.Luciferase activity assay indicated that luciferase reporter plasmid pMYOZ1-116/+61 had significantly higher luciferase activity than negative control pGL3-Basic,indicating that it may contain core promoter region of bovineMYOZ1 gene.Luciferase activity of pMYOZ1-1 628/+61 was significantly higher than its downstream plasmid pMYOZ1-1 430/+61 (P<0.01),indicating that the -1 628/-1 430 region upstream ofMYOZ1 gene promoter might contain transcriptional enhancer factors. Bioinformatics analysis found that pMYOZ1-116/+61 might contain transcriptional combining sites such as SP1,GC Box and CAAT,and pMYOZ1-1 628/-1 430 might contain SP1 and MyoD.【Conclusion】 The luciferase report gene vectors of bovineMYOZ1 gene promoter were successfully constructed and the core promoter of bovineMYOZ1 gene was determined within -116/+61 region.
bovine;MYOZ1 gene;5′ RACE;promoter;dual-luciferase reporter
时间:2016-08-0909:40DOI:10.13207/j.cnki.jnwafu.2016.09.001
2015-02-11
“十二五”国家“863”计划项目(2013AA102505,2011AA100307-02);国家现代农业产业技术体系建设专项(CARS-38);“十二五”国家科技支撑计划项目(2011BAD28B04-03)
王明明(1989-),男,山东鱼台人,在读硕士,主要从事肉牛基因转录调控研究。E-mail:785336874@qq.com
昝林森(1963-),男,陕西扶风人,教授,博士生导师,主要从事肉牛奶牛遗传改良与健康养殖研究。
E-mail:zanlinsen@163.com
S823
A
1671-9387(2016)09-0001-09
网络出版地址:http://www.cnki.net/kcms/detail/61.1390.S.20160809.0940.002.html

