加入放归个体后的栗子坪保护区大熊猫小种群遗传结构研究
陈子君, 黄蜂,2, 何可, 冯菲菲,古晓东,杨旭煜,戴强, 齐敦武,杨志松
(1.西华师范大学 西南野生动植物资源保护教育部重点实验室, 四川 南充 637002;2.四川栗子坪国家级自然保护区管理处, 四川 石棉 625400;3.四川省野生动物资源调查保护管理站, 四川 成都 610081;4.中国科学院成都生物研究所, 四川 成都 610041;5.成都大熊猫繁育研究基地, 四川 成都 610086)
加入放归个体后的栗子坪保护区大熊猫小种群遗传结构研究
陈子君1, 黄蜂1,2, 何可1, 冯菲菲1,古晓东3,杨旭煜3,戴强4, 齐敦武5,杨志松1
(1.西华师范大学 西南野生动植物资源保护教育部重点实验室, 四川南充637002;2.四川栗子坪国家级自然保护区管理处, 四川石棉625400;3.四川省野生动物资源调查保护管理站, 四川成都610081;4.中国科学院成都生物研究所, 四川成都610041;5.成都大熊猫繁育研究基地, 四川成都610086)
小相岭山系是现有大熊猫种群数量最少的山系之一。国家林业局拟通过在该山系的栗子坪国家级自然保护区放归大熊猫“泸欣”、“淘淘”、“张想”来复壮大熊猫小种群。为评估加入3只放归个体后的小种群遗传结构,2012~2013年在研究区域采集大熊猫粪便样品92份,鉴定出14只个体(含放归个体)。放归个体加入后,测得放归个体与本地大熊猫小种群亲缘关系较远。种群年龄组成为青年组8只、成年组4只、未知年龄个体2只。平均观测杂合度(Ho)为0.557,平均期望杂合度(He)为0.581,遗传多样性处于中等偏上水平。5个位点上共计增加6个等位基因,平均等位基因数(MNA)达到3.667。遗传基因簇由1簇增加为4簇,出现新的基因频率。以上结果显示放归个体加入后该区大熊猫小种群遗传多样性有所提高,放归成效初显。
大熊猫;小种群;遗传多样性;放归;栗子坪国家级自然保护区
放归是促进小种群复壮,恢复破碎化栖息地的有效方法[1-2]。国内外已有不少成功案例,国内的如普氏野马(Equus ferus przewalskii)、麋鹿(Elaphurus davidianus )、扬子鳄(Alligator sinensis)等。国外的如加拿大盘羊(Ovis canadensis)、美洲野牛(Bison bison)、美洲黑熊(Ursus americanus)等。然而,也有少数放归活动因近交[3]、遗传多样性丢失[4]或者人类干扰、天敌捕食[5]而成为失败的尝试。因此,及时分析放归成效,总结经验、教训,对放归工作的进一步开展有着极为重要的意义。
小相岭山系是现有大熊猫种群数量最少的山系之一,种群生存力分析表明,如果与其他种群没有遗传交流或者无外来个体补充,该小种群极有可能在80年内会趋于灭绝[6]。为提高该区大熊猫小种群生存能力,促进大熊猫小种群的复壮,(截止到本研究开展的2013年)国家林业局在小相岭山系的栗子坪国家级保护区先后放归了3只大熊猫(Ailuropoda melanoleuca):“泸欣”已放归超过6年,“淘淘”已放归超过3年,“张想”也已放归超过2年。然而,加入放归个体后,栗子坪自然保护区大熊猫小种群遗传结构是否有所改善,目前仍不清楚。本研究利用野外调查收集的大熊猫粪便,通过粪便咬节测量、粪便DNA性别鉴定以及微卫星分析,确定了放归地大熊猫种群的年龄结构、性比和种群遗传结构,并从种群遗传上对放归成效进行了评估。这一评估结果有助于确定将来大熊猫放归和小种群复壮项目的方向和重点,对其他濒危珍稀野生动物的小种群复壮工作也有很好的参考价值。
1 研究对象及区域
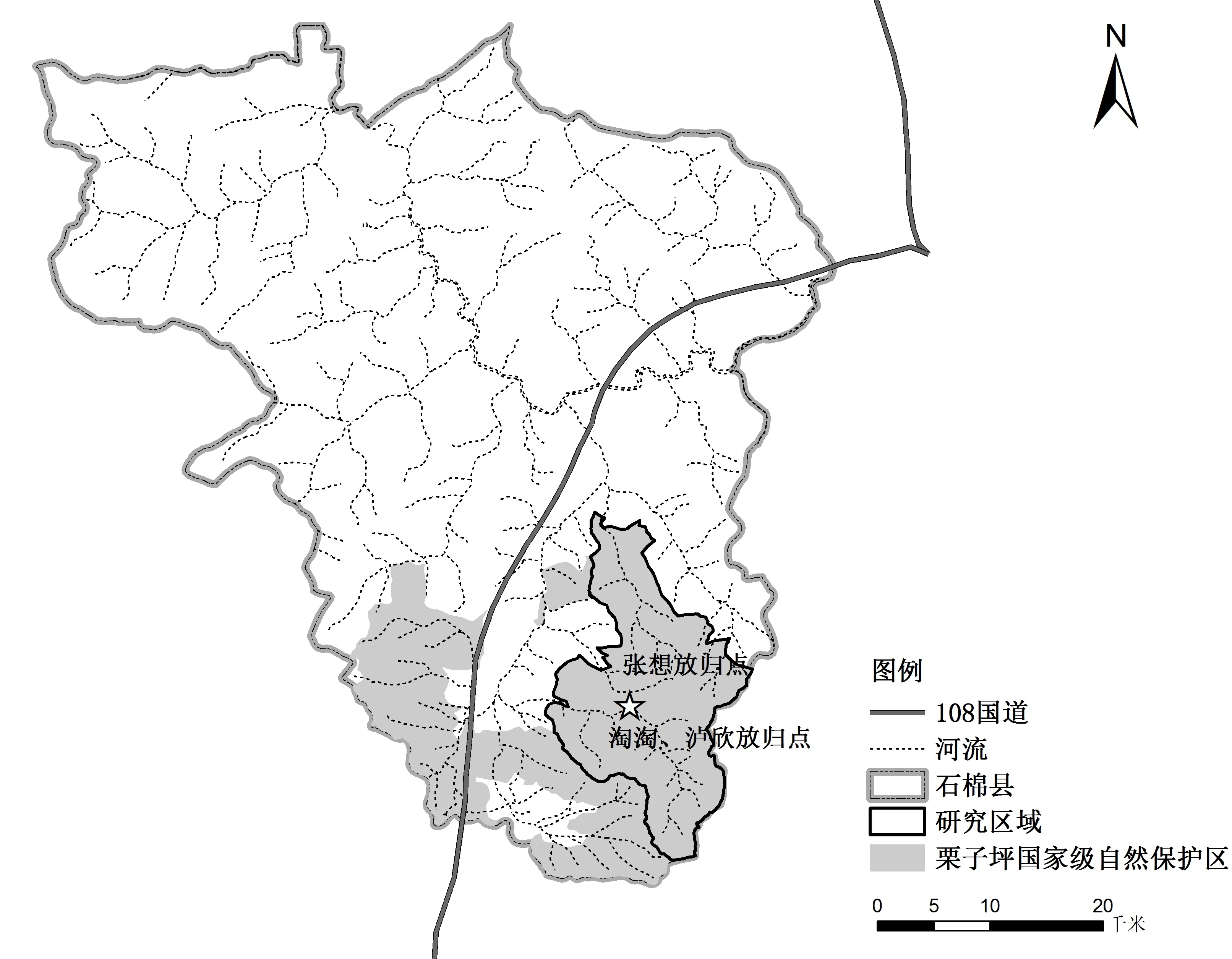
图1 研究区域区位示意图
栗子坪自然保护区位于四川石棉县境内,地理位置为E 102°10′33″~102°29′07″,N 28°51′02″~29°08′42″;南北长23 km,东西宽17.8 km,总面积47 940 hm2。区内最高海拔为4 551 m,最低海拔1 330 m,最大相对高差达3 221 m,平均相对高差大于2 000 m。保护区是小相岭山系大熊猫种群的核心分布区和关键栖息地,也是小相岭山系大熊猫种群遗传基因交流的关键走廊带和重要集中分布区。
本项目研究区域位于栗子坪自然保护区内公益海保护站周边,面积约为23 251.45 hm2。“泸欣”、“淘淘”、“张想”先后放归于该区(详见图1和表1),遗传分析所用粪便也采集自这一区域。这一区域的大熊猫栖息地与周边大熊猫栖息地存在较为严重的隔离[7-8],第四次全国大熊猫调查也认为该区域大熊猫种群是一个相对独立的局域种群[9]。

表1 放归大熊猫个体信息
2 研究方法
2.1野外采样
野外样品于2012年4月至2013年12月采集自保护区内公益海保护站周边(见图1)。 研究区域内调查样线的抽样强度为1 条/200 hm2,采用非损伤性遗传取样方法在野外收集粪便样品[10]。 在采样同时,对所有的样品采集点均进行GPS定位并记录。
2.2大熊猫年龄组划分
测量采集到的大熊猫粪便的咬节长度,采用“离差平方和法”对咬节长度进行聚类,参照胡锦矗的年龄组划分标准[11]确定个体年龄段:幼年组(小于1.5 岁),咬节值小于30 mm,粪便直径30 mm以下;青年组(大于等于1.5岁,并且小于5.5 岁),咬节值在30~35 mm(包含30 mm) 之间, 粪便直径30~55 mm(包含30 mm);成年组(大于等于5.5岁,并且小于20 岁),咬节长度在35~40 mm(包含35 mm)之间,粪便直径55~65 mm (包含55 mm);老年组(大于等于20 岁),咬节长度在40 mm(包含40 mm)以上,粪便直径在65 mm(包含65 mm)以上。
2.3粪便微卫星DNA提取与分析
使用QIAGEN公司的QIAamp DNA Stool kit试剂盒进行大熊猫粪便DNA的提取,性别鉴定PCR、微卫星PCR以及结果判定参考胡义波[12]、詹祥江[10]的方法,我们挑选了12对微卫星引物(Ame -μ10, Ame -μ11, Ame -μ22, Ame -μ13, Ame -μ15, Ame -μ24, Ame -μ26, Ame -μ27以及AY161179, AY161195, AY161213, AY161217)来对大熊猫粪便DNA进行微卫星扩增[12]。
因大熊猫粪便中DNA含量较低,为避免因引物选取、PCR扩增、数据读取等引起的误差,我们利用MICRO-CHECKER软件[13]检测基因分型数据中是否存在无效等位基因(Null alleles)、大片段等位基因丢失(Large allele dropout)及鬼影峰误判(Stutter scoring error)等分型错误。利用GIMLET 1.3.3软件[14]计算了12个座位的联合P(ID)值,以确定选择的微卫星引物数量是否能将影子效应(Shadow effect)[15]减少到一个不显著的水平。
2.4个体鉴定与种群数量估计
利用MStools插件[16],寻找基因分型数据中相匹配的基因型。判断为同一个个体的标准是:所有座位上的基因型都相同,或者只有一个座位上的一个等位基因存在差别[17]。由于我们采样分析是针对放归成效的研究,粪便采集也集中在放归大熊猫主要活动的区域(依据放归大熊猫佩戴的GPS项圈确定),可以认为出现在此区域外或是很少出现在该区域的个体被捕捉概率很低,因此我们选择CAPWIRE软件里TIRM(Two Innate Rates Model)模型[10]进行种群数量估计。
2.5种群遗传多样性及亲缘关系分析
利用ARLEQUIN 3.5[18]和GENEPOP 3.4[19]计算期望杂合度(He, expected heterozygosity)、表观杂合度(Ho, observed heterozygosity)、等位基因数(A),测算总体和各个位点是否符合哈温平衡(Hardy-Weinberg)。利用FSTAT 2.9.3.2[20]来检验位点是否存在连锁不平衡(Linkage disequilibrium)。利用STRUCTURE方法[21]检测研究区域所有个体中存在的遗传基因簇。利用Kingroup v2.08[22]来测算个体间的亲缘关系远近,其中个体间的亲缘关系值(pairwise relatedness, r)参考Wang[23]的研究。
3 结果
3.1种群数量、性比及年龄结构
本研究从92 份样品(含粪便样品及放归个体血样)中得到52个可靠基因型。TIRM模型分析表明,研究区域大熊猫种群数量(含3 只放归个体)为25 只(95%置信范围:14~28)。种群数量最低估计值(lowCI)为14 只(与实验室分析鉴定结果一致),因此研究区域中大熊猫种群最保守的数量应该是14 只。14只个体分布在公益海保护站区域,海拔集中在2 291~3 510 m(见图2),分别是8 只雌性、6只雄性,雌雄比例为4∶3。其中“泸欣”、“淘淘”和“张想”为放归个体,年龄有准确记录,无需通过粪便咬节确定年龄,另有两只个体未能获得有效咬节数据,无法判断年龄,其余个体的年龄段均根据咬节法测得。结果表明,本地种群的年龄组成为:青年组6 只,成年组3 只,未知年龄个体2只(见表2)。从年龄结构上来看,青年组个体数明显多于成年组个体数,年龄组成为增长型。 加入放归个体后的大熊猫种群年龄组成为:青年组8 只、成年组4只,未知年龄个体2只(见表2),与放归前比较,年龄组结构的增长型特征更为明显。

表2 本研究大熊猫种群年龄组组成
3.2本地小种群遗传结构
MICRO-CHECKER检验表明,本研究基因分型数据结果未受无效等位基因、大片段等位基因丢失及鬼影带误判等分型错误的显著干扰。GIMLET分析显示,12 个微卫星座位的联合区分率很高,两只个体具有相同基因型的无偏概率P(ID)为3.286×10-9,即使为双胞胎,判断错误的概率P(sib) 也仅为0.04778%,小于0.01[15]。哈温平衡检验结果显示研究区域本地大熊猫小种群未偏离哈温平衡(P>0.05)。连锁不平衡分析结果显示本研究采用的12 个位点,对当地大熊猫小种群而言不存在连锁遗传。12个微卫星位点分析结果显示研究区域本地大熊猫小种群的平均观测杂合度(Ho)为0.635,平均期望杂合度(He)为0.608。平均等位基因数(MNA)为3.455(见表3)。STRUCTURE 分析结果显示K=1时(图3A),LnP(D)具有最大的平均值,图3B表明研究区域本地大熊猫小种群整体上是一个遗传基因簇。

表3 研究区域本地大熊猫小种群(代号A)与加入放归个体后的种群(代号B)遗传多样性信息
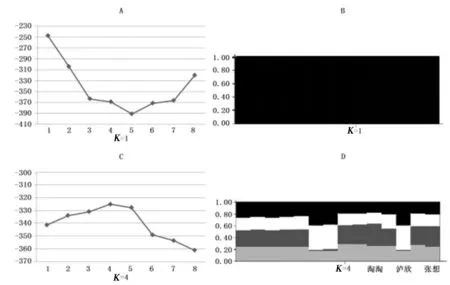
图3 LnP(D)值及遗传基因簇图图A:本地小种群LnP(D)值;图C:放归个体加入后的种群LnP(D)值;图B图D分别为K=1、K=4的遗传基因簇图
3.3加入放归个体后小种群遗传结构
3.3.1放归个体和本地大熊猫亲缘关系
亲缘关系分析结果显示(见表4),“泸欣”、“淘淘”、“张想”和整个本地小种群的亲缘关系都较远,与其它13 只个体的亲缘系数(r)平均值平均为-0.07、-0.21、-0.23。
虽然“泸欣”和lzp6、lzp7、lzp10、lzp11、“张想”的亲缘系数(r)>0,但是这五只个体均为雌性,不存在因亲缘关系近而带来的近交风险。“淘淘”和lzp2、lzp7、“张想”的亲缘系数(r)分别为0.05、0.10、0.10,但是r<0.125,“淘淘”和这三只个体的亲缘关系水平低于双重表亲(Double first cousins),不存在明显的因亲缘关系较近而带来的近交风险。“张想”和lzp6、“淘淘”、“泸欣”的亲缘系数(r)分别为0.03、0.10、0.11,r<0.125,“张想”和这三只个体的亲缘关系水平也低于双重表亲(Double first cousins),不存在明显的因亲缘关系近而带来的近交风险。因lzp7是雌性大熊猫,虽然它和“张想”亲缘系数(r)为0.30,属于亲子/全同胞(parent-offspring/full sibs)的水平,但也不存在因亲缘关系近的近交风险。
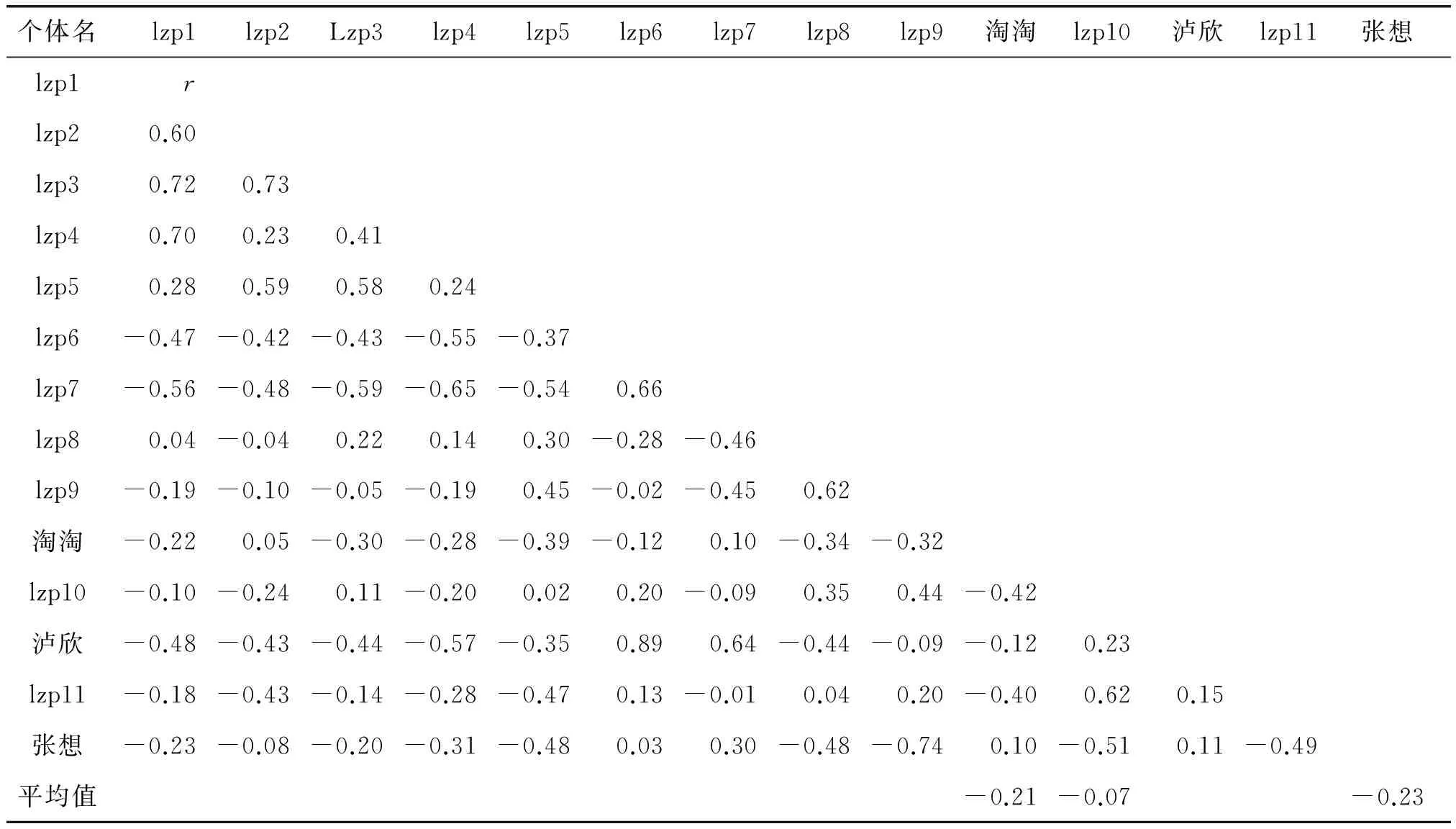
表4 研究区域大熊猫个体亲缘关系
3.3.2加入放归个体后的遗传结构
根据哈温平衡检验分析结果,加入放归个体后的大熊猫小种群仍未偏离哈温平衡(p>0.05)。连锁不平衡分析结果显示对于加入放归个体后的大熊猫小种群,也不存在连锁遗传。12个微卫星位点分析结果显示:加入放归个体后种群平均观测杂合度(Ho)为0.557,平均期望杂合度(He)为0.581(见表3)。相比Zhang等[24]的研究,加入放归个体后的遗传多样性仍处于中等偏上的水平。平均等位基因数(MNA)增加到3.667,表现在Ame-μ11(Locus1)、Ame-μ22(Locus3)、Ame-μ24(Locus7)、Ame-μ13(Locus10)和Ame-μ27(Locus12)5个位点上,共计增加6 个等位基因。 STRUCTURE分析结果表明,K=4时(见图3C),LnP(D)具有最大的平均值,遗传基因簇由一簇变为了四簇(见图3D),这表明放归个体加入后,出现了新的基因频率。
4 讨 论
三只放归个体加入当地种群后,研究区域大熊猫小种群的等位基因数有所增加,同时也出现了新的基因频率,这表明该区大熊猫小种群的遗传多样性确实呈增长趋势。遗传多样性的高低在一定程度上反映了大熊猫种群的进化潜力[25],因此放归个体的加入有助于研究区域大熊猫小种群的种群遗传多样性的提高,将对该种群的复壮带来积极影响。
本研究分析结果显示放归个体与研究区域本地大熊猫小种群亲缘关系较远。杨波等[26]的研究表明:亲缘关系越远,释放个体与本地种群组成的混合种群内近交率会越低,从而减少近交衰退的风险,更有利于改善野生种群遗传水平。因此这三只放归个体的加入,能有效提高研究区域本地大熊猫小种群的遗传多样性水平。
迄今,“泸欣”、“淘淘”、“张想”已放归分别超过6年、3年、2年,它们在大熊猫放归地——栗子坪国家级自然保护区生存状况良好。年龄组结构分析显示,加入放归个体后,研究区域大熊猫小种群年龄结构得到进一步改善。此外,该区域的红外相机监测结果显示,“泸欣”已成功带崽,表明放归个体已经初步融入当地种群,放归工作取得初步成效。若其余两只放归个体也能成功繁殖,将进一步促进该地区大熊猫小种群的复壮。
目前,小相岭山系大熊猫种群数量仍然很少,成功放归三只大熊猫远不足以复壮当地小种群。针对这一问题,该山系还需进一步持续开展大熊猫放归工作。而在放归个体的选择上,需要比较放归个体和当地小种群的遗传背景,优先选择能有效提高当地小种群遗传多样性的个体。
[1]ZHU L F, ZHAN X J, MENG T, et al. Landscape features influence gene flow as measured by cost-distance and genetic analyses: a case study for giant pandas in the Daxiangling and Xiaoxiangling mountains [J]. BMC genetics, 2010(1):72.
[2]ZHU L F, ZHANG S N, GU X D, et al. Significant genetic boundaries and spatial dynamics of giant pandas occupying fragmented habitat across southwest China [J]. Molecular Ecology, 2011, 20(6):1122-1132.
[3]BIEBACH I, KELLER L F. Inbreeding in reintroduced populations: the effects of early reintroduction history and contemporary processes [J]. Conservation Genetics, 2010(2):527-538.
[4]ALCAIDE M., NEGRO J J., SERRANO D, et al. Captive breeding and reintroduction of the lesser kestrel Falco naumanni: a genetic analysis using microsatellites [J]. Conservation Genetics, 2010(11): 331-338.
[5]GREY-ROSS R, DOWNS C T, KIRKMAN K.. Reintroduction Failure of Captive-Bred Oribi (Ourebia ourebi) [J]. South African Journal of Wildlife Research, 2009, 39(1): 34-38.
[6]朱磊, 吴攀文, 张洪, 等. 小相岭山系大熊猫种群生存力分析 [J]. 西华师范大学学报( 自然科学版), 2008, 29(2): 112-116.
[7]ZHU L F, ZHAN X J, WU H, et al. Conservation implications of drastic reductions in the smallest and most isolated populations of giant pandas [J]. Conservation Biology, 2010, 24(5): 1299-1306.
[8]青菁, 胥池, 杨彪, 等. 小相岭山系大熊猫廊道规划[J]. 生态学报, 2016, 36(4): 1-9.
[9]四川省林业厅. 四川的大熊猫[M]. 成都:四川科学技术出版社,2015.
[10]詹祥江.利用非损伤性遗传取样研究大熊猫的种群数量和扩散模式 [D]. 中国科学院动物研究院, 2006.
[11]胡锦矗. 从野外大熊猫的粪便估计年龄及其种群年龄结构的研究 [J]. 兽类学报, 1987, 7 (2): 81-84.
[12]胡义波. 凉山山系大熊猫的种群历史、景观与空间遗传格局研究 [D]. 北京:中国科学院研究生院, 2008.
[13]VAN OOSTERHOUT C, HUTCHINSON W F, WILLS D P M, et al. Micro-checker: software for identifying and correcting genotyping errors in microsatellite data [J]. Molecular Ecology Notes, 2004(3): 535-538.
[14]VALIERE N. GIMLET: a computer program for analysing genetic individual identification data [J]. Molecular Ecology Notes, 2002, 2: 377-379.
[15]MILLS L S, CITTA J J, LAIR K P, et al. Estimating animal abundance using noninvasive DNA sampling: Promise and pitfalls [J]. Ecological Applications, 2000, 10(1):283-294.
[16]PARK S D E. Trypanotolerance in west African cattle and the population genetic effects of selection [D]. Dublin:University of Dublin, 2001.
[17]BELLEMAIN E, SWENSON J E, TALLMON D, et al. Estimating population size of elusive animals with DNA from hunter-collected feces: Four methods for brown bears [J]. Conservation Biology, 2005, 19(1): 150-161.
[18]EXCOFFIER L, LAVAL G, SCHNEIDER S. Arlequin (version 3.0): an integrated software package for population genetics data analysis [J]. Evolutionary Bioinformatics Online, 2005, 1: 47-50.
[19]RAYMOND M, ROUSSET F. Genepop (Version1.2): Population Genetics Software for Exact Tests and Ecumenicism [J]. Journal of Heredity, 1995,86(3):248-249.
[20]GOUDET J. FSTAT, a program to estimate and test gene diversities and fixationindices (version 2.9.3). 2001. Updated from Goudet J. FSTAT (Version 1.2): A computer program to calculate F-statistics [J]. Journal of Heredity, 1995, 86: 485-486.
[21]PRITCHARD J K, STEPHENS M, DONNELLY P. Inference of population structure using multilocus genotype data [J]. Genetics, 2000, 155(2): 945-959.
[22]KONOVALOV D A, MANNING C, HENSHAW M T. Kingrouf: a program for pedigree relationship reconstruction and kin group assignments using genetic markers [J]. Molecular Ecology Notes, 2004(4): 779-782.
[23]WANG J. COANCESTRY: a program for simulating, estimating and analysing relatedness and inbreeding coefficients [J]. Molecular Ecology Resources, 2011, 11(1): 141-145.
[24]ZHANG B W, LI M, ZHANG Z J, et al. Genetic Viability and Population History of the Giant Panda, Putting an end to the "Evolutionary dead end"? [J]. Molecular Biology and Evolution, 2007, 24(18): 1801-1810.
[25]张泽钧, 张陕宁, 魏辅文, 等. 移地与圈养大熊猫野外放归的探讨 [J]. 兽类学报, 2006, 26 (3): 292-299.
[26]杨波, 杨承忠, 涂飞云, 等. 大熊猫野化放归中的遗传学分析 [J]. 四川动物, 2013, 32(1):149-155.
[Abstract]The population of giant panda in Xiaoxiangling is one of the smallest . In order to rejuvenate this small panda population, the State Forestry Administration has tried to release three giant pandas(Ailuropoda melanoleuca): “Luxin”, “Taotao” and “Zhangxiang” in the Liziping National Nature Reserve. To assess the genetic structure of this small population after reintroduction, 92 stools of giant pandas were collected in research area from 2012 to 2013, and 14 individuals(including three reintroduced individuals)were identified. After reintroduced the three individuals, measurement proved that the kinship between the reintroduced individuals and local small population was far. The population age composition was: eight individuals in youth group,four individuals in adult group and two individuals in unknown group.The mean observed heterozygosity (Ho) was 0.557. The mean expected heterozygosity (He) was 0.581. Genetic diversity was in the medium level.Five sites increased six alleles.And the mean allele number (MNA) achieved 3.667. What’s more ,the genetic cluster increased to four meaning that new gene frequency had appeared. All these results suggest that the genetic diversity of the small giantpanda population in this region is improved. The reintroduction is beginning to bear results.
[Key words]giant panda(Ailuropoda melanoleuca); small population; genetic diversity; reintroduction; Liziping National Nature Reserve
[责任编辑刘景平]
The Genetic Structure of Small Giant Panda Population in the Liziping Nature Reserve after Reintroduction
CHEN Zi-jun1, HUANG Feng1,2, HE Ke1, FENG Fei-fei1, GU Xiao-dong3, YANG Xu-yu3,DAI Qiang4, QI Dun-wu5, YANG Zhi-song1
(1.Key Laboratory of Southwest China Wildlife Resource Conservation, China West Normal University,Nanchong, Sichuan 637002, China;2.Sichuan Liziping Nature Reserve Management Office, Shimian, Sichuan 625400, China;3.Sichuan Station of Wildlife Survey and Management, Chengdu, Sichuan 610081, China;4.Chengdu Institute of Biology, Chinese Academy of Sciences, Chengdu, Sichuan 610041, China;5.Chengdu Research Base of Giant Panda Breeding, Chengdu, Sichuan 610086, China)
Q953
A
1672-9021(2016)02-0001-07
陈子君(1989-),女,四川绵阳人,西华师范大学生命科学学院硕士研究生,主要研究方向:大熊猫保护生物学;杨志松(1975-),男,四川南充人,博士,西华师范大学生命科学学院副教授,硕士生导师,主要研究方向:大熊猫保护生物学、分子系统地理学、 鸟类生态学。
香港海洋公园保育基金资助项目(GP07_1213)。
2016-03-01

