犬副流感病毒的分离鉴定及部分基因序列分析
孙 明,刘巧荣,秦亚嫚,邓小雨,杨欣艳,刘伯华,陈西钊,2
(1. 北京世纪元亨动物防疫技术有限公司,北京 100085;2.中国动物疫病预防控制中心,北京 100125)
研究报告
犬副流感病毒的分离鉴定及部分基因序列分析
孙明1,刘巧荣1,秦亚嫚1,邓小雨1,杨欣艳1,刘伯华1,陈西钊1,2
(1. 北京世纪元亨动物防疫技术有限公司,北京100085;2.中国动物疫病预防控制中心,北京100125)
【摘要】目的分离并鉴定犬副流感病毒(canine parainfluenza virus,CPIV),并对该病毒N、HN和F基因进行序列分析,研究其遗传变异情况;为CPIV诊断、治疗以及预防奠定分子生物学基础。方法用Vero细胞接种感染CPIV阳性犬肺组织,盲传3代,观察病变情况,收集72 h培养液进行PCR鉴定、血凝特性以及形态学观察。同时,用3对特异性引物,扩增N、HN和F全基因,进行序列测定和分析,并制作系统进化树。结果从犬肺中成功分离出一株犬副流感病毒,并命名为QF20100726。该病毒能凝集豚鼠、猪、鸡和人O型血,电镜观察病毒呈圆形、长丝状等形态多样、大小不等的颗粒状;序列分析表明,与人源、犬源等10株有代表性的副流感病毒基因相比,N基因核苷酸同源性为 95.7%~99.8%,氨基酸同源性为97.4%~99.6%,其中有两处氨基酸发生新的变异,分别是第257位由V变成了A,第301位由A变成了T;HN基因核苷酸同源性为95.5~99.8%,氨基酸同源性为96.3~99.6%,F基因核苷酸同源性为94.7%~99.6%,氨基酸同源性为 95.6%~99.3%。有两处发生特异变异,分别是56位由T变成了S,89位由T变成了M。结论成功分离犬副流感病毒QF20100726分离株,其血凝特性及超微形态特征均符合CPIV特性,N、HN和F全基因生物进化树显示,该分离株与犬副流感病毒分离株08-1990(登录号:KC237063)亲缘关系最近;N基因氨基酸在第257位(由V变成了A)和第301位(由A变成了T),F基因氨基酸在第56位(由T变成了S)和89位(由T变成了M)发生了特异性变异。这些数据将为CPIV的防控奠定基础。
【关键词】CPIV;病毒分离;序列分析
犬副流感病毒感染(canine parainfluenza virus infection)是由犬副流感病毒(canine parainfluenza virus,CPIV)引起的犬的一种接触性传染病,主要表现为发热、流涕和咳嗽。CPIV是犬传染性呼吸系统疾病(即“犬窝咳”)的主要病源之一[1]。常引起犬的支气管炎和支气管肺炎,损害呼吸道上皮细胞,给其他病原体的感染创造了条件[2,3]。1967年Binn等用犬肾细胞首次从患有呼吸道病的犬体内分离到犬副流感病毒,此后在世界各地均有CPIV的报道,严重影响着世界各国养犬业的发展。
CPIV是单股、不分节段的负链RNA病毒,约为15246 bp[4]。CPIV基因组主要编码6种蛋白:大蛋白(L)、血凝素神经氨酸酶(HN)、核衣壳蛋白(NP)、融合蛋白(F)、磷蛋白(P)和基质蛋白(M),其中NP蛋白是核衣壳蛋白的主要成分,与病毒RNA直接结合,高度保守,在病毒感染时可引起强烈的抗体反应,HN蛋白和F蛋白突出于囊膜形成纤突,分别介导病毒吸附和穿入细胞。
该病毒在分类上属副粘病毒科(Paramyxoviridae),副粘病毒亚科(Paramyxovirinae)茹布拉病毒属(Rubulavirus)。与同属的猴病毒V型(SV5)、人SV5分离株及人副流感病毒- II 型(hPIV-II)抗原性密切相关[5]。目前,市场虽有此类疫苗,该病仍然时有发生,可能与该病毒的不断变异有关,故分离当前的流行变异株,了解序列变异情况对于预防该病的流行、诊断和治疗等具有重要意义。
1材料和方法
1.1病料
临床诊断犬副流感病毒病死京巴犬,年龄约1岁。临床表现为患犬精神萎顿、不喜食;咳嗽、流涕、喘息等呼吸道症状,伴有发烧。治疗无效死亡,取肺脏进行处理。
1.2细胞系
Vero细胞为本实验室保存。
1.3载体、菌株、试剂
pGEM-T-easy购自Promega公司。DH5a感受态细胞购自北京全式金生物技术有限公司。氨苄青霉素、购自Sigma公司。病毒RNA提取试剂盒和胶回收试剂盒均购自Omega公司。DMEM培养液、新生牛血清为Gibco产品。
1.4病毒分离
参照白文彬等介绍的方法[6],进行病毒分离,并将病毒命名为QF20100726。
1.5病毒鉴定
1.5.1血凝试验:参考姚火春介绍的方法[7],分别采集豚鼠、鸡、猪抗凝血,用PBS (pH 7.4)洗涤红细胞四次,1 500 rpm离心10 min,用PBS(pH 7.2)配成1%红细胞悬液进行血凝试验(HA)。
1.5.2RT-PCR鉴定:用引物对:5′-AGTCAGAGTAGTTCAATAAGGACCTATC-3‘和5’-TGGTGAATTGAGATGGACTTCAGG-3′对所分离的病毒进行RT-PCR鉴定,扩增程序:42℃ 45 min,95℃ 3 min,95℃ 30 sec,55℃ 45 sec,72℃ 1.5 min,35个循环后72℃延伸10 min。
1.5.3病毒的电镜观察:参考殷震、金昌德等介绍的方法[3,8],对细胞培养液负染色,进行电镜观察。
1.6N、HN和F基因的克隆测序
1.6.1引物设计与合成:依据GenB上的CPIV基因组序列,分别针对N基因、HN基因和F基因设计了引物。如下:
N1:5′-AGTCAGAGTAGTTCAATAAGGACCTATC-3′
N2:5′-TGGTGAATTGAGATGGACTTCAGG-3′
HN1:5′-TTGCTGTCCTAACACCTGCTATAG-3′
HN2:5′-TGGTGAATTGAGATGGACTTCAGG-3′
F1:5′-CCAATAACTGGAATCACCAGCTTG-3′
F2:5′-CGAGACGGTTCTTTCAATACTAGTTTC-3′
三对引物分别可扩增大小为1 678 bp、1 964 bp、和1 830 bp的片段。引物由上海英骏生物技术有限公司合成。
1.6.2扩增及克隆:
1.6.2.1病毒RNA的提取: 参照Omega公司的病毒RNA提取试剂盒操作说明提取PRV总DNA。
1.6.2.2RT-PCR扩增: 42℃ 45 min,95℃ 3 min,95℃ 30 s,55℃ 45 s,72℃ 2 min,35个循环后72℃延伸10 min。
1.6.2.3PCR产物电泳、克隆和序列测定:PCR扩增产物进行琼脂糖凝胶电泳鉴定,用Omega 公司胶回收试剂盒回收PCR产物,克隆到pGEM-T-easy,经PCR鉴定阳性的重组质粒送上海英骏生物技术有限公司测序。
2结果
2.1病毒分离培养
病料接种到Vero细胞第5代24 h后出现典型细胞病变,至第5 天收毒时,大部分细胞融合,裂解死亡,培养液中碎片增多,相同条件下的正常细胞无此变化(图1和图2)。
2.2血凝试验
病毒在25℃条件能凝集豚鼠、猪、鸡和人O型红细胞。
2.3RT-PCR鉴定结果
对Vero细胞培养病毒进行犬副流感病毒的RT-PCR检测,结果显示, Vero细胞培养的病毒为CPIV阳性。
2.4电镜观察
电镜照片显示,CPIV为圆形,有囊膜,直径80 nm左右,囊膜上有直径8 nm~10 nm的纤突。病毒因囊膜的破损而形态不规则,呈多形性、长丝状等长丝状核衣壳见图3。
2.5N、HN和F基因克隆、测序
用设计的N、F和HN基因引物扩增的产物经琼脂糖电泳显示,分别可见约1 680 bp、1 970 bp、和1 830 bp左右的3个基因片段(图4),大小与N、F和HN蛋白基因相符,预测结果证实所克隆的基因正确。
图1 正常对照vero细胞Fig.1 Normal control vero cells
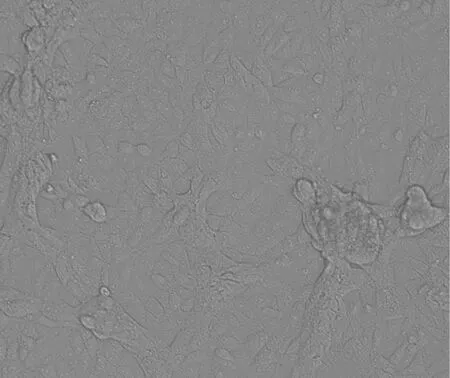
图2 CPIV接种vero后24 h细胞变化Fig.2 Vero cells at 24 hours after CPIV inoculation
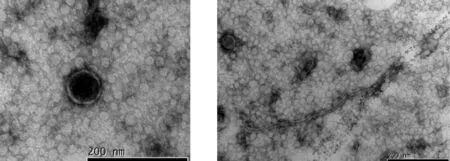
图3 电镜下不同形态的病毒粒子(标尺=200 nm)Fig.3 Different morphology of the virus particles observed under electron microscope(Bar=200 nm)
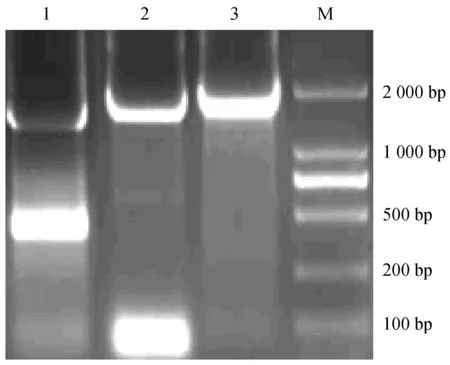
1. N基因, 2. F基因, 3. HN基因 M. Marker图4 N、F和HN基因扩增1. N gene, 2. F gene, 3. HN gene, M. markerFig.4 Amplification of N, F and HN genes
2.6N序列同源性分析
与人源、犬源等10株有代表性的副流感病毒N基因相比,核苷酸同源性为 95.7%~99.8%,氨基酸同源性为97.4%~99.6%。其中有两处氨基酸发生新的变异,分别是第257位由V变成了A,第301位由A变成了T。其中与人源副流感病毒分离株LN(登录号:JQ743324)同源性最低为97.4%,与犬源副流感病毒分离株08-1990(登录号:KC237063)和D277(登录号:KC237065)同源性最高均为99.6%。用DNAMan生物软件绘制系统进化树见图5。

图5 N基因系统进化树Fig.5 The phylogenetic tree based on N gene
2.7F基因序列同源性分析
与人源、犬源等10株有代表性的副流感病毒F基因相比。核苷酸同源性为94.7%~99.6%,氨基酸同源性为 95.6%~99.3%。有两处发生特异变异,分别是56位由T变成了S,89位由T变成了M。其中与人源副流感病毒分离株LN(登录号:JQ743324)同源性最低为95.6%,与犬源副流感病毒分离株08-1990(登录号:KC237063)同源性最高为99.3%。F基因系统进化树显示,该分离株与分离株D277和08-1990在一个分支上(图6)。
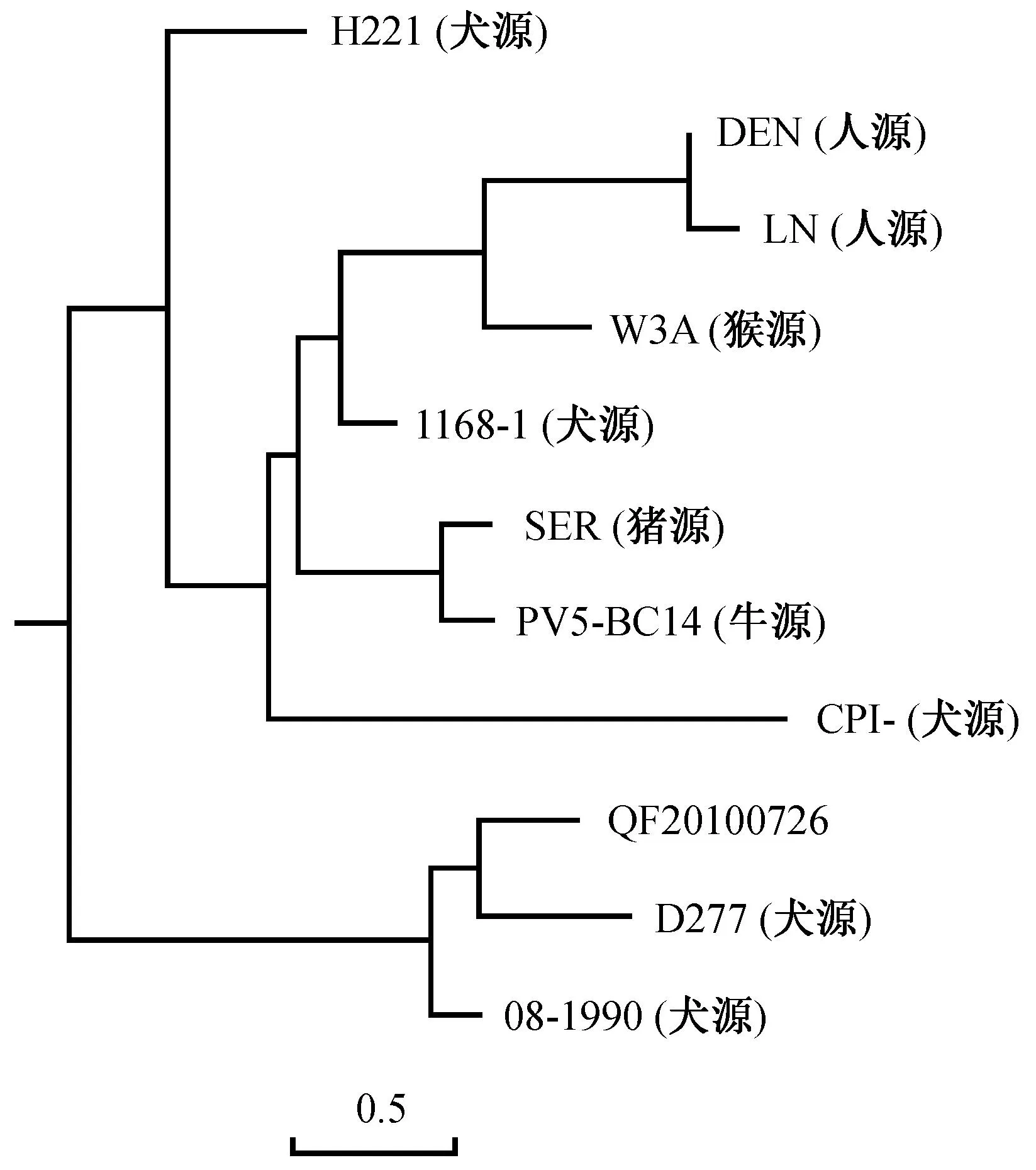
图6 F基因系统进化树Fig.6 The phylogenetic tree based on F gene
2.6HN基因序列同源性分析
与人源、犬源等10株有代表性的副流感病毒H基因相比,核苷酸同源性为95.5~99.8%,氨基酸同源性为96.3~99.6%,其中与人源副流感病毒分离株LN(登录号:JQ743324)同源性最低为95.5%,与犬源副流感病毒分离株D277(登录号:KC237065)同源性最高为99.6%。HN基因系统进化树显示,该分离株与D277和08-1990分离株在一个分支上(图7)。
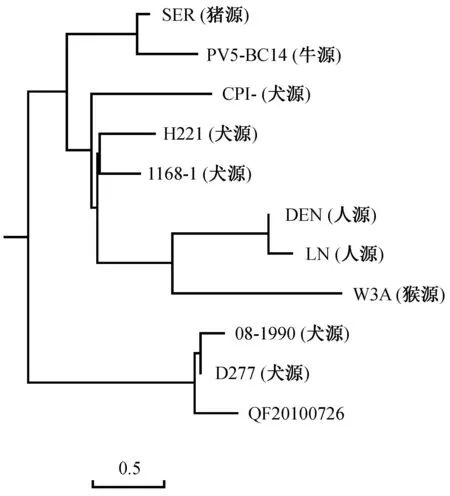
图7 HN基因系统进化分析Fig.7 The phylogenetic tree based on HN gene
3讨论
血清学调查表明,CPIV 在世界各国普遍存在[9-11],是危害犬业重要传染病之一。该病毒广泛感染玩赏犬、实验犬、警犬和军犬。主要表现为发热、流涕和咳嗽,在军犬和警犬中长导致嗅觉障碍。另外CPIV也可引起急性脑脊髓炎和脑内积水,临床表现为后驱麻痹和运动失调等症状,严重危害犬类的健康生长。近年来,随着养犬业的不断扩大,犬副流感病毒感染越来越受到人们的重视。北京某动物医院诊治一例临床诊断为疑似CPIV感染京巴犬,表现为精神萎顿、不喜食;咳嗽、流涕、喘息等呼吸道症状,伴有发烧,治疗无效死亡。自然条件下犬单独感染CPIV的情况不为多见,究其死亡原因,可能与细菌、支原体和病毒等混合感染有关[12]。为了分析CPIV变异情况,本研究从此患犬肺脏组织中分离得到一株CPIV,命名为QF20100706,并对其部分基因序列进行分析,研究基因变异情况。
该病毒在4℃条件下能凝集豚鼠、猪、鸡和人“O”型红细胞,这与CPIV血凝特性一致[13]:4℃或 25℃条件下能凝集鸡、猪、兔、犬、豚鼠、羊、人“O”型血红细胞;电镜观察期超微结构呈园形、长丝状等不规则形态,与金昌德报道的一致[10]。
本研究从患犬组织中分离CPIV,并对N、HN和F基因进行扩增和测序,分析其变异情况。核苷酸和氨基酸同源性分析和系统进化关系显示,与10株有代表性的副流感病毒N基因相比,核苷酸同源性为 95.7%~99.8%,氨基酸同源性为97.4%~99.6%。其中有两处氨基酸发生新的变异,分别是第257位由V变成了A,第301位由A变成了T。其中与人源副流感病毒分离株LN(登录号:JQ743324)同源性最低为97.4%,与犬源副流感病毒分离株08-1990(登录号:KC237063)同源性最高为99.6%。HN基因分析显示,与10株有代表性的副流感病毒H基因相比,核苷酸同源性为95.5~99.8%,氨基酸同源性为96.3~99.6%,其中与人源副流感病毒分离株LN(登录号:JQ743324)同源性最低为95.5%,与犬源副流感病毒分离株D277(登录号:KC237065)同源性最高为99.6%。与10株有代表性的副流感病毒F基因相比,该毒株F基因核苷酸同源性为94.7%~99.6%,氨基酸同源性为 95.6%~99.3%。有两处发生特异变异,分别是56位由T变成了S,89位由T变成了M。其中与人源副流感病毒分离株LN(登录号:JQ743324)同源性最低为95.6%,与犬源副流感病毒分离株08-1990(登录号:KC237063)同源性最高为99.6%。F基因的遗传变异对于CPIV的预防有着重要影响,特别是重要抗原位点的变化可能导致疫苗的免疫保护不全面,那么本分离株F基因的变异是否能导致疫苗的免疫保护不全或者增加CPIV毒力变化值得进一步研究。N、HN和F基因序列分析还表明,犬副流感病毒流行毒株之间、氨基酸序列之间变化不大,但与其他相关毒株间差异相对较大,这与李长磊等研究结果一致[14]。
成功分离一株犬副流感病毒,该病毒在25℃条件能吸附豚鼠、鸡、猪和人O型血红细胞。电镜观察显示病毒为圆形、长丝状等形态多样,大小不等的病毒粒子。N、HN和F基因序列分析显示,该分离株与犬副流感病毒分离株08-1990(登录号:KC237063)亲缘关系最近;N基因氨基酸在第257位(由V变成了A)和第301位(由A变成了T),F基因氨基酸在第56位(由T变成了S)和89位(由T变成了M)发生了特异性变异。
参考文献:
[1]Appel MJ. Virus Infections of Carnivores [M]. New York. Elesevier Science Publishing Co. Inc. 1987: 125-132.
[2]Damian M, Morales E, Salas G, et al. Immunohistochemical detection of antigens of distemper, adenovirus and parainfluenza viruses in domestic dogs with pneumonia [J]. J Comp Pathol, 2005, 133(4): 289-293.
[3]金昌德, 李六金, 李秦, 等. 犬副流感病毒弱毒株的分离与鉴定 [J].第四军医大学学报, 2000, 21(6): 721-724.
[4]Lamb R , Kolakofsky D. Paramyxoviridae: the viruses and their replication. In: Knipe DM, Howley PM. Fields Virology [M]. 4thed. Philadelphia, PA: Lippincott-Raven, 2001: 1305-1340.
[5]Ueland K. Serological, bacteriological and clinical observations on an outbreak of canine infectious tracheobronchitis in Norway [J]. Vet Rec, 1990, 126: 481-483.
[6]白文彬, 于康震. 动物传染病诊断学 [M]. 北京:中国农业出版社, 2002: 106-110.
[7]姚火春.兽医微生物学实验指导(第2版)[M]. 北京:中国农业出版社, 2002: 105-107.
[8]殷震, 刘景华主编. 动物病毒学(第二版)[M]. 北京:科学出版社,1997:750-751.
[9]闫喜军, 夏咸柱, 柴秀丽, 等. 犬副流感病毒血清抗体流行病学调查 [J]. 黑龙江畜牧兽医, 2001,(5): 75-76.
[10]Englund L, Jacobs AA, Klingeborn B, et al. Seroepidemiological survey of Bordetella bronchiseptica and canine parainfluenza-2 virus in dogs in Sweden [J].Vet Rec, 2003,152(9): 251-254.
[11]Ellis J, Anseeuw E, Gow S. et al. Seroepidemiology of respiratory (group 2) canine coronavirus, canine parainfluenza virus, andBordetellabronchisepticainfections in urban dogs in a humane shelter and in rural dogs in small communities [J]. Can Vet J, 2011, 52(8): 861-868.
[12]刘腾飞. 犬副流感病毒的检测和NP基因的克隆、表达及其重组蛋白的免疫活性的研究 [D]. 乌鲁木齐, 新疆农业大学, 2010: 1-2.
[13]中国农业科学院哈尔滨兽医研究所. 兽医微生物学 [M]. 北京: 中国农业出版社, 1998: 414-415.
[14]李长磊, 赵福广, 于亚洲. 犬副流感病毒的分离鉴定及F基因的遗传分析 [J]. 经济动物学报, 2013, 17(2): 86-89.
Isolation and identification of canine parainfluenza virus and partial gene sequence analysis of the isolate
SUN Ming1, LIU Qiao-rong1, QIN Ya-man1, DENG Xiao-yu1, YANG Xin-yan1, LIU Bo-hua, CHEN Xi-Zhao1,2
(1. Beijing ANHEAL Laboratories Co. Ltd, Beijing 100094, China; 2. China Animal Disease Control Center, Beijing 100125)
【Abstract】ObjectiveTo isolate and identify canine parainfluenza virus (CPIV) from the a dog suspected of CPIV infection, then to analyze the gene sequence variation of N,HN and F genes and provide a molecular biology basis for the diagnosis, treatment, prevention and control of CPIV infection. Methods Samples of the lung tissue was taken from a dead dog suspected of CPIV infection, and were inoculated into Vero cells. CPE were observed after three blind passages, then the cell culture fluid after 72-hour-cultuere was assayed for further PCR identification, blood coagulation characteristics and morphological observation. Moreover, 3 pairs of oligonucleotide primers were designed, and N, HN and F complete genes of CPIV were amplified by RT-PCR from positive CPIV culture fluid. The genetic variation of the three genes were further analyzed by biological software, and phylogenetic trees were produced. ResultsOne strain of CPIV was isolated and named QF20100726. The results showed that the CPIV strain could agglutinate Guinea pig, pig,chicken and human o type blood cells,and had the basic ultrastructural chatacteristics of parainfluenza virus. Compared with 10 representative CPIV genes, N, HN and F gene phylogenetic analysis of the QF20100726 CPIV gene showed 95.7% to 99.8%, 94.7% to 99.6% and 94.7% to 99.6% nucleotide identity, respectively, and 97.4% to 99.6%, 96.3 to 99.6%, 95.6% to 99.3% amino acid identity, respectively. Of these aa substitutions, 2 substitutions (V208A, A301T) occurred in the N open reading frame (ORF), and 2 substitutions (T56S, T89M) occurred in the F open reading frame (ORF). ConclusionsOne strain QF20100726 of CPIv is successfully isolated, and the phylogenetic trees of N, HN and F genes from the QF20100726 CPIV strain show close phylogenetic relationship with other CPIV isolates. The data provide a valuable molecular biology basis for the studies on prevention and control of CPIV infection.
【Key words】CPIV, Viral isolation, Sequence analysis
doi:10.3969.j.issn.1671-7856. 2016.02.016
【中图分类号】R-33
【文献标识码】A
【文章编号】1671-7856(2016) 02-0077-06
[作者简介]孙明(1963-),男,博士,研究方向:分子病毒学诊断技术。Email: sunming@anheal.com。[通讯作者]陈西钊(1965-),男,博士,研究员,研究方向:动物传染病与预防兽医学。Email: chenxizhao@anheal.com。

