肽核酸钳制-PCR检测K-ras突变法的阳性判断阈值及检测下限的研究
·论著·
肽核酸钳制-PCR检测K-ras突变法的阳性判断阈值及检测下限的研究
丁佳寅高军李泉江金晶吴洪玉满晓华李兆申
【摘要】目的确定肽核酸钳制-PCR(PNA-PCR)检测K-ras突变的阳性判断阈值(Cut off)和检测下限。方法将含K-ras基因 12或13密码子突变的胰腺癌细胞株PANC1、SW1990基因组DNA以不同的浓度分别与K-ras野生型的胎盘DNA混合配制成含不同突变率(0、0.1%、0.2%、0.4%、0.8%、1.6%、3.1%、6.25%、12.5%、25%、50%)的样本,并制备1%突变率PANC1细胞及30%突变率SW1990细胞DNA总量分别为50、20、5、1 ng和50、10、5、1 ng的样本。应用PNA-PCR方法检测样本中的K-ras基因12、13密码子突变,收集它们的突变Ct值、总体Ct值,并计算出ΔCt值(突变Ct值-总体Ct值)。实验重复10次。采用受试者工作特征(ROC)曲线分析ΔCt值,确定K-ras基因突变的最适Cut off值,计算诊断阳性率及确定诊断下限。结果所有不同突变率的PANC1细胞12密码子突变以及SW1990细胞13密码子突变的突变Ct值及ΔCt值与阴性对照样本的差异均有统计学意义(P值均<0.05),而总体Ct值与阴性对照样本的差异无统计学意义。诊断K-ras基因12密码子突变的ROC曲线下面积(AUC)为0.926,最适Cut off值为11,相应的敏感度和特异度为84%和100%,检出下限为0.4 ng;诊断K-ras基因13密码子突变的AUC为0.906,最适Cut off值为9.5,相应的敏感度和特异度为71%和100%,检测下限为1.5 ng。固定突变率的检测结果进一步确定上述的检测下限。结论成功确立了PNA-PCR法检测K-ras基因12、13密码子突变的Cut off值和检测下限,达到临床应用的要求。
【关键词】K-ras基因;突变;肽核酸类;聚合酶链反应
DOI:10.3760/cma.j.issn.1674-1935.2015.01.008
基金项目:国家自然科学基金重大国际合作项目(30910103911),上海市重点科技攻关项目(11441901800)
收稿日期:(2014-10-31)
Identification of cut off value and lower limit of detection for K-ras mutation by PNA-PCR clamping methodDingJiayin,GaoJun,LiQuanjiang,JinJing,WuHongyu,ManXiaohua,LiZhaoshen.DepartmentofGastroenterology,ChanghaiHospital,SecondMilitaryUniversity,Shanghai200433,China
Correspondingauthor:GaoJun,Email:13816012151@163.com
Abstract【】ObjectiveTo determine the lower limit of detection (LLOD) and cut off values of K-ras mutation detection by peptide nucleic acid (PNA) clamping-PCR. MethodsThe genomic DNA of pancreatic cancer cell lines (PANC1 and SW1990) with codon12, 13 mutation and the genomic DNA of placenta with K-ras wild type were mixed and diluted serially into samples with different mutation rate (0, 0.1%, 0.2%, 0.4%, 0.8%, 1.6%, 3.1%, 6.25%, 12.5%, 25%, 50%), PANC1 cells with 1% mutation rate and SW1990 cells with 30% mutation rate and 4 samples with the quantity of DNA was 50, 20, 5, 1 ng and 50, 10, 5, 1 ng was prepared. Codon 12, 13 mutation of K-ras was determined by PNA-PCR, and the mutation Ct values, overall Ct values were collected, and the ΔCt values (mutation Ct values-overall Ct values) were calculated, and the tests were repeated for 10 times. ROC curve was used to analyze the ΔCt values and determine the best cut off values for K-ras mutation, and the positive diagnostic rate, LLOD was evaluated. ResultsThe mutation Ct, ΔCt values of codon 12 mutation of PANC1 and codon 13 mutation of SW1990 of all the different mutation rates were statistically significantly different (P<0.05) when compared with negative

作者单位:200433上海,第二军医大学长海医院消化内科
通信作者:高军,Email:13816012151@163.com
control group, but the overall Ct values were not statistically significantly different from that of negative control group. For detection of K-ras codon 12 mutation by ROC curve, the relevant area of ROC curve (AUC) was 0.926, the optimum cut off value of ΔCT was 11, the sensitivity and specificity were 84% and 100%, respectively, and the LLOD was 0.4 ng. For detection of K-ras codon 13 mutation by ROC curve, the relevant AUC was 0.906, the optimum cut off value of ΔCT was 9.5, the sensitivity and specificity were 71% and 100%, respectively, and the LLOD was 1.5 ng. The mutation detection results of fixed rate further confirmed the LLOD. ConclusionsThis study successfully defines LLOD and cut off value of PNA clamping-PCR/K-ras method in detection of K-ras 12 and 13 codon mutations. This method meets the requirement of clinical application.
【Key words】K-ras gene;Mutation;Peptide nucleic acids;Polymerase chain reaction
K-ras基因在胰腺癌中的突变率高达90%以上[1],在结直肠癌和肺癌中为30%~40%[2-4]。在K-ras基因突变中,2号外显子的12、13密码子突变占90%以上[5-8]。目前,K-ras基因突变检测已成为肿瘤患者进行表皮生长因子受体(epithelia groth factor receptor, EGFR)单克隆抗体治疗时的必检项目[9-11],且K-ras基因的突变对患者生存及预后有重要影响[12]。由于临床样本中提取的DNA往往夹杂大量的野生DNA,为避免野生DNA的干扰以获得较高的灵敏度和特异度,本课题组前期建立了肽核酸(PNA)钳制-PCR(PNA-PCR)检测K-ras突变的方法,即利用PNA钳制样本中野生DNA的扩增。本研究进一步明确该方法的检测下限及阳性判断标准,为今后检测临床样本提供实验依据。
材料和方法
一、K-ras基因突变的细胞株及野生型K-ras基因组织的DNA抽提
PANC1细胞株为K-ras基因12密码子突变,SW1990细胞株为K-ras基因13密码子突变。两细胞株为上海长海医院消化内科实验室保存,常规培养、传代,使用细胞株DNA提取试剂盒(天根生物公司)抽提PANC1和SW1990细胞总DNA,按试剂盒说明书操作。
胎盘组织委托Invitrogen公司进行测序分析,证实胎盘DNA中的K-ras基因为野生型,故以胎盘组织DNA作为阴性对照。取适量冻存的胎盘组织,在液氮中捣碎,使用组织DNA提取试剂盒(天根生物公司)抽提胎盘DNA,按说明书操作。
二、K-ras基因突变DNA参考样本制备
将两株细胞DNA从50 ng/μl倍比稀释至0.1 ng/μl,共11个梯度。将突变DNA与突变+野生DNA比例为50%、25%、12.5%、6.25%、3.1%、1.6%、0.8%、0.4%、0.2%、0.1%、0的每个浓度梯度的细胞DNA与胎盘DNA混合作为参考样本,DNA总量为50 ng。另外,将PANC1 DNA与胎盘DNA混合比例为1%的样本DNA总量分为50、20、5、1 ng 4种参考样本;将SW1990 DNA与胎盘DNA混合比例为30%的样本DNA总量分为50、10、5、1 ng 4种参考样本。
三、PNA-PCR法检测K-ras基因突变
将上述各个K-ras基因突变DNA样本分别加入含检测12、13密码子突变和DNA总量的MGB探针(Invitrogen公司)的PCR反应体系中进行反应。PCR总反应体系:参考样本2.5μl,Master Mix 7.5 μl,引物(10 pmol/μl)1.5 μl,PNA1(75 pmol/μl)或PNA2(100 pmol/μl)或鲑精液(20 ng/μl)2.5 μl,MGB探针1 μl,总体积为15 μl。其中PNA1加入检测12密码子突变反应体系,PNA2加入检测13密码子突变反应体系,鲑精液加入检测DNA总量反应体系。引物和PNA序列拟申请专利保护,未列出。反应条件:95℃ 10 min,95℃ 10 s、70℃ 10 s、45℃ 5 s、72℃ 30 s,50个循环。在每个循环末收集荧光信号,通过仪器自带软件获取突变Ct值、总体Ct值及ΔCt值(突变Ct值-总体Ct值)。实验独立重复10个批次。
四、统计学处理
采用SPSS 15.0软件进行统计分析。各K-ras基因突变参考样本的突变Ct值、总体Ct值及ΔCt值与阴性样本胎盘DNA之间的比较采用非参数检验。采用受试者工作特征(ROC)曲线分析法选定阳性判断阈值(Cut off)。检测下限和阳性检出率的确定以及检测下限的验证采用交叉表法。P<0.05为差异具有统计学意义。
结果
一、K-ras基因突变样本与阴性对照样本Ct值、总体Ct值及ΔCt值的比较
所有不同突变率的PANC1细胞12密码子突变以及SW1990细胞13密码子突变的突变Ct值及ΔCt值与阴性对照样本的差异均有统计学意义(P值均<0.05),而总体Ct值与阴性对照样本的差异无统计学意义(图1、2)。
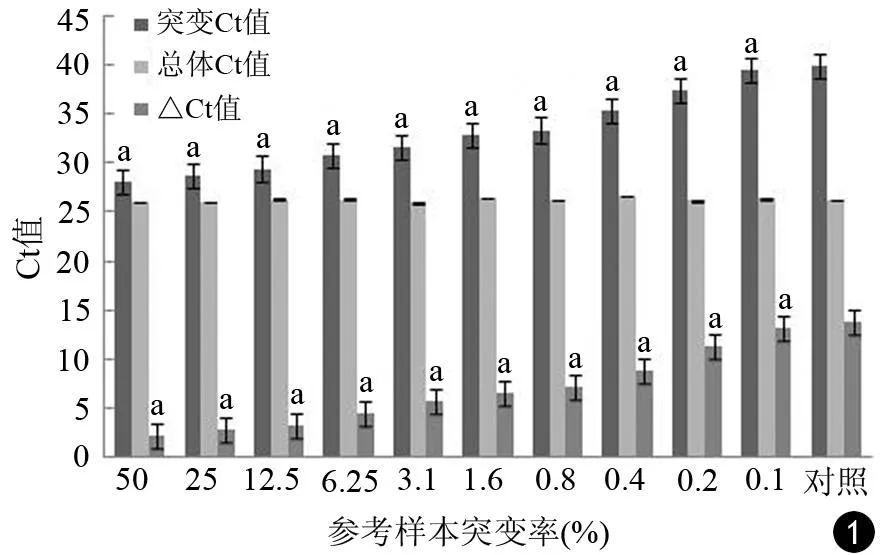
与对照样本比较, aP<0.05 图1 PANC1细胞不同的K-ras 基因12密码子突变率样本及胎盘对照样本的突变Ct值、总体Ct值及ΔCt值
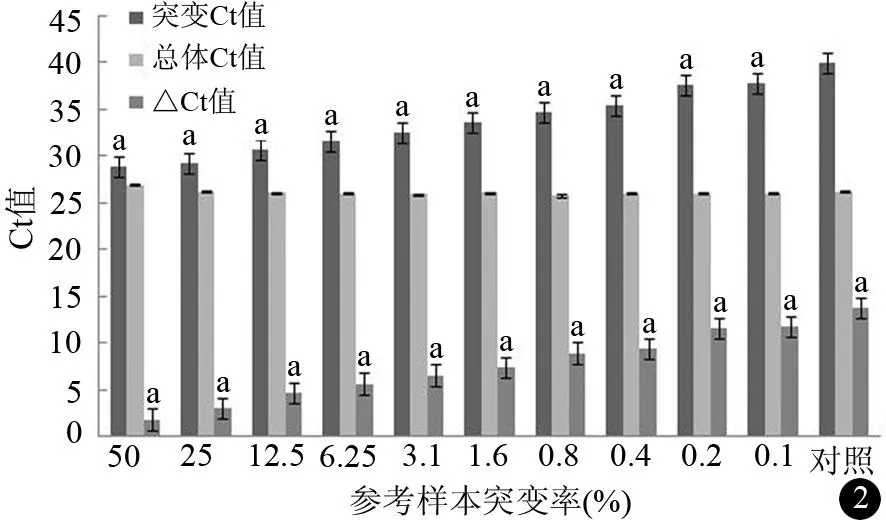
与对照样本比较, aP<0.05 图2 SW1990细胞不同的K-ras基因13密码子突变率样本及胎盘对照样本的突变Ct值、总体Ct值及ΔCt值
二、阳性诊断标准及检查下限的确定
PANC1细胞K-ras 基因12密码子突变的ROC曲线见图3,相应的曲线下面积(AUC)为0.926,对应的Cut off值为11,诊断12密码子突变的敏感度和特异度分别为84%(84/100)和100%(100/100)。对0、0.1%、0.2%、0.4%、0.8%、1.6%、3.1%、6.25%、12.5%、25%、50%突变率参考样本的阳性检出率分别为0、10%、40%、90%、100%、100%、100%、100%、100%、100%、100%,检测下限为0.8%的突变率(表1),即能检出≥0.4 ng的12密码子突变DNA。
SW1990细胞K-ras基因13密码子突变的ROC曲线见图4,相应的AUC为0.906,相应的Cut off值为9.5,诊断13密码子突变的敏感度和特异度分别为71%(71/100)和100%(100/100)。 在上述各突变率梯度参考样本的阳性检出率分别为0、10%、10%、60%、60%、70%、100%、100%、100%、100%、100%,检测下限为3.1%突变率(表2),即能检出≥1.5 ng的13密码子突变DNA。
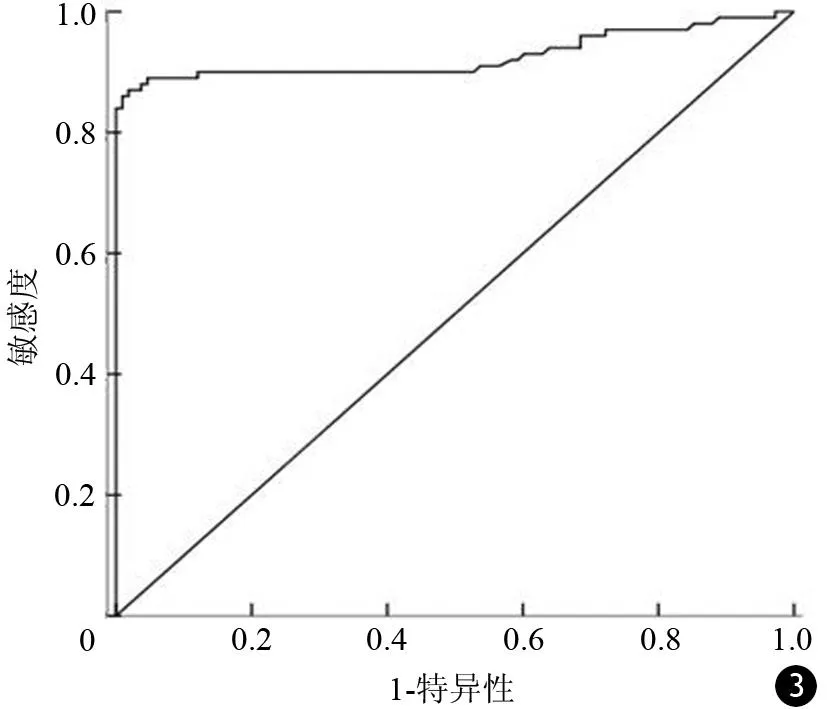
图3 PANC1细胞K-ras基因12密码子突变ΔCt值诊断价值的ROC曲线
表1PANC1细胞K-ras基因12密码子突变的阳性检出率
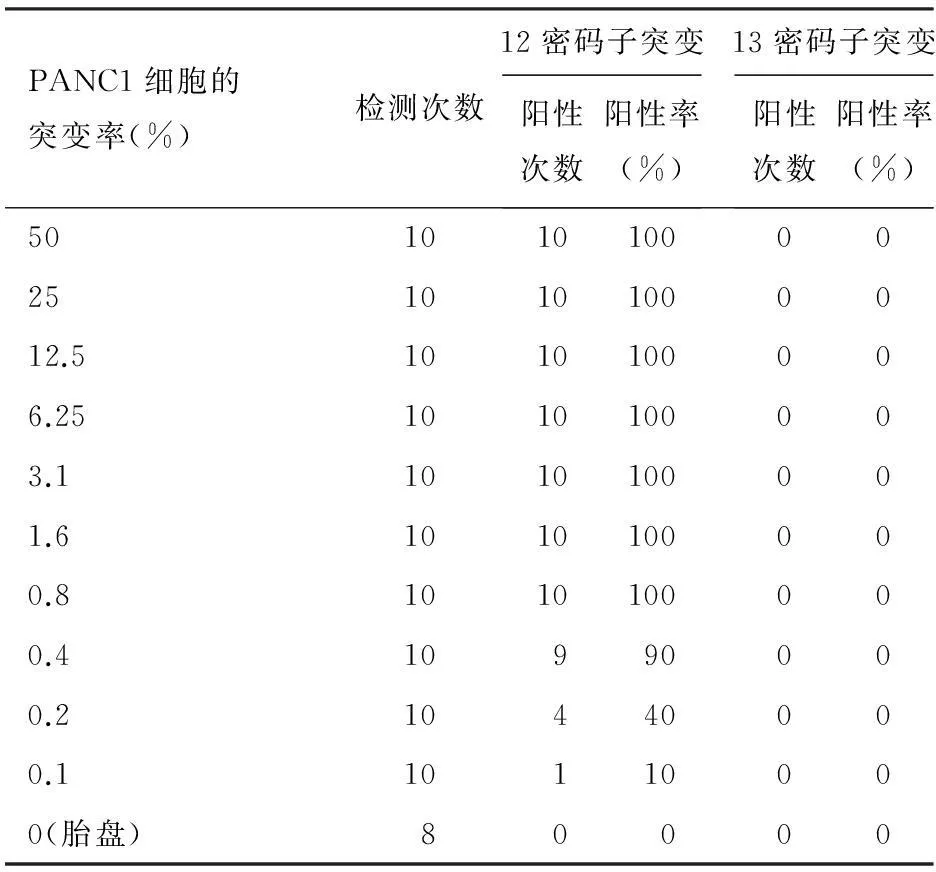
PANC1细胞的突变率(%)检测次数12密码子突变阳性次数阳性率(%)13密码子突变阳性次数阳性率(%)501010100002510101000012.51010100006.251010100003.11010100001.61010100000.81010100000.410990000.210440000.110110000(胎盘)80000
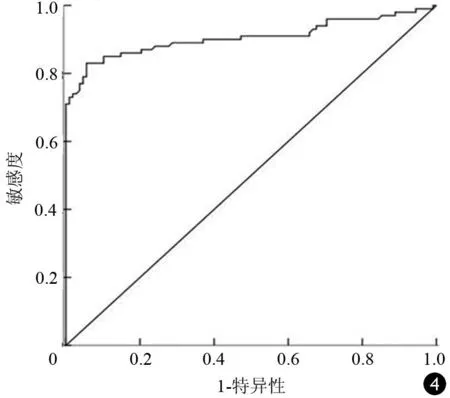
图4 SW1990细胞K-ras基因13密码子突变ΔCt值诊断价值的ROC曲线
表2SW1990细胞K-ras基因13密码子突变的阳性检出率
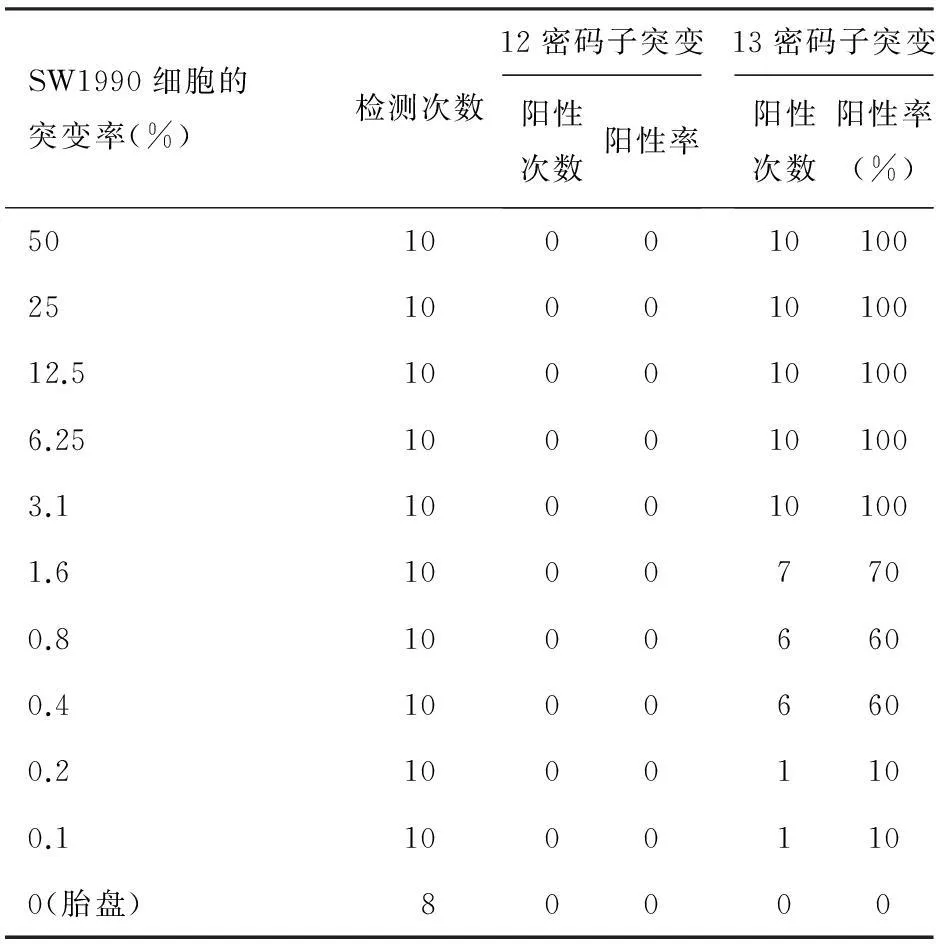
SW1990细胞的突变率(%)检测次数12密码子突变阳性次数阳性率13密码子突变阳性次数阳性率(%)501000101002510001010012.51000101006.251000101003.11000101001.610007700.810006600.410006600.210001100.110001100(胎盘)80000
通过对固定突变率而DNA总量不同的参考样本的检测,能检出突变率为1%、DNA总量为50 ng的PANC1参考样本,即0.5 ng的突变DNA(表3);能检出突变率为30%,DNA总量为5 ng的SW1990参考样本,即1.5 ng的突变DNA(表4)。
表31%突变率PANC1细胞不同DNA总量的K-ras基因12密码子突变阳性检出率
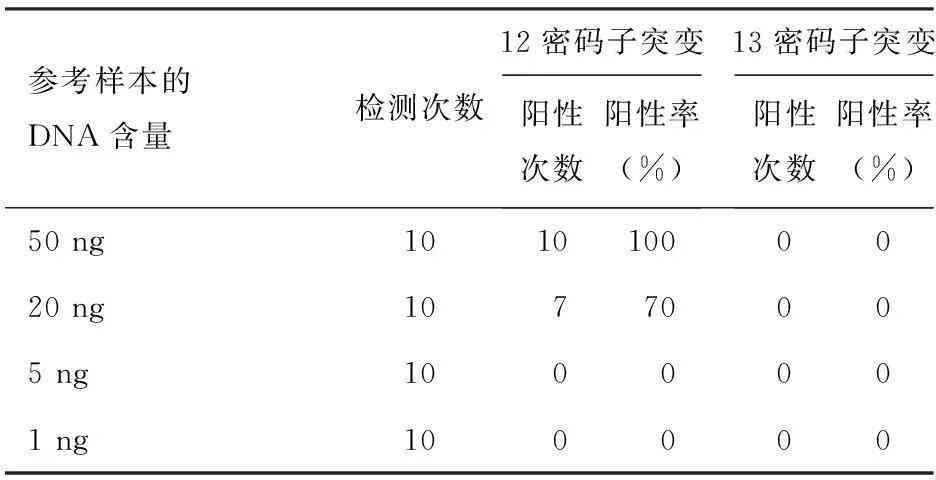
参考样本的DNA含量检测次数12密码子突变阳性次数阳性率(%)13密码子突变阳性次数阳性率(%)50ng10101000020ng10770005ng1000001ng100000
讨论
近年来,K-ras基因突变检测已广泛应用于指导肿瘤患者进行靶向药物治疗。所有转移性结直肠癌和非小细胞肺癌患者使用EGFR抑制剂(如西妥昔单抗和帕尼单抗)之前都需要进行K-ras基因突变检测,只有K-ras基因为野生型的患者才能接受EGFR抑制剂靶向治疗[13-14]。既往报道的K-ras基因突变检测方法较多,主要有直接测序法、焦磷酸测序法、突变特异性扩增系统(ARMS)、高分辨率熔解曲线分析法(HRM)和StripAssay法。这些方法的灵敏度各不相同,但都存在操作繁琐、灵敏度不够、对设备要求高、成本过高等缺陷,难以在临床推广普及。Jancik等、Tsiatis等和Ogino等[15-17]用含突变型和野生型K-ras基因的肿瘤细胞株DNA配制成不同拷贝数的梯度模板,测定了上述5种K-ras突变检测法的检测下限:直接测序法为25%~30%,焦磷酸测序法为5%~10%,ARMS法为1%,HRM法为5%~10%,StripAssay法小于1%。Tsiatis等[16]分析了上述各方法的检测下限,认为测序法、HRM法和StripAssay法在样品检测值接近检测下限时易受读者主观因素的干扰,焦磷酸测序法和ARMS法虽然受读者主观因素干扰小,但灵敏度有待提高。上述文献中只使用了K-ras 12密码子突变的细胞株DNA确立检测方法的灵敏度和检测下限,并未提及检测的DNA总量。本研究除了确立K-ras 12密码子突变细胞株DNA的检测灵敏度和检测下限之外,还对K-ras 13密码子突变细胞株DNA的检测灵敏度和检测下限进行了研究,并且对DNA总量的投入做了限定,并用参考样本对检测下限进行了验证。由于本研究将含突变型基因的细胞株DNA与K-ras野生型的组织DNA混合制成参考样本,可以最大限度地模拟临床样本,所以确立的细胞株DNA检测下限和最适Cut off值更具实用价值,为临床样本的检测提供了重要的参数。
表430%突变率SW1990细胞不同DNA总量的K-ras基因13密码子突变阳性检出率
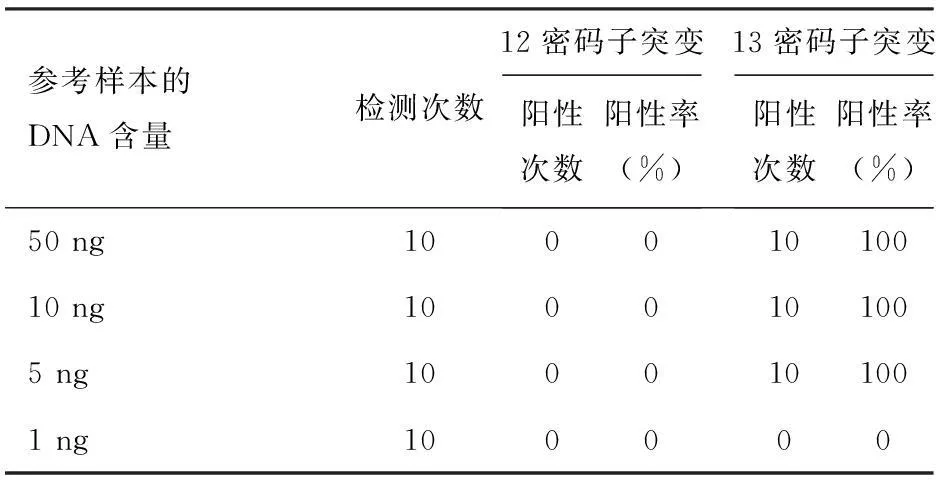
参考样本的DNA含量检测次数12密码子突变阳性次数阳性率(%)13密码子突变阳性次数阳性率(%)50ng10001010010ng1000101005ng1000101001ng100000
参考文献
[1]Almoguera C,Shibata D,Forrester K,et al. Most human carcinomas of the exocrine pancreas contain mutant c-K-ras genes[J].Cell,1988,53(4):549-554.
[2]De Roock W,De Vriendt V,Normanno N,et al.KRAS,BRAF,PIK3CA and PTEN mutations:implications for targeted therapies in metastatic colorectal cancer[J].Lancet Oncol,2011,12(6):594-603.
[3]Kalikaki A,Koutsopoulos A,Trypaki M,et al.Comparison of EGFR and K-ras gene status between primary tumours and corresponding metastases in NSCLC[J].Br J Cancer,2008,99(6):923-929.
[4]Rajagopalan H,Bardelli A,Lengauer C,et al.Tumorigenesis:RAF/RAS oncogenes and mismatch-repair status[J].Nature,2002,418(6901):934.
[5]Bos JL.Ras oncogenes in human cancer:a review[J].Cancer Res,1989,49(17):4682-4689.
[6]Adjei AA.Blocking oncogenic Ras signaling for cancer therapy[J].J Natl Cancer Inst,2001,93(14):1062-1074.
[7]Ma ES,Wong CL,Law FB,et al.Detection of KRAS mutations in colorectal cancer by high resolution melting analysis[J].J Clin Pathol,2009,62(10):886-891.
[8]Whitehall V,Tran K,Umapathy A,et al.A multicenter blinded study to evaluate KRAS mutation testing methodologies in the clinical setiing[J].J Mol Diagn,2009,11(6):543-552.
[9]Allegra CJ,Jessup JM,Somerfield MR,et al.American Society of Clinical Oncology provisional clinical opinion:testing for KRAS gene mutations in patients with metastatic colorectal carcinoma to predict response to anti-epidermal growth factor receptor monoclonal antibody therapy[J].J Clin Oncol,2009,27(12):2091-2096.
[10]Wierzbicki R,Jonker DJ,Moore MJ,et al.A phase Ⅱ,multicenter study of cetuximab monotherapy in patients with refractory,metastatic colorectal carcinoma with absent epidermal growth factor receptor immunostaining[J].Invest New Drugs,2011,29(1):167-174.
[11]Krens LL,Baas JM,Gelderblom H,et al.Therapeutic modulation of K-ras signaling in colorectal cancer[J].Drug Discov Today,2010,15(13-14):502-516.
[12]Jancik S, Drabek J, Radzioch D, et al. Clinical relevance of KRAS in human cancers[J]. J Biomed Biotechnol, 2010, 2010: 150960.
[13]De Roock W, Claes B, Bemasconi D, et al. Effects of KRAS, BRAF, NRAS, and PIK3CA mutations on the efficacy of cetuximab plus chemotherapy in chemocherapy-refractory metastatic colorectal cancer: a retrospective consortium analysis[J]. Lancet Oncol, 2010, 11(8):753-762.
[14]Jancik S, drabek J, berkovcova J, et al. A comparison of Direct sequencing. Pyosequencing. High resolution meltinganalysis. TheraScreen Dxs, and the K-ras Strip Assay for detecting KRAS mutations in non small cell lung carcinomas[J]. J Exp Clin Cancer Res, 2012,31:79.
[15]Jancik S,Drabek J,Berkovcova J,et al.A comparison of Direct sequencing,Pyrosequencing,High resolution melting analysis,TheraScreen DxS and the K-ras StripAssay for detecting KRAS mutations in non small cell lung carcinomas[J].J Exp Clin Cancer Res,2012,31:79.
[16]Tsiatis AC,Norris-Kirby A,Rich RG,et al.Comparison of Sanger sequencing,pyrosequencing and melting curve analysis for the detection of KRAS mutations:diagnostic and clinical implications[J].J Mol Diagn,2010,12(4):425-432.
[17]Ogino S,Kawasaki T,Brahmandam M,et al.Sensitive sequencing method for KRAS mutation detection by Pyrosequencing[J].J Mol Diagn,2005,7(3):413-421.
(本文编辑:屠振兴)
欢迎订阅《中华胰腺病杂志》

