lpxA/lpxC/lpxD 基因突变致鲍曼不动杆菌耐多黏菌素作用机制
张文利,Joan-Miquel Balada-Llasat,Vijay Pancholi ,Bhowmik Aurosree,Bhavani Gopalakrishnan,Preeti Pancholi
鲍曼不动杆菌是医院获得性菌血症、重症肺炎、脑膜炎、尿路感染、血流和身体其他部位感染的主要病原菌[1]。近年来,多重耐药鲍曼不动杆菌(MDRAB)感染的急剧增加是一个非常棘手的问题[2-4]。由于抗生素选择性压力导致MDR-AB的出现[5],抗菌药物普遍耐药致使MDR-AB 的治疗面临极大挑战。
多黏菌素往往是最后可以选择用于治疗MDR铜绿假单胞菌和不动杆菌属的抗生素[6-7]。多黏菌素是环状多肽的多聚阳离子抗生素,依靠与细菌外膜脂多糖上阴离子相互作用,使膜溶解[8]。
耐多黏菌素现象罕见,但鲍曼不动杆菌耐多黏菌素已被报道[9-11]。而多黏菌素耐药鲍曼不动杆菌的出现[12]已成为一个令人越来越担忧的问题。
肠道沙门菌和铜绿假单胞菌对多黏菌素的耐药机制主要包括细菌外膜脂多糖(LPS)上脂质A 的修饰、缺失或完全丧失,从而减少或去除与多黏菌素相互作用的负电荷[13-14]。此外,负责脂质A 生物合成的lpxA/lpxD/lpxC 的基因突变[15-16]会导致耐多黏菌素。鲍曼不动杆菌对多黏菌素耐药的机制不断 有文献报道[17-18]。我们的 研究表明,lpxA、lpxC和lpxD 基因的突变会导致细胞膜脂质A 生物合成的成分改变,使得MDR-AB对多黏菌素耐药性增强。了解多黏菌素耐药机制可以提供研发治疗MDR-AB感染新药的依据。
1 材料与方法
1.1 材料
1.1.1 菌株来源 分离自俄亥俄州立大学Wexner医学中心临床患者的25 株鲍曼不动杆菌用Micro Scan(Siemens Healthcare Diagnostics,IL,USA)鉴定,药敏试验采用Micro Scan和(或)E试验法(bioMérieux,Marcy l′Etoile,France)。其中18株是耐多黏菌素株(Col-R)(R1、R2、R4、R5、R6、R7、R9、R10、R11、R14、R15、R16、R17、R18、R19、R20、R22、R23),3株是多黏菌素依赖株(Col-D)(R3D、R12D、R13D)[19],4株是多黏菌素敏感株(Col-S)(S1、S2、S3、S5)。这些初选的Col-R 和Col-D 菌株是基于E 试验测试结果最低抑菌浓度(MIC)≥4 mg/L 被选出的。质控菌株为鲍曼不动杆菌ATCC19606。
1.1.2 培养基和抗菌药物 药敏试验用MH 琼脂培养基。抗菌药物多黏菌素E 试验药敏条为AB Biodisk公司产品。
1.2 方法
1.2.1 抗生素的MIC测定 根据美国临床和实验室标准化协会(CLSI)指南[20],用Micro Scan和E试验法检测抗生素的MIC 值。用E 试验法检测25株临床分离鲍曼不动杆菌对多黏菌素的MIC值,多黏菌素耐药被定义为MIC≥4 mg/L。铜绿假单胞菌ATCC27853和鲍曼不动杆菌ATCC19606(多黏菌素MIC为0.125 mg/L)作为对照。
1.2.2 薄层透射电子显微镜(TEM)观察细胞壁和细胞膜结构 6 株鲍曼不动杆菌(S1、S2、R5、R9、R3D、R13D)接种在新鲜的5 mL 无任何抗菌药物TSB培养基和含有10 mg/L 多黏菌素TSB中生长(Teknova,Fisher Scientific,USA),直到光密度在600 nm(A600)时为1.0。随后细菌在室温下3 100 转/min离心10 min。菌液用1×PBS缓冲液洗2次后混悬在1 mL含4%多聚甲醛和2.5%戊二醛的缓冲液中。混悬液再用琼脂糖包埋制成薄层切片后用俄亥俄州立大学Wexner医学中心的FEI Tecnai TEM(The FEI TecnaiTM,OR,USA)和成像设备(CMIF)观察细胞壁和细胞膜结构。
1.2.3 气相色谱-质谱仪(GC-MS)对细胞脂肪酸进行分析 对数生长期的鲍曼不动杆菌用1 mL PBS洗后离心去除上清液,细菌沉淀称重后提交到俄亥俄州立大学 Wexner 医学中心化学仪器中心(CCIC)用GC-MS(Finnigan Trace,TX,USA)的质谱及蛋白质组学方法分析细胞脂肪酸。样品脂肪酸各组分含量与Sherlock模式识别软件的存储数据库对比后得到。
1.2.4 脂质A 生物合成基因突变的检测 用基因组DNA 提取试剂盒(UltraClean Mcrobial DNA Isolation Kit,MO BIO Laboratories,Inc.)提取鲍曼不动杆菌基因组DNA,通过聚合酶链反应(PCR)扩增脂质A 的生物合成基因lpxA、lpxC 和lpxD,3种引物序列见表1。PCR 扩增产物由西雅图生物研究所进行基因测序并由序列分析仪进行分析(Seattle Biomedical Research Institute,USA)。用美国国家生物技术信息中心网站(http://www.ncbi.nlm.nih.gov/BLAST/)中BLASTN 程序比对同源序列。
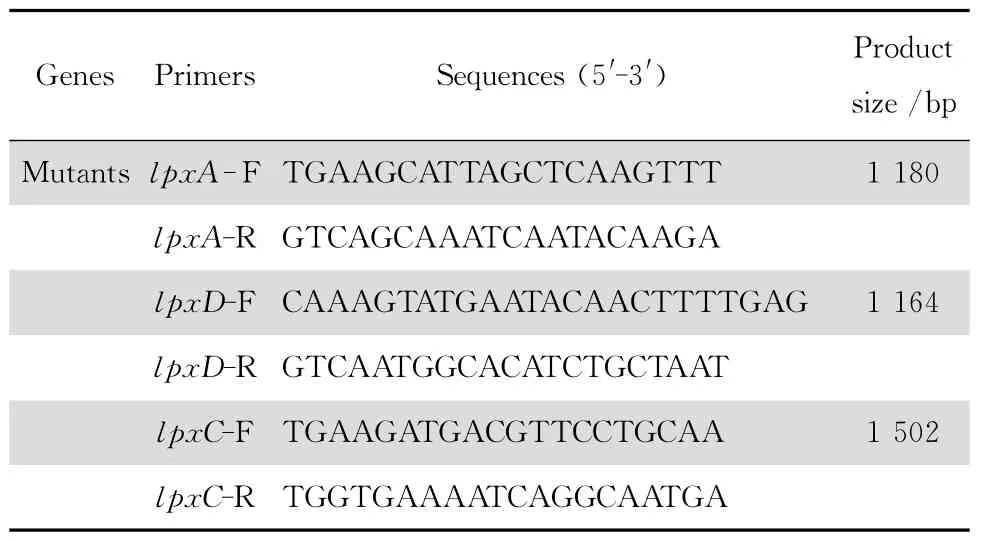
表1 检测基因突变所用的PCR 引物Table 1 Primers used in PCR detection of gene mutations
2 结果
2.1 多黏菌素MIC值
用E 试验检 测25 株MDR-AB 临床分 离株对多黏菌素的MIC 值。18 株Col-R 菌和3株Col-D菌对多黏菌素MIC的范围为8~256 mg/L(图1、表2)。
2.2 Col-R 和Col-D 株的细胞膜改变
应用TEM 观察了2株Col-S,2 株Col-R 和2株Col-D 共6株鲍曼不动杆菌的内、外膜结构,比较Col-S株、Col-R 株和Col-D 株细胞膜之间的成像差异。TEM 显示2株Col-S菌株具有完整、厚和均匀的内、外膜。2 株Col-R 菌株的外侧和内侧膜的完整性破坏,失去了膜的均匀性,多数似串珠样,甚至有些膜还失去了一些外周胞质空间。2株Col-D 菌株的外膜和内膜出现分叉,丧失完整性,外周胞质空间缺乏均一性(图2)。
图1 Col-R和Col-D鲍曼不动杆菌Figure 1 Col-R and Col-D A.baumannii strains
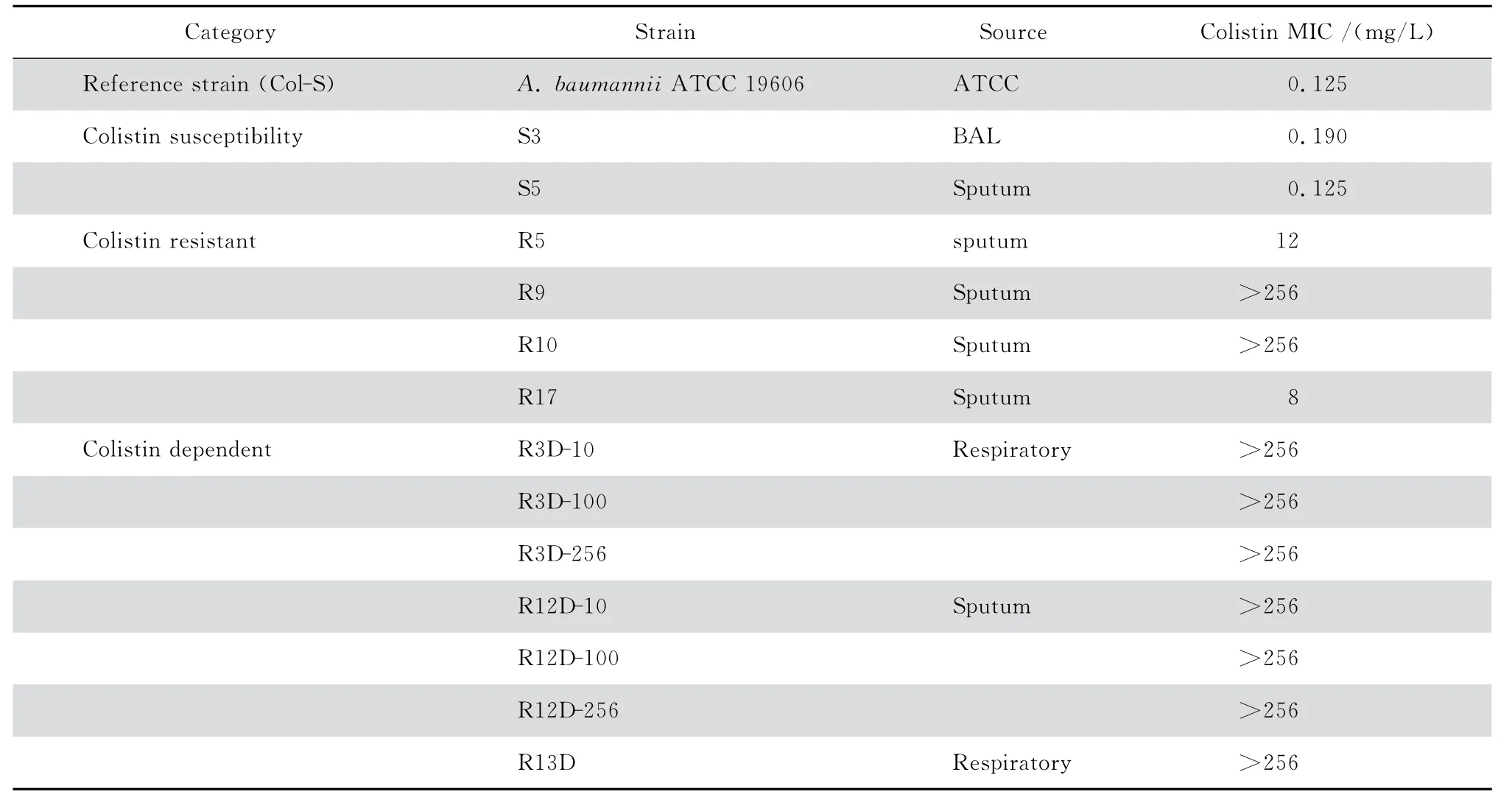
表2 鲍曼不动杆菌不同临床株对多黏菌素的MIC值Table 2 Minimum inhibitory concentrations of colistin against different strains of Acinetobacter baumannii
2.3 脂质A 的脂肪酸成分分析
我们用GC-MS 法定量分析脂肪酸组成,发现Col-R 菌株和Col-D 菌株的脂肪酸组成成分改变与鲍曼不动杆菌脂质A 合成缺乏或丧失相关(图3)。与Col-S组相比,Col-R 组和Col-D 组菌株脂质正常成分的合成降低并有错误成分合成,特别是在低碳链上总脂质组分中的脂肪酸里缺少了乙醇成分,还有许多未确定的脂肪酸峰也出现在Col-R 组和Col-D 组菌株的脂质合成中。
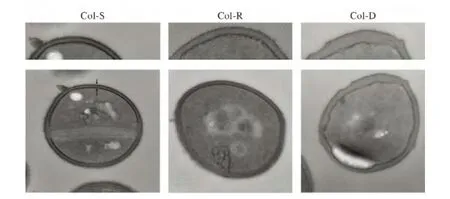
图2 鲍曼不动杆菌细胞膜TEM 观察结果Figure 2 Images obtained by transmission electron microscopy of A.baumannii
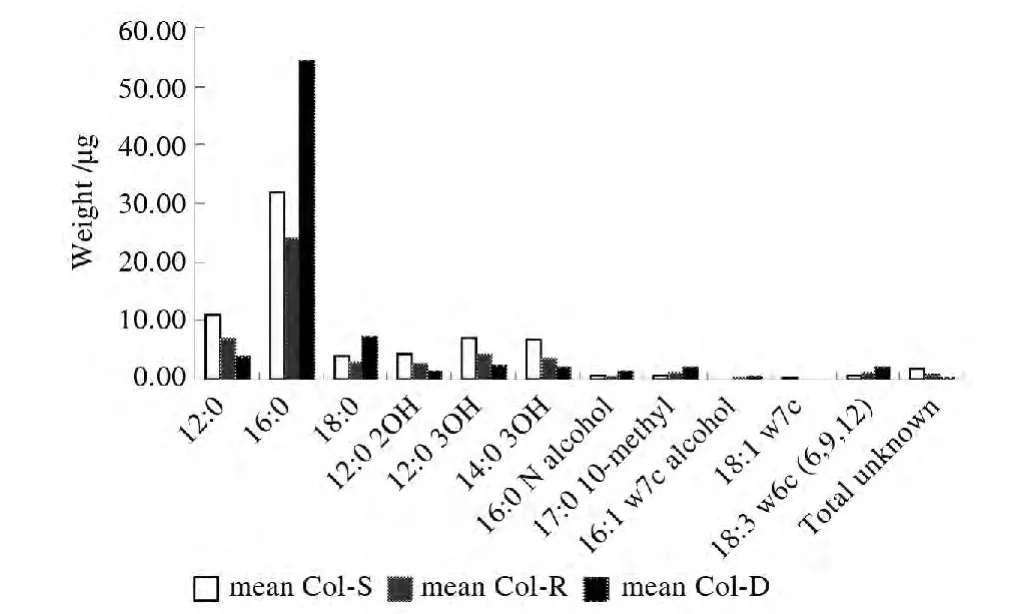
图3 GC-MS数据分析Figure 3 Gas chromatography-mass spectrometry data analysis
2.4 Col-R 和Col-D 菌株基因突变分析
对2株Col-S、18 株Col-R 和3 株Col-D 鲍 曼不动杆菌lpxA、lpxC 和lpxD 基因序列分析发现,与标准株ATCC19606相比,Col-R 和Col-D 菌株中都有部分基因发生了突变,突变范围从单点突变直至大片段缺失(表3)。在lpxA 基因,R3D 有11bp核苷酸缺失导致23位点缬氨酸(V)之后的氨基酸移码。R12D 有一个核苷酸的插入突变导致242位点后氨基酸错译。Col-S、ATCC19606 与其他Col-R 菌株之间无差异。在lpxC 基因,分别有2 个核苷酸的替换导致氨基酸的改变和1个插入突变导致氨基酸错译。同样,lpxD 基因分别在14 株Col-R中和3株Col-D 中有3个核苷酸的替换和1株Col-R 中1个碱基删除导致氨基酸移码。

表3 多黏菌素耐药鲍曼不动杆菌脂质A 生物合成基因突变Table 3 Mutations identified in the lipid A biosynthesis genes of colistin-resistant A.baumannii
3 讨论
本研究收集的所有多黏菌素耐药鲍曼不动杆菌株均来自临床感染患者MDR分离株,其中还发现了3株多黏菌素依赖株(Col-D)[19]。作为一种老药,多黏菌素重新用于临床治疗MDR-AB的感染。在治疗过程中多黏菌素耐药性以及体外黏菌素或多黏菌素依赖性已被发现和报道[19,21]。多黏菌素E试验测试显示,所有这3株Col-D菌株产生的MIC均超过256 mg/L,其他Col-R 菌株的MIC 范围分别从8~256 mg/L不等。
为了评价鲍曼不动杆菌对黏菌素耐药机制,我们将25株临床感染鲍曼不动杆菌菌株分为3组:4株Col-S为一组,18株Col-R 为一组,3株Col-D 为一组。几种多黏菌素耐药机制的研究数据显示,Col-S与Col-R、Col-D 之间均存在明显差异。
多黏菌素耐药最常见的机制就是多黏菌素作用的第一个位点——细胞膜构成成分LPS 的修改[22-24]。我们通 过TEM 观察了2 株Col-S,2 株Col-R 和2 株Col-D 菌 株,发现在Col-S 和Col-R、Col-D 之间有一些令人关注的差异。例如,我们发现2株Col-S菌株有着完整、厚和均匀的内膜与外膜,然而,2株Col-R 菌株和2株Col-D 内外膜的完整性均被破坏,失去了均匀性、出现分叉的膜、甚至大多数还出现串珠 状膜,所有Col-R 和Col-D 菌体细胞内外膜间都失去了部分间隙。这些Col-R 和Col-D 株内外细胞膜合成被干扰和再加工,改变了细胞膜通透性和抗生素结合位点,结果对多黏菌素耐药性增强[22,25]。
鲍曼不动杆菌脂肪酸成分显示3-羟基十四烷酸(3-hydroxytetradecanoic acid,3-C14OH)、3-羟基月桂酸(3-hydroxydodecanoic acid,3-C12OH)、2-羟基十四烷酸(2-hydroxytetradecanoic acid,2-C14OH)和月桂酸(dodecanoic acid,C12)、棕榈酸(hexadecanoic acid,C16)、硬脂酸(octadecanoic acid,C18)是脂质A 的重要化学成分[24]。为了判析临床Col-R和Col-D 菌株不完整的细胞膜是否是源于脂肪酸成分的定量改变,我们通过GC-MS比较了Col-S株与Col-R、Col-D 菌株全部脂肪酸的组成,确切的数据显示,长碳链饱和脂肪酸关键成分,包括C12、C16、C18有明显的数量改变。从Col-S株的平均值分别与Col-R株和Col-D株的值比较,C12所占比重依次降低,但C16、C18所占比重依次增高;在乙醇侧链和不饱和脂肪酸成分的生物合成上也有明显区别,多种成分生物合成减少,但其他成分增加,如一些多黏菌素耐药和依赖的菌株显示低碳链脂肪酸上几乎没有乙醇侧链,未知脂肪酸的生物合成有降低趋势。GC-MS检测证实这些多黏菌素耐药和依赖的菌株增加了对长链脂肪酸、不饱和脂肪酸的修改以及丢失了许多不明脂肪酸成分。在3组鲍曼不动杆菌详细的脂肪酸组成比较中,认识到Col-R和Col-D菌株组成成分可随多黏菌素暴露浓度改变而有巨大改变这一点是非常重要的。
在细菌脂质A 生物合成途径中,lpxA、lpxC 和lpxD 基因参与了关键的前三步——LPS的疏水性锚合成[22-23],而多黏菌素发挥作用的靶位是细菌外膜的脂质双分子层,即抗菌药物带正电的肽与细菌带负电的LPS内毒素成分脂质A 间的静电相互作用。对于耐药菌株来说,脂质A 发生的各种修饰可以减少或消除负电荷从而影响与抗菌药物的静电相互作用。脂质A 的修饰也许并不影响细菌基础生长,但却可以加强病原体的渗透屏障和影响毒力[22,25]。为了观 察lpxA、lpxC 和lpxD 基因突 变对脂质A 生物合成的影响,用Col-S 和标准菌株ATCC19606做对照,我们运用测序方法分析了Col-R 和Col-D 临床鲍曼株中这3种基因的突变情况。在18株Col-R 和3株Col-D 的3种基因里发现了一定数量的不同基因突变,范围从单点突变到大片段核苷酸缺失。本研究中,3株多黏菌素依赖株中的1株——R3D的lpxA 基因从位点68开始有11个核苷酸被删除导致lpxA 翻译成氨基酸过程中位点23的缬氨酸之后翻译过早终止,结果,脂质A 不能生成从而使LPS缺损,不仅使临床菌株对多黏菌素产生了耐药而且还产生了依赖,这一证据已通过TEM 试验图像和GC-MS脂肪酸数值得到确认。这一发现表明,对多黏菌素产生耐药性的鲍曼不动杆菌临床株存在lpxA 的突变和LPS 的缺失。另一株Col-D(R12D)在lpxC 基因上有3个核苷酸的插入导致脂质A 生物合成第二步的氨基酸错译从而不能产生完整的脂质A;其他的突变是属于单点突变导致单个氨基酸的改变。这些基因突变中,发生数量最多的是多黏菌素耐药株和依赖株(10株Col-R和2株Col-D)在lpxD 基因上发生碱基A 替代G 导致谷氨酸变成赖氨酸(E138K)。序列分析表明,几乎所有被分析的Col-R和Col-D菌株都有lpxA、lpxC 或lpxD 基因的1个或多个突变。
总之,本研究数据清晰地显示,临床Col-R 和Col-D鲍曼不动杆菌对多黏菌素耐药的主要机制是由于脂质A 生物合成涉及的前3个关键合成基因——脂质A 生物合成基因突变导致LPS合成缺失所致。随着多黏菌素在临床上的应用越来越多,我们担心对多黏菌素的耐药性会由于多黏菌素的使用增加而被广泛传播。因此,需要对临床鲍曼不动杆菌的多黏菌素耐药机制进一步研究,为临床有效控制耐药菌克隆株传播及治疗新药的开发提供科学依据。
[1]Munoz-Price LS,Weinstein RA.Acinetobacter infection[J].N Engl J Med,2008,358(12):1271-1281.
[2]Singh H,Thangaraj P,Chakrabarti A.Acinetobacter baumannii:A Brief Account of Mechanisms of Multidrug Resistance and Current and Future Therapeutic Management[J].J Clin Diagn Res,2013,7(11):2602-2605.
[3]Giamarellou H,Antoniadou A,Kanellakopoulou K.Acinetobacter baumannii:a universal threat to public health?[J].Int J Antimicrob Agents,2008,32(2):106-119.
[4]Michalopoulos A,Falagas ME.Treatment of Acinetobacter infections[J].Expert Opin Pharmacother,2010,11(5):779-788.
[5]Snitkin ES,Zelazny AM,Montero CI,et al.Genome-wide recombination drives diversification of epidemic strains of Acinetobacter baumannii[J].Proc Natl Acad Sci USA,2011,108(33):13758-13763.
[6]Nation RL,Li J.Colistin in the 21st century[J].Curr Opin Infect Dis,2009,22(6):535-543.
[7]Rolain JM,Roch A,Castanier M,et al.Acinetobacter baumannii resistant to colistin with impaired virulence:a case report from France[J].J Infect Dis,2011,204(7):1146-1147.
[8]Murray CK,Hospenthal DR.Treatment of multidrug resistant Acinetobacter[J].Curr Opin Infect Dis,2005,18(6):502-506.
[9]Henry R,Vithanage N,Harrison P,et al.Colistin-resistant,lipopolysaccharide-deficient Acinetobacter baumannii responds to lipopolysaccharide loss through increased expression of genes involved in the synthesis and transport of lipoproteins,phospholipids,and poly-β-1,6-N-acetylglucosamine [J].Antimicrob Agents Chemother,2012,56(1):59-69.
[10]Peck KR,Kim MJ,Choi JY,et al.In vitro time-kill studies of antimicrobial agents against blood isolates of imipenem-resistant Acinetobacter baumannii,including colistin-or tigecyclineresistant isolates[J].J Med Microbiol,2012,61(3):353-360.
[11]López-Rojas R,Jiménez-Mejías ME,Lepe JA,et al.Acinetobacter baumannii resistant to colistin alters its antibiotic resistance profile:a case report from Spain[J].J Infect Dis,2011,204(7):1147-1148.
[12]Durante-Mangoni E,Zarrilli R.Global spread of drug-resistantAcinetobacter baumannii:molecular epidemiology and management of antimicrobial resistance[J].Future Microbiol,2011,6(4):407-422.
[13]Jochumsen N,Liu Y,Molin S,et al.A Mig-14-like protein(PA5003)affects antimicrobial peptide recognition in Pseudomonas aeruginosa[J].Microbiology,2011,157(Pt 9):2647-2657.
[14]Sun S,Negrea A,Rhen M,et al.Genetic analysis of colistin resistance in Salmonella enterica serovar Typhimurium [J].Antimicrob Agents Chemother,2009,53(6):2298-2305.
[15]Opiyo SO,Pardy RL,Moriyama H,et al.Evolution of the Kdo2-lipid A biosynthesis in bacteria[J].BMC Evol Biol,2010,10:362.
[16]Vuorio R,Vaara M.Comparison of the phenotypes of the lpxA and lpxD mutants of Escherichia coli[J].FEMS Microbiol Lett,1995,134(2-3):227-232.
[17]García-Quintanilla M,Pulido MR,Moreno-Martínez P,et al.Activity of host antimicrobials against multidrug-resistant Acinetobacter baumannii acquiring colistin resistance through loss of lipopolysaccharide[J].Antimicrob Agents Chemother,2014,58(5):2972-2975.
[18]Beceiro A,Llobet E,Aranda J,et al.Phosphoethanolamine modification of lipid A in colistin-resistant variants of Acinetobacter baumannii mediated by the pmrAB two-component regulatory system[J].Antimicrob Agents Chemother,2011,55(7):3370-3379.
[19]Hawley JS,Murray CK,Jorgensen JH.Development of colistindependent Acinetobacter baumannii-Acinetobacter calcoaceticus Complex[J].Antimicrob Agents Chemother,2007,51(12):4529-4530.
[20]Clinical and Laboratory Standards Institute.Performance standards for Antimicrobial susceptibility testing[S].2013,M100-23.
[21]Olaitan AO,Morand S,Rolain JM.Mechanisms of polymyxin resistance:acquired and intrinsic resistance in bacteria[J].Front Microbiol,2014,26(5):643.
[22]Moffatt JH,Harper M,Harrison P,et al.Colistin Resistance in Acinetobacter baumannii is mediated by complete loss of lipopolysaccharide production[J].Antimicrob Agents Chemother,2010,54(12):4971-4977.
[23]Bainbridge BW,Karimi-Naser L,Reife R,et al.Acyl chain specificity of the acyltransferases LpxA and LpxD and substrate availability contribute to lipid A fatty acid heterogeneity in Porphyromonas gingivalis[J].J Bacteriol,2008,190(13):4549-4558.
[24]Williamson JM,Anderson MS,Raetz CR.Acyl-acyl carrier protein specificity of UDP-GlcNAc acyltransferases from gramnegative bacteria:relationship to lipid A structure[J].J Bacteriol,1991,173(11):3591-3596.
[25]De Castro C,Parrilli M,Holst O,et al.Microbe-associated molecular patterns in innate immunity:Extraction and chemical analysis of gram-negative bacterial lipopolysaccharides[J].Methods in Enzymology,2010,480:89-115.

