抗莠去津抗体基因构建及蛋白结构模拟
东 琴,魏松红*,李平生,王海宁
(沈阳农业大学植物保护学院,辽宁 沈阳 110866)
抗莠去津抗体基因构建及蛋白结构模拟
东琴,魏松红*,李平生,王海宁
(沈阳农业大学植物保护学院,辽宁 沈阳110866)
构建抗莠去津单链抗体基因,采用DNAStar软件对单链抗体基因进行氨基酸序列推导,用生物信息学在线网站预测莠去津单链抗体基本特性及二、三级结构。结果表明,抗莠去津单链抗体基因全长744 bp,对应248 个氨基酸,单链抗体相对分子质量约为26 061.9,等电点预测值7.81,为碱性蛋白质;二级结构中α-螺旋17 处,延伸链85 处,β-转角19 处,无规卷曲127 处;三级结构建模显示,重链和轻链的6个环区共同组成了抗体的抗原结合区,符合单链抗体的结构特点,具有抗原结合位点的空间构象。该研究为抗莠去津单链抗体的进一步表达、纯化和活性研究奠定了基础。
莠去津;单链抗体;结构预测
莠去津又名阿特拉津,是一种三氮苯类选择性除草剂,自上世纪50年代末投入生产后在世界范围内得到广泛应用[1]。国内外的研究表明莠去津对水生动植物、两栖类生物、哺乳动物、人类细胞都有不同程度的损害作用[1-4]。莠去津在环境中的残留不断被检测到,研究莠去津残留的检测方法显得尤为重要。
酶联免疫吸附分析(enzyme-linked immunosorbent assay,ELISA)作为近年来快速发展的一种检测技术已经开始应用到农药残留的检测方面,具有操作简便快速、前处理简单、检测周期短、成本低、检测样本容量大等特点[5-6]。目前已有多种农药的残留检测试剂盒研制成功[7-9]。抗体作为免疫分析技术的核心试剂,化学农药抗体的制备一直是免疫分析技术在农药残留分析领域应用的瓶颈因素:抗体制备过程中的半抗原不易合成、抗体制备周期长、动物选择要求较高,而且动物个体间的差异导致批次间稳定性差,不易于大量制备[10]。高效率、低成本制备特异性强的抗体成为努力探索的目标之一。重组抗体尤其是噬菌体展示技术的运用,使快速获得具有高亲和性及特异性的抗体成为可能[11-12]。单链抗体(single-chain variable fragment,scFv)技术将抗体基因进行加工、改造和重新装配,然后克隆到合适的表达载体中,保持了亲本抗体的抗原亲和活性和特异性[13-14]。因此,构建目标抗体基因,对基因序列进行分析,并推导预测抗体的结构及理化性质对于重组抗体的进一步表达和在免疫检测中的应用具有重要意义。
本研究以三氮苯类选择性除草剂莠去津为研究对象,通过莠去津免疫抗原免疫Balb/C小鼠,构建抗莠去津scFv基因[15],并对其编码蛋白的理化性质、二级和三级结构进行预测[16],为该基因进一步表达莠去津scFv及抗体分子的修饰改造奠定基础,为实现ELISA法快速检测莠去津提供一种获得抗莠去津特异性抗体的新途径。
1 材料与方法
1.1动物、材料与试剂
雌性Balb/c小鼠(6~8 周龄)辽宁长生生物技术有限公司;莠去津免疫抗原沈阳农业大学植物保护学院农药学实验室自主制备。
弗氏完全佐剂、弗氏不完全佐剂美国Sigma公司;Trizol试剂美国Invitrogen公司;质粒提取试剂盒、胶纯化试剂盒、引物、Linker生工生物工程(上海)股份有限公司;反转录聚合酶链反应(reverse transcription polymerase chain reaction,RT-PCR)试剂盒、限制性内切酶NotI、sfiI和T4DNA连接酶宝生物工程(大连)有限公司;质粒pCANTAB-5E、大肠杆菌TG1杭州远方生物科技有限公司;辅助噬菌体M13KO7、酶标二抗HRP/Anti-M13美国NEB公司。
1.2仪器与设备
96孔酶标板美国Corning公司;Versa Max连续波长酶标仪美国Molecular Devices公司;DK-S26型电热恒温水浴锅上海精宏实验设备有限公司;Biofuge Stratos高性能台式冷冻离心机德国贺利氏有限公司;PCR仪、凝胶成像系统美国Bio-Rad公司;NanoDrop ND-1000微量分光光度计美国Thermo公司;RS-1涡旋混合器北京昊诺斯科技有限公司;DYY-12型电脑三恒多用电泳仪北京六一仪器厂。
1.3方法
1.3.1引物及Linker设计合成

表1 引物序列Table 1 Primer sequenceess
表1为鼠源抗体重链(heavy chain,VH)及轻链(light chain,VL)可变区的扩增引物序列[17],以编码柔性肽(Gly4Ser)3的基因序列为Linker。
1.3.2动物免疫及总RNA的提取
莠去津免疫抗原[18]与弗氏完全佐剂等量混合对小鼠进行首次免疫,随后换弗氏不完全佐剂,隔两周加强免疫一次,共3 次,在取小鼠脾脏前3 d加强免疫一次。采用间接竞争ELISA法测定抗体效价,确定免疫效果;取免疫效果好的小鼠的脾脏,用Trizol法提取总RNA,用微量分光光度计检测RNA浓度,琼脂糖凝胶电泳鉴定RNA的完整性。
1.3.3cDNA第一链的合成
采用RT-PCR试剂盒,将RNA逆转录合成cDNA第一链。取0.5 μL RNA,加0.5 μL OligodT接头引物,0.25 μL RNA酶抑制剂,1.0 μL AMV反转录酶,1.0 μL 10 mmol/L dNTP,1.0 μL 10×RT缓冲液,2.0 μL MgCl2,4.25 μL RNase Free ddH2O至10 μL,反应条件:第一阶段反应温度55 ℃,反应时间25 min;第二阶段反应温度95 ℃,反应时间5 min。反应产物置于冰上。
1.3.4VH和VL基因的扩增
以cDNA第一链为模板,分别以表1中VH和VL引物PCR扩增VH和VL基因。
VH基因片段的扩增。取cDNA第一链2.0 μL,10×PCR Buffer 5.0 μL, dNTP(2.5 mmol/L)4.0 μL,VH1back引物(10 μmol/L)1 μL,VH1-2for引物(10 μmol/L)1 μL,加ddH2O至50 μL,涡旋混匀,短暂离心后进行PCR反应。反应条件为第一阶段94 ℃预变性5 min;第二阶段,94 ℃变性30 s;65 ℃条件下退火30 s;72 ℃条件下延伸30 s,共进行30 个循环;第三阶段,72 ℃条件下修复延伸10 min。反应产物进行琼脂糖凝胶电泳检测并进行凝胶回收纯化。
VL基因片段的扩增。以Vκ2back(10 μmol/L)1 μL,MJκfor(10 μmol/L)1 μL为引物,PCR反应体系同上。PCR扩增反应条件:第一阶段:94 ℃条件下预变性5 min;第二阶段:94 ℃条件下变性30 s;60 ℃条件下退火30 s;72 ℃条件下延伸30 s,共进行30 个循环;第三阶段:72 ℃条件下修复延伸10 min。反应产物进行琼脂糖凝胶电泳检测并进行凝胶回收纯化。
1.3.5scFv基因的拼接和扩增
将回收纯化的VH、VL和Linker片段按比例混合,采用重叠延伸PCR方法拼接成scFv基因,第一次PCR反应条件为:第一阶段:94 ℃预变性5 min;第二阶段:94 ℃变性1 min;63 ℃退火1 min;共进行15 个循环;第三阶段:72 ℃条件下修复延伸10 min。随后以上下游带有酶切位点的引物(VH1backSfi I和Jκ1 Not I、Jκ2 Not I、Jκ4 Not I、Jκ5 Not I)进行二次PCR扩增,反应条件:第一阶段:94 ℃条件下预变性5 min;第二阶段:94 ℃条件下变性30 s;57 ℃条件下退火45 s;72 ℃条件下延伸45 s,共进行33 个循环;第三阶段:72 ℃条件下修复延伸10 min。反应产物进行琼脂糖凝胶电泳,凝胶回收目的片段。
1.3.6scFv基因库的构建及鉴定
用SfiI、Not I双酶切单链抗体基因片段,酶切产物与经相同两种酶切的pCANTAB5E载体连接,电转化大肠杆菌TG1,涂布SOBAG平板,挑选克隆,提取质粒,进行PCR扩增鉴定,并将克隆进行测序。
1.3.7抗莠去津scFv基因结构分析
1.3.7.1基因测序及氨基酸序列推导
挑取经鉴定的重组克隆进行质粒抽提并送交生工生物工程(上海)股份有限公司测序。根据起始密码子“ATG”的位置,采用软件DNAStar进行氨基酸序列推导。同时,根据Kabat法则,对抗体可变区VH、VL的骨架区(framework region,FR)及互补决定区(complementarity-determining region,CDR)进行划分。
1.3.7.2编码蛋白的理化性质分析
根据推导所得到的抗莠去津scFv氨基酸序列,采用瑞士生物信息学研究所提供的 ExPASyProteomics tools在线工具中的子程序ProtParam(http://web.expasy.org/ protparam/)对抗体蛋白的氨基酸组成、相对分子质量、理论等电点(isoelectric point,PI)值、半衰期、不稳定系数等物理化学参数进行理论计算[19]。
1.3.7.3编码蛋白二级、三级结构预测
根据推导所得到的抗莠去津单链抗体氨基酸序列,采用ExPASyProteomics tools在线工具中的子程序SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat. pl?page=npsa_sopma.html)对抗体蛋白的二级结构进行预测[20],分析抗体蛋白的α-螺旋、β-转角、延伸链、无规卷曲等;将抗体蛋白提交在线工具ExPASyProteomics tools中的子程序 Swiss Model(http://swissmodel.expasy. org/)进行三维结构模拟[21-22],预测莠去津scFv蛋白的三级结构。
2 结果与分析
2.1抗体可变区基因的PCR扩增
本实验利用提取到的小鼠脾脏RNA反转录合成cDNA第一链,然后以此cDNA第一链为模板,利用根据抗体VH可变区和VL可变区基因框架区的保守性而设计的鼠源通用VH引物和VL引物,经PCR扩增,分别获得VH、VL片段(图1),大小分别约为350 bp和320 bp。经PCR扩增获得的VH和VL产物经胶回收后,进行重叠延伸PCR,通过Linker连接得到scFv基因片段,结果如图2所示,scFv基因片段约为750 bp左右,且条带清晰,与预期相符。

图1 VH基因和VL基因的PCR扩增结果Fig.1 PCR products of VH and VL
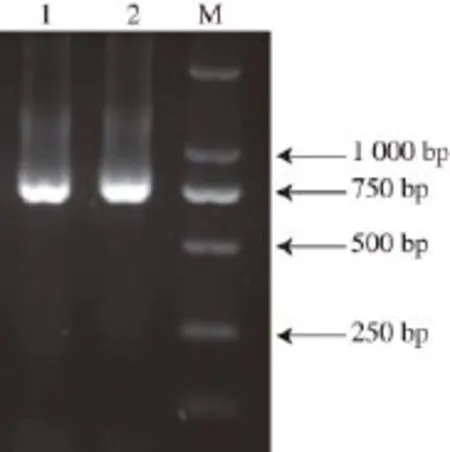
图2 scFv基因的PCR扩增结果Fig.2 Ampli.ed fragment of scFv gene
2.2抗莠去津scFv基因结构和性质分析
2.2.1scFv基因序列测定
抗莠去津scFv基因序列全长为744 bp(图3),其中VH片段长度为369 bp,连接肽长度为45 bp,VL片段长度为330 bp。两端划线部分分别为Sfi Ⅰ、NotⅠ双酶切位点,中间划线部分为Linker序列,其基因构成完全符合scFv基因的组成。

图3 3 scFvscFv的基因序列Fig.3 Gene sequence of scFv
2.2.2抗莠去津scFv基因编码蛋白氨基酸序列推导
对抗莠去津scFv基因编码蛋白进行氨基酸序列推导,VH与VL的氨基酸序列以及FR、CDR的划分如图4所示。VH含123 个氨基酸,VL含110 个氨基酸。scFv基因编码各种氨基酸的含量如表2所示。其中含量最高的是甘氨酸和丝氨酸,分别占14.10%;其次为苏氨酸、丙氨酸和亮氨酸,分为10.10%、7.30%和5.20%;含量最少的有天冬氨酸、半胱氨酸和组氨酸,均仅占1.60%。用在线工具IGMT/V-Quest分别对抗体基因的VH和VL进行同源性比对分析,结果表明,VH与鼠源抗体AC160473 IGHV1-74的同源率达86.06%;VL与鼠源抗体AJ231229 IGKV-4-91同源性最高,同源率达98.58%。

图4 VH、VL的氨基酸序列Fig.4 Amino acid sequences of VH and VL
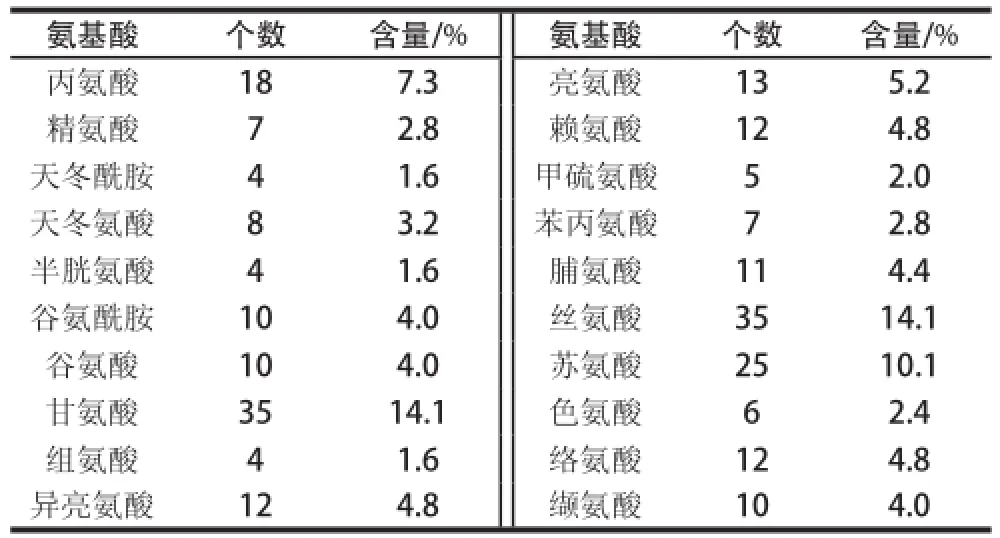
表2 抗莠去津scFv蛋白氨基酸组成Table 2 Amino acid composition of scFv against atrazine
2.2.3抗莠去津scFv蛋白的性质分析
在线分析工具ProtParam对抗莠去津scFv蛋白理化性质的预测结果表明,该蛋白质相对分子质量为26 061.9,分子式为C1144H1755N309O371S9,理论pI值为7.81,酸性氨基酸残基(天冬氨酸+谷氨酸)18 个,碱性氨基酸残基(精氨酸+亮氨酸)19 个,该蛋白质为碱性蛋白质;不稳定指数为38.66,该抗体蛋白属于稳定蛋白质[23];脂肪指数为58.27,总平均亲水性值为-0.340;在体外哺乳类网状细胞的半衰期为30 h,在酵母体内半衰期大于20 h,在埃希菌属大肠杆菌体内的半衰期大于10 h。
2.2.4抗莠去津scFv蛋白的结构分析
在推导莠去津scFv蛋白氨基酸序列分析其同源性、FR区和CDR区及理化性质的基础上,对抗体蛋白的二级和三级结构进行预测和分析。蛋白质二级结构预测不仅是联系其一级结构和三级结构的桥梁和纽带,而且是从其一级结构预测其三级结构的关键步骤,有助于确定蛋白质空间结构与功能的关系[24]。SOPMA软件对该单链抗体的二级结构预测(图5)显示,该抗体的二级结构含α-螺旋17 处,占总二级结构的6.85%;延伸链85 处,占总二级结构的34.27%;β-转角19 处,占总二级结构的7.66%;无规卷曲127 处,占总二级结构的51.21%,由此可推测,该抗体蛋白的二级结构以无规卷曲和延伸链为主要结构元件。用Swiss Model在线软件预测抗莠去津scFv的三维结构,如图6所示,抗体的VH和VL通过Linker折叠在一起,可变区的6 个环区(CDR区)共同组成抗体的抗原结合区,该三维模拟图符合典型scFv的结构。
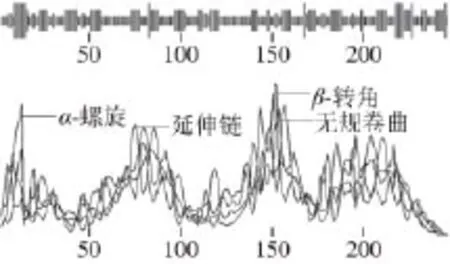
图5 抗莠去津scFv蛋白质二级结构分析Fig.5 Secondary structure analysis of anti-atrazine scFv protein

图6 抗莠去津scFv蛋白三维结构模拟图Fig.6 Simulated 3D structure of scFv against atrazine
3 结 论
本研究从抗莠去津小鼠中成功构建了抗莠去津scFv基因,并应用生物信息学方法对抗莠去津scFv基因编码的蛋白质进行分析。结果显示,抗莠去津scFv基因全长744 bp,其中VH为369 bp,VL为330 bp,连接肽为45 bp;抗莠去津scFv蛋白含248 个氨基酸,相对分子质量为26 061.9,分子式为C1144H1755N309O371S9,理论pI值为7.81,为碱性蛋白质;其二级结构以无规卷曲和延伸链为主要结构元件。本研究成功构建了抗莠去津scFv基因,并将其克隆入pCANTAB5E质粒载体,为抗莠去津scFv基因表达、蛋白纯化和活性研究等奠定了实验基础。
[1]万年升, 顾继东, 段舜山. 阿特拉津生态毒性与生物降解的研究[J].环境科学学报, 2006, 26(4): 552-560.
[2]GFRERER M, WENZL T, QUAN X, et al. Occurrence of triazines in surface and drinking water of Liaoning Province in Eastern China[J]. Journal of Biochemical Biophysical Methods, 2002, 53(1/3): 217-228.
[3]BRUSICK D J. An assessment of the genetic toxicity of atrazine:relevance to human health and environmental effects[J]. Mutation Research, 1994, 317(2): 133-144.
[4]PRASAD T A, REDDY D C. Atrazine toxicity on hydromineral balance of fish, Tilapia mossambicus[J]. Ecotoxicology and Environmental Safety, 1994, 28(3): 313-316.
[5]周培, 陆贻通. 农药残留的酶联免疫检测技术研究进展[J]. 环境污染与防治, 2002, 24(4): 248-251.
[6]王兆守, 刘丽花, 邵宗泽, 等. 农药残留检测新方法研究进展[J]. 化工进展, 2008, 27(9): 1370-1374.
[7]魏松红, 东琴, 逄若霖, 等. 检测莠去津残留的间接竞争ELISA试剂盒的研制[J]. 沈阳农业大学学报, 2014, 45(5): 543-547.
[8]YANG Jinyi, WANG Hong, JIANG Yueming, et al. Development of an enzyme-linked immunosorbent assay (ELISA) method for carbofuran residues[J]. Molecules, 2008(13): 871-881.
[9]魏松红, 逄若霖, 王翀, 等. 甲拌磷残留检测间接竞争ELISA试剂盒的研制[J]. 食品科学, 2011, 32(4): 284-287.
[10] 何扩, 张秀媛, 杜欣军, 等. 氧氟沙星单链抗体库的构建, 筛选及蛋白结构模拟[J]. 现代食品科技, 2014, 30(3): 108-113.
[11] 潘阳, 王露楠. 噬菌体抗体库技术的构建及应用研究进展[J]. 标记免疫分析与临床, 2011, 18(3): 207-211.
[12] 尹惠琼. 单链抗体的研究进展综述[J]. 动物医学进展, 2003, 24(2):24-26.
[13] HOLLIGER P, HUDSON P J. Engineered antibody fragments and the rise of single domains[J]. Nature Biotechnology, 2005, 23(9):1126-1136.
[14] ALCOCER M J, DOYEN C, LEE H A, et al. Functional Scfv antibody sequences against the organophosphorus pesticide chlorpyrifos[J]. Journal of Agricultural and Food Chemistry, 2000, 48(2): 335-337.
[15] KATO M, HANYU Y. Construction of an scFv library by enzymatic assembly of VLand VHgenes[J]. Journal of Immunological Methods,2013, 396(1/2): 15-22.
[16] 王弘, 刘细霞, 潘科, 等. 抗克伦特罗单链抗体基因构建及蛋白结构模拟[J]. 食品科学, 2009, 30(13): 227-231.
[17] 沈倍奋, 陈志南, 刘民培. 重组抗体[M]. 北京: 科学出版社, 2005:18-97.
[18] 魏松红, 逄若霖, 李兴海, 等. 莠去津人工抗原的合成与鉴定[J]. 沈阳农业大学学报, 2011, 42(1): 102-105.
[19] GASTEIGER E, HOOGLAND C, GATTIKER A, et al. Protein identification and analysis tools on the ExPASy server[M]. New York:Humana Press, 2005: 571-607.
[20] GEOURJON C, DELEAGE G. SOPMA: significant improvement in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Computer Applications in the Bioinformatics,1995, 11(6): 681-684.
[21] ARNOLD K, BORDOLI L, KOPP J, et al. The SWISS-MODEL workspace: a web-based environment for protein structure homology modeling[J]. Bioinformatics, 2006, 22(2): 195-201.
[22] ZHOU Chun, SHEN Guanxin, ZHU Huifen, et al. Construction and analysis of three-dimensional graphic model of single-chain Fv derived from an anti-human placental acidic isoferritin monoclonal antibody by computer[J]. Journal of Tongji Medical University, 2000, 20(1):23-25.
[23] 郭宇立, 倪逸声. 从一级结构预测蛋白质稳定性: Guruprasad, Reedy和Pandit方法[J]. 生物化学与生物物理进展, 1993, 20(2): 95-99.
[24] 王志学. 蛋白质结构预测的现状与展望[J]. 生命的化学, 1998,18(6): 19-22.
Gene Construction and Protein Structure Prediction of Single-Chain Variable Fragment Antibody against Atrazine
DONG Qin, WEI Songhong*, LI Pingsheng, WANG Haining
(Plant Protection College, Shenyang Agricultural University, Shenyang110866, China)
In order to obtain the gene encoding single-chain variable fragment (scFv) antibody against atrazine, the amino acid sequence of scFv was deduced by DNAStar software and its physical and chemical properties, and secondary and tertiary structures of its encoding protein were analyzed according to bioinformatics online websites. The results showed that the full-length scFv gene had 744 bp and encoded 248 amino acids. The relative molecular weight of scFv was 26061.9, and the antibody had isoelectric point (pI) of 7.81 and belonged to alkaline and stable protein. The predicted secondary structure comprised 17 α-helices, 85 extended strands, 19 β-turns and 127 random coils. The mimic tertiary structure model of scFv indicted that all six complementarity-determining regions (CDRs) of light chain variable (VL) and heavy chain variable (VH)region contributed to the antigen-binding sites. This research can lay the foundation for further studies on the expression,purification and bioactivity of genetically engineered antibodies.
atrazine; single-chain variable fragment (scFv); structure prediction
TS207.3
A
1002-6630(2015)23-0195-05
10.7506/spkx1002-6630-201523036
2014-12-25
辽宁省自然科学基金项目(201202195)
东琴(1990—),女,硕士研究生,主要从事农药毒理学研究。E-mail:qdadongqin@126.com
魏松红(1974—),女,教授,博士,主要从事农药学研究。E-mail:songhongw125@163.com

