黄瓜根际土壤细菌群落的16S rDNAPCR-DGGE分析
赵柏霞 闫建芳 刘 秋 于基成 赵秀香
(1大连市农业科学研究院,辽宁大连 116036;2沈阳农业大学植物保护学院,辽宁沈阳 110161;3大连民族大学生命科学学院,辽宁大连 116600)
黄瓜枯萎病〔Fusarium oxysporum(Schl) f. sp.cucumerinum〕是黄瓜生产上的一种顽固土传病害,是目前造成我国瓜类生产上大面积减产的主要原因之一,是黄瓜病害中最为严重、造成经济损失最大的病害之一(张宏宇 等,2010,闫霜 等,2011)。其致病菌可以在土壤中存活多年,给该病害的防治带来很大的困难。黄瓜枯萎病的发生及发病程度的轻重,是病原菌和土壤中各种微生物相互作用的结果(邓晓 等,2012)。土壤微生物多样性与植物土传病害抑制水平密切相关,根际微生物在抑制土传病害和促进植物生长过程中具有重要的作用(Garbeva et al.,2004a,2004b; 申卫收和林先贵,2011)。PCR-DGGE技术作为一种指纹分析技术,适合用来分析环境微生物种群多样性。近些年来,DGGE技术已经在环境微生物研究领域中广泛应用(李怀 等,2008;马俊孝 等,2008;潘雪莲 等,2009;Chong et al.,2009;刘晓燕 等,2014)。
为揭示枯萎病菌对黄瓜根际细菌群落的影响,本试验以黄瓜根际土壤总DNA为模板,采用PCRDGGE分析方法监测接种枯萎病菌后黄瓜根际土壤细菌的变化,试图阐述在枯萎病菌影响条件下土壤细菌群落的演替规律,为从根际微生物生态角度理解枯萎病问题,进而为开展生物防治奠定 基础。
1 材料与方法
1.1 材料
1.1.1 供试病原菌 试验所用的黄瓜枯萎病病原菌——尖孢镰孢菌黄瓜专化型(Fusarium oxysporumf. sp.cucumerinum),由大连民族大学实验室保存。
1.1.2 供试土壤样品采集 试验田设在大连民族大学的温室内,2012年 4月,将从大连当地种子店购买的黄瓜品种碧秀的种子催芽后,播种在直径30 cm的大花盆中。待黄瓜3~4片叶时,接种黄瓜枯萎病菌。采用抖落法(张学利 等,2005)分别采集黄瓜种植前的土壤(ZQ,4月18日取样)、接种黄瓜枯萎病菌前的土壤(JQ,5月20日取样)、接菌后感染枯萎病菌的黄瓜根际土壤(JB,6月17日取样)、接菌后未发病的黄瓜根际土壤(JJ,6月17日取样)及接种清水的黄瓜根际土壤(CK,6月17日取样)共5份黄瓜根际土壤样品。每份样品由5株黄瓜根际土壤充分混匀。样品取回后置于阴凉通风处干燥,过20目筛后备用。
1.2 方法
1.2.1 样品总 DNA 的提取 采用碱裂解法提取供试土壤样品总 DNA(Hu et al.,2010),用 1.0% 的琼脂糖凝胶电泳检验,以λ-Hind Ⅲ作为 Marker。
1.2.2 PCR扩增 采用两轮PCR反应,引物见表1。第1轮PCR扩增以提取的土壤总DNA为模板,采用细菌 16 S rDNA 的通用引物 F27/R1492 进行扩增。反应体系及程序参考韦超英等(2011)的方法。第2轮PCR反应,采用 F338-GC/R518引物扩增细菌 16 S rDNA V3 区。反应体系 50μL,参照杨尚东等(2014)的方法进行。PCR反应程序:94 ℃ 预变性 5 min;94 ℃变性 30 s,63 ℃(后 20 个循环每个循环降 0.5 ℃直至降到 53 ℃)退火 30 s,72 ℃ 延伸 30 s,共 40 个循环;72 ℃延伸 7 min;扩增产物4 ℃保存。
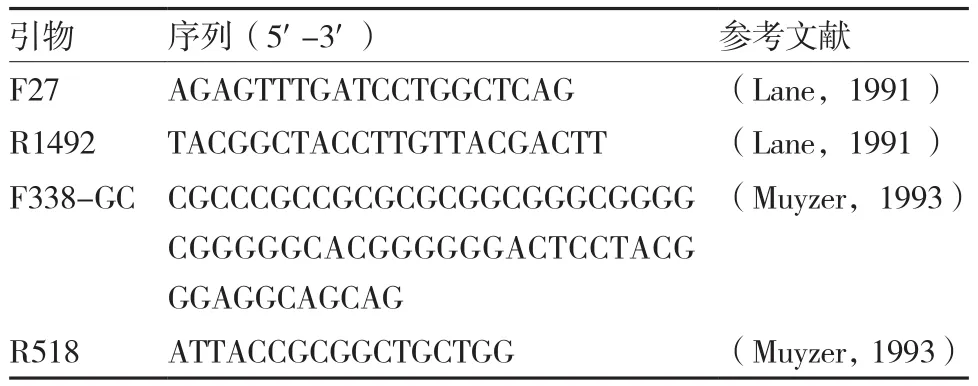
表1 PCR 引物
1.2.3 DGGE 分析 采用 Bio-Rad 电泳系统。变性剂梯度为 30%~60%的 8%聚丙烯酰胺凝胶,60 ℃、80 V 条件下电泳 10 h。胶用 sybr green Ⅰ染色 30 min,DGGE 条带用 Quantity One 软件分析。
2 结果与分析
2.1 DNA 提取
采用碱裂解法提取的总DNA,通过与λ-Hind Ⅲ Marker比较,可以看出由该方法获得的5个土壤样品的总DNA片段大小超过23.1 kb,形成了较亮的清晰条带,并且DNA片段的产量大,可以进行后续试验(图1)。
2.2 PCR 扩增
第1轮PCR反应以F27/R1492为引物,得到大小约为1 500 bp的目的片段(图2),PCR产物的特异性较好。第2轮PCR反应以F338-GC/R518为引物,扩增得到大小约为230 bp的目的片段(图3)。
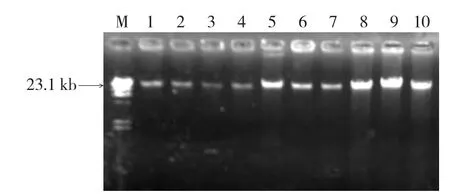
图1 根际土壤总DNA电泳结果
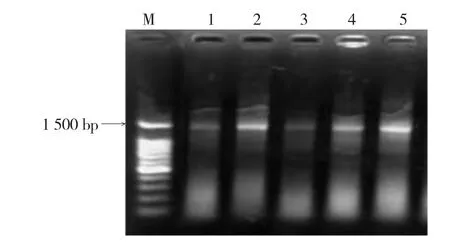
图2 各样品16S rDNA扩增结果
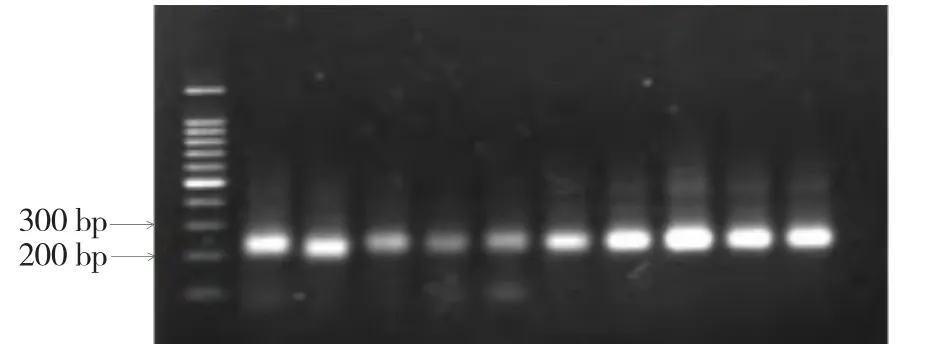
图3 各样品16S rDNA基因V3区扩增结果
2.3 DGGE分析
对各样品 16S rDNA V3 区 PCR 产物进行 DGGE 分析(图4)。每个土壤样品都检测到了不同数目的条带,从15个条带(CK)到24个条带(JJ)不等,它们之间的带型也各不相同。接种清水的对照土壤样品(CK)有15个条带,细菌多样性最低,接菌后未发病的土壤样品(JJ)条带数最多,细菌多样性最高。
采用Quantity one软件分析表明,只有少数条带为个别样品的特异性条带,如条带E1、E5及E6;多数条带如条带E2、E4,在各个样品中普遍存在。这说明样品之间既存在着相似的细菌种群,又具有其特异的细菌种群。
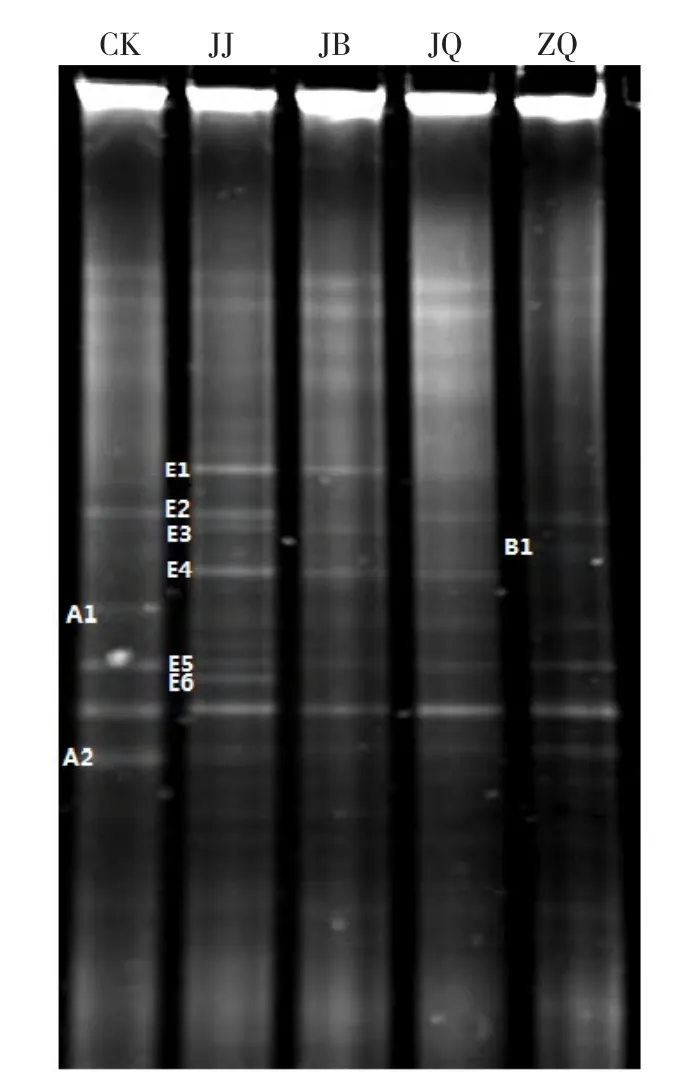
图4 PCR 产物的 DGGE 电泳图谱
2.4 序列测定
从5份土壤样品总DNA扩增得到不同数目的 16S rDNA V3 区条带,选择各样品特有的或共有的、并有一定亮度的9 个条带,回收测序,测序后与 GenBank 数据库序列进行比对。由表2可知,条带 A1、A2、E2、E4、E5的序列均与未培养细菌种属相似性较高,分别属于酸杆菌门(Acidobacteria)、 拟 杆 菌 门(Bacteroidetes)、芽单胞菌门(Gemmatimonadetes)、放线菌门(Actinobacteria),这些未培养种群大多从污染土壤、植物根际土壤中获得。其余几个条带与可培养细菌相似度较高,B1与Sphingomonassp.相似性达 100%。结合图4可以看出,B1条带所对应的细菌是在种植前的土壤样品(ZQ)中数量较多,在接种枯萎病菌后的各土壤样品(JB、JJ)中数量则有所降低。E3、E6的序列都和Clostridiumsp.相似性较高。本试验中,接种枯萎病菌后Rhodococcus

表2 DGGE主要条带的测序比对结果
phenolicus(E1)、Clostridiumsp.(E3、E6) 以 及3种未培养细菌(E2、E4、E5)数量增加,另外2种未培养细菌(A1、A2)数量减少。
3 结论与讨论
本试验中,从不同样品的DGGE电泳图谱可以看出,每个样品均可以分离到15~24个不等条带,不同的条带代表不同细菌的基因片段,表明每个样品都存在着丰富的微生物种类。条带最少的是接种清水的对照土壤(CK),条带最多的是接种枯萎病菌后未发病的土壤样品(JJ),表明接种后未发病的土壤样品中细菌多样性较高。分析其原因,可能是接种枯萎病菌后诱导了一些微生物数量的增加,进而使土壤中微生物的丰富性升高;接菌后枯萎病菌与土壤中其他微生物产生了竞争作用,最终未发病可能是有益微生物的数量和种类占了优势。这一结果与张钰等(2013)的研究报道相似,即对黄瓜幼苗施用菌剂后,能使根围和根际中细菌和放线菌的数量升高。
DGGE 条带数目可以近似地体现细菌种群的数量,而条带亮度则反映该种细菌数量的多少(Hu et al.,2004)。本试验中,接种枯萎病菌使根际土壤部分细菌的数量发生了明显变化,这体现在条带亮度增强或减弱上,如条带E1、E4在样品JB、JJ中亮度增强,这说明接种枯萎病菌后使得E1、E4所代表的细菌种群在根部富集。
DGGE 指纹图谱提供了细菌群落结构变化趋势,为了进一步了解细菌群落结构的组成,对DGGE 特殊条带进行切胶回收测序。从9个 DGGE 条带测序结果看,这些条带对应同源性最高的微生物分别属于变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、酸杆菌门(Acidobacteria)、芽单胞菌门(Gemmatimonadetes)以及放线菌门(Actinobacteria)。这些微生物多为未培养微生物,表明接种枯萎病菌对土壤中大量未培养细菌的影响较大,但由于这些细菌还未被培养或不可培养,因此很难鉴定它们在原位生态环境中的生理生化特性及其所具备的生态功能意义。
从DGGE图谱可以看出,接菌前(JQ)和对照(CK)中E4、A2条带发生了变化,由于对照是在接种清水后、在植株生长后期取的样品,这就说明随着黄瓜的生长发育,也就是说黄瓜的不同生育期,根际土壤中细菌群落会发生改变。接种病原菌(JJ、JB)使得E1、E3、E4、E5条带亮度增强,也会引起A1、A2条带亮度的降低。其中E1、E3分 别 与Rhodococcus phenolicus、Clostridiumsp.相似性达到99%,文献报道Rhodococcus phenolicus是一种硝化细菌(张光亚 等,2003),Clostridiumsp.是一种典型的产氢菌(许继飞 等,2009),这两种菌和植物病害的具体关系均未见报道。
在除了ZQ样品以外的4个样品中,Sphingomonassp.(B1)数量减少,这说明种植黄瓜后引起了该种细菌的数量变化,Sphingomonas属的一个显著特征是可以降解多环或单环芳香族化合物如苯甲酸、水杨酸等,它能利用这些芳香族化合物为唯一碳源进行生长(Basta et al.,2005),而苯甲酸、水杨酸及其衍生物是黄瓜根系分泌物中的重要化感物质(Pramanik et al.,2000)。本试验得到的细菌群落 DGGE 图谱与胡元森等(2007)研究结果不一致,胡元森研究发现,黄瓜连作引起土壤中Sphingomonassp.数量增加。本试验后期接入了枯萎病菌,而胡元森研究的是连作黄瓜的根际土壤,这似乎说明根际土壤中细菌群落受病菌影响较大。
有研究表明,根系分泌物是由多种物质组成,包括各种低分子有机物、各种离子等(罗永清 等,2012)。根系分泌物会改变根际细菌群落组成(Kozdroj & van Elsas,2000),添加根系分泌物能有效促进细菌的生长(T é cher et al.,2011)。而根系分泌物也受到各种因素的影响,如芳香族化合物等有机物质(Hoang et al.,2011)、真菌侵染(Norman & Hooker,2000)、金属元素(Meier et al.,2012)等。本试验中,DGGE 9个主要条带所代表的细菌并未发现与黄瓜枯萎病的发生有着明显的关联,但是,枯萎病菌的接入可能是通过改变黄瓜根系分泌物的产生或者其中的各种离子成分等进而改变了黄瓜根际的生态环境,从而影响了根际的细菌群落组成,还有待进一步研究。
邓晓,李勤奋,侯宪文,武春媛,李光义.2012.香蕉枯萎病不同感病级别植株根际与非根际土壤微生物物种多样性研究.中国农学通报,28(30):239-248.
胡元森,吴坤,李翠香,贾新成.2007.黄瓜连作对土壤微生物区系影响Ⅱ——基于 DGGE方法对微生物种群的变化分析.中国农业科学,40(10):2267-2273.
李怀,关卫省,欧阳二明,王晓慧,张杰.2008.DGGE技术及其在环境微生物中的应用.环境科学与管理,(10): 93-96,99.
刘晓燕,张磊,韦泽秀,朱丹,王晓锋,吴先勤,丁红利.2014.用PCR-DGGE研究菌肥对西藏青稞土壤微生物群落多样性的影响.西南大学学报:自然科学版,36(7):39-48.
罗永清,赵学勇,李美霞.2012.植物根系分泌物生态效应及其影响因素研究综述.应用生态学报,23(12):3496-3504.
马俊孝,孔健,季明杰.2008.利用PCR-DGGE技术分析微生态制剂在传代过程中的菌群变化.山东大学学报:理学版,43(7):56-60.
潘雪莲,黄晟,方昊,徐军,郭晓峰,陈旸,崔益斌.2009.黄土高原土壤中细菌群落结构多样性的PCR-DGGE分析.生态与农村环境学报,25(3):39-43,48.
申卫收,林先贵.2011.三种不同利用方式土壤上黄瓜根际微生物群落差异.土壤学报,48(3):654-658.
韦超英,杨滨银,方新湘,娄恺,晁群芳.2011.适用变性梯度凝胶电泳分析的石油污染土壤微生物DNA模板制备的研究.安徽农业科学,39(16):9676-9679,9722.
许继飞,任南琪,邱颉,苏东霞.2009.木糖发酵产氢菌的筛选及其生长产氢特性研究.微生物学通报.36(4):467-472.
闫霜,吴洪生,周晓冬,张富存,刘小雪,王增辉,孔祥云,刘正柱.2011.黄瓜枯萎病生物防治研究进展.山东农业科学,43(1):86-92.
杨尚东,赵久成,郭伊娟,吴俊,龙明华.2014.番茄青枯病罹病植株和健康植株根际土壤细菌群落结构的初步分析.中国蔬菜,(8):25- 29.
张光亚,陈美慈,韩如旸,闵航.2003.一株异养硝化细菌的分离及系统发育分析.微生物学报,42(2):156-161.
张宏宇,陈霞,左洪波,张艳菊,秦智伟,周秀艳.2010.中国现行推广黄瓜品种及种质资源对枯萎病的抗病性评价.东北农业大学学报,41(5):36-41.
张学利,杨树军,张百习,袁春良.2005.不同林龄樟子松根际与非根际土壤的对比.福建林学院学报,25(1):80-84.
张钰,孙锦,郭世荣.2013.基质中添加微生物制剂对黄瓜幼苗生长和枯萎病抗性的影响.西北植物学报,33(4): 780-786.
Basta T,Buerger S,Stolz A.2005.Structural and replicative diversity of plasmids from sphingomonads that degrade polycyclic aromatic compounds and xenobiotics.Microbiology,151: 2025-2037.
Chong C W,Dunn M J,Convey P,Tan G Y A,Wong R C S,Tan I K.2009.Environmental influences on bacterial diversity of soils on Signy Island,maritime Antarctic.Polar Biology,32(11):1571-1582.
Garbeva P,van Veen J A,van Elsas J D.2004a.Assessment of the diversity and antagonists towardsRhizoctonia solaniAG3 ofPseudomonasspecies in soil from different agricultural regimes.FEMS Microbiological Ecology,47:51-64.
Garbeva P,van Veen J A,van Elsas J D.2004b.Microbial diversity in soil:selection of microbial populations by plant and soil type and implications for soil suppressiveness.Annual Review of Phytopathology,42:243-270.
Hoang H,Yu N,Toyama T,Inoue D,Sei K,Ike M.2010.Accelerated degra-dation of a variety of aromatic compounds bySpirodela polyrrhiza-bacterial associations and contribution of root exudates released fromS.polyrrhiza.Journal of Environmental Sciences,22:494-499.
Hu Y C,Liu Z H,Yan J F,Qi X H,Li J,Zhong S Q,Yu J C,Liu Q.2010.Developed DNA extraction method for different soil samples.Journal of Basic Microbiology,50:401- 407.
Hu Y S,Wu K,Liu N,Chen H G,Jia X C.2004.Dynamics of microbial communities in bulk and developing cucumber rhizosphere soil.Agricultural Sciences in China,3(5):376-383.
Kozdroj J,van Elsas J D.2000.Response of the bacterial community to root exudates in soil polluted with heavy metals assessed by molecular and cultural approaches.Soil Biology and Biochemistry,32:1405-1417.
Lane D J.1991.16S/23S rDNA sequencing// Stackebrandt E,Goodfellow M.Nucleic acid techniques in bacterial systematics.New York:Wiley:115 -175.
Meier S,Alvear M,Borie F,Aguilera P,Ginocchio R,Cornejo P.2012.Influence of copper on root exudate patterns in some metallophytes and agricultural plants.Ecotoxicology and Environmental Safety,75:8-15.
Muyzer G,de Waal E C,Uitterlinden A G.1993.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA.Applied Environmental Microbiology,59(3):695-700.Norman J R,Hooker J E.2000.Sporulation ofPhytophthora fragariaeshows greater stimulation by exudates of non-mycorrhizal than by mycorrhizal strawberry roots.Mycological Research,104:1069-1073.Pramanik M H R,Nagai M,Asao T,Matsui Y.2000.Effects of temperature and photoperiod on phytotoxic root exudates of cucumber (Cucumis sativus) in hydroponic culture.Journal of Chemical Ecology,26(8):1953-1966.
T é cher D,Laval-Gilly P,Henry S,Bennasroune A,Formanek P,Martinez-Chois C,D′Innocenzo M,Muanda F,Dicko A,Rejšek K,Falla J.2011.Contribution ofmiscanthus×giganteusroot exudates to the biostimulation of PAH degradation:an in vitro study.Science of the Total Environment,409:4489-4495.

