基因芯片技术在结核分枝杆菌耐药检测及菌种鉴定中的应用
石国民 喻容 彭雪峰 石燕 聂英 齐志强 陈拥军 向延根 刘忠泉
·论著·
基因芯片技术在结核分枝杆菌耐药检测及菌种鉴定中的应用
石国民 喻容 彭雪峰 石燕 聂英 齐志强 陈拥军 向延根 刘忠泉
目的 应用基因芯片技术快速检测结核分枝杆菌的耐药基因型以及分枝杆菌菌种基因型,探讨基因芯片技术检测结核分枝杆菌耐药性和基因型的临床价值。方法 应用菌种分型基因芯片技术和间隔区寡核苷酸分型(spacer oligonucleotide typing, Spoligotyping)技术对长沙市中心医院2011年1月至2012年12月的临床标本分离菌株137株进行菌种鉴定。对鉴定为结核分枝杆菌的菌株应用绝对浓度法进行利福平和异烟肼药物敏感性检测。并进一步对结核分枝杆菌临床分离株(利福平耐药、异烟肼耐药、敏感株)应用耐药基因芯片技术对rpoB、katG、inhA基因的野生型位点及各突变位点进行检测。即rpoB基因中1个或多个位点的碱基突变为利福平耐药,katG、inhA基因任何基因中的1个或多个位点的碱基突变为异烟肼耐药。Spoligotyping法和基因芯片法菌种鉴定比较应用Kappa检验,绝对浓度法和基因芯片法药物敏感性比较采用χ2检验和Kappa检验,均以P<0.05为差异有统计学意义。结果 (1)与绝对浓度法比较,对利福平敏感和异烟肼敏感的菌株45株,耐药基因芯片法检测鉴定为敏感株、即野生型的rpoB基因42株,符合率为93.3%(42/45)(不符合的1株为511T-C突变,1株为531C-T突变, 1株为516A-T突变);katG基因45株,符合率为100.0%(45/45);inhA基因43株,符合率为95.6%(43/45)(不符合的2株为inhA-15C-T突变)。(2)与绝对浓度法比较,对利福平轻度耐药的耐药菌株18株,耐药基因芯片检测鉴定为rpoB基因突变型的16株,符合率为88.9%(16/18),且与531、516、526、511位点突变相关。对利福平高度耐药的耐药菌株19株,耐药基因芯片检测全部鉴定为rpoB基因突变型,符合率为100.0%(19/19),且与531、516、526、511位点突变相关。(3)与绝对浓度法比较,对异烟肼轻度耐药的耐药菌株13株,耐药基因芯片检测鉴定为katG基因突变型的12株,符合率为92.3%(12/13),与315G-C和315G-A位点突变相关。inhA基因突变型0株。对异烟肼高度耐药的耐药菌株9株,耐药基因芯片检测鉴定为katG基因全部为突变型,符合率为100.0%(9/9),与315G-C和315G-A位点突变相关。inhA基因突变型0株。(4)与Spoligotyping法比较,137株临床分离株中104株分离株基因芯片法检测鉴定为结核分枝杆菌复合群,33株基因芯片法检测出7株鸟分枝杆菌、15株胞内分枝杆菌、1株偶然分枝杆菌、10株龟或脓肿分枝杆菌,与Spoligotyping法比较,结核分枝杆菌复合群一致性为100.0%(104/104),鸟分枝杆菌一致性为77.8%(7/9),胞内分枝杆菌一致性为93.8%(15/16),偶然分枝杆菌一致性为0.0%(0/0),龟或脓肿分枝杆菌一致性为100.0%(8/8)。经Kappa检验,Kappa=0.95,U=30.6,P<0.05。结论 耐药基因芯片检测法与绝对浓度法有高度的一致性,且利福平耐药与rpoB基因的531、516、526、511位点突变相关。异烟肼耐药与katG基因的315位点突变相关,没有发现inhA相关的耐药位点突变。基因芯片方法可快速、准确地检测临床分离株的菌种基因型和耐药性。
结核分枝杆菌; 抗药性, 细菌; 微生物敏感性试验; 寡核苷酸序列分析
近年来由于耐药结核病、特别是耐多药结核病的流行,使得为数不多的一线抗结核药物很难有效,甚至二线和三线药物也用到了治疗结核病上。有文献表明耐多药结核病患者应用抗结核药物治疗的失败率在15%~77%[1]。目前结核分枝杆菌耐药检测方法有表型检测和基因型检测两大类,而表型检测相对时间较长。Lee等[2]的研究表明结核分枝杆菌耐利福平的特性95%与rpoB基因的-85 bp范围基因位点突变相关,耐异烟肼结核分枝杆菌大多与katG、inhA、aphC、kasA基因中1个或多个基因位点的突变相关。基因型检测方法目前有DNA测序、单链构像多态性(SSCP)、双脱氧指纹法、异源双链核酸分子分析、错位配子分析、线型扩增、分子标记序列分析、耐药芯片检测等。基因芯片技术是近年发展起来的一种新的分析方法,它有快速、简便、高通量等特点,一次能检测十几至几十个基因位点,适用于多位点分析。因此笔者应用基因芯片技术对本院近期结核分枝杆菌临床分离菌株进行了耐药基因检测。旨在发现本地区耐利福平rpoB基因,耐异烟肼katG、inhA基因的突变特征。
材料和方法
一、材料
1.菌株来源:收集长沙市中心医院2011年1月至2012年12月所有培养阳性且抗酸染色阳性的临床分离株。培养方法通过Bactec MGIT 960分枝杆菌培养检测系统获得临床分离株。共获得结核分枝杆菌复合群临床分离株104株,非结核分枝杆菌临床分离株33株。来源患者均为本院住院患者,104株肺结核患者均符合《临床诊疗指南 结核病分册》[3]肺结核的诊断标准;临床上已排除其他病因;经病理或细菌培养证实明确诊断为肺结核患者,在随后观察中对抗结核治疗有效。33株非肺结核患者均符合《临床诊疗指南 结核病分册》肺结核的诊断标准;临床上已排除其他病因;经病理或细菌培养证实诊断为肺结核患者,但在随后观察中对抗结核治疗无效。其中男86例,女51例;年龄20~65岁,平均(46±12.5)岁;137株临床分离株均为经痰培养阳性,并抗酸染色阳性,137株临床分离株经Spoligotyping 菌型鉴定,并进行基因芯片法菌种鉴定。鉴定为结核分枝杆菌复合群的临床分离株104株再进行绝对浓度法药物敏感性试验(简称“药敏试验”)、耐药基因芯片检测。
2.抗结核药物、试剂以及仪器来源:分枝杆菌分离培养采用美国BD公司Bactec MGIT 960分枝杆菌培养检测系统进行培养;绝对浓度法药敏试验采用自制改良罗氏培养基;利福平、异烟肼标准粉剂,均购自美国Sigma公司);分枝杆菌菌种鉴定试剂盒Mini-blotter-MN45(Spoligotyping法)为荷兰Isogen Life Science 公司产品;耐药基因芯片检测仪器(核酸快速提取仪、芯片杂交仪、芯片洗干仪、激光扫描仪)与检测试剂盒[晶芯®分枝杆菌菌种鉴定试剂盒(基因芯片法)、晶芯®结核分枝杆菌耐药检测试剂盒(基因芯片法)]均为北京博奥生物有限公司产品,基因扩增采用美国ABI 7300基因扩增仪。
二、方法
(一)菌种鉴定
1.Spoligotyping分型检测:对137株临床分离株采用Spoligotyping法菌种鉴定试剂盒进行检测,严格按照说明书进行操作。以结核分枝杆菌基因组内DR区和16S rDNA为靶序列, 设计特异性引物, 5′末端采用地高辛(digoxigenin, DIG)标记。PCR 反应条件: 96 ℃ 预变性3 min,96 ℃变性1 min, 55 ℃退火 1 min, 72 ℃延伸 30 s,20个循环, 72 ℃ 延伸5 min,扩增整个DR区和16S rDNA。将PCR产物与结合有间隔区寡核苷酸探针及非结核分枝杆菌特异性探针的Biodyne 尼龙转印C膜进行杂交。地高辛化学发光检测系统进行检测。通过间隔区寡核苷酸探针斑点的显色和非结核分枝杆菌特异性探针斑点显色来判断分枝杆菌菌型。Mycobacterium tuberculosis strain H37Rv和Mycobacterium bovis BCG P3标准株作为阳性控制,蒸馏水(double distilled water,DDW)为阴性控制。
2.基因芯片分型检测:采用北京博奥生物有限公司菌种检测试剂盒进行检测(DNA微阵列芯片法)进行检测,操作严格按照说明书进行。取培养状况良好的临床分离株配制菌液(浓度为:1×106CFU/ml)后,加入核酸提取液采用核酸快速提取仪提取结核分枝杆菌DNA,取试剂盒中PCR反应体系18 μl加入2 μl样本DNA,PCR扩增结核分枝杆菌基因组DNA上的重复元件DR之间的序列,引物为DRa:5′-GGTTTTGGGTCTGACGAC-3′; DRb:5′-CC-GAGAGGGGACGGAAAC-3′,按照扩增程序进行PCR扩增;PCR 反应条件: 96 ℃ 预变性5 min,96 ℃变性1 min, 55 ℃退火 1 min, 72 ℃延伸 30 s,30个循环, 72 ℃ 延伸5 min,将扩增产物变性(96 ℃ 变性5 min)后,取10 μl与杂交缓冲液配制杂交混合物后加入芯片点阵中进行杂交(表1);杂交后的芯片用芯片洗干仪洗涤、甩干后,运用激光扫描仪,运行分枝杆菌菌种鉴定基因芯片判别系统,进行芯片扫描和根据配套软件分析杂交结果。北京博奥生物有限公司菌种检测试剂盒内包含表面化学质控探针(QC)、杂交阳性外对照探针(EC)、空白对照(BC)、阴性对照探针(NC)、内对照探针(IC)作为芯片质量控制。
我国城市化建设速度,呈现出日益上升的趋势,在经济不断发展,人们生活水平不断提高的新时期,我国电力企业为了适应时代发展的要求,其施工技术与施工机械,也在逐步的变革中,电力自动化在电力工程中的运用,使电力工程的工作效率得到了很大的提高。但是,受到种种因素的制约,电力自动化系统,在运行时,还存在一些问题。这些问题对我国电力系统的整体质量产生了不同程度影响,因此,国家有关部门应该高度重视起来,在电力工程实现电力自动化的过程中,不断的加大工程监管力度和施工人员的技术水平,使得我国的电力工程自动化进程可以不断的加快。我国居民也会因此得到实惠,工业以及各项社会事业的建业也会因此得到更好的发展。

表1 分枝杆菌菌种鉴定芯片的探针排布
注 QC:表面化学质控探针;EC:杂交阳性外对照探针;BC:空白对照;NC:阴性对照探针;IC:内对照探针。探针1、探针2:芯片中两排不同突变位点检测探针;1、2、3、4、5代表做5次平行试验
(二)104株结核分枝杆菌复合群药物敏感性试验
1. 绝对浓度法:药物检测浓度、具体的检测方法和结果判定标准均参照文献[4]。药敏检测培养基药物浓度:异烟肼(1 μg/ml,10 μg/ml),利福平(50 μg/ml,250 μg/ml)。耐药结果的判定:异烟肼抑菌浓度,1 μg/ml以下细菌生长为敏感株;1~10 μg/ml为轻度耐药株;10 μg/ml以上为高度耐药株。利福平抑菌浓度,50 μg/ml以下细菌生长为敏感株; 50~250 μg/ml为轻度耐药株; 250 μg/ml以上为高度耐药株。
2.基因芯片法:采用北京博奥生物有限公司的基因芯片法药敏检测试剂盒进行此项检测,芯片探针分布见表2和表3,操作严格按照说明书进行。对104株结核分枝杆菌复合群提取DNA进行检测,检测基因包括:rpoB基因(引物为R1 5′-AGGACGTGGAGGCGATCA-3′; R2 5′-AACGGGTTGACCCGCGCGTA-3′);katG基因(引物:K1 5′-GCGGCGGTCGACATT-3′;K2 5′-CTCGAGGA-AACTGTTGTCCC-3′);inhA基因(引物为I1 5′-CGCAGCCAGGGCCTCGCTG-3′;I2 5′-CTCCGGTAACCAGGACTGA-3′)。芯片扫描后根据配套软件分析杂交结果,以突变信号值超过野生信号值2倍以上为阳性。北京生物有限博奥公司基因芯片法药敏检测试剂盒内包含QC、EC、BC、NC、IC作为芯片质量控制。

表2 利福平耐药相关rpoB基因的探针排列
注 QC:表面化学质控探针;EC:杂交阳性外对照探针;BC:空白对照;NC:阴性对照探针;IC:内对照探针;WT:野生型。探针1、探针2:芯片中两排不同突变位点检测探针;1、2、3、4、5代表做5次平行试验
(三)质量控制
绝对浓度法和Spoligotyping法均设有H37Rv作为阳性对照,作为质量控制。每份标本分二批做同一实验,检验实验方法的重复性。批内每个样本设2个复孔。基因芯片法有QC、EC、BC、NC、IC作为质量控制。并且每块芯片中每个探针重复做 5次平行试验。
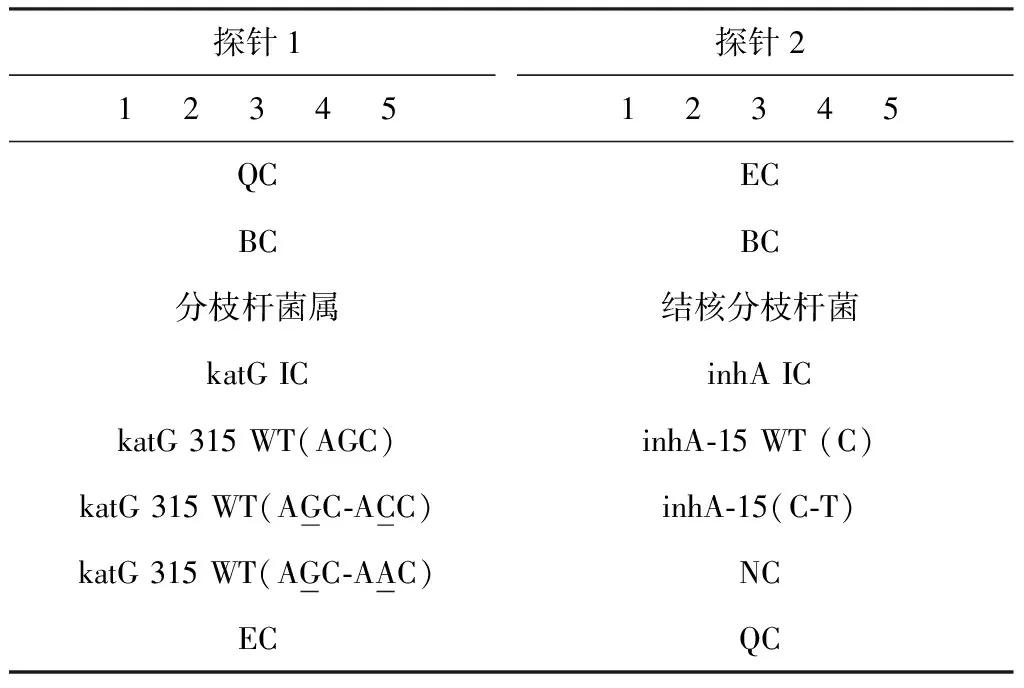
表3 异烟肼耐药相关katG基因和 inhA基因的探针排列
注 QC:表面化学质控探针;EC:杂交阳性外对照探针;BC:空白对照;NC:阴性对照探针;IC:内对照探针;WT:野生型。探针1、探针2:芯片中两排不同突变位点检测探针;1、2、3、4、5代表做5次平行试验
(四)统计学方法
采用SPSS 18.0统计学软件进行统计学分析,Spoligotyping法和基因芯片法菌种鉴定比较应用Kappa检验,绝对浓度法和基因芯片法药物敏感性比较采用χ2检验和Kappa检验。均以P<0.05为差异有统计学意义。
结 果
1.基因芯片法检测104株结核分枝杆菌复合群对利福平、异烟肼耐药的结果。基因芯片扫描图谱见图1、2。
2.基因芯片检测法与Spoligotyping法基因型分析的比较:具体见表4。137株临床分离株中,104株分离株基因芯片法检测鉴定为结核分枝杆菌复合群,与Spoligotyping法检测比较(以Spoligotyping法为金标准),符合率为100.0%(104/104)。 基因芯片法检测鉴定7株鸟分枝杆菌,与Spoligotyping法检测为9株比较,符合率为77.8%(7/9)。15株胞内分枝杆菌,与Spoligotyping法检测为16株比较,符合率为93.8%(15/16)。基因芯片法检测鉴定出1株偶然分枝杆菌,而Spoligotyping法未检测出偶然分枝杆菌,符合率为0.0%(0/0)。基因芯片法检测鉴定出10株龟或脓肿分枝杆菌,而Spoligoty-ping 法仅检测出其中8株,符合率为100.0%(8/8)。经Kappa检验,Kappa=0.95,U=30.6,P<0.05。表明两方法结果具有极强的一致性。
3.药敏试验基因芯片检测法与绝对浓度法对利福平耐药检测结果:104株结核分枝杆菌复合群中绝对浓度法有45株为利福平和异烟肼敏感株;37株为利福平耐药株;22株为异烟肼耐药株。共82株结核分枝杆菌分离株,其中绝对浓度法和基因芯片法均为利福平耐药分离株有35株,与之相关的rpoB基因突变位点分布(表5)表明:531位点突变42.9%(15/35),其中531 TCG→TTG 为37.1%(13/35);516位点突变54.3%(19/35),其中516 GAC→GTC为31.4%(11/35);511 CTG→CCG 14.3%(5/35);526位点突变17.1%(6/35),其中CAC→TAC为 8.6%(3/35)。
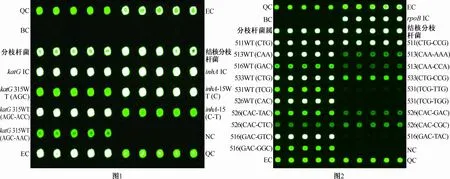
QC:基因芯片法有表面化学质控探针;EC:杂交阳性外对照探针;BC:空白对照;NC:阴性对照探针;IC:内对照探针。白色斑点为强信号(阳性),绿色斑点为弱信号,无色斑点为无信号(阴性)。CAC-CTC表示碱基A突变为碱基C,其他依此类推图1,2 图1为katG、inhA基因芯片扫描图谱;图2为rpoB基因芯片扫描图谱
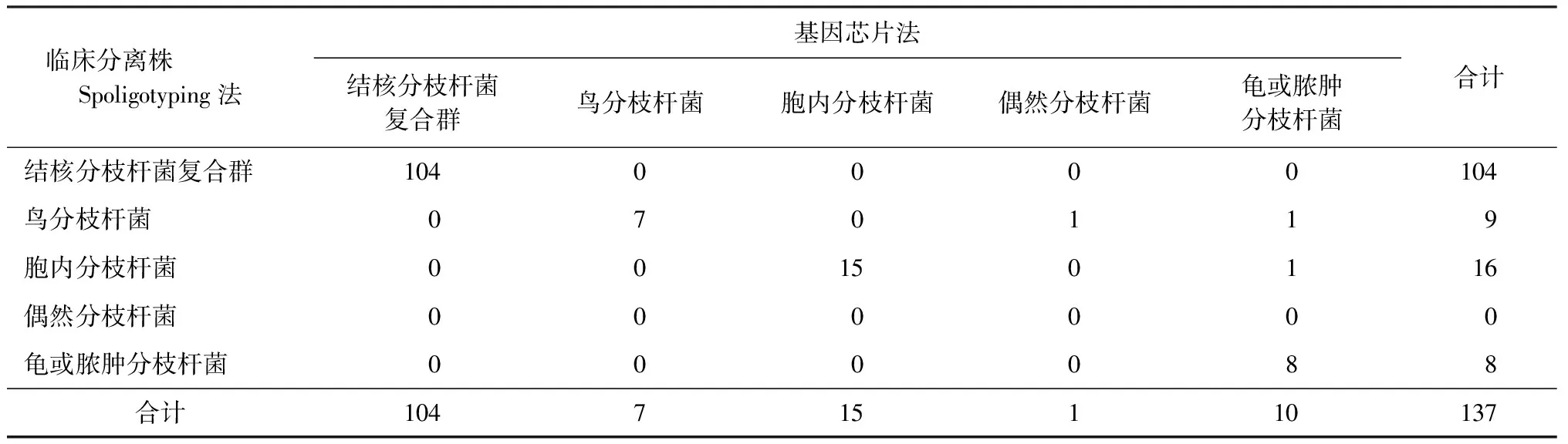
表4 基因芯片法与Spoligotyping法对137株临床分离株的菌种鉴定比较(株)
药敏试验基因芯片检测法与绝对浓度法对利福平耐药检测结果的比较具体见表6。表明18株绝对浓度法轻度耐药株中有16株基因芯片法rpoB基因突变,符合率即敏感度为88.9%(16/18)。19株高度耐药菌株中全部基因芯片法rpoB基因突变,符合率即敏感度为100.0%(19/19)。45株绝对浓度法敏感株中有42株基因芯片法为野生型,特异度93.3%(42/45),经χ2检验,绝对浓度法检测结果与基因芯片法检测耐药株检测结果相比差异有统计学意义(χ2值分别为41.3、51.6,P值均<0.01)。经Kappa检验,Kappa=0.81,U=10;Kappa=0.887,U=14.8,P<0.05。表明两方法结果具有极强的一致性。
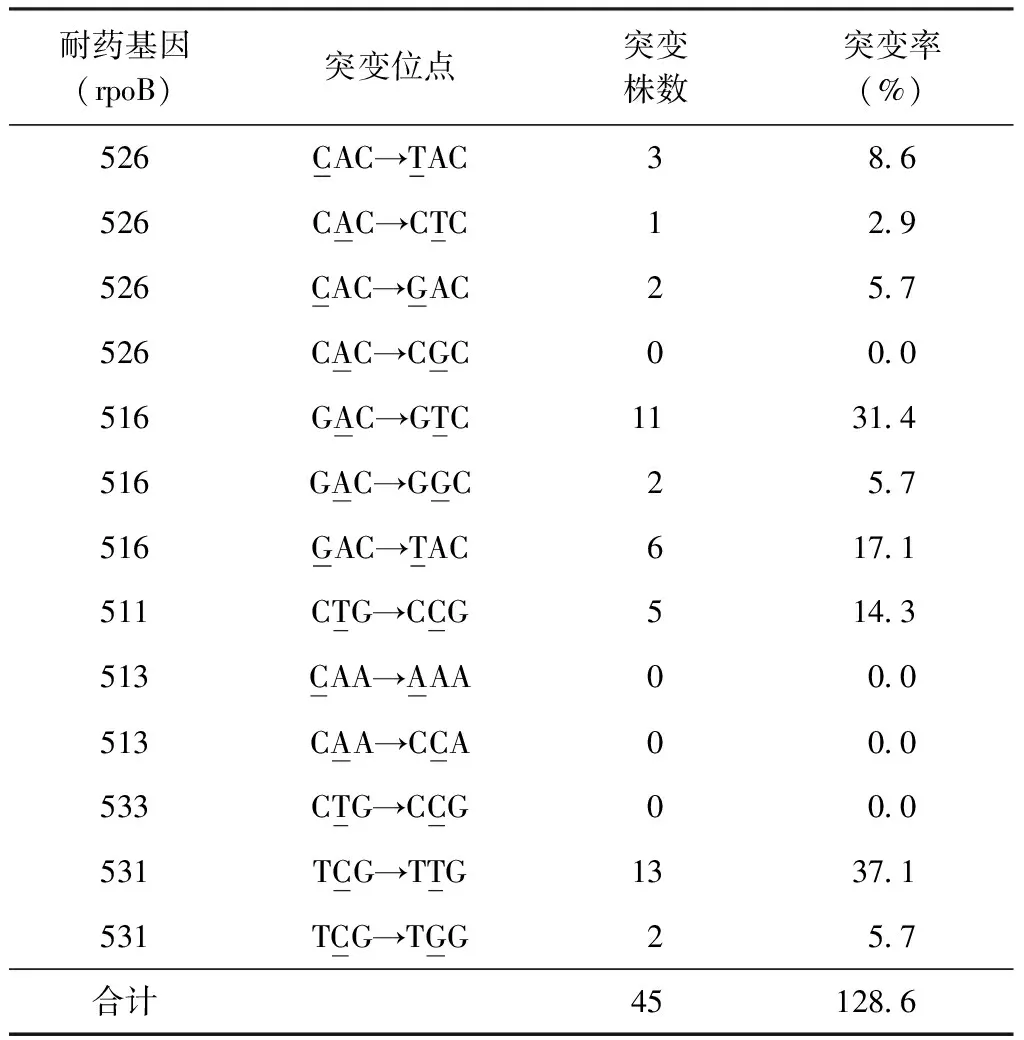
表5 35株利福平耐药相关的rpoB基因突变位点分布
注 突变率=耐药菌株某位点突变株数/耐药菌株株数×100%
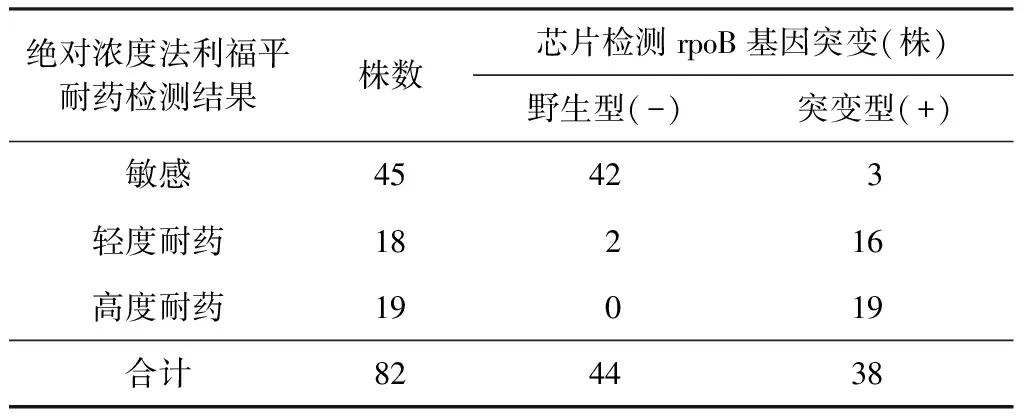
表6 基因芯片法与绝对浓度法对利福平耐药检测结果的比较
注 “+”:耐药;“-”:敏感
4.药敏试验基因芯片检测法与绝对浓度法对异烟肼耐药性检测的结果:104株结核分枝杆菌复合群中绝对浓度法有45株为利福平和异烟肼敏感株;22株为异烟肼耐药株;37株为利福平耐药株。共67株结核分枝杆菌分离株,其中绝度浓度法和基因芯片法均为异烟肼耐药分离株有21株, 与异烟肼耐药相关的katG、inhA基因突变位点分布(表7):katG基因315位点突变率100.0%(21/21),其中katG315 AGC→ACC突变率为85.7%(18/21);inhA基因15位点突变率0.0%(0/21),15 C→T。
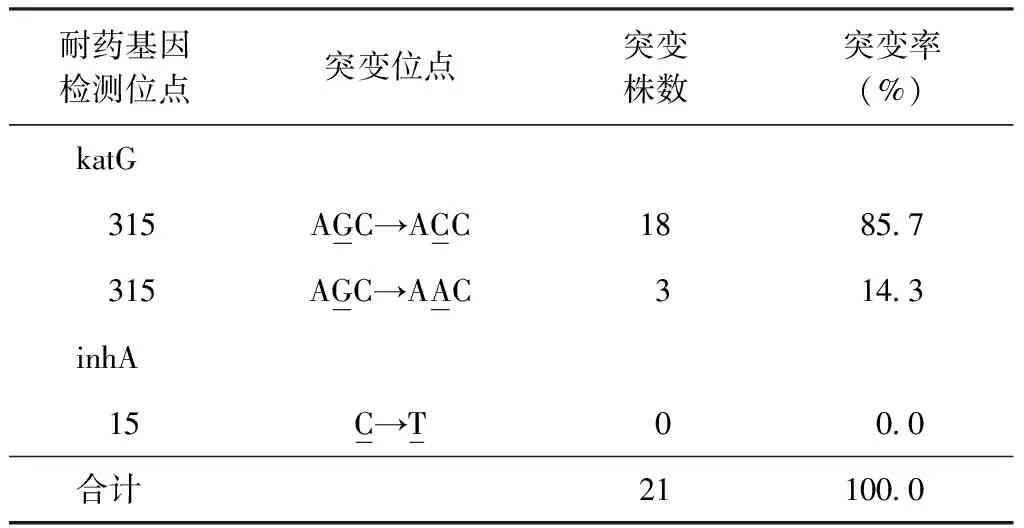
表7 21株异烟肼耐药株(绝度浓度法和基因芯片法)相关的katG、inhA基因突变位点分布
注 突变率=耐药菌株某位点突变株数/耐药菌株株数×100%
药敏试验基因芯片检测法与绝对浓度法对结核分枝杆菌临床分离株异烟肼耐药性检测的结果的比较具体见表8。13株绝对浓度法轻度耐药株(1~10 μg/ml)中有12株基因芯片法katG基因突变,敏感度92.3%,0株基因芯片法inhA基因突变,敏感度0.0%。9株绝对浓度法检测为高度耐药株(10 μg/ml以上)中全部基因芯片法katG基因突变,敏感度100.0%;0株基因芯片法inhA基因突变,敏感度0.0%。45株绝对浓度法检测为敏感株中,45株基因芯片法katG基因为野生型,特异度为100.0%;43株基因芯片法inhA基因为野生型,特异度95.6%。基因芯片法katG基因突变与绝对浓度法比较,经χ2检验,基因芯片法检测与绝对浓度法比较差异有统计学意义(χ2值分别为46.9、36.5,P值均<0.01)。经Kappa检验,Kappa=0.94,U=17.1;Kappa=1,U=∞,P值均<0.05;表明两种方法结果一致性好。经χ2检验,基因芯片法inhA突变与绝对浓度法检测结果相比较差异无统计学意义(χ2值分别为0.01、0,P值均>0.05。经Kappa检验,Kappa=-0.08,U=-0.33;Kappa=0,U=0,P值均>0.05)。
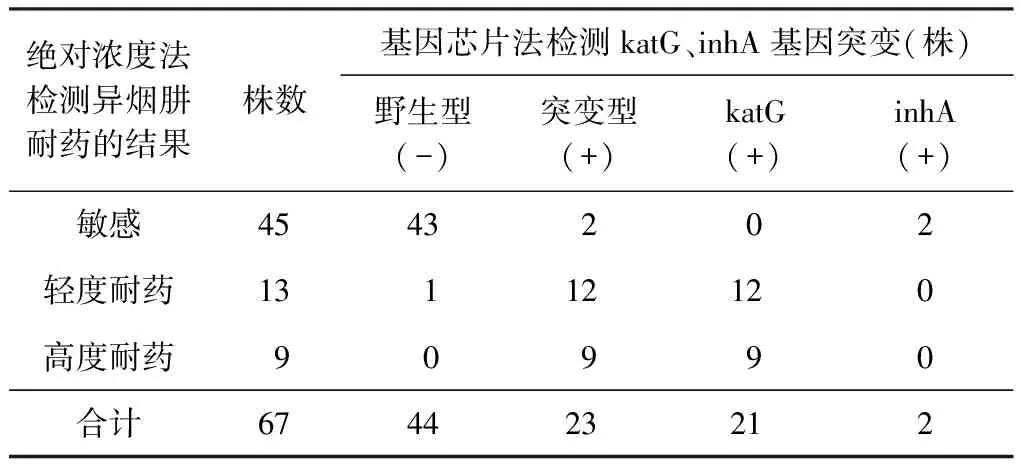
表8 基因芯片法与绝对浓度法对异烟肼耐药的检测结果比较
注 “+”:耐药;“-”:敏感
讨 论
基因突变是导致结核分枝杆菌产生耐药性的重要原因,有研究表明95%的结核分枝杆菌利福平耐药株有RNA聚合酶β亚单位编码基因rpoB507-533位密码子发生碱基突变[5],至高度保守的氨基酸置换,空间构象发生变化,最常见突变位点为531、526、516。结核分枝杆菌异烟肼耐药株主要有KatG基因和inhA基因碱基突变[6-7],常见的为KatG基因315位密码子AGC-ACC的突变,从而引起细胞过氧化氢-过氧化物酶活性降低,导致异烟肼活化效率降低,甚至不能活化。分枝杆菌酸细胞壁的合成所需成份之一为NADH依赖的enoyl-ACP还原酶,即inhA基因编码的脂肪酸转动蛋白,活化的异烟肼能与NADH结合,抑制依赖的enoyl-ACP还原酶,从而阻止分枝杆菌酸细胞壁的合成,导致细胞的死亡。如果inhA基因碱基突变,enoyl-ACP还原酶分泌受阻,活化的异烟肼不能与NADH结合,而导致耐药。
基因芯片技术始创于20世纪90年代,是目前结核分枝杆菌菌种分型和耐药分子诊断的先进方法之一,很多学者应用基因芯片方法报道了不同地区结核分枝杆菌菌种类型和耐药基因突变位点,并与测序比较有很好的一致性,大多数是对耐利福平的rpoB基因和耐异烟肼的KatG基因和inhA基因突变位点的检测。
笔者应用基因芯片技术对收集的绝对浓度法耐利福平耐药株37株,异烟肼耐药株22株,全敏感株45株进行了rpoB(19个位点)、katG(4个位点)、inhA(3个位点)检测,表明利福平轻度耐药株敏感度为88.9%,高耐药株敏感度为100.0%,特异度为93.3%,rpoB基因位点突变主要有42.9%的为531,17.1%为526,54.3%为516,14.3%为511位点突变。突变位频率较高有531TCG→TTG 37.1%,516GAC→GTC 31.4%,511CTG→CCG 14.3%,与Huang等[5]对中国、日本、韩国等国家大多数结核分枝杆菌耐利福平菌株rpoB基因突变位点在Ser-531,Yao等[8]对重庆地区结核分枝杆菌临床株rpoB、katG、inhA基因位点突变检测表明突变频率较高的基因位点为rpoB 531、rpoB 513,Mokrousov等[9]对北京地区结核分枝杆菌临床株rpoB基因位点突变主要有58.9%的为531,12.4%为526,2.7%为516位点,Zhang等[10]对河南省耐多药结核分枝杆菌菌株基因突变检测表明53.8%为rpoB 531点突变,Deng等[11]对武汉地区60株确诊为肺结核患者的临床分离株应用基因芯片技术分析rpoB基因型,结果表明531TCG→TTG突变率为47.3%,526CAC→TAC突变率为12.7%,516GAC→GTC突变率为5.5%比较有高度的一致性,表明各地区之间结核病利福平耐药rpoB基因突变位点和频率区别不大。
基因芯片法检测表明异烟肼轻度耐药敏感度为92.3%,高耐药敏感度为100.0%,特异度为95.6%,katG基因位点突变有95.4%的为315位点突变,突变位点频率较高有315AGC→ACC 81.8%。本研究的结果与报道的重庆、北京、河南、武汉结核分枝杆菌异烟肼耐药的敏感度、特异度相似[8-11]。
实验中2株inhA基因15突变均为敏感株,而耐药株无inhA基因15突变,因此可以说INH耐药至少在本地区可能与inhA基因15位突变无关,也可能为绝对浓度法定义的耐药浓度高于比率法原因所致。与Yao等[8]报道的18%的inhA-15突变不一致,这可能是我国各地区结核病流行菌型差异所至。与Aragón 等[12]报道的31.6% inhA-15、10.5% inhA-8突变相差很大,可能是与西班牙巴塞罗那地区所在的国际环境结核分枝杆菌流行趋势大相径庭的缘故。也有中国研究人员在对本地区耐多药结核病耐药基因检测研究中没有把inhA基因选入检测中[13],这大概也是inhA在中国某些地区突变率低的缘故。表明异烟肼耐药可能与inhA突变无关,也可能为绝对浓度法定义的耐药浓度高于比率法的原因。
本研究对高耐药结核分枝杆菌(利福平250 μg/ml以上;异烟肼10 μg/ml以上)芯片法检测发现全部菌株都有耐药基因突变。因此可以认为高耐药与结核分枝杆菌耐药基因突变呈强相关。
王峰等[14]利用基因芯片法检测了21株结核分枝杆菌标准株和50 株临床分离株,结果表明:与PCR测序方法比较,两种方法具有很好的一致性,且基因芯片鉴定分枝杆菌具有快速、特异、准确率高的特点,笔者应用北京博奥生物有限公司基因芯片与Spoligotyping方法对照检测结果与王峰等的结果一致。可见基因芯片检测方法与PCR测序和Spoligotyping方法均有较好的一致性,且基因芯片方法有检测时间短(当天可以出结果)、人为影响因素少、重复性好等特点,更适合临床应用。
张俊仙等[15]利用基因芯片法检测了30株利福平、异烟肼敏感株和50株利福平、异烟肼耐药株,结果表明,与基因测序比较,敏感株特异度为100%,利福平耐药株rpoB基因突变率为86%,异烟肼耐药株katG基因突变率为62%、inhA基因突变率仅为10%。本研究利用基因芯片法与传统绝对浓度法检测利福平和异烟肼耐药性,比较结果与张俊仙等的结果一致,且本研究检测inhA基因突变率为0%,与张俊仙等的仅10%突变率结果相近,表明异烟肼耐药与inhA基因突变相关性不大。且基因芯片法结果的快速、准确更是值得在临床检测中应用。施美华等[16]应用北京博奥生物有限公司基因芯片检测了21株临床标本对INH的药敏试验表明基因芯片法检测菌株INH耐药与传统药敏检测方法比较,准确率为73.8%,与DNA测序比较,芯片法检测katG基因突变准确率为88.9%。笔者基因芯片法检测为绝对浓度法轻度耐药株(1~10 μg/ml)中katG基因突变的敏感度92.3%(12/13),绝对浓度法高耐药株(10 μg/ml以上)中全部芯片法katG基因突变,敏感度100.0%(9/9)。高于施美华等的检测结果。表明绝对浓度法结果比罗氏耐药培养结果更准确,因此新方法的验正应以绝对浓度法为金标准。
总之,基因芯片法有较高的敏感度和特异度,且检测时间短,检测费用一样。基因芯片法是值得推广应用的一种耐药检测方法。但基因芯片法需要昂贵的芯片、芯片杂交仪、芯片洗干仪、激光扫描仪等耗材和设备,不易在基层推广,需要进一步改进。
[1] Heymann SJ, Brewer TF, Wilson ME,et al. The need for global action against multidrug-resistant tuberculosis. JAMA,1999,281(22):2138-2140.
[2] Lee AS, Lim IH, Tang LL, et al. Contribution of kasA analysis to detection of isoniazid-resistant Mycobacterium tuberculosis in Singapore. Antimicrob Agents Chemother, 1999,43(8):2087-2089.
[3] 端木宏谨,屠德华,张培元. 临床诊疗指南结核病分册. 北京:人民卫生出版社,2005:10-11.
[4] 中国防痨协会. 结核病诊断细菌学检验规程. 中国防痨杂志,1996,18(1):28-31.
[5] Huang H, Jin Q, Ma Y, et al. Characterization of rpoB mutations in rifampicin-resistant Mycobacterium tuberculosis isolated in China. Tuberculosis (Edinb), 2002,82(2/3): 79-83.
[6] Cade CE,Dlouhy AC,Medzihradszky KF,et al.Isoniazid-resis-tance conferring mutations in Mycobacterium tuberculosis KatG:catalase,peroxidase,and INH-NADH adduct formation activities. Protein Sci,2010,19(3):458-474.
[7] Luckner SR, Liu N, am Ende CW, et al. A slow, tight bin-ding inhibitor of InhA,the enoyl-acyl carrier protein reductase from Mycobacterium tuberculosis.J Biol Chem,2010,285(19):14330-14337.
[8] Yao C, Zhu T, Li Y, et al. Detection of rpoB, katG and inhA gene mutations in Mycobacterium tuberculosis clinical isolates from Chongqing as determined by microarray. Clin Microbiol Infect, 2010,16(11):1639-1643.
[9] Mokrousov I, Jiao WW, Sun GZ,et al.Evaluation of the rpoB macroarray assay to detect rifampin resistance in Mycobac-terium tuberculosis in Beijing China. Eur J Clin Microbiol Infect Dis, 2006,25(11):703-710.
[10] Zhang SL, Shen JG, Xu PH, et al.A novel genotypic test for rapid detection of multidrug-resistant Mycobacterium tuberculosis isolates by a multiplex probe array. J Appl Microbiol, 2007,103(4):1262-1271.
[11] Deng JY, Zhang XE, Lu HB, et al. Multiplex detection of mutations in clinical isolates of rifampin-resistant Mycobacterium tuberculosis by short oligonucleotide ligation assay on DNA chips. J Clin Microbiol, 2004,42(10):4850-4852.
[12] Aragón LM, Navarro F, Heiser V,et al. Rapid detection of specific gene mutations associated with isoniazid or rifampicin resistance in Mycobacterium tuberculosis clinical isolates using non-fluorescent low-density DNA microarrays. J Antimicrob Chemother, 2006,57(5):825-831.
[13] 李锋,李凫坚,陈园园,等. 应用DNA芯片法检测耐多药结核分枝杆菌的研究. 医学研究杂志,2011, 40(1):115-117.
[14] 王峰,朱玉梅,桂静,等. PCR测序和基因芯片快速鉴定分枝杆菌菌种的应用研究. 中国防痨杂志,2011,33(11):713-717.
[15] 张俊仙,吴雪琼,阳幼荣,等. 应用基因芯片方法检测结核分枝杆菌利福平和异烟肼的耐药性. 中国防痨杂志,2011,33(10):680-685.
[16] 施美华,唐佩军,叶志坚,等. 基因芯片法对苏州市结核临床分离株INH耐药性快. 结核病与肺部健康杂志,2013,2(3):164-168.
(本文编辑:王然 张晓进)
Identification ofMycobacteriumtuberculosisand its drug resistance with gene chip
SHI Guo-min, YU Rong, PENG Xue-feng, SHI Yan, NIE Ying, QI Zhi-qiang, CHEN Yong-jun, XIANG Yan-gen,LIU Zhong-quan.
Clinical Laboratory, Changsha Central Hospital, Changsha 410004, China
Corresponding author: LIU Zhong-quan, Email: Liuzq6608@tom.com
Objective Study on gene chip technique in detection of drug resistance of Mycobacterium tuberculosis and its clinical value. Methods (1) One hundred and thirty-seven strain identification of Mycobacterium clinical isolates from Changsha Central Hospital by gene chip and Spoligotyping technology from 2011-01 to 2012-12. (2)Drug-susceptibility test of rifampin and isoniazid to Mycobacterium tuberculosis clinical isolates by absolute concentration method. (3) Drug-susceptibility test of rifampin and isoniazid to Mycobacterium tuberculosis clinical isolates by DNA chips for rpoB, KatG, inhA. Rifampin resistant is one or a plurality of site mutation of rpoB gene, isoniazid resistance is one or a plurality of site mutation of katG gene and inhA gene. Compared Spoligotyping with gene chip method of species identification byKappatest, Compared the absolute concentration method with gene chip method of drug sensitivity byχ2test andKappatest. Results (1)For 45 rifampin sensitive (under 50 μg/ml) and isoniazid sensitive(under 1 μg/ml) isolates tested by absolute concentration method, 42 are wild type for rpoB gene with the sensitivity of 93.3%(1 for 511T-C mutations, 1 for 531C-T mutations, 1 for 516A-T mutations), 45 are wild type for KatG gene with the sensitivity of 100.0% and 43 are wild type for inhA gene with the sensitivity of 95.6%(2 for15C-T mutation) by DNA chip method, respectively. (2)For 18 rifampin-resistant (50-250 μg/ml) isolates tested by absolute concentration method, 16 has mutation in rpoB gene with the sensitivity of 88.9% by DNA chip method. Those mutations happened at 531,516,526,511 amino acid sites of rpoB. For 19 rifampin-resistant (above 250 μg/ml) isolates tested by absolute concentration method, 19 has mutation in rpoB gene with the sensitivity of 100% by DNA chip method. Those mutations happened at 531,516,526,511 amino acid sites of rpoB. (3)Compare the DNA chips with the absolute concentration method, for 13 isoniazid- resistance(1-10 μg/ml) isolates, 12 has mutation in KatG gene with the sensitivity of 92.3% by DNA chip method, which mainly happened at 315 site. No mutation related to the drug resistance was found in inhA gene. Compare the DNA chips with the absolute Concentration method, For 9 isoniazid- resistance(above 10 μg/ml) isolates, 9 has mutation in KatG gene with the sensitivity of 100% by DNA chip method, which mainly happened at 315 site. No mutation related to the drug resistance was found in inhA gene. (4)For 137 clinical isolates tested by Spoligotyping, 104 are Mycobacterium tuberculosis with the sensitivity of 100.0%(104/104), 7 are Mycobacterium avium complex with the sensitivity of 77.8%(7/9), 15 are Mycobacterium intracellulare with the sensitivity of 93.8%(15/16), 1 are Mycobacterium fortuitum with the sensitivity of 0.0%(0/0), 10 are Mycobacterium chelonei with the sensibility of 100.0%(8/8) by DNA chip method, respectively.Kappa=0.95,U=30.6,P<0.05. Conclusion The drug-sensitive test by DNA chips method matched the absolute concentration method very well. RFP-resistance was related to the mutations at the sites of 531,516,526,511 for rpoB gene, and INH-resistance was related to the mutations at the site of 315 for KatG gene. No mutation was found in inhA gene in INH-resistant isolates. DNA chip method might be a rapid and effective method for the detection of Mtb drug-resistant isolates and mycobacterium species.
Mycobacterium tuberculosis; Drug resistance, bacterial; Microbial sensitivity tests; Oligonucleotide array sequence analysis
10.3969/j.issn.1000-6621.2015.01.013
“十二五”国家科技重大专项(2012ZX10005011-003-001);首都卫生发展科研专项(2011-1010-01)
410004 长沙市中心医院检验科(石国民、喻容、彭雪峰、石燕、聂英、齐志强、陈拥军、向延根);北京市结核病胸部肿瘤研究所 耐药结核病北京市重点实验室 (刘忠泉)
刘忠泉,Email:Liuzq6608@tom.com
2014-05-04)

