基于β-连环蛋白基因的分子标记甄别海蜇(Rhopilema esculentum)和沙海蜇(Nemopilema nomurai)*
杨傲傲 朱 玲① 周春娅 杨 洪 骆晓蕊 刘春胜 庄志猛
(1. 上海海洋大学水产与生命学院 上海 201306; 2. 农业部海洋渔业可持续发展重点实验室 山东省渔业资源与生态环境重点实验室 中国水产科学研究院黄海水产研究所 青岛 266071)
海蜇(Rhopilema esculentum)和沙海蜇(Nemopilema nomurai)同属于刺胞动物门(Cnidaria)、钵水母纲(Scyphozoa)、根口水母目(Rhizostomeae)、根口水母科(Rhizostomatidae), 广泛分布于西北太平洋沿岸海域, 在我国主要分布于东海北部和黄、渤海海域(董婧等, 2012)。海蜇隶属于海蜇属(Rhopilema), 目前作为国际公认品质最好的可食用大型水母, 具有极高的经济价值。同时, 海蜇也是我国重要海洋渔业捕捞、增养殖和放流对象(谢忠明等, 2004)。而沙海蜇属于沙蜇属(Nemopilema), 经济价值较低, 但是作为世界上最大的水母之一, 是我国近海暴发的主要大型灾害性水母(程家骅等, 2004)。近年来, 沙海蜇频繁暴发于黄海海域及东海北部海域。沙海蜇的大量暴发性增殖, 一方面严重改变了自然海洋生态系统的结构与功能, 另一方面也严重影响了其它海洋渔业资源产量以及正常的海洋渔业生产, 而且对滨海旅游和工业活动等都产生了重要影响(孙松等, 2012)。
海蜇和沙海蜇的生活史复杂, 从受精卵到成体,经历浮浪幼虫、螅状体、横裂体、碟状体和水母体五个发育阶段, 不同发育阶段之间个体形态、结构差异显著(董婧等, 2012)。已有的研究结果表明: 沙海蜇与海蜇的水母体阶段可以根据伞径大小、外伞表面特征和附属器形态等直接用肉眼区分(洪惠馨, 2002)。但是在浮浪幼虫、螅状体、横裂体和碟状体阶段, 海蜇和沙海蜇的形态、结构特征极为相似, 几乎难以区别。尽管孙明等(2010)在实验室条件下对沙海蜇与海蜇晚期碟状体的形态特征进行研究, 认为可以通过比较生殖腺、胃丝和口腕等的细微形态差异进行区分(孙明等, 2010)。但由于水母具有含水量多, 采集易破碎, 保存易变形、失真等特点, 导致这种方法在野外采样后难以应用。
本文基于海蜇转录组454 GS FLX测序与PCR技术, 在海蜇和沙海蜇中分别克隆了包含 EST-SSR和EST-SNP标记的β-连环蛋白(β-catenin) 基因的目的片段, 并进行了生物信息学分析, 期望从分子水平上寻找一种简单、准确甄别海蜇和沙海蜇的分子标记,为海蜇和沙海蜇种群的栖息地调查、资源量或潜在暴发量的评估奠定理论基础。
1 材料与方法
1.1 海蜇转录组 454 GS FLX测序与β-连环蛋白基因的筛选
在浙江杭州湾采集野生性成熟的海蜇, 立即运回山东荣成养殖场进行海蜇人工繁育。分别选取海蜇不同发育阶段的个体(螅状体、横裂体、碟状体和幼蛰)提取总RNA, 并等比例混合, 然后采用SMARTTMcDNA Library Construction (Clontech)试剂盒构建海蜇cDNA文库。由美国夏威夷大学的ASGPB实验小组利用 Roche Next Generation Sequencer GS FLX System (a.k.a. 454 Sequencer)进行半板454 GS FLX测序。生物信息学分析后, 筛选与其它生物β-连环蛋白基因具有高度相似性的海蜇β-连环蛋白基因序列。
1.2 海蜇和沙海蜇样品的采集与基因组DNA提取
海蜇和沙海蜇样品分别采自青岛沙子口和胶州湾。液氮保存返回实验室后, 采用酚-氯仿法提取基因组DNA: 每个个体分别取约200mg液氮保存的样品进行研磨, 研磨至粉末状后, 立即加入 600μL的DNA提取液[其中Tris-HCl (pH 8.0) 100mmol/L, EDTA(pH 8.0) 100mmol/L, 1% SDS 50μL, 20mg/mL 蛋白酶K 8μL], 充分混匀, 55°C水浴 1—2h, 待组织充分裂解完全。加入等体积的酚-氯仿试剂[其中饱和酚、氯仿、异戊醇以体积比为25︰24︰1混合], 静置10min,12000r/min离心10min, 除去上清。如此反复抽提三次。然后加入0.6体积的异丙醇静置5min, 12000r/min离心 5min, 除去上清。加入 600μL已预冷的无水乙醇, 沉淀 DNA约 30min后, 70%乙醇洗涤沉淀两次,自然干燥2min, 加入20μL超纯水溶解。提取的DNA经1.5%琼脂糖电泳进行检测。
1.3 海蜇和沙海蜇β-连环蛋白基因目的片段的克隆与测序
利用软件SSRhunter 1.3 (http: //www.bio-soft.net/dna/SSRHunter.htm)识别海蜇β-连环蛋白基因序列中的微卫星重复的位置和重复单位的形式。然后利用Primer Premier 5.00在微卫星重复序列的两翼设计一对特异性引物β-catenin-F1 (5'-TATCAAACCAGGC ATGTCCACC-3')和β-catenin-R1 (5'-GCCCAGACCT ACCAAGATGA A-3'), 分别扩增海蜇和沙海蜇β-连环蛋白基因的目的片段。50μL的PCR反应体系包括:5μL 10×PCR buffer, 4μL dNTP (25mmol/L), 3μL MgCl2(25mmol/L), 上下游引物各 2μL (10μmol/L),0.4μL (1U) DNA Taq 酶, 2μL DNA 模板, 31.6μL PCR水; PCR反应条件为: 94°C预变性5min, 36个PCR循环(包括: 94°C变性45s, 53°C退火45s, 72°C延伸45s),最后 72°C总延伸10min。反应结束后, 用1.5%的琼脂糖凝胶电泳检测 PCR扩增产物, 对目的片段进行切胶回收后, 与 pMD18-T载体(TaKaRa)连接, 转化到大肠杆菌感受态细胞(Top10)中。最后通过蓝白斑筛选阳性克隆, 对阳性克隆送华大基因公司进行测序(每个物种随机选取10个体); 剩余PCR扩增产物用于聚丙烯酰胺凝胶电泳。
1.4 海蜇和沙海蜇β-连环蛋白基因片段的生物信息学分析
运用 BLAST技术(http: //www.ncbi.nlm.nih.gov/blast)和蛋白分析系统(http: //www.expasy.org/), 作者分析了海蜇β-连环蛋白基因的cDNA和氨基酸序列。用ClustalW (http: //www.ebi.ac.uk/clustalw/)对海蜇和沙海蜇群体的β-连环蛋白基因片段进行多序列比对, 识别海蜇与沙海蜇的SSR特征序列和单核苷酸多态位点。
1.5 聚丙烯酰胺凝胶电泳与图谱分析
PCR扩增产物通过6%聚丙烯酰胺凝胶垂直电泳55W, 2.5h, 然后银染, 显色, 拍照, 根据DNA长度标准 pBR322/MspI, 确定等位基因的长度范围。采用Genepop 4.0统计等位基因数, 计算表观杂合度和期望杂合度, 进行哈迪-温伯格平衡背离测试, 并进行成对标记间连锁不平衡测试。
2 结果与分析
2.1 海蜇β-连环蛋白基因的筛选与同源性分析
生物信息学分析表明, 在半板海蜇454 GS FLX转录组测序结果中, 共有7个Est序列与其它生物β-连环蛋白基因具有高度相似性。将上述 EST序列拼接后, 共获得长1833 bp的海蜇β-连环蛋白基因片段。推导的氨基酸序列经 BLAST分析发现, 海蜇β-连环蛋白基因片段与来自刺胞动物门的 Hydractinia echinata (ACZ51403)、Podocoryna carnea (ABI74628)、Hydra magnipapillata (AFI99111)和Hydra vulgaris(AAQ02885)的β-连环蛋白基因具有很高的同源性,其氨基酸序列一致性高达 84%—89%。与长牡蛎Crassostrea gigas (AFL93714)、海鞘 Ciona savignyi(BAA32789)和 文 昌 鱼 Branchiostoma floridae(BAD12593)的氨基酸序列一致性也分别高达 79%、78%和82%。
2.2 海蜇β-连环蛋白基因微卫星筛选与PCR扩增
利用软件 SSRhunter 1.3搜索拼接后的海蜇β-连环蛋白基因片段, 在序列 342—360bp处发现一个微卫星序列, 其重复单位为(TGC)6。分别以海蜇和沙海蜇的基因组 DNA为模板, 用海蜇β-连环蛋白基因片段的一对特异性引物β-catenin-F1和β-catenin-R1进行PCR扩增并测序, 结果显示: 10个海蜇个体的β-连环蛋白基因扩增片段的大小分别为166bp和169bp, 其中169bp与已知的β-连环蛋白cDNA序列的大小相同,表明扩增的目标片段中没有内含子存在。而10个沙海蜇个体的β-连环蛋白基因扩增片段的大小分别为157bp和160bp。
2.3 海蜇和沙海蜇β-连环蛋白基因目的片段特征分子标记分析
海蜇和沙海蜇β-连环蛋白基因的目的片段核苷酸多序列比对见图1。比对结果显示, β-连环蛋白基因在海蜇和沙海蜇中是高度保守的, 海蜇个体β-连环蛋白基因目的序列之间, 除了微卫星重复差异外, 只有一个碱基的差异; 而沙海蜇个体之间除了微卫星重复差异外, 其余完全一致。海蜇和沙海蜇的β-连环蛋白基因目的片段之间, 在相同位置上都存在微卫星重复, 但所包含的微卫星重复单元截然不同, 海蜇为:(TGC)4-6(TGT)1-2(TGC)4-5, 而海蜇为(TGT)5-6。除此之外,海蜇和沙海蜇的β-连环蛋白基因目的片段之间还存在 14个能够甄别两物种的单核苷酸多态位点, 分别为: (T/C)1, (T/C)2, (C/T)3, (C/T)4, (C/T)5, (T/G)6,(G/C)7, (T/G)8, (A/G)9, (C/T)10, (G/A)11, (A/G)12,(C/T)13, (A/T)14。

表1 海蜇和沙海蜇β-连环蛋白基因的多态微卫星位点的特征Tab.1 Characteristics of SSR from β-catienin in R. esculentum and N. nomurai
2.4 微卫星位点的多态性分析
根据 DNA长度标准 pBR322/MspI, 在聚丙烯酰胺凝胶上, 海蜇和沙海蜇群体的 PCR扩增产物长度主要分布在166 bp和160 bp, 见图2。遗传学分析显示: 该位点在海蜇和沙海蜇群体中的期望杂合度分别为 0.4998和 0.7482, 表观杂合度分别为 0.5208和0.7083, 等位基因数均为2。通过哈迪-温伯格平衡背离测试, Bonferroni校正后均未发现偏离现象(表1)。
3 讨论
随着分子生物技术和生物信息学的快速发展,利用DNA分子标记对海蜇与沙海蜇进行物种鉴定、探讨物种分类地位及种间亲缘关系已经成为传统分类学的重要辅助手段。刘敏等(2010, 2011)分别利用18S rDNA序列和COI序列对东黄海沙海蜇与口冠水母分类关系进来了辨析, 认为东黄海野生沙海蜇与越前水母的亲缘关系很近, 应为同一物种, 与海蜇的亲缘关系较远, 与口冠水母的关系更远。张姝等(2009)也利用16S rDNA和COI基因序列对海蜇和沙海蜇进行了分子鉴定, 认为线粒体基因作为分子标记可能是解决水母幼体分类困扰的有效途径之一。本研究在海蜇转录组454 GS FLX测序基础上, 通过对EST序列的深度解析, 在β-连环蛋白基因序列中发掘出同时包含EST-SSR和EST-SNP两种分子标记的目的片段,只需要设计一对引物, 分别从海蜇和沙海蜇的基因组 DNA中扩增出目的片段, 经聚丙烯酰胺凝胶电泳即可进行海蜇和沙海蜇的物种甄别。该方法不但简单、快速、准确, 同时也为EST-SSR和EST-SNP分子标记在物种鉴定和亲缘关系分析中的应用提供了一种新思路。

基于EST序列开发的EST-SSR标记与从基因组开发的SSR标记相比, 具有以下优点: a) EST-SSR是在已有的 EST序列中进行筛选, 所以该开发过程不仅简单, 成本也低; b) EST-SSR标记通常来自较为保守的转录区, 故而开发出的分子标记在近缘物种间具有较高的通用性和可转移性; c) EST-SSR 代表基因的编码部分, 因此可作为功能基因的“绝对”的标记, 也可用于直接鉴定一些决定重要表型性状的等位基因; d) 通过序列同源性比对, 可以分析获得EST-SSR标记的某种可能功能(Scott et al, 2000;Areshehenkova et al, 2002)。本研究所获得的β-连环蛋白基因分子标记正是利用了 EST-SSR的这些优点。β-连环蛋白作为 Wnt/β-catenin信号通路的核心成员,几乎参与了生物体从胚胎到成体所有的发育过程(Peifer, 1997)。例如调控细胞的增殖和生长(Logan et al, 2004)、保障各种干细胞的功能(Willert et al, 2005)、参与细胞的分化(Prestwich et al, 2007)、控制胚胎早期发育(Klaus et al, 2008)等。通过对目前已在GenBank注册的β-连环蛋白进行生物信息学分析发现: 这个位于β-连环蛋白基因C末端的SSR重复序列仅存在于刺胞动物海蜇、沙海蜇及 H. echinata(ACZ51403)中, 其它动物类群均不存在。尽管目前尚不知道这个微卫星序列对β-连环蛋白的生物功能的影响, 但越来越多证据表明, 位于编码区的SSR序列或者直接影响基因编码产物功能(Ellegren, 2000;Huang et al, 2002), 或者通过与转录因子结合进而调控基因表达(Bachtrog et al, 2000), 或者通过影响局部染色质结构等发挥着重要作用(Dupuy et al, 2004)。已有研究表明, β-连环蛋白的C末端与核转录因子淋巴样增强因子-1/T-细胞因子的结合, 形成转录复合物,进入细胞核后激活下游基因的表达, 发挥转录因子的作用(Kuhl et al, 2001), 因此推测该微卫星序列可能通过与转录因子结合进而调控基因表达, 从而参与刺胞动物的发育过程(Bachtrog et al, 2000)。

图1 海蜇和沙海蜇β-连环蛋白基因目的片段的核苷酸多序列比对Fig.1 Multiple alignment of β-catienin target nucleotide sequences from R. esculentum and N. nomurai
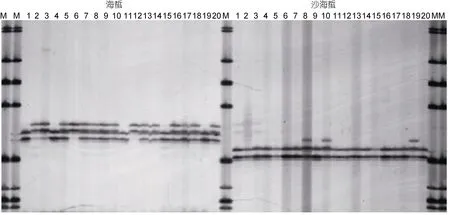
图2 海蜇和沙海蜇β-连环蛋白基因的多态微卫星图谱Fig.2 SSR profile from β-catienin in R. esculentum and N. nomurai populations
刘 敏, 马凌波, 凌建忠等, 2011. 东黄海沙海蜇与口冠水母分类关系的辨析——基于核糖体18S rDNA基因序列. 海洋渔业, 33(2): 131—137
刘 敏, 程家骅, 马凌波等, 2010. 东黄海沙海蜇群体线粒体COI基因序列多态性. 海洋渔业, 32(2): 120—124
孙 松, 于志刚, 李超伦等, 2012. 黄、东海水母暴发机理及其生态环境效应研究进展. 海洋与湖沼, 43(3): 401—405
孙 明, 董 婧, 赵 云等, 2010. 沙蜇与海蜇晚期碟状体的形态学研究. 渔业科学进展, 31(1): 48—53
张 姝, 张 芳, 刘 媛等, 2009. 我国海域两种大型水母的分子鉴定. 海洋与湖沼, 40(1): 94—101
洪惠馨, 2002. 水母和海蜇. 生物学通报, 37(2): 13—16
董 婧, 孙 明, 赵 云等, 2012. 中国北部海域灾害水母沙蜇(Nemopilema nomurai)及其它钵水母繁殖生物学特征与形态比较. 海洋与湖沼, 43(3): 550—555
程家骅, 李圣法, 丁峰元等, 2004. 东、黄海大型水母暴发现象及其可能成因浅析. 现代渔业信息, 19(5): 10—12
谢忠明, 王永顺, 黄鸣夏, 2004. 海蜇增养殖技术. 北京: 金盾出版社, 107—118
Areshehenkova T, Ganal M W, 2002. Comparative analysis of Polymorphism and chromosomal location of tomato microsatellite markers isolated from different sources. Theor Appl Genet, 104(2—3): 229—235
Bachtrog D, Agis M, Imhof M et al, 2000. Microsatellite variability differs between dinucleotide repeat motifs-evidence from Drosophila melanogaster. Mol Biol Evol, 17(9): 1277—1285
Dupuy B M, Stenersen M, Egeland T et al, 2004. Y-chromosomal microsatellite mutation rates: differences in mutation rate between and within loci. Hum Mutat, 23(2): 117—124
Ellegren H, 2000. Microsatellite mutations in the germline:implications for evolutionary inference. Trends Genet,16(12): 551—558
Huang Q Y, Xu F H, Shen H et al, 2002. Mutation patterns at dinucleotide microsatellite loci in humans. Am J Hum Genet,70(3): 625—634
Klaus A, Birchmeier W, 2008. Wnt signalling and its impact on
development and cancer. Nat Rev Cancer, 8(5): 387—398 Kuhl M, Geis K, Sheldahl L C et al, 2001. Antagonistic regulation of convergent extension movements in Xenopus by Wnt/beta-catenin and Wnt/Ca2+signaling. Mech Dev,106(1—2): 61—76
Logan C Y, Nusse R, 2004. The Wnt signaling pathway in development and disease. Annu Rev Cell Dev Biol, 20: 781—810
Peifer M, 1997. β-catenin as oncogene: the smoking gun. Science,275(5307): 1752
Prestwich T C, Macdougald O A, 2007. Wnt/β-catenin signaling in adipogenesis and metabolism. Curr Opin Cell Biol, 19(6):612—617
Scott K D, Eggler P, Seaton G et al, 2000. Analysis of SSRs derived from grape ESTs. Theor Appl Genet, 100(5): 723—726
Willert K, Brown J D, Danenberg E et al, 2003. Wnt proteins are lipid-modified and can act as stem cell growth factors.Nature, 423(6938): 448—452

